牦牛HSFY基因的克隆及其结构分析
2013-09-13曾贤彬王永刘仲娜马志杰海汀钟金城
曾贤彬 王永 刘仲娜 马志杰 海汀 钟金城
热休克转录因子HSFY(heat shock transcription factor,Y chromosome)是哺乳动物4种热休克因子(HSF1、HSF2、HSF4和HSFY)之一,控制热休克蛋白表达[1]。HSFY 与其它 HSFs(HSF1和HSF2)具有不同的功能[2]。Tessari等[3]将 HSFY作为人精子发生障碍的候选基因之一。Giovanna[4]、Shinka[2]、Ferlin[5]等报道 HSFY 基因缺失与无精子、少精子症紧密相关。
Tessari[3]、Christine[6]等对人、恒河猴、普通牛、家猫、挪威鼠的HSFY蛋白质序列进行了多序列比对,结果显示,普通牛与家猫亲缘关系最近,两物种间的该基因编码区和与DNA结合的结构域的序列一致性分别为58%和81%。目前认为,HSFY基因是Y染色体上的多拷贝基因。人有6个拷贝,其中两个功能拷贝;家猫有8个功能拷贝,拷贝数未知;普通牛约有73个拷贝,功能基因数未知[3,6,7]。人、普通牛的HSFY主要在睾丸中特异表达[3,8],即HSFY仅在支持细胞和精母细胞中表达[2],其mRNA表达水平与精原细胞和精母细胞标记(UCHL1和 TRPC2)mRNA 表达水平显著相关(P<0.0001)[6]。用抗HSFY抗体证实,正常精子生成时,HSFY在曲细精管的生殖细胞和支持细胞中表达。精原细胞期、精母细胞细线期均无HSFY表达信号;偶线期染色质周围有强HSFY表达信号,但在精子成熟后HSFY蛋白量减少[9],表明其与精子生成有关,但其生物学功能尚待研究[2-4,10]。
有关牦牛HSFY基因的研究尚未见报道,本研究对牦牛的HSFY基因进行克隆测序、结构和密码子偏好性分析以及与普通牛等其他物种相应基因进行比对研究,以期为进一步研究牦牛HSFY基因的结构和功能提供理论依据。
1 材料与方法
1.1 材料
在四川省龙日种畜场选取健康成年公牦牛两头,采取耳组织,置于75%乙醇中带回实验室,保存于-20℃冰箱中备用。
1.2 方法
1.2.1 基因组DNA的提取 按试剂盒(天根公司)操作说明提取基因组DNA。
1.2.2 牦牛HSFY基因克隆测序 根据普通牛HSFY2序列(NC_016145.1)设计引物,上下游引物分别为F:5'-TCACAGCCTTTTGACTTTG-3'、R:5'-CTCTTTTCCCTTGCTTCTC-3';引物由上海Invitrogen公司合成。
25μL PCR反应体系:基因组 DNA 1μL、上下游引物各1μL、ddH2O 9.5μL 和 2×long Taq酶(博奥维新公司)12.5μL 。PCR反应程序:95℃预变性3min;95℃变性30s,56℃复性30s,72℃延伸1.5min,共30个循环;72℃终延伸10min,4℃保存。
经胶回收试剂盒回收PCR产物,与pMD19-T(购自TaKaRa公司)于16℃过夜连接,其产物转化大肠杆菌(DH5α)感受态细胞,涂布于含50μg/mL氨苄青霉素、200mg/mL IPTG及20mg/mL X-Gal的LB平板,过夜培养。经PCR筛选阳性克隆进行测序。
1.2.3 序列分析 测序结果用DNAMAN4.0、DNAssist1.0等生物信息学软件进行拼接、比对分析,用 GENSCAN(http://genes.mit.edu/GENSCAN.html)预测蛋白的氨基酸序列,并与普通牛(NP_001070-474)、瘤牛(AFV09891)、人(hHSFY1:NP_1490-99.2;hHSFY2:NP_714927.1)、挪威鼠(NP_00101-2132.1)、家短尾负鼠(ACT35160)、恒河猴(ACL-51668.1)、家猫(NP_001035212.1)等的 HSFY 蛋白的序列用 Clustal W[11]进行多序列比对[6],用MEGA5软件构建系统进化树。在UCSC基因组浏览器(http://genome.ucsc.edu/)的普通牛基因组中,以HSFY引物对搜索,选取位于Y染色体上的HSFY相似序列。GENSCAN预测外显子区域,condon W(http://mobyle.pasteur.fr/cgi-bin/portal.py?#forms::codonw)分析其密码子偏好性和有效密码子数等。用 SWISS-MODEL(http://swissmodel.expasy.org/)同源建模构建HSFY蛋白三维结构。
2 结果
2.1 PCR扩增结果
经PCR扩增,电泳检测为一清晰条带(图1)。结合普通牛HSFY2序列(NC_016145.1),通过DNA分子量标准和测序结果分析得知,牦牛HSFY基因PCR产物大小为2000bp左右,与预期结果一致。

图1 牦牛HSFY基因的PCR扩增电泳图
2.2 牦牛HSFY基因的核苷酸序列及其比对
本研究得到的牦牛HSFY基因由2个外显子和1个内含子组成,与普通牛基因组中的相应序列进行比对,其一致性高达99.54%。第一外显子未出现插入/缺失,但在1012-1020bp(Ⅰ)处有AAAGAAGA/TG等9个核苷酸缺失,位于内含子内;在1071-1073bp(Ⅱ)、1249-1251bp(Ⅲ)处分别有AAG、CCA/C等3个核苷酸缺失,位于第二外显子内,分别导致赖氨酸和组氨酸的缺失;在1 708-1 710bp(Ⅳ)处有CAA 3个核苷酸缺失,位于3'-URT。在普通牛HSFY基因的84条序列中Ⅰ、Ⅱ、Ⅲ、Ⅳ插入/缺失分别有29、3、2、1次,未发现Ⅱ、Ⅲ同时缺失的拷贝,但Ⅱ、Ⅲ缺失均伴有Ⅰ缺失。Ⅰ、Ⅱ、Ⅲ、Ⅳ插入/缺失的序列与其相邻序列极为相似(图2)。

图2 两条牦牛HSFY基因序列与普通牛部分相应序列的比对
HSFY基因在许多物种中为多拷贝基因,但在物种内序列的相似性极高,人的两个功能拷贝的氨基酸序列相似性达100%。本研究得到的牦牛HSFY序列1仅有Ⅲ缺失,且预测的基因剪切方式也与普通牛中具有功能的HSFY基因剪切方式一致。相对序列2含有Ⅰ、Ⅲ、Ⅳ3处缺失,序列1预测的氨基酸序列更能反映牦牛HSFY蛋白质信息。其与人、恒河猴、普通牛、瘤牛、家猫、挪威鼠、家短尾负鼠的HSFY蛋白的氨基酸序列进行多序列比对,构建系统进化树(图3)。牦牛与普通牛、瘤牛首先聚为一类,再相继与家猫、家短尾负鼠、人等聚类,聚类结果与动物学分类结果基本一致。
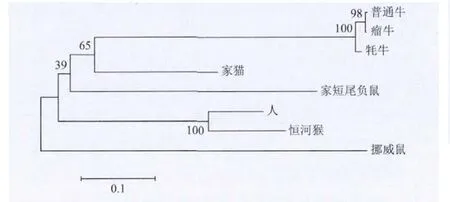
图3 牦牛与普通牛等物种HSFY的系统进化关系
2.3 牦牛HSFY蛋白质的三维结构
以牦牛序列1的氨基酸序列为基础序列,研究HSFYⅡ、Ⅲ插入/缺失与蛋白的三维结构变化情况。结果(图4)表明,Ⅱ与Ⅲ的单独缺失与Ⅱ和Ⅲ均缺失的三维结构基本一致,但与Ⅱ、Ⅲ均未缺失的结构差异较大。Ⅱ、Ⅲ插入/缺失对应的带正电荷的赖氨酸、组氨酸比较倾向于位于蛋白表面,通过静电作用等维持HSFY蛋白的立体结构,Ⅱ、Ⅲ任一缺失均使HSFY蛋白的立体结构塌陷。Ⅱ、Ⅲ插入/缺失可能会使HSFY蛋白的功能发生变化进而影响精子发生。
2.4 牦牛HSFY基因的密码子偏好性
以牦牛HSFY序列1作为密码子偏好性分析,GENSCAN预测得到长1131bp的蛋白编码区。A、T、C和G分别占33.24%、29.27%、20.34%和17.15%,A3s、T3s、G3s和C3s分别为30.77%、37.93%、15.91%和15.38%,其中G+C、G3s+C3s含量分别为37.19%和31.30%。有效密码子数为46.82。可见,HSFY基因偏好使用A、T和以A、T结尾的密码子,UUU、UUG、CUU、AUU、GUG、UAU、CAU、CAA、GAA、GAU、UCU、UCA、CCU、CCA、ACU、ACA、GCU、GCA、UGU、AGA、AGG、GGC和 GGA等 23个密码子RSCU(相对密码子使用度)均大于1,为牦牛HSFY偏好性密码子;Asn、Lys、Arg、Ser 4个氨基酸没有密码子偏好现象;除编码Leu、Val、Arg、Gly的UUG、GUG、AGG、GGC外,避免密码子后两位以G或C结尾,尤其以CG结尾如CCG、ACG、GCG的 RSCU为 0(表1)。

图4 牦牛HSFY蛋白质的Ⅱ、Ⅲ插入/缺失的三维结构
3 讨论
3.1 关于牦牛HSFY基因的拷贝数
在人体中,研究发现Yq11区存在与精子发生相关的基因,这些基因或基因家族又称为无精子症因子(azoospermia facter,AZF),目前已经确定AZF由相互叠加的4个区域组成,即AZFa、AZFb、AZFc、AZFd[12,13]。HSFY 是位于 AZFb 区无精子症因子候选因子之一。在许多物种中发现是由多拷贝组成的基因,人有6个拷贝,家猫有8个功能拷贝,普通牛约有73个拷贝[3,6,7]。以往的研究表明,牦牛与普通牛具有较近的亲缘关系,在许多基因组成和基因结构上都有相似性,本研究得到的2条牦牛HSFY基因序列虽在核苷酸组成上有一些差异,但相似性很高。因此认为牦牛的HSFY基因是一个多拷贝基因。

表1 牦牛HSFY序列1的RSCU
3.2 关于牦牛HSFY基因的结构与遗传变异
Hamilton等[6]研究普通牛HSFY基因的拷贝数在67-84范围波动,基因表达和56天NRR系数(反映受精比率)均无显著性差异。与Shaw-Smith[14]、Inoue[15]、Lupski[16]等研究表明的基因微量删除和重复严重影响生物学功能现象不一致,推测是由多拷贝和基因调控序列区域变化两种调节机制确保进行精子发生。该机制可能受到严格的自然选择。本研究得到2条牦牛的HSFY序列,并搜索得到普通牛的HSFY序列84条,与Hamilton[6]研究结果基本一致,表明引物结合处比较保守,可以用于检测HSFY基因缺失状态。比对发现牦牛、普通牛有4处插入/缺失,其序列均分别与其相邻序列极为相似,可能由染色体内同源重组导致缺失的发生[17]。Ⅰ、Ⅳ缺失虽然不引起蛋白结构的变化,但估计起转录调控作用,以便精细调节细胞中HSFY蛋白浓度变化弥补个别基因因突变或缺失造成的功能缺失。Ⅱ、Ⅲ插入/缺失对应的赖氨酸和组氨酸的零深度相对倾向因子分别为30.850、15.312[18],使均带正电荷的赖氨酸、组氨酸倾向于位于蛋白表面,HSFY蛋白的立体结构塌陷。Ⅱ、Ⅲ插入/缺失可能会使HSFY蛋白的功能发生变化进而影响精子发生。普通牛中Ⅱ和Ⅲ缺失均伴有Ⅰ缺失,很可能Ⅰ插入/缺失参与该基因的精确表达调控,但是使得该基因更易发生缺失突变。当然作者的这些推测还有待于进一步的生物试验证实。
3.3 关于牦牛HSFY蛋白的结构与功能
热休克蛋白(heat shock protein,HSP)是指所有生物细胞在应激原刺激后,发生热休克反应时产生的一类细胞伴侣蛋白的总称。该类蛋白在细胞生命过程中可参与蛋白的折叠、装配及运输等活动,对调控细胞的生长、存活及分化起着重要作用。在哺乳动物中,HSP家族参与精子发生、精子获能及受精等一系列活动,与雄性生殖过程密切相关[19]。研究表明,人和猫Y染色体上多拷贝基因都在睾丸组织中表达并行使功能[8,20-22],则进一步推测牦牛HSFY在睾丸中表达和行使生物学功能。HSF成员HSF1、HSF2在精子发生中也起到了重要作用[23,24]。Ahn 等[25]报道,HSFY 和其它 HSFs(HSF1、HSF2、HSF4)的结构不同,后者的中心螺旋型是由α-螺旋2和3组成,α-螺旋3是识别的DNA的螺旋结构,然而HSFY有类似HSFs的DNA结合区,但没有α-螺旋2和3。推测在精子发生活动中,HSFY可能具有与其它HSFs不同的调控HSP表达途径。
蛋白质序列变化[26]与其生物学功能变化密切相关。本研究中,牦牛、普通牛HSFY基因未发现Ⅱ、Ⅲ同时缺失,且Ⅱ、Ⅲ任意一处缺失与均缺失的三维结构基本一致且有5个序列,占5.95%,可能与精子发生受阻相关。因缺失的比例较小和调控其它正常基因,确保了生物体精子发生正常。可通过Ⅱ、Ⅲ两端的保守序列设计引物进一步研究其数量变化与生物活动变化之间的规律。值得一提的是基因密码子的使用与基因表达的生理功能有密切联系[27,28]。其不仅调控基因表达[29],而且与翻译的准确性和效率有关[30]。本研究以牦牛序列1预测的编码区偏好使用A、T和以A、T结尾的密码子。考虑到该基因在小鼠精子发生过程中有瞬时高表达现象,估计牦牛HSFY翻译比较低的准确度,更有利于瞬时高表达行使功能。但其生物学机制需要进一步研究。
4 结论
牦牛HSFY基因由2个外显子和1个内含子组成,是在睾丸中表达的多拷贝基因,与普通牛基因组中的相应序列的一致性较高,属于同源性的基因。
牦牛、普通牛的该基因具有4处插入/缺失,且序列与其相邻序列极为相似。牦牛HSFY基因编码的蛋白质偏好使用以A或T结尾的密码子。
[1] Pirkkala L, Nykanen P, Sistonen L.Roles of theheat shock transcription factors in regulation of theheat shock response and beyond [J].FASEB J, 2001, 15(7):1118-1131.
[2] Shinka T, Sato Y, Chen G, et al.Molecular characterization ofheat shock-like factor encoded on thehuman Y chromosome, and implications formale infertility [J].Biol Reprod, 2004, 71(1):297-306.
[3] Tessari A, Salata E, Ferlin A, et al.Characterization of HSFY, a novel AZFb gene on the Y chromosome with a possible role inhuman spermatogenesis [J].Mol Hum Reprod, 2004, 10(4):253-258.
[4] Vinci G, Raicu F, Popa L, et al.A deletion of a novelheat shock gene on the Y chromosome associated with azoospermia [J].Mol Hum Reprod, 2005, 11(4):295-298.
[5] Ferlin A, Moro E, Rossi A, et al.Thehuman Y chromosomes azoospermia factor b(AZFb)region:squence, structure and deletion analysis infertilemen [J].J Med Genet, 2003, 40:13-24.
[6] Hamilton CK, Revay T, Domander R, et al.A large expansion of the HSFY gene family in cattle shows dispersion across Yq and Testisspecific expression [J].PLoS One, 2011, 6(3):e17790.
[7] Pearks Wilkerson AJ, Raudsepp T, Graves T, et al.Gene discovery and comparative analysis of X-degenerate genes from the domestic cat Y chromosome [J].Genomics, 2008, 92(5):329-338.
[8] Skaletsky H, Kuroda-Kawaguchi T, Minx PJ, et al.Themale-specific region of thehuman Y chromosome is amosaic of discrete sequence classes [J].Nature, 2003, 423:825-837.
[9] Sato Y, Yoshida K, Shinka T, et al.Altered expression pattern ofheat shock transcription factor, Y chromosome(HSFY)may be related to altered differentiation of spermatogenic cells in testes with deteriorated spermatogenesis[J].Fertility and Sterility, 2006, 86(3):612-618.
[10] Kinoshita K, Shinka T, Sato Y, et al.Expression analysis of amouse orthologue of HSFY, a candidate for the azoospermic factor on thehuman Y chromosome [J].J Med Invest, 2006, 53:117-122.
[11] Thompson JD, Higgins DG, Gibson TJ.CLUSTAL W:Improving the sensitivity of progressivemultiple sequence alignment through sequence weighting, position-specific gap penalties and weightmatrix choice [J].Nucleic Acids Res, 1994, 22:4673-4680.
[12] Vogt PH.Human chromosome deletions in Yq11, AZF candidate genes andmale infertility:History and update [J].Mol Hum Reprod, 1998, 4(8):739-744.
[13] Kent-First M, Muallem A, Shultz J, et al.Defining regions of the Y chromosome responsible formale infertility and identification of a fourth AZF region(AZFd)by Y-chromosomemicrodeletion detection [J].Mol Reprod Dev, 1999, 53(1):27-41.
[14] Shaw-Smith C, Redon R, Rickman L, et al.Microarray based comparative genomichybridisation(array-CGH)detects submicroscopic chromosomal deletions and duplications in patients with learning disability/mental retardation and dysmorphic features[J].J Med Genet, 2004, 41(4):241-248.
[15] Inoue K, Lupski JR.Molecularmechanisms for genomic disorders[J].Annu Rev Genomics Hum Genet, 2002, 3:199-242.
[16] Lupski JR, Stankiewicz P.Genomic disorders:molecularmechanisms for rearrangements and conveyed phenotypes [J].PLoS Genet, 2005, 1(6):e49.
[17] Vogt PH.Molecular genetic ofhumanmale infertility:from genes to new therapeutic perspectives [J].Curr Pharm Des, 2004, 10(5):471-500.
[18] 沈世锰, 胡刚, 张华.氨基酸在蛋白质结构中的深度倾向性因子[J].生物数学学报, 2007, 22(2):305-310.
[19] 朱晔敏, 丁之德.雄性生殖中热休克蛋白作用的研究进展[J].国际生殖健康/计划生育杂志, 2010, 29(1):18-21.
[20] Murphy WJ, Pearks Wilkerson AJ, Raudsepp T, et al.Novel gene acquisition on carnivore Y chromosomes [J].PLoS Genet, 2006, 2:e43.
[21] Lahn BT, Pearson NM, Jegalian K.Thehuman Y chromosome, in the light of evolution [J].Nat Rev Genet, 2001, 2:207-216.
[22] Lahn BT, Page DC.Functional coherence of thehuman Y chromosome [J].Science, 1997, 278(5338):675-680.
[23] Nakai A, Suzuki M, Tanabe M.Arrest of spermatogenesis inmice expressing an activeheat shock transcription factor [J].EMBO J,2000, 19:1545-1554.
[24] Kallio M, Chang M, Manuel M, et al.Brain abnormalities defectivemeiotic chromosome synapsis and female subfertility in HSF2 nullmice [J].EMBO J, 2002, 21:2591-2601.
[25] Ahn SG, Liu PCC, Klyachko K, et al.The loop domain ofh eat shock transcription factor 1dictates DNA-binding specificity and responses toheat stress [J].Genes Dev, 2001, 15:2134-2145.
[26] Redon R, Ishikawa S, Fitch KR, et al.Global variation in copy number in thehuman genome [J].Nature, 2006, 444(7118):444-454.
[27] Karlin S, Campbell AM, Mrázek J.Comparative DNA analysis across diverse genomes [J].Annu Rev Genet, 1998, 32:185-225.
[28] Kennedy SP, Ng WV, Salzberg SL.Understanding the adaptation of Halobacterium species NRC-1 to its extreme environment through computational analysis of its genome sequence [J].Genome Res,2001, 11(10):164-165.
[29] Carlini DB, Stephan W.In vivo introduction of unpreferred synonymous codons into the Drosophila Adh gene results in reduced levels of ADH protein [J].Genetics, 2003, 163(1):239-243.
[30] Sharp PM, Matassi G.Codon usage and genome evolution [J].Curr Opin Genet Dev, 1994, 4(6):851-860.
