5个黄牛群体垂体转录因子基因变异研究*
2013-01-07钱林东李卓然张自芳苗永旺
钱林东,唐 贺,李卓然,张自芳,袁 峰,钱 坤,苗永旺*
(1.云南农业职业技术学院 畜牧兽医系,云南 昆明650212;2.云南农业大学 动物科学学院,云南 昆明650201 3.湖州师范学院,浙江 湖州313000)
垂体转录因子(Pituitary-specific transcription factor,POU1F1)是POU结构域中同源异型蛋白之一[1]。在哺乳动物中,POU1F1够促进生长激素(growth hormone,GH)细胞系、催乳素(prolactin,PRL)细胞系和促甲状腺素β亚单位(thyroid stimulating hormoneβ,TSHβ)细胞系的生长和发育,并对GH、PRL和TSHβ基因的表达起到正向调控的作用,其对哺乳动物有提高生长速度、降低脂肪沉积等广泛的生物学功能。POU1F1基因只激活相关细胞中特异基因表达,同时抑制其他激素基因不适当转录,表现出强烈组织特异性,在哺乳动物胚胎分化和生长发育过程中起着关键作用[1]。
牛的POU1F1基因位于1号染色体上,由调控区、6个外显子和5个内含子构成,全长19 654 bp,其mRNA全长1 502 bp,编码291个氨基酸[2]。其中POU特异性区域主要由第3和第4外显子编码;POU同源区由第5和第6外显子编码。研究发现,牛的POU1F1基因不仅在牛的胚胎发育中至关重要[3],而且与奶牛的产奶量、乳成分等生产性状以及肉牛的体重、腰肉厚度等生产性状都存在相关性[4-5]。可见,POU1FI基因与牛的养殖生产有着重要的关系。
云南本地黄牛多为没有经过系统选育的原始品种。昭通黄牛主要分布于滇东北地区,属于西南山地型黄牛;迪庆黄牛主要分布于滇西北高原,属于高原型黄牛[6]。过去,对云南本地黄牛POU1F1基因的研究工作开展的很少,本研究采用DNA序列分析和PCR-RFLP技术,以云南昭通黄牛和迪庆黄牛为研究对象,并以引进品种短角牛、荷斯坦奶牛和安格斯牛为对照,对POU1F1基因的第5内含子和第6外显子进行了基因克隆测序和群体变异检测分析,以揭示POU1F1基因的特征,为今后云南黄牛的选育提供了依据。
1 材料与方法
1.1 试验材料
研究样品来自4个黄牛群体,其中包括3个云南本地群体和2个国外引进品种。云南本地各群体样品为:昭通黄牛(ZT)62头、迪庆黄牛(DQ)22头、云南荷斯坦奶牛(HOS)12头;引进品种样品为:短角牛(SH)45头、安格斯牛(ANG)10头。采样方式为随机采样,每头牛颈静脉采血10 m L,肝素钠抗凝,然后与等体积的DNA保存液混合,带回实验室-20℃冰箱保存。
1.2 基因组DNA的提取
参照《分子克隆》中的方法提取基因组总DNA[7],经紫外分光光度法重检测其纯度和浓度,稀释成50 ng/μL浓度作为PCR扩增的模板。
1.3 引物设计
引物由上海生工合成。根据文献[8],引物序列如 下:上 游 引 物:5'-AAACCATCATCTCCCTTCTT-3';下游引物:5'-AATGTACAATGTGCCTTCTGAG-3'。
1.4 PCR-RFLP分析
1.4.1 PCR引物及反应体系 PCR扩增采用高保真DNA聚合酶(PrimeSTAR HS DNA Polymerase)按照厂家说明进行。PCR反应体系为15μL,其中含有基因组DNA约50 ng,引物20 pmol/L,d NTPs 10 mmol/L,MgCl21.5 mmol/L,r Taq酶0.5 U。PCR扩增条件为:95℃预变性5 min;然后35个循环:94℃变性30 s,62℃复性30 s,72℃延伸30 s;最后72℃延伸7 min。预期扩增出POU1F1基因的第5内含子和第6外显子中包括突变位点的一段序列,长度为451 bp。
1.4.2 酶切消化和基因分型 将PCR产物使用限制性内切酶HinfⅠ(Fermentas公司)在37℃水浴锅中消化8 h,然后使用3.0%琼脂糖凝胶对酶切产物进行检测。
1.5 PCR产物测序
PCR产物经琼脂糖凝胶回收纯化后,使用TAKARA公司(大连宝生物)质粒连接试剂盒将之连接到p MD18-T Vector载体上,转化感受态大肠杆菌。将转化之后的大肠杆菌在加氨苄青霉素的平板培养基上培养,挑取单克隆菌落在液体培养基中培养。经PCR检测后,选取呈阳性的菌液提取质粒DNA,送上海生工测序。各种基因型分别选1个个体进行测序。
1.5 数据分析
用PopGen32和Gen AlEx软件计算POU1F1基因等位、频率和遗传距离等指标,进行聚类分析。
2 结 果
2.1 PCR-RFLP检测结果
对检测的群体采用HinfⅠ酶切,酶切产物经琼脂糖凝胶检测结果见图1。PCR产物全长为451 bp,经HinfⅠ酶切后,可以得到A和B这2个等位基因,AA、BB和AB共3种基因型。A等位基因的PCR产物没有HinfⅠ酶切位点,经消化后检测没有变化。B等位基因的PCR产物有1个HinfⅠ酶切位点,经消化后检测得到长度分别为244 bp和207 bp的2条带。AA基因型仅可检测到长度为451 bp的1条带;BB基因型可检测到长度为244 bp和207 bp的2条带;AB基因型可检测到长度为451 bp、244 bp和207 bp的3条带。

图1 POU1F1基因的3种基因型1.AA型;2-5.BB型;6.AB型Fig.1 3 genetypes of the POU1F1 gene1.Genetype AA;2-5.Genetype BB;6.Genetype AB
2.2 POU1F1基因的遗传多态性指标
研究的5个群体中,都发现了A和B两种基因,各群体基因频率和基因型频率见表1。在荷斯坦牛中没有发现AA型个体;在短角牛和安格斯牛中没有发现BB型个体。昭通黄牛、迪庆黄牛和荷斯坦牛中,B等位基因是优势等位基因;短角牛和安格斯牛中,A等位基因是优势等位基因。经x2检测,各群体都处于Hardy-Weinberg平衡状态。

表1 5个群体POU1F1-HinfⅠ基因座的基因频率和基因型频率Table 1 Gene and genotype frequencies at POU1F1-HinfⅠlocus in 5 populations
5个群体的POU1F1基因的遗传多态性信息见表2。昭通黄牛和迪庆黄牛的杂合度高于短角牛和荷斯坦牛,但是低于安格斯牛。昭通黄牛、迪庆黄牛和安格斯牛的有效等位基因数和多态信息含量都高于短角牛和荷斯坦牛。
2.3 群体间遗传差异
5个群体间POU1F1基因的遗传差异与相似系数见表3。由遗传距离构建的聚类图见图2。
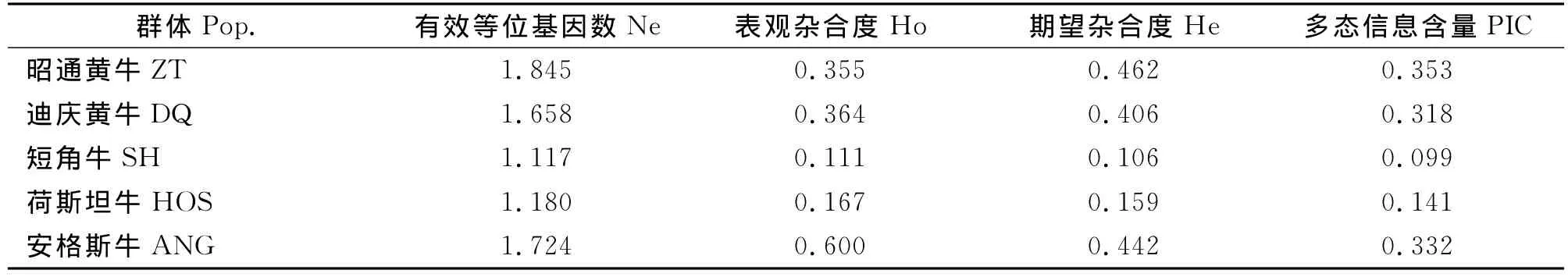
表2 5个群体POU1F1-HinfⅠ基因座遗传多态性Table 2 Genetic diversity indices at POU1F1-HinfⅠlocus in 5 populations

表3 5个黄牛群体Nei's遗传距离及相似系数Table 3 Genetic identity and genetic distance between the 5 cattle breeds

图2 5个牛群体的Nei's(1978)遗传距离聚类图Fig.2 Dendrogram of Nei's(1978)genetic distance among 5 cattle breeds
2.4 PCR产物测序结果
测序发现A等位基因和B等位基因的PCR产物存在2处差异。A等位基因产物在207 bp处为A,在281 bp处为T。与A等位基因相比,B等位基因的突变为A207G和T281G。其中207处的突变造成了B等位基因产物上出现HinfⅠ酶切位点(GANTC)。
3 讨 论
根据国外对意大利荷斯坦公牛[8]、利木赞牛[9]和波兰黑白花奶牛[4]的POU1F1基因该段等位基因的研究结果,B等位基因在这3种牛中都是优势等位基因,其基因频率分别为0.812、0.7269和0.75。国内对秦川牛和中国荷斯坦牛的研究也发现,B等位基因在这2个群体中是优势等位基因,分别为0.768和0.868[10]。已经研究的品种包括了奶牛和肉牛,而这2种用途的牛中,B等位基因都是优势等位基因,据此推测B等位基因可能对产奶和产肉性能都有正效应。
在本研究所使用的荷斯坦牛中,B等位基因频率达到了0.917,这与前人对荷斯坦牛的研究结果相符,说明该荷斯坦牛群体受到了更强的人工选择。而在本文的短角牛和安格斯牛中,A等位基因为优势等位基因,其基因频率分别为0.944和0.700,与前人对肉用牛的研究结果不完全一致,揭示短角牛和安格斯牛POU1F1基因具有不同的群体遗传特征。云南本地黄牛多为没有经过系统选育的原始品种。本研究中,昭通黄牛和迪庆黄牛的B等位基因频率分别只有0.645和0.727。而杂合度和多态信息含量较高。这说明这2个地方品种虽然受到了人工选择,但是选择强度并不大,提示它们为原始地方牛种,应当进一步加强选育,淘汰生产性能不高的个体,继续提高这2个群体的生产性能。至于本研究中安格斯牛的杂合度也较高,提示该牛可能存在一定的血统混杂,而短角牛、荷斯坦奶牛的杂合度相对较低,提示它们选育程度较高。
本研究结果可为利用POU1F1基因为候选基因进行黄牛的肉用和奶用选育利用提供依据和遗传背景资料。
[1] Ingraham H A,Albert V R,Chen R,et al.A family of POU-domain and PIT-1 tissue-specific transcription factors in pituitary and neuroendocrine development[J].Annual Review Physiology,1999,52:773-791.
[2] Woollard J,Tuggle C K,Ponce de Leon A.Rapid communication:Loca ization of Pit-l to bovine,ovine,and caprine lq21-22[J].J Anim Sci,2000,78:242-243.
[3] Joundy E M,Lechniak D,Petrik J,et al.Expression of growth hormone and its transcription factor,Pit-1,in early bovine development[J].Molecular Reproduction and Development,2003,64:275-283.
[4] Zwierzehowski L,Krzyzewski J,Strzalkowska N,et al.Effect of polymorphisms of growth hormone(GH),Pit-1,and leptin(LEP)genes,cow's age,lactation stage and somatic cell count on milk yield and composition of Polish Black-and-White cows[J].Anim Sci Papers Reports,2002,20:213-227.
[5] Di Stasio L,Sartotore S,Albera A.Lack of Association of GH 1 and POU1F1 gene variants with meat production traits in Piemontese cattle[J].Animal Genetics,2002,33(1):61-64.
[6] 云南省畜牧局.云南省家畜家禽品种志[M].昆明:云南科技出版社,1987:229-232,246-249.
[7] Sambrook J,Russell D W.Molecular Cloning:A Laboratory Manual[M].3 Edition.Cold Spring Harbor:CSHL Press,2002:483-485.
[8] Renaville R,Gengler N,Vrech E,et al.Pit-1 gene polymorphism,milk yield,and conformation traits for Italian Holstein-Friesian bulls[J].Journal of Dairy Science,1997,80(12):3 431-3 438.
[9] Dybus A,Kmiec M,Sobek Z,et al.Associations between polymorphisms of growth hormone releasing hormone(GHRH)and pituitary transcription factor 1 (PIT1)genes and production traits of Limousine cattle[J].Arch Tierz Dummerstorf,2003,46(6):527-534.
[10] 严林俊,刘 波,房兴堂,等.秦川牛和中国荷斯坦牛POU1F1基因多态性研究[J].遗传,2006,28(11):1 371-1 375.
