拟南芥At PLC4基因RNAi载体的构建及遗传转化
2012-11-07岳智亮马惠斌侯名语潘延云
岳智亮,马惠斌,侯名语,潘延云
(河北农业大学 生命科学学院,河北 保定 071001)
拟南芥At PLC4基因RNAi载体的构建及遗传转化
岳智亮,马惠斌,侯名语,潘延云
(河北农业大学 生命科学学院,河北 保定 071001)
利用RNAi技术,构建了AtPLC4基因的RNAi双元载体,并进行了拟南芥的遗传转化,通过抗性筛选及PCR鉴定,获得20株遗传转化株系.进一步通过RT-PCR检测,得到3株AtPLC4基因表达降低的转基因植株,为运用反向遗传学的方法深入研究AtPLC基因家族的生物学功能提供基础.
拟南芥;AtPLC4;RNAi
肌醇磷脂依赖性磷脂酶C(phosphoinositidespecific phospholipase C,PI-PLC,简称为PLC)是肌醇磷脂信号系统中的关键组分之一.PLC受到外界刺激时可被受体激活,水解膜组分PIP2,产生双信使分子IP3和DAG,调节细胞中Ca2+的浓度和PKC的活性,影响一系列靶蛋白和靶酶的活性,参与多种生理过程[1-2].1995年第1个植物PLC的cDNA在拟南芥中被克隆,随后在烟草、百合等多个物种中也相继被克隆[3-6].植物PLC基因的生物功能是多方面的,除对生长发育进行调节外[7-10],还参与了众多生物和非生物刺激的胁迫应答反应,如伤害[11],厌氧[12],病原侵染[13],热激[14]、乙烯、ABA等激素因子和冷、盐、干旱等渗透胁迫因子[15-17].但迄今对植物PLC基因的作用机制还远未澄清.
植物PLC在许多物种中都含有多个亚型(isoform),不同的亚型可能分担不同的生理功能.拟南芥基因组计划完成后,检索发现有9个AtPLC亚型[17],基因芯片、实时定量PCR和GUS报告基因等方法研究的结果发现,不同亚型不仅有各自的组织表达模式,而且在不同胁迫环境中被不同程度地诱导表达[17].拟南芥作为一种模式植物已经被广泛地应用于基因功能的研究,ABRC的建立使人们可以利用反向遗传学的方法,直接从已知基因序列入手,通过功能缺失突变体的表型效应,研究目的基因的功能.但ABRC中缺乏部分亚型如AtPLC4的T-DNA插入基因突变体.AtPLC4基因在多种胁迫环境中被不同程度地诱导表达,如在盐、ABA、MS、冷和干旱胁迫环境下其表达量提高了1.5~6倍[17].为了进一步研究AtPLC4在非生物胁迫中的作用,本研究利用RNAi技术,通过载体构建和遗传转化获得AtPLC4基因表达下调的转基因植株,为深入研究AtPLC4的生物学功能和作用机制奠定基础.
1 材料与方法
1.1 实验材料
拟南芥(Arabidopsisthaliana)Columbia生态型(Col)、RNAi载体p TCK303均为马力耕教授惠赠;大肠杆菌菌株DH5α、农杆菌菌株GV3101为本实验室保存.
1.2 实验方法
1.2.1 拟南芥的种植
拟南芥种子经深层消毒(25 g/L次氯酸钠,10 min,无菌水洗3~4次)后,4℃暗中春化3 d,播种在固体MS培养基(30 g/L蔗糖,8 g/L琼脂粉,1×Murashige and Skoog盐和维生素,p H用NaOH调至5.8)上,4℃暗中春化3 d,转移到拟南芥培养室(22℃,16 h/8 h光暗周期,光照强度6 000~8 000 lx)中培养.10~14 d后(幼苗长出2~4片真叶),将幼苗移栽到浸透Hoagland营养液的蛭石中,相同温度和光照条件继续培养,适时适量向蛭石中加入Hoagland营养液.
1.2.2 RT-PCR及目的片段的扩增
选取拟南芥莲座叶片,利用Trizol试剂提取总RNA,用Ta KaRa公司的AMV反转录试剂盒合成cDNA.提取及反转录方法按照试剂盒说明操作.以cDNA为模板,用引物5′-GAGCTCGGATCCAGGTCCATCAAAACATGGA-3′(下 划 线 部 分 为 酶 切 位 点SacⅠ,BamHⅠ )和5′-ACTAGTGTCGACTACTCTTTTGGAGGTTTTGTTG-3′(下划线部分为酶切位点SpeⅠ,SalⅠ )进行PCR,反应体系为50μL,包括模板5.0μL,Buffer 5.0μL,d NTPs 4.0μL,引物分别为4.0μL,Taq聚合酶0.25μL,Pfu聚合酶0.25μL,H2O 27.5μL.扩增条件为:95℃预变性5 min;94℃ 变性30 s,60℃退火30 s,72℃延伸1 min,35个循环;72℃延伸10 min.PCR扩增产物经琼脂糖凝胶检测后,回收目的片段,回收产物分别与p MD19-T simple vector连接,转化大肠杆菌DH5α,进行蓝白斑筛选,对阳性克隆进行质粒提取及酶切验证,对含有目的片段的质粒进行测序,将测序正确的质粒命名为P4X-T.
1.2.3 p TCK303-PLC4i载体构建
拟南芥p TCK303-PLC4i载体构建策略如图1所示.以SacⅠ和SpeⅠ酶切将目的片段反向插入p TCK303质粒,T4连接酶连接转化DH5α感受态细胞,获得中间载体p TCK303 4-,鉴定无误后再以SalⅠ和BamHⅠ酶切的目的片段正向插入该中间载体,再次进行T4连接酶的连接反应和DH5α的转化,获得p TCK303-PLC4i载体.构建好的载体经酶切验证后,转化农杆菌GV3101感受态细胞,用于拟南芥的遗传转化.

图1 pTCK303-PLC4i表达载体示意Fig.1 Construction of pTCK303-PLC4ivector
1.2.4 拟南芥的遗传转化及转基因植株的筛选
拟南芥的遗传转化采用浸花法[18],收获T0代种子后,播种于潮霉素(50 mg/L)的MS固体培养基上进行抗性筛选.选择长出2片真叶并生根的幼苗,按照方法1.2.1继续培养.p TCK303载体中既有潮霉素抗性筛选基因,也有GUS报告基因(图1).因此转基因植株进一步通过GUS报告基因进行检测,以获取阳性株系.GUS染色液的配制及染色方法参考文献[19].
1.2.5 转基因植株的RT-PCR分析
按照方法1.2.2获得转基因株系的cDNA,以此为模板,以野生型为对照检测AtPLC4的转录水平的变化,引物分别为5′-GAGGGTGCGGTTATGTCAAG-3′和5′-AAGAGTGGAACAGCGCGTAT-3′.以Actin基因为内参,引物为5′-GTTAGCAACTGGGATGATATGG-3′和5′-TCCACATCACACTTCATGAT-3′.PCR反应体系:cDNA模板1.0μL,Buffer 1.0μL,d NTPs 0.8μL,上游引物0.8μL,下游引物0.8μL,Taq聚合酶0.1μL,H2O 5.5μL.PCR扩增条件为:94℃预变性5 min;94℃变性30 s,60℃退火30 s,72℃延伸50 s,30个循环;72℃延伸10 min.扩增产物用10 g/L的琼脂糖凝胶检测.
2 结果与分析
2.1AtPLC4目的片段的获得
选择AtPLC4 cDNA中一段不含SacⅠ,BamHⅠ,SpeⅠ和SalⅠ等酶切位点,长度约500 bp的序列,按照方法1.2.2进行PCR和琼脂糖凝胶电泳检测,获得大小为478 bp的扩增条带(图2).将该条带进行回收,与p MD19-T simple vector连接.连接产物转化DH5α感受态细胞.提取质粒用BamHⅠ+SalⅠ进行双酶切验证,结果如图3示,显示鉴定正确,测序结果显示序列准确无误,命名为P4X-T,可用于p TCK303-PLC4i双元载体的构建.

图2AtPLC4基因目的片段的扩增Fig.2 PCR product of theAtPLC4

图3 P4X-T载体的酶切验证Fig.3 Identification of P4X-T vector by restriction digestionp
2.2 pTCK303-PLC4i双元载体的构建
根据p TCK303载体的酶切位点,按照方法1.2.3构建p TCK303-PLC4i双元表达载体.图4显示中间载体p TCK303 4-的酶切鉴定结果,用SacⅠ+SpeⅠ切出466 bp的片段,显示中间载体连接正确.图5显示最终载体p TCK303-PLC4i的酶切鉴定结果,泳道1为SacⅠ+SpeⅠ双酶切结果,泳道2为SalⅠ+BamHⅠ双酶切鉴定结果,均切出预计大小条带;泳道3为用SacⅠ+BamHⅠ对载体进行双酶切,结果显示酶切产物条带大小与2条目的片段及Rice Intron长度总和一致,证明p TCK303-PLC4i载体构建成功.

图4 p TCK303 4-载体的酶切鉴定Fig.4 Identification of pTCK303 4-vector by restriction digestion

图5 RNAi载体的酶切鉴定Fig.5 Identification of RNAi vector by restriction digestion
2.3 转基因植株的抗性筛选及鉴定
将构建好的p TCK303-PLC4i载体按照方法1.2.4转化Col生态型拟南芥,成熟后收取T0代种子.然后将种子播种于MS(Hyg+)培养基中进行筛选.由于载体带有抗性基因,转基因幼苗能够在Hyg培养基中正常生长,10 d后能长出2片真叶并且正常生根,而未转化的幼苗无法正常生长(图6).本文共获得20株具有抗性的转基因植株,随机选取其中5株提取基因组DNA进行PCR鉴定,同时取叶片进行GUS染色鉴定.PCR的引物和参数同方法1.2.2,结果如图7所示.野生型对照中,PCR可以扩出623 bp片段(含有内元)(图7泳道1),5株具有抗性的阳性苗中,均能扩出2条条带,分别为623 bp和454 bp,说明转基因植株中基因组中插入了目的序列(图7泳道2~6).

图6 转基因植株在平板上的生长情况Fig.6 Phenotype of positive transgenic plant on Petri dish

图7 拟南芥转基因植株的PCR检测Fig.7 PCR amplification of in transgenic plants
由于p TCK303-PLC4i载体中带有GUS报告基因,转基因植株可进行GUS染色分析,染色结果如图8.转基因阳性植株的叶片经GUS染色后呈蓝色,而野生型植株的叶片则不能被着色.以上结果表明p TCK303-PLC4i载体成功转化拟南芥植株.
2.4 转基因植株的RT-PCR检测
选取上述3株经PCR和GUS染色鉴定后的转基因阳性植株,提取总RNA,检测各株系AtPLC4基因的转录水平,结果如图9所示.3个转基因株系与野生型相比,AtPLC4基因表达水平均降低,但沉默效率彼此不同,其中第1株沉默效率最佳.后续实验将着重采用该株系进行.在上述3个株系中,本文还检测了At-PLC家族中与AtPLC4同源性较高的另一个基因AtPLC8基因的表达,发现该基因表达量无明显变化.表明p TCK303-PLC4i载体能够特异性地引起AtPLC4基因表达下调.
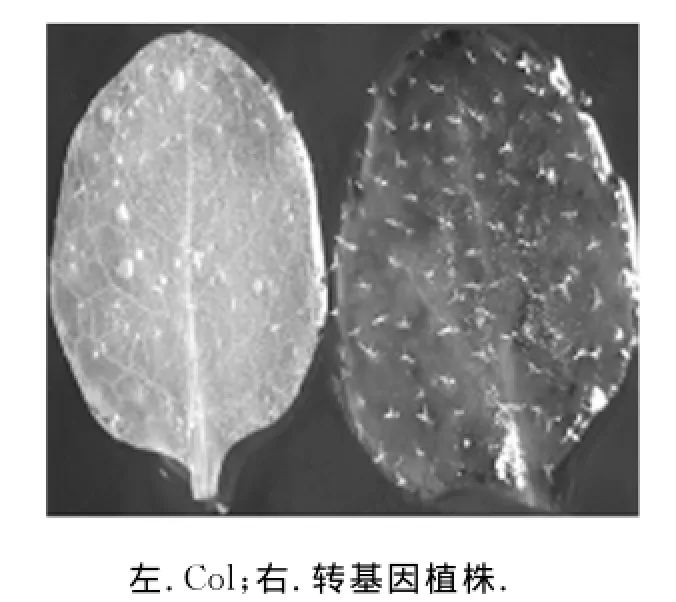
图8 转基因植株GUS染色结果Fig.8 GUS staining of transgenic plants
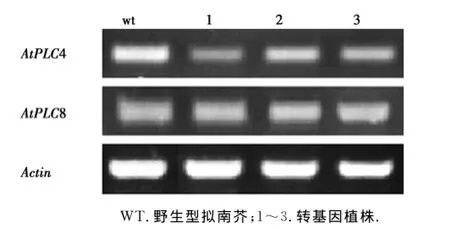
图9 转基因植株的RT-PCR检测Fig.9 Analysis of transgenic plants by RT-PCR
3 讨论
本实验中,RNAi载体构建的策略是利用同一条扩增序列分别正向和反向连接到p TCK303载体上(图1).操作时,首先连接到载体上的是反向目的片段,这一步的顺序问题是至关重要的.因为正向片段中包含SalⅠ,BamHⅠ位点,这2个酶切位点是用于连接反向片段的.如果先将正向片段与p TCK303载体进行连接,在连接反向目的片段时,用SalⅠ+BamHⅠ对中间载体进行酶切回收,会同时将正向目的片段一同切除,而先连接反向片段,正向片段的酶切不会影响已经连接到载体上的反向片段.按照这种策略,本课题组顺利地构建了RNAi载体p TCK303-PLC4i,使用不同的双酶切组合对其进行鉴定,结果如图5所示.泳道1为用SacⅠ+SpeⅠ双酶切载体p TCK303-PLC4i结果,切出与正向目的片段大小(478 bp)一致的条带;泳道2为SalⅠ+BamHⅠ双酶切鉴定结果,切出与反向目的片段大小(466 bp)一致的条带;泳道3为用SacⅠ+Bam HⅠ对载体进行双酶切,结果显示酶切产物条带大小与2条目的片段及Rice Intron长度(496 bp)总和(1 440 bp)一致.双酶切结果说明2个目的片段已经以正确的方式连接到p TCK303载体上,可以用于拟南芥的遗传转化.
RNAi作为基因特异性敲除的有效方法,已经广泛应用于动、植物基因功能研究.拟南芥基因组中的9个At PLCs基因亚型,不同亚型间可能存在着功能冗余,因此单个基因的突变不足以引起明显的表型变化,这对利用反向遗传学进行基因功能的研究带来困扰,因此,本室拟利用杂交结合RNAi的方法获得多个亚型的多基因突变体.本文采用RNAi的方法获得了AtPLC4亚型的基因表达缺陷突变株,该突变体将进一步用于表型和生理分析及与其他亚型的突变体进行杂交,探索AtPLCs基因家族的生物学功能.
[1] BERRIDGE M J.Inositol trisphosphate and calcium signaling[J].Nature,1993,361:315-325.
[2] RHEE S G,BAE Y S.Regulation of phosphoinositide-specific phospholipase C isozymes[J].Jour Biol Chem,1997,272:15045-15048.
[3] SHI J,GONZALES R A,BHATTACHARYYA M K.Characterization of a plasma membrane-associated phopshinosit-ide-specific phospholipase C from soybean[J].Plant Jour,1995,8:381-390.
[4] KOPKA J,PICAL C,GRAY J E,et al.Molecular and enzymatic characterization of three phosphoinositide-specific phospholipase C isoforms from potato[J].Plant Physiol,1998,116:239-250.
[5] PICAL C,KOPKA J,MULLER R B,et al.Isolation of c DNA clones for phosphoinositide-specific phospholipase C from epidermal peels(accession no.X95877)and guard cells(accession no.Y11931)of Nicotiana rustica(PGR 97-086)[J].Plant Physiol,1997,114:747-749.
[6] DOED P E,COURSOL S,SKIRPAN A L,et al.Petunia phospholipase C1 is involved in pollen tube growth[J].Plant Cell,2006,18(6):1438-1453.
[7] PAN Yanyun,WANG Xin,MA Ligeng,et al.Characterization of phosphatidylinositol-specific phospholipase C(PIPLC)from lilium daviddi pollen[J].Plant Cell Physiol,2005,46:1657-1665.
[8] HELLING D,POSSART A,COTTIER S,et al.Pollen tube tip growth depends on plasma membrane polarization mediated by tobacco PLC3 activity and endocytic membrane recycling[J].Plant Cell,2006,18(12):3519-3534.
[9] HUANG C H,CRAIN R C.Phosphoinositide-specific phospholipase C in oat roots:association with the actin cytoskeleton[J].Planta,2009,230(5):925-933.
[10] KOMIS G,GALATIS B,QUADER H,et al.Phospholipase C signaling involvement in macrotubule assembly and activation of the mechanism regulating protoplast volume in plasmolyzed root cells of Triticum turgidum[J].New Phytol,2008,178(2):267-282.
[11] NAKAMURA K,SANO H.A plasma-membrane linker for the phosphoinositide-specific phospholipase C in tobacco plants[J].Plant Signal Behav,2009,4(1):26-29.
[12] REGGIANI R,LAORETI P.Evidence for the involvement of phospholipase C in the anaerobic signal transduction[J].Plant Cell Physiol,2000,41(12):1392-1396.
[13] VOSSEN J H,ABD A,FRADIN E F,et al.Identification of tomato phosphatidylinositol-specific phospholipase-C(PI-PLC)family members and the role of PLC4 and PLC6 in HR and disease resistance[J].Plant J,2010,62(2):224-239.
[14] ZHENG Shuzhi,LIU Yuliang,LI Bing,et al.Phosphoinositide-specific phospholipase C9 is involved in the thermotolerance ofArabidopsis[J].Plant J,2012,69(4):689-700.
[15] MUNNIK T,TESTERINK C.Plant phospholipid signaling:“in a nutshell”[J].J Lipid Res,2009(Supp):260-265.
[16] WANG Xuemin.Plant Phospholipases[J].Annu Rev Plant Physiol Plant Mol Biol,2001,52:211-231.
[17] TASMA I M,BRENDEL V,WHITHAM S A,et al.Expression and evolution of the phosphoinsitide-specific phospholipase C gene family inArabidopsisthaliana[J].Plant Physiol Biochem,2008,46(7):627-637.
[18] CLOUGH S J,BENT A F.Floral dip:a simplified method forAgrobacterium-mediated transformation ofArabidopsisthaliana[J].Plant J,1998,16:735-743.
[19] JEFFERSON R A.Assaying chimeric in genes in plants:the GUS gene fusion system[J].Plant Mol Biol,1987(5):387-405.
(责任编辑:赵藏赏)
Construction and genetic transformation of RNAi vector for AtPLC4 gene of Arabidopsis
YUE Zhi-liang,MA Hui-bin,HOU Ming-yu,PAN Yan-yun
(College of Life Sciences,Agricultural University of Hebei,Baoding 071001,China)
The RNAi vector forAtPLC4 gene was constructed and transformed intoArabidopsis.Twenty transgenic plants were identified by Hyg resistance screening and PCR.Three lines that lower expression ofAtPLC4 gene were identified by RT-PCR.Those transgenic plants will be used to study the functions of theAtPLCgene family by the reverse genetics method.
Arabidopsisthaliana;AtPLC4;RNAi
O658
A
1000-1565(2012)04-0393-06
2012-01-06
国家自然科学基金资助项目(30570993);河北省自然科学基金资助项目(C2008000292)
岳智亮(1986-),男,山西平定人,河北农业大学在读硕士研究生.
潘延云(1963-),女,山东章丘人,河北农业大学教授,博士,主要从事植物分子生物学研究.E-mail:pyycell@163.com
