芸薹属重要作物的比较基因组研究进展
2012-09-19谭永俊唐志东
罗 宝,谭永俊,唐志东
(湖南农业大学油料作物研究所,长沙410128)
比较基因组学就是利用共同的标记构建图谱或对不同物种基因组相应部分(或全部)区域进行测序,从结构关系、相对位置、基因数目等方面进行分析,以揭示不同物种之间的基因家族成员数目和排列的异同[1]。1930年,基于种间杂种的细胞遗传学分析建立了芸薹属作物的禹氏三角,遗传作图分析进一步验证了6个芸薹属栽培种的亲缘关系[2~5]。三角关系的建立,显示了芸薹属各物种及其变种的亲缘关系,从而为种间基因交流及远缘杂交提供了理论基础,指导并加快了重要芸薹属作物的遗传改良。笔者主要总结了甘蓝型油菜(B.napus,AACC,2n=38)与白菜(B.rapa,AA,2n=20)、甘蓝(B.oleracea,CC,2n=18)3个物种间的基因组比较研究进展。
1 A基因组和C基因组比较细胞遗传研究
Morinaga等[6]早期对芸薹属二倍体和异源四倍体作物单倍体杂交的减数分裂过程研究发现,3个基因组有共同的起源,比较发现A、C基因组的染色体的亲缘关系更近[7]。Harrison和 Heslop-Harrison[8]利用FISH技术从甘蓝和白菜中获得的近着丝粒区域的两端DNA分子的重复序列做探针,对3个基本种和3个复合种进行了定位分析,结果表明DNA重复序列均可以定位在白菜和甘蓝的大部分染色体上,可是信号在黑芥上则显得很微弱,同样表明了A、C基因组的染色体的亲缘关系更近。Snowdown等[9~11]用GISH 技术发现在甘蓝型油菜中很难区分A基因组和C基因组,说明两个亚基因组有很高的同源性;同期使用FISH技术,用rDNA做探针,分别与白菜、甘蓝、甘蓝型油菜有丝分裂的中期的基因组杂交,发现在3个物种中每条染色体都能发现信号,并且在复合体中可以区分两个亚基因组,但统计发现,复合体中的信号数少于两个基本种的信号数之和,由此表明,两个基因组自然加倍后发生了染色体变异;之后使用5srDNA和25srDNA做探针,结合DAPI染色法,可更加清晰地将两个基因组在复合种中区分开来,并且还可以看到二者的同源区域,与二倍体比较发现在复合种中有些rDNA增加或者丢失了一些拷贝。很显然细胞学观察不仅费时费力,也很容易出错,因此必须用简单可靠的技术方法来推动比较基因组学的研究。
2 A和C基因组比较遗传作图进展
20世纪90年代末期,相关学者使用同套类型的标记对十字花科芸薹族的3个二倍体物种的基因组及由它们之间衍生出来的多倍体基因组进行了比较作图研究,结果如表 1。Slocum 等[12]、Song等[13]、Landry 等[14]最早分别以 RFLP 标记发表了甘蓝、白菜、甘蓝型油菜的遗传图谱,图谱之间发现同一个基因内35%~40%的探针有多个座位。使用同套探针在不同物种进行遗传作图发现,白菜和甘蓝之间在碱基序列和基因拷贝数之间存在很高的相似性,相关比较作图还发现这两个二倍体物种与甘蓝型油菜对应的亚基因组有很高的共线性[5,15]。Lagercrantz[16]研究发现3个基因组有广泛的共线性,并且同一基因组内每个区段一般可以在其他物种找到几个相应的同源区段。Parkin等认为A基因组与C基因组亲缘关系最近,与B基因组最远(图1)。以上结果阐明了芸薹属A、C基因组在个别物种的保守性,也揭示基因组进化过程中发生的多倍化事件和广泛的基因重排现象。
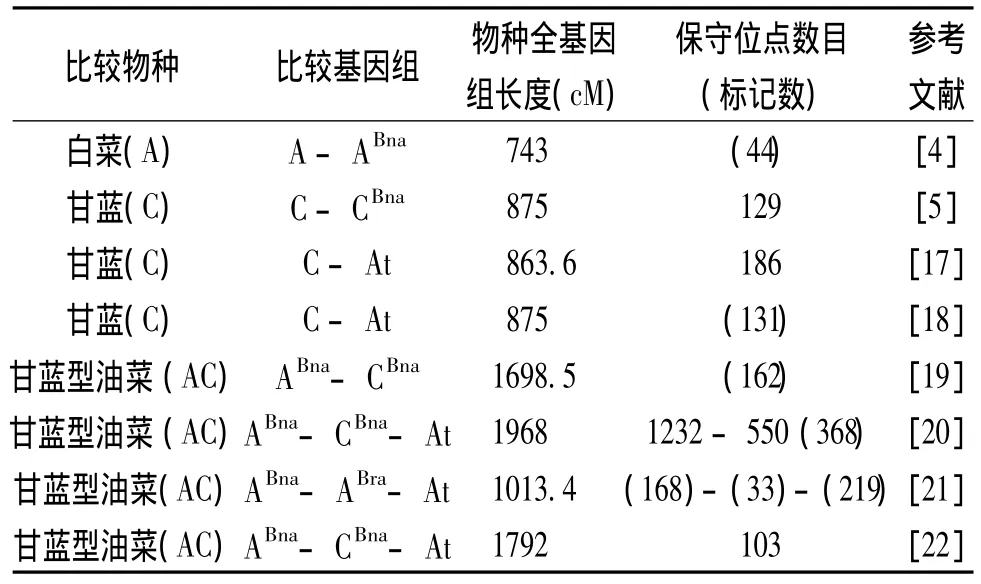
表1 芸薹属白菜、甘蓝与甘蓝型油菜比较作图的部分研究结果
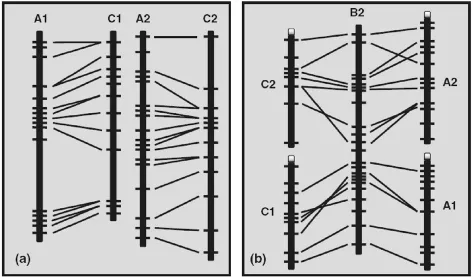
图1 A、B、C基因组比较遗传作图常见的3种情况
作为模式植物的拟南芥与芸薹属作物同一家族,亲缘关系非常近。2000年,拟南芥基因组完成测序[24],从芸薹属种间比较作图延伸到与拟南芥基因组的比较做图,这样有利于加快研究芸薹属作物的基因组结构特征和进化过程。通过对甘蓝与拟南芥的比较作图研究,Kowalski等[25]发现,尽管自从甘蓝与拟南芥分化后发生了染色体大规模的重排,两者之间发现了11个3.7~49.6 cM不等的保守区段,甘蓝基因组的29.9%区域及拟南芥基因组的24.6%区域都是由这些保守区段组成的,在甘蓝基因组中还发现部分拟南芥区段有3个重复,还发现至少有9个倒位和17个易位存在。进一步研究发现,甘蓝和拟南芥之间存在广泛的共线性,但共线性区域的重复次数目前还无法确定,大部分研究认为共线性区域在二倍体基本种中倾向于有3个拷贝,从而大致认为芸薹属由一个六倍体祖先演化而来[18,19,26,27]。Schranz 等[28]在芸薹属作物比较研究中发现,拟南芥(Arabidopsis lyrata,n=8)和白芥(Capsella rubella,n=8)更适合作芸薹属比较基因组研究的祖先种,这一点与Parkin等[20]结果不一致。他们将拟南芥基因组划分24个保守区段,古染色体组在进化过程中发生了染色体融合、易位及倒位、片段重排等事件,形成了染色体数为5且含一套24区段的拟南芥,以及染色体数为10并且含大约3套24区段的白菜。Parkin等[20]使用经过测序的1 000个多个RFLP标记构建好的甘蓝型油菜图谱与拟南芥基因组进行比对,发现若将拟南芥基因组划分21个区段,则每个区段平均有6个重复区段在油菜遗传图谱上。孙思龙[29]对白菜、甘蓝和拟南芥比较研究发现,与白菜类似,甘蓝基因组同样发生过三倍化事件,在甘蓝中找到3个甚至更多个重复的拟南芥保守区段。二者比较研究表明,甘蓝基因组部分染色体结构与白菜类似,这暗示着与白菜分开进化之后甘蓝基因组并未发生大的结构变化,另一部分染色体结构与白菜相比却发生了大的变化,这极有可能与甘蓝中染色体的断裂、融合重排有关。最近,Wang等[30]对甘蓝的物理图谱与拟南芥的比较发现,67%的重叠群能够与拟南芥基因组比对成功,其中96%在拟南芥基因组的常染色质区域,3.5%(含重复序列)在近着丝粒区域,另外发现在拟南芥与甘蓝分化后,染色体组可能发生过39次断裂事件。Jiang等[31]通过将白菜的BCA重叠群锚定到甘蓝型油菜的遗传图谱进而分别从结构和功能分析了白菜A基因组和甘蓝型油菜An之间的共线性关系,研究表明祖先种在共线性区域每个区段在A基因组都有3份重复,此外在两物种中,只发现5%的基因组区域发生了染色体倒位、染色体间和染色体内的易位事件,由此认为二者在基因组结构上并未发生较大的变化。
3 比较作图与重要农艺品质性状QTL定位
Osborn等[32]在白菜、甘蓝型油菜和拟南芥开花基因的比较定位上发现,油菜两个主效QTL同样能够在白菜中找到,并认为甘蓝型油菜需要春化的基因来源于白菜;与拟南芥基因组的比较QTL定位发现,两个主效QTL区域与拟南芥5号染色体顶端区域呈共线性关系,而此区域包含了FLC,FY和CO基因,另一对QTL区间则与拟南芥4号染色体同源,由此认为芸薹属控制开花的基因可能是拟南芥调控开花的基因(FLC和FRI)。Long等[33]在研究油菜控开花期QTL的定位中发现,拟南芥的28%花期同源基因可以通过电子定位到甘蓝型油菜QTL的置信区间内,9%的同源基因则可比对到置信区间邻近两侧,另外发现一个BnFLC10分别在春油菜种植环境和冬油菜种植环境分别定位到N10和N16的QTL簇中。Zhao等[34]利用拟南芥脂肪酸代谢途径的同源基因开发特异标记定位到油菜的遗传图谱上,比较作图找到33个共线性区域,将近一半的同源位点落在线性区域内,结合含油量的QTL定位发现,14个与脂肪酸代谢相关的基因位点落在QTL的置信区间内,这表明拟南芥与油菜在脂肪酸调控位点上广泛保守,Arti Sharma等[35]认为在拟南芥、白菜、蓖麻和大豆中控制脂肪酸合成的主效基因如硬脂酰脱氢酶、FatB、FAD2、FAD3、DGAT存在着丰富的结构变异,这也导致这些物种在脂肪酸组成上的差异。Ding等[36]在甘蓝型油菜产量和磷吸收等相关性状的QTL定位发现,161个与拟南芥同源的基因与定位的45个QTL有关,其中4个与磷吸收有关的基因标记定位在QTL置信区间。Xia等[37]在甘蓝型油菜两个隐性核不育基因BnMs4和BnRf的精细定位研究发现,拟南芥3号染色体上存在与这两个基因微观共线性的区域。Wang等[38]在全基因组水平比较白菜与拟南芥中硫苷代谢途径相关基因的同源性,分别发现了白菜中102个候选基因和拟南芥52个直系同源基因与硫苷合成有关,并且大部分基因在白菜只有一个拷贝,与其他报道一样,两者之间在硫苷代谢途径存在广泛共线性。在抗病研究方面,Saito 等[39]和 Suwabe 等[40]在白菜抗根肿病QTL定位中发现,2个QTL(Crr1,Crr2)位置都含有拟南芥4号染色体的一个同源区段,而这个区段包含了大量的基因簇MRCs,另外一个QTL则位于拟南芥3号染色体一个臂的端部,以上结果表明白菜抗根肿病基因与拟南芥基因同源。Mayerhofer等[41]在抗黑胫病基因的定位研究中发现,LmR1和ClmR1两个基因在甘蓝型油菜基因组中有严格的基因组间和基因组内重复,通过微观共线性发现,位于第7连锁群的位点与拟南芥1号染色体末端区域同源,只是在拟南芥中该区域发生了倒位。其他方面,Muangprom等[42]在研究白菜类作物矮化基因定位发现,位于白菜基因组R6连锁群末端的DWF2基因与拟南芥2号染色体的顶端区域具有同源性。至今已有数以百计的控制各种农艺品质性状QTL被精确定位在高质量的遗传图上(http://www.gramene.org/qtl/),但是由于目前芸薹属作物只有白菜基因组刚完成,各实验室利用的分子标记没有通用性,构建的图谱密度也不均一,利用的群体也都集中于常规人工群体(如F2、RIL、NIL),因此比较QTL定位还只是初步阶段。基于此,一方面应该利用通用性标记构建更加高密度高质量的遗传图谱,同时利用拟南芥和白菜基因组信息,构建白菜全基因组物理图谱和转录图谱,进而加快白菜基因组信息的注释和结构分析,另一方面则对数量性状遗传模型进行研究,结合生物数学,环境因子作用,生化代谢机制,对各种数据类型都能分析并且准确易操作的QTL定位软件,可以预见,芸薹属作物的分子设计育种将迎来一个崭新活跃的时机。
4 小结与展望
早期芸薹属物种细胞学的研究奠定了物种间亲缘关系的基本构架,使各物种间远缘杂交能够获得理论支撑。20世纪90年代开始,分子标记技术的应用在物种遗传作图、分子标记辅助育种、QTL定位、比较作图、染色体结构与进化等研究方面发挥了巨大的作用,2000年拟南芥基因组的测序[43]、2011年4月琴叶拟南芥的全基因组测序[44]、2011年白菜的基因组完成测序并释放全部信息[45],都提供了海量的基因组信息资源。常见的芸薹属基因组资源网站有:英国Rothamsted Research(http://www.brassica.info/);中国农业科学院花卉蔬菜所(http://brassicadb.org/brad/index.php);英国 John Inner Center(http://brassica.bbsrc.ac.uk/);美国JCVI中心(http://www.jcvi.org/cms/404)。加快这些资源向栽培作物转移则变得极为紧要,现全球范围内芸薹属学者正在合作的项目“国际芸薹属基因组项目”(Multinational Brassica Genome Project,简称 MBGP),“芸薹科植物图谱比对项目”(Brassicaceae Map Alignment Project,简称BMAP)正是顺应这些问题来开展研究的。可以预见,芸薹属作物将和禾本科植物水稻一样,重要的染色体结构进化机制得到解析,重要的农艺品质性状基因被克隆,为芸薹属分子育种改良提供坚固的平台。
[1] 李媛媛,傅廷栋,马朝芝.芸薹属植物比较基因组学研究进展[J].植物学通报,2007,24(2):200-207.
[2] Parkin I,Sharpe A,Keith D,et al.Identification of the A and C genomes of amphidiploid Brassica napus(oilseed rape)[J].Genome,1995,38(6):1122 -1131.
[3] Panjabi P,Jagannath A,Bisht NC,et al.Comparative mapping of Brassica juncea and Arabidopsis thaliana using Intron Polymorphism(IP)markers:homoeologous relationships,diversification and evolution of the A,B and C Brassica genomes[J].BMC Genomics,2008,9(1):113.
[4] Suwabe K,Morgan C,Bancroft I.Integration of Brassica A genome genetic linkage map between Brassica napus and B.rapa[J].Genome,2008,51(3):169 -176.
[5] Bohuon EJR,Keith D,Parkin IAP et al.Alignment of the conserved C genomes of Brassica oleracea and Brassica napus[J].Theoretical and Applied Genetics,1996,93(5):833-839.
[6] Morinaga T.Interspecific hybridization in Brassica.IV.The cytology of F1 hybrid of B.carinata and some other species with 10 chromosomes[J].Cytologia,1931,3:77-83.
[7] Attia T,R9bbelen G.Cytogenetic relationship within cultivated Brassica analyzed in amphihaploids from the three diploid ancestors[J].Canadian Journal of Genetics and Cytology,1986,28(3):323-329.
[8] Harrison G,Heslop-Harrison J.Centromeric repetitive DNA sequences in the genus Brassica[J].Theoretical and Applied Genetics,1995,90(2):157-165.
[9] Snowdon R,K9hler W,Friedt W,et al.Genomic in situ hybridization in Brassica amphidiploids and interspecific hybrids[J].Theoretical and Applied Genetics,1997,95(8):1320-1324.
[10] Snowdon R,K9hler W,KÖhler A.Chromosomal localization and characterization of rDNA loci in the Brassica A and C genomes[J].Genome,1997,40(4):582 -587.
[11] Snowdon R,Friedrich T,Friedt W,et al.Identifying the chromosomes of the A-and C-genome diploid Brassica species B.rapa(syn.campestris)and B.oleracea in their amphidiploid B.napus[J].Theoretical and Applied Genetics,2002,104(4):533-538.
[12] Slocum M,Figdore S,Kennard W,et al.Linkage arrangement of restriction fragment length polymorphism loci in Brassica oleracea[J].Theoretical and Applied Genetics,1990,80(1):57-64.
[13] Song K,Suzuki J,Slocum M,et al.A linkage map of Brassica rapa(syn.campestris)based on restriction fragment length polymorphism loci[J].Theoretical and Applied Genetics,1991,82(3):296-304.
[14] Landry BS,Hubert N,Etoh T,et al.A genetic map for Brassica napus based on restriction fragment length polymorphisms detected with expressed DNA sequences[J].Genome,1991,34(4):543-552.
[15] Hoenecke M,Chyi Y.Comparison of Brassica napus and B.rapa genomes based on restriction fragment length polymorphism mapping[A].Proceedings of the 8thInternational rapeseed congress[C].1988:1102 -1107.
[16] Lagercrantz U,Lydiate DJ.Comparative genome mapping in Brassica[J].Genetics,1996,144(4):1903.
[17] Lan TH,Paterson AH.Comparative mapping of quantitative trait loci sculpting the curd of Brassica oleracea[J].Genetics,2000,155(4):1927.
[18] Lukens L,Zou F,Lydiate D,et al.Comparison of a Brassica oleracea genetic map with the genome of Arabidopsis thaliana[J].Genetics,2003,164(1):359 -372.
[19] Parkin IAP,Sharpe A,Lydiate D.Patterns of genome duplication within the Brassica napus genome[J].Genome,2003,46(2):291-303.
[20] Parkin IAP,Gulden SM,Sharpe AG,et al.Segmental structure of the Brassica napus genome based on comparative analysis with Arabidopsis thaliana[J].Genetics,2005,171:765 -781.
[21] Xu J,Qian X,Wang X,et al.Construction of an integrated genetic linkage map for the A genome of Brassica napus using SSR markers derived from sequenced BACs in B.rapa[J].BMC Genomics,2010,11(1):594.
[22] Wang J,Lydiate DJ,Parkin IAP,et al.Integration of linkage maps for the Amphidiploid Brassica napus and comparative mapping with Arabidopsis and Brassica rapa[J].BMC Genomics,2011,12(1):101.
[23]Parkin I.Chasing ghosts:comparative mapping in the Brassicaceae[J].Genetics and Genomics of the Brassicaceae,2011:153-170.
[24]Arabidopsis GI.Analysis of the genome sequence of the flowering plant Arabidopsis thaliana[J].Nature,2000,408:796.
[25] Kowalski SP,Lan TH,Feldmann KA,et al.Comparative mapping of Arabidopsis thaliana and Brassica oleracea chromosomes reveals islands of conserved organization[J].Genetics,1994,138(2):499.
[26] Lan TH,DelMonte TA,Reischmann KP,et al.An EST-enriched comparative map of Brassica oleracea and Arabidopsis thaliana[J].Genome Research,2000,10(6):776.
[27] Babula D,Kaczmarek M,Barakat A,et al.Chromosomal mapping of Brassica oleracea based on ESTs from Arabidopsis thaliana:complexity of the comparative map[J].Molecular Genetics and Genomics,2003,268(5):656-665.
[28] Schranz ME,Lysak MA,Mitchell-Olds T.The ABC’s of comparative genomics in the Brassicaceae:building blocks of crucifer genomes[J].Trends in Plant Science,2006,11(11):535-542.
[29] 孙思龙.白菜,甘蓝与拟南芥比较基因组分析[D].北京:中国农业科学院,2011.
[30] Wang X,Torres M,Pierce G,et al.A physical map of Brassica oleracea shows complexity of chromosomal changes following recursive paleopolyploidizations[J].BMC Genomics,2011,12(1):470.
[31] Jiang C,Ramchiary N,Ma Y,et al.Structural and functional comparative mapping between the Brassica A genomes in allotetraploid Brassica napus and diploid Brassica rapa[J].Theoretical and Applied Genetics,2011,123(6):927-941.
[32] Osborn T,Kole C,Parkin I,et al.Comparison of flowering time genes in Brassica rapa,B.napus and Arabidopsis thaliana[J].Genetics,1997,146(3):1123.
[33] Long Y,Shi J,Qiu D,et al.Flowering time quantitative trait loci analysis of oilseed Brassica in multiple environments and genomewide alignment with Arabidopsis[J].Genetics,2007,177(4):2433.
[34] Zhao J,Huang J,Chen F,et al.Molecular mapping of Arabidopsis thaliana lipid-related orthologous genes in Brassica napus[J].Theoretical and Applied Genetics,2011,24(2):1 -15.
[35] Sharma A,Chauhan RS.In Silico identification and comparative genomics of candidate genes involved in biosythesis and accumulation of seed oil in plants[J].Comparatine and functional genomics,2012,(1):1155.
[36] Ding G,Zhao Z,Liao Y,et al.Quantitative trait loci for seed yield and yield-related traits,and their responses to reduced phosphorus supply in Brassica napus[J].Annals of Botany,2012.
[37] Xia S,Cheng L,Zu F,et al.Mapping of BnMs4 and BnRf to a common microsyntenic region of Arabidopsis thaliana chromosome 3 using intron polymorphism markers[J].Theoretical and Applied Genetics,2012,124(7):1193 -1200.
[38] Wang H,Wu J,Sun S,et al.Glucosinolate biosynthetic genes in Brassica rapa[J].Gene,2011,487(2):135-142.
[39] Saito M,Kubo N,Matsumoto S,et al.Fine mapping of the clubroot resistance gene,Crr3,in Brassica rapa[J].Theoretical and Applied Genetics,2006,114(1):81 -91.
[40] Suwabe K,Tsukazaki H,Iketani H,et al.Simple sequence repeat-based comparative genomics between Brassica rapa and Arabidopsis thaliana:the genetic origin of clubroot resistance[J].Genetics,2006,173(1):309.
[41] Mayerhofer R,Wilde K,Mayerhofer M,et al.Complexities of chromosome landing in a highly duplicated genome:toward map-based cloning of a gene controlling blackleg resistance in Brassica napus[J].Genetics,2005,171(4):1977.
[42] Muangprom A,Osborn T.Characterization of a dwarf gene in Brassica rapa,including the identification of a candidate gene[J].Theoretical and Applied Genetics,2004,108(7):1378-1384.
[43] Kaul S,Koo HL,Jenkins J,et al.Analysis of the genome sequence of the flowering plant Arabidopsis thaliana[J].Nature,2000,408:796.
[44] Hu TT,Pattyn P,Bakker EG,et al.The Arabidopsis lyrata genome sequence and the basis of rapid genome size change[J].Nature Genetics,2011,43(5):476 -481.
[45] Wang X,Wang H,Wang J,et al.The genome of the mesopolyploid crop species Brassica rapa[J].Nature Genetics,2011,43(10):1035-1039.
