杨树锈菌表达序列微卫星分析及EST-SSR标记开发1)
2012-08-08万志兵王小龙管宏伟仰皖旭尹佟明张新叶
万志兵 王小龙 管宏伟 仰皖旭 尹佟明 张新叶
(江苏省杨树品种改良与种质创新重点实验室(南京林业大学),南京,210037) (湖北林业科学研究院)
杨树(Populus)是我国适生范围较广的树种之一,是重要的速生丰产工业用材、四旁绿化、农田林网和防风固沙树种。随着我国木材用量的不断增加,杨树大面积推广,全国杨树人工林的栽培面积超过700万hm2,其中大部分为单一品种构成的纯林。虽然大面积纯林可为工业利用提供品质较为均一的原材料,但在营林方面暴露出一些弱点,特别是容易导致病虫害的暴发流行,造成严重减产。杨树锈病是杨树生产中的一种主要病害[1]。夏秋季节,叶片出现大量黄色锈粉物(夏孢子),严重发生时会布满整个叶片和林带,导致提早落叶,严重影响树木光合和生长。防治该病经济、有效和环境安全的方法是利用抗病品种。然而,由于杨树锈菌具有高度的变异性和种植抗病品种造成的选择压力,杨树锈菌种群变异很快,毒力结构十分复杂[2]。杨树锈菌致病变异性一般是通过接种不同杨树品种进行毒力检测来评价[3]。用致病性作为病原菌遗传变异的指标存在一些缺点,如费工费时、需要很大的温室空间、受温湿度及接种技术等的影响大,而且由于抗病性和感病性的分类受主观性因素影响,表型变异往往不稳定。因此,需要利用更加有效的手段来进行杨树锈菌的鉴定和研究杨树锈菌的变异情况。
分子标记技术已成为植物病原真菌遗传变异研究和检测的重要手段。一些基于DNA的分子标记,如rDNA-ITS,RFLP,RAPD等技术也已应用于杨树锈菌的鉴定、遗传变异及多样性、无毒基因作图等研究[4-8]。与RFLP及RAPD等分子标记相比,SSR是一种更加有效的标记技术。SSR标记虽然在一些植物和动物中已被广泛开发和应用,但在真菌中却开发得很少[9-12]。简单序列重复(simple sequence repeats,SSRs)或微卫星(microsatellites)是以1~6个碱基为基元的串联重复序列,它们普遍存在和随机分布于大多数真核生物的基因组中,重复数具有高频率的变异,这种专化位点的多态性可以十分容易地通过设计特异引物进行PCR扩增检测[13]。SSR标记具有多态性高、多等位性、共显性、容易用PCR检测、重复性高等特点,是一种理想的分子标记,但开发SSR标记需要事先知道基因组序列信息,对没有基因组序列的物种而言,开发SSR标记是昂贵和低效的[13-14]。随着表达序列标签(expressed sequence tag,EST)技术的发展,EST数据库为开发SSR标记提供了快速、有效和廉价的途径[15]。EST是通过对cDNA文库中随机挑取的克隆进行大规模测序所获得的cDNA的5’或3’端序列,长度一般为150~500 bp。近年来随着功能基因组的研究,大量的EST数据收录于NCBI(National Center for Biotechnology Information,美国国家生物信息技术中心),为EST-SSR标记的开发提供了丰富的序列资源。由于EST-SSR标记开发是基于基因组中的表达序列,因此标记要近缘物种间有很高的通用性[16-19],再加上现在高度发展的信息技术为从EST序列中开发SSR标记提供了丰富的软件[20],这一切都使得EST-SSR标记成为当今热门的分子标记技术。一些植物如葡萄、小麦的EST-SSR标记已开发利用[21-24]。在植物病原真菌中,Kaye 等人[9]也开发了少量水稻稻瘟病菌(Magnaporthe grisea)的EST-SSR标记,朱振东等[25]报道了从大豆疫霉菌(Phytophthora sojae Kaufmann&Gerdemann)的EST序列中开发的EST-SSR标记。
迄今,在GenBank中已有6万多条杨树锈菌EST序列。本研究利用GenBank数据库中杨树锈菌的EST序列,对这些序列进行了拼接和组装,在此基出上,开展了这些序列所含微卫星的查找,并对杨树锈菌基因组中基因转录序列所含微卫星重复序列的特征和组成情况进行了分析,然后根据所发现的微卫星位点设计SSR引物,并随机选取了部分引物对进行了试验分析,以期为杨树锈菌的研究提供有价值的遗传工具和分子标记资源。
1 材料与方法
1.1 杨树锈菌EST-SSR序列的检索
从GenBank的dbEST数据库(http://www.ncbi.nlm.nih.gov/dbEST/index.html)以 FASAT 格式下载杨树锈菌EST序列。序列拼接的计算机为Power Edge T710(内存60G),使用的软件为GS De nove Assembler,拼接参数采用默认值。采用C语言编辑的 Sputnik(C.Abajian,University of Washington)程序进行所有重复单元长度为2~5个碱基的微卫星的查找,程序默认阈值最小Score设定为9。
不同重复单元微卫星密度的计算:D=N/L。D为不同重复单元微卫星密度;N为各重复单元微卫星数量;L为杨树锈菌EST序列拼接片段总长。
将所有回纹重复序列及其互补的序列归为一类,如 AAG 基序代表所有 AAG、AGA、GAA、CTT、TCT和TTC的SSR。据此,2核甘酸重复序列归为4类,3核甘酸重复序列归为10类,4核甘酸重复序列归为33类,5核甘酸重复序列归为102类。
1.2 EST-SSR 引物设计
应用引物设计软件Primer Premier 5.0和Oligo6.0设计杨树锈菌EST-SSR引物。Primer Premier 5.0主要用于引物的自动检索,其基本检索条件为:EST序列长度大于100 bp;EST-SSR序列的开始和结束分别距5’和3’端不少于20 bp;引物长度18~24 bp;GC含量40% ~60%;理论退火温度(Tm)40~60℃;预计PCR扩增产物长度控制在100~500 bp;尽量避免引物二级结构dimer、hairpin、falseprimer以及连续6个碱基配对的出现。Oligo 6.0主要用于对检索到的引物进行评价分析:正反引物的Tm值相差不能超过5℃,5’端Tm稍微高于3’端Tm。设计的引物上传至网址 ftp://202.119.210.12,读者可与作者联系下载引物数据库。我们从表中随机挑选了30对引物(数据库中红色标记部分)用于引物的试验验证,这些引物由上海捷瑞生物工程有限公司合成。
1.3 杨树锈菌DNA提取与EST-SSR引物验证
以南京林业大学树木园采集的杨树锈菌为材料,利用北京天恩泽基因科技有限公司玻璃珠法柱式真菌DNAout提取锈菌总DNA。
PCR扩增反应体系采用10 μL反应体系,其中含有 1×buffer,Mg2+2.5 mmol/L,dNTPs 0.2 mmol/L,引物0.4 μmol/L,Taq 酶1.0 U,模板DNA 50 ng。反应程序为:94℃预变性85 s,94℃变性35 s,引物退火温度60 s,72℃延伸90 s,30个循环;72℃终延伸10 min,4℃保存。扩增产物用1.0%的琼脂糖电泳检测,120 V电压电泳40 min后,用Gel Image System Tanon 2500凝胶成像系统成像。有扩增产物的样品采用6%的聚丙烯酰胺凝胶电泳和银染法显色检测DNA条带的多态性。
2 结果与分析
2.1 微卫星丰度及分布密度分析
在杨树锈菌的64498条EST中,经过拼接得到1998个拼接片段,进行2~5个核苷酸重复的SSR位点搜索发现604个SSR位点,拼接片段含有SSR位点的频率为30.23%,总共有420个拼接片段含有SSR位点,平均1个片段含有1.44个SSR位点,1个片段含有的SSR位点数最多的是6个,69.05%的拼接片段只含有1个SSR位点,见表1。拼接片段的总长度是444 907 bp,平均736.6 bp含有1个SSR位点。

表1 拼接片段中含有SSR位点数
本研究查找的EST序列中所含微卫星为二核苷酸至五核苷酸重复,其中三核苷酸重复的数量最多,占全部SSR的44.70%,为优势重复基序。二核苷酸、四核苷酸、五核苷酸所含微卫星所占比例分别为 17.05%、17.72%、20.53%。不同重复单元微卫星由于数量差异较大,所以分布密度的变化也很大,见表2。
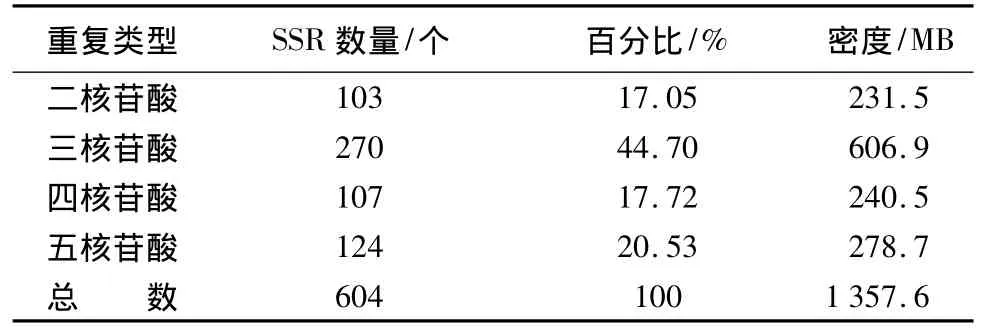
表2 不同长度重复单元微卫星所占百分比及分布密度
2.2 微卫星优势重复单元碱基组成
在二碱基重复类型中,AG基序重复的数量最多,共41 个,占39.8%;其次是 AC,33 个,占32.0%;AT基序重复的数量也达28个,占27.2%;而CG基序只有一个。在10种三碱基重复类型中,AGT基序数量最多,共69个,占25.6%;其次为AAG(22.6%)和ACT(14.4%),其他重复基序则相对较少。四碱基重复类型共20种,AAAG基序数量最大,共19个占17.76%;AATC(14.95%)、AAAC(14.02%)次之;这3种类型基序占了四碱基重复类型总量的46.73%。24种五碱基重复类型中AAAAC基序数量最大,共21 个占16.9%;其次为AAAAG(12.9%),见表3。从以上分析可以看出,在杨树锈菌的EST中,检测到的四和五碱基重复的SSR的种类都不多,可能是在不同物种中,SSR出现的种类存在偏好。
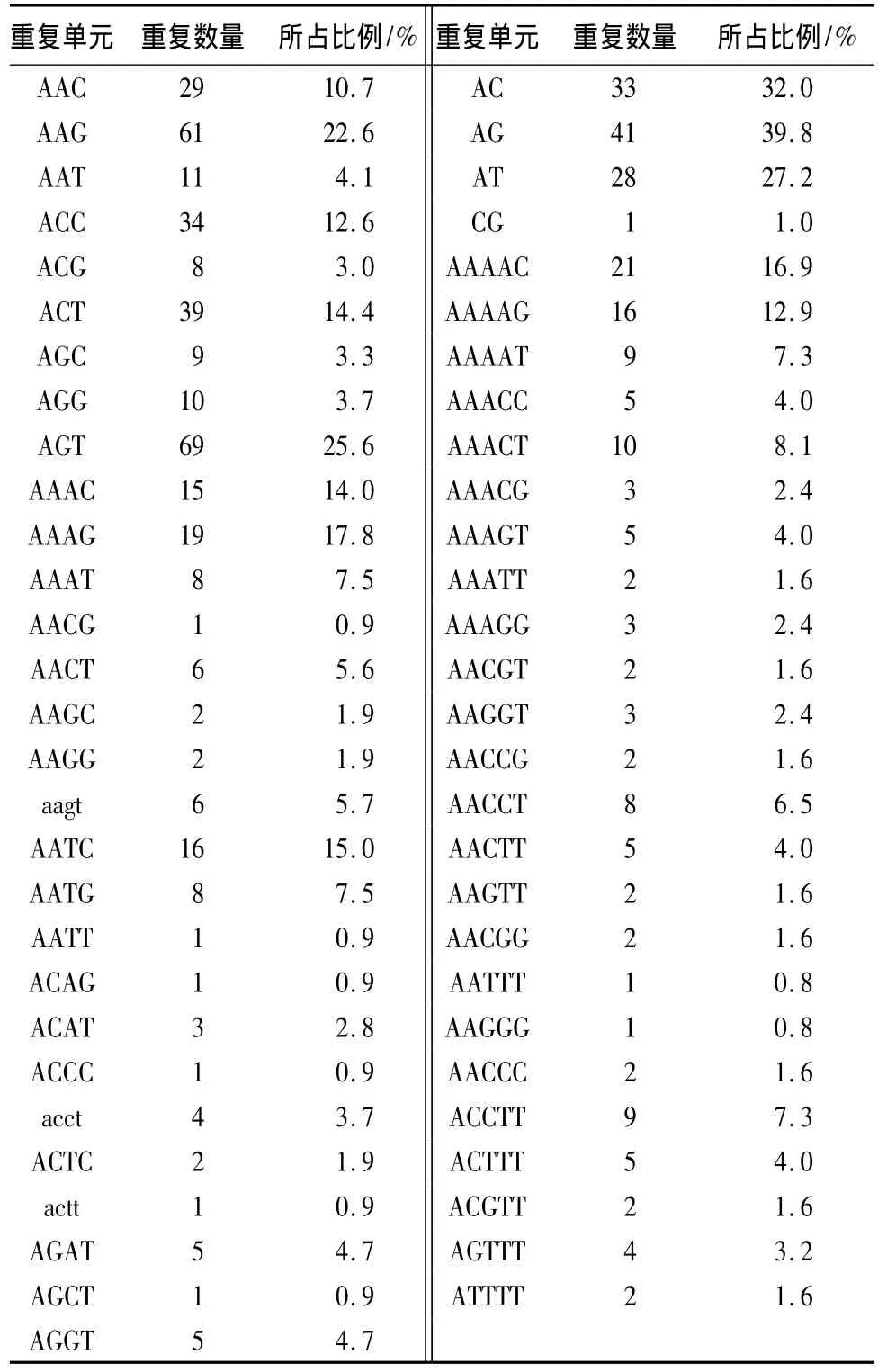
表3 不同长度重复单元微卫星中各重复单元含量比例
总体而言,在杨树锈菌EST中最丰富的微卫星类型是三碱基重复微卫星,其次为四碱基重复微卫星,最主要的优势重复序列分别是AGT、AAG以及AG类。而在四碱基、五碱基重复类型中,(AAAN)n和(AAAAN)n分别比同类型其他组合的基序数量大。这些处于优势数目的重复拷贝类型富含A和T碱基。
2.3 微卫星长度分布及变异分析
本研究中杨树锈菌EST序列中微卫星的平均长度为26.07 bp,最长的为58 bp,最短11 bp。长度大于20 bp的微卫星仅占总数的15.07%(表4)。
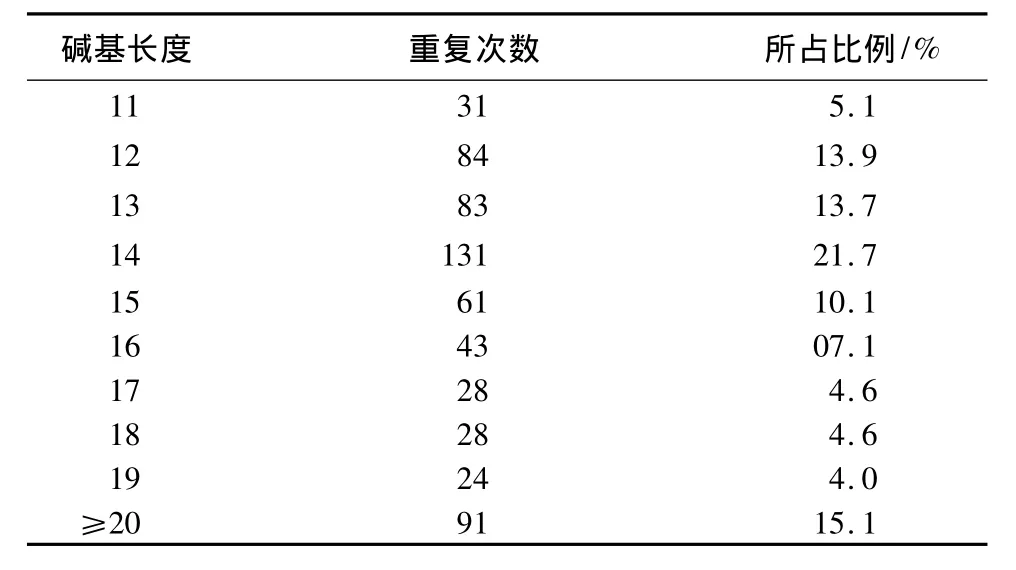
表4 杨树锈菌微卫星长度分布及不同长度微卫星频率
基序重复次数的变异引起位点长度的变化是产生EST-SSR位点多态性的主要原因。进一步对不同长度重复单元的杨树锈菌微卫星的长度变异情况进行了分析(表5),从扇形部分数量的变化可以看出,三碱基重复微卫星重复单元的变化次数显著高于其他重复类型微卫星的重复单元变化次数。长度变异程度最高的为三碱基重复微卫星,变异程度最低的为五碱基重复微卫星。
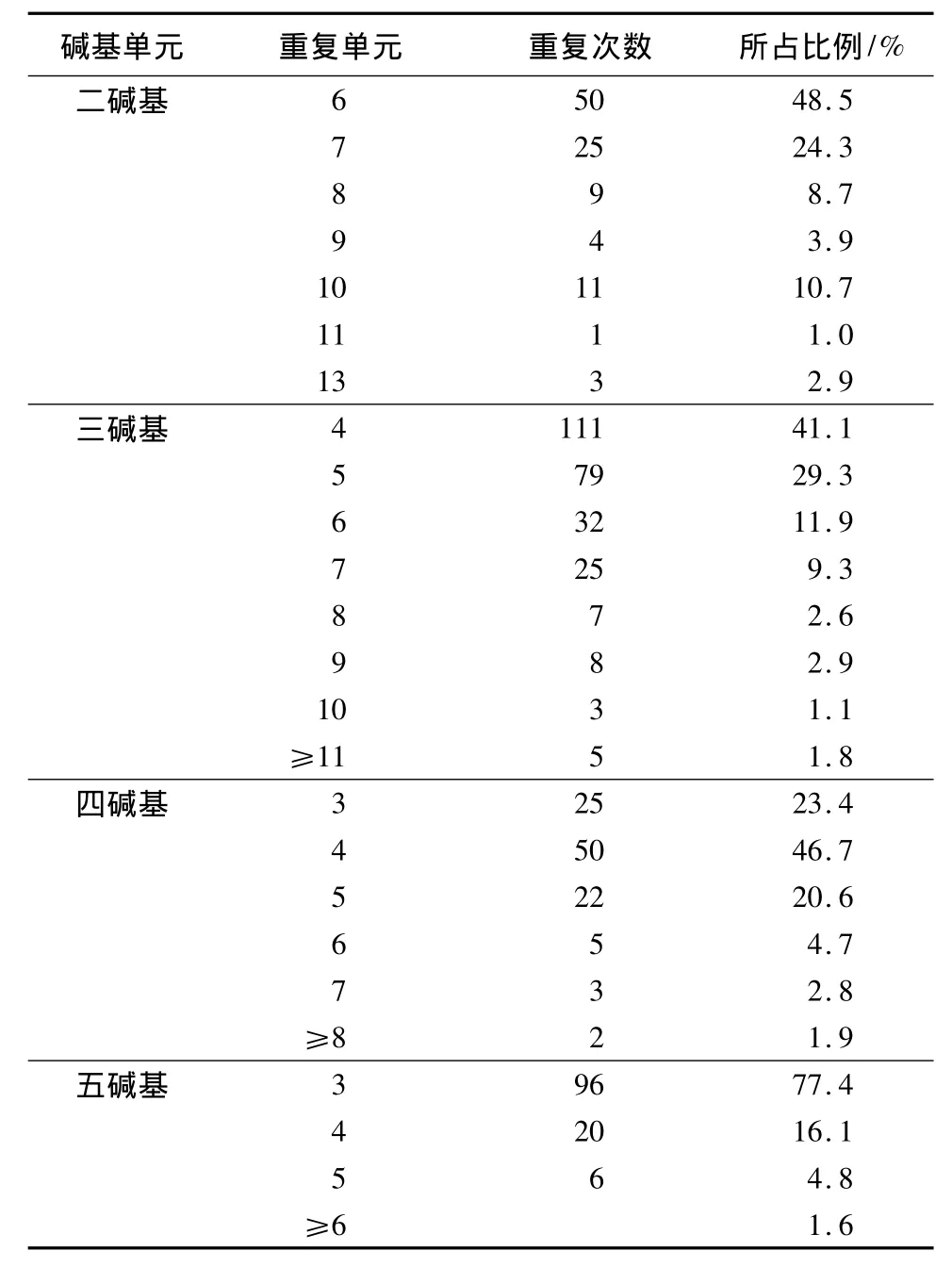
表5 不同长度重复单元微卫星变异情况
2.4 杨树锈菌EST-SSR引物设计及筛选
根据SSR位点,共设计了455对SSR引物,随机抽取了30对SSR引物,对杨树锈菌基因组DNA进行扩增,除3对引物未产生扩增产物外,其余27对引物(90.0%)均扩增出SSR特征条带(图1)。
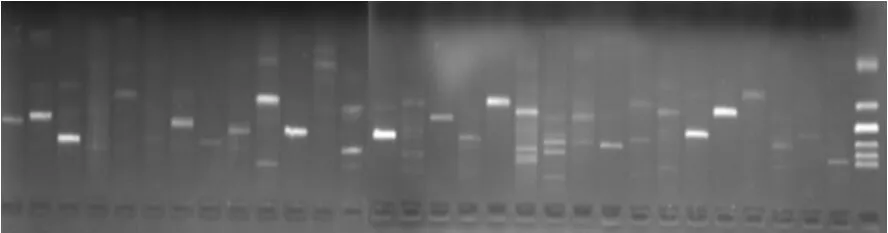
图1 部分引物对杨树锈菌基因组DNA扩增产物
在27个功能性引物对中,有19个引物对扩增出1条产物带,其中有13个引物对的扩增产物在预期片段大小范围之内,6个引物对的扩增产物大于预期片段大小;有8个引物对扩增出2条或2条以上条带,都存在扩增出在预期片段大小范围之内的的条带。共有21个引物对扩增出在预期片段大小范围之内的的条带,占功能性引物对的77.8%。
3 结论与讨论
对64 498条杨树锈菌EST进行筛选组装,拼接成1 998个片段,共发掘了604个SSR位点,筛选组装的杨树锈菌片段序列平均每736.6 bp含有1个SSR。采用同样的参数和分析方法,其他真菌中,香菇的EST序列平均每25.924 kb含有1个SSR位点[26],大豆疫霉菌的EST序列平均每8.9 kb含有1个SSR[25],表明杨树锈菌含有SSR的频率很高,SSR十分丰富。由于微卫星序列是生物基因组中变异最为迅速的一类序列,杨树锈菌表达序列中微卫星含量很高,将导致杨树锈菌基因有很高的变异频率,我们推测微卫星序列是杨树锈菌基因变异迅速的重要驱动力。在鉴定的杨树锈菌EST-SSR中以三核苷酸重复基元最丰富,占鉴定总数的50.1%。本研究的结果与以前报道的在一些植物中的研究结果一致[23,27-28]。在基因编码区主要存在三核苷酸重复类型可能是三核苷酸重复数的变异不会引起移码突变,从而减少突变的压力。Gao等人[23]发现许多三核苷酸与具有重要功能的基因相关,如CCG重复涉及许多基因的功能,如胁迫抗性、转录调控、信号传导等。
Rota L R等人[29]曾提出若EST-SSR数目足够大和无偏倚性,则4种不同的碱基随机组合将产生4种二核苷酸、10种三核苷酸、33种四核苷酸和102种五核苷酸基本重复基元类型。杨树锈菌ESTSSR分析的结果表明,杨树锈菌不同重复基元出现的类型表现出很大的偏倚性。二核苷酸中GC重复单元只出现一次,远远低于其他重复单元。在桃树[30]、拟南芥[31]、大豆[23]等植物的研究中也表明存在很大的偏倚性,没有出现GC重复基元。
按照Temnykh[32]的划分,按照长度将微卫星分为两大类:长度大于等于20 bp的SSR和长度大于12 bp但小于20 bp的SSR。与第二类SSR相比,第一类SSR序列长度更长,具有更高的突变率,因而更不稳定。本实验对604个微卫星长度进行分析发现,杨树锈菌EST序列所含微卫星在长度上存在极显著的变异,微卫星长度从11~58 bp不等,微卫星平均长度为26.07 bp。微卫星主要为长度较短的重复序列,长度大于20 bp的序列仅占微卫星总数的15.07%。在简单重复序列中,序列越长变异就越大,其多态性就可能越丰富,就越有利于EST-SSR标记的开发[33-34]。在杨树锈菌 EST-SSR 位点中重复最长的是五核苷酸中长度为58的一段SSR。EST-SSR的多态性主要是由于重复长度和重复次数造成的[35]。SSR一般认为是由于DNA复制过程中聚合酶瞬时脱离、碱基错配引起的,杨树锈菌EST序列中SSR重复基元的重复次数为1~111,最大的重复次数出现在三核苷酸中。詹少华等[36]首次统计了SSR基元重复次数变异范围,推测SSR基元重复次数变异范围越大,分子标记的多态性越高,而且也认为SSR重复基元重复次数变异的发生也与SSR形成有关。研究杨树锈菌EST-SSR重复基元的重复次数对于杨树锈菌EST-SSR引物扩增多态性分析有重要的意义。
随着植物病原菌基因组研究的发展,多种不同类型植物病原菌的EST已被开发。从EST数据库中发掘SSR标记,为植物病原菌SSR标记的发展提供了一种便利的途径。SSR一般都有2~5个等位位点,在植物病原菌研究中不但可以用于种群遗传多样性、近缘种的比较及系谱研究[37-38],而且还可用于不同寄主的分离物之间的比较[39-40],或是菌株、小种之间的比较[37-38,41]。本研究从杨树锈菌EST中发掘SSR标记,证明了这种途径的有效性和高效性。虽然EST-SSR标记与其他一些从非编码区分离的标记相比在种内检测的多态性水平要低,但他们检测的是基因组表达部分的变异,是真正与性状连锁的标记,这将可能对决定重要表型性状的等位基因进行直接鉴定。另外,EST-SSR标记具有很高的通用性,一旦开发,可以在近缘种(属)中应用。因此,杨树锈菌EST-SSR标记的开发利用,为杨树锈菌及近缘种属的鉴别、遗传变异等研究提供了重要的标记资源。杨树锈菌EST-SSR的开发也将会促进杨树抗锈病育种的研究。
[1] 曾士迈,杨演.植病病害流行学[M].北京:农业出版社,1984.
[2] Mendgen K,Hahn M.Plant infection and the establishment of fungal biotrophy[J].Trends Plant Sci,2002,7(8):352-356.
[3] 刘春海,曹支敏,余仲东.中国落叶松-杨栅锈菌生理小种鉴定[J].西北林学院学报,2008,23(2):105-108.
[4] 余仲东,刘小勇,曹支敏.松杨栅锈菌的ITS序列和微卫星分析[J].中国农业科学,2006,39(6):1159-1165.
[5] 余仲东,张振,曹轩峰,等.松杨栅锈菌两菌群RAPD特异序列的标记转换[J].中国农业科学,2009,42(1):349-354.
[6] 陈建珍.落叶松-杨栅锈菌DNA多态性和同工酶分析[D].杨凌:西北农林科技大学,2006.
[7] 魏明.落叶松-杨栅锈菌生理小种的SCAR标记[D].杨凌:西北农林科技大学,2007.
[8] Gérard pr,Husson C,Pinon J,et al.Comparison of genetic and virulence diversity of Melampsora larici-populina populations on wild and cultivated poplar and influence of the alternate host[J].Phytopathology,2006,96(10):1027-1036.
[9] Kaye C,Milazzo J,Rozenfeld S,et al.The development of simple sequence repeat markers for Magnaporthe grisea and their integration into an established genetic linkage map[J].Fungal Genet Biol,2003,40(7):207-214.
[10] Sirjusingh C,Kohn M K.Characterization of microsatellites in fungal plant pathogen,Sclerotinia sclerotiorum[J].Mol Ecol Notes,2001(1):267-269.
[11] Barnes I,Gaur A,Burgess T,et al.Microsatellite markers reflect intra-specific relationships between isolates of the vascular wilt pathogen Ceratocystis fimbriata[J].Mol Plant Pathol,2001,510(2):319-325.
[12] Chen W,Shi X,Chen Y C.Microsatellite markers and clonal genetic structure of the fungal pathogen Phialophora gregata[J].MycolRes,2002,106:194-202.
[13] Zane L,Bargelloni L,Patarnello T.Strategies for microsatellite isolation:A review[J].Mol Ecol,2002,11(1):1-16.
[14] Dutech C,Enjalbert J,Fournier E,et al.Challenges of microsatellite isolation in fungi[J].Fungal Genet Biol,2007,44(7):933-949.
[15] Squirrell J,Hollingsworth P M,Woodhead M,et al.How much effort is required to isolate nuclear microsatellites from plants?[J].Mol Ecol,2003,12(2):1339-1348.
[16] Varshney R K,Graner A,Sorrells M E.Genic microsatellite marers in plants:features and applications[J].Trends Biotechnol,2005,23(1):48-55.
[17] Tang J F,Gao L F,Cao Y S,et al.Homologous analysis of SSRESTs and transferability of wheat SSR-EST marers across barley,rice and maize[J].Euphytica,2006,151(1):87-93.
[18] Slate J,Hale M C,Birkhead T R.Simple sequence repeats in zebrafinch(Taeniopygia guttata)expressed sequence tags:a new resource for evolutionary genetic studies of passerines[J].BMC Genomics,2007,8(1):52-64.
[19] Yin T M,Zhang X Y,Gunter L E,et al.Microsatellite primers resource developed from the mapped sequence scaffolds of Nisqually-1 genome[J].New Phytol,2009,181(2):498-503.
[20] Ewell E,Robinson A,Savage D,et al.SSR Primer and SSR Taxonomy Tree:Biome SSR discovery[J].Nucleic Acids Res,2006,34(2):656-659.
[21] Scott K D,Eggler P,Seaton G,et al.Analysis of SSRs derived from grape ESTs[J].Theor Appl Genet,2000,100:723-726.
[22] Gupta P K,Rustgi S,Sharma S,et al.Transferable EST-SSR markers for the study of polymorphism and genetic diversity in bread wheat[J].Mol Genet Genomics,2003,270(5):315-323.
[23] Gao L,Tang J,Li H,et al.Analysis of microsatellites in major crop assessed by computional and experimental approaches[J].Mol Breed,2003,12(11):245-261.
[24] Eujayl I,Sledge M K,Wang L,et al.Medicago truncatula ESTSSRs reveal cross-species genetic markers for Medicago spp[J].Theor Appl Genet,2004,108(4):414-422.
[25] 朱振东,霍云龙,王晓鸣,等.从大豆疫霉菌ESTs中发掘SSR标记[J].科学通报,2004,49(17):1749-1754.
[26] 刘春滟,李南羿,张玉琼.香菇EST2SSR标记的开发及应用[J].食用菌学报,2010,17(2):1-6.
[27] Cardle L,Ramsay L,Milbourne D.Computational and experimental characterization of physically clustered simple sequence repeats in plants[J].Genetics,2000,156(3):847-854.
[28] Kantety R V,Rota M L,Matthews D E,et al.Data mining for simple sequence repeats in expressed sequence tags from barley,maize,rice,sorghum,and wheat[J].Plant Mol Biol,2002,48(4):501-510.
[29] Rota L R,Kantety R V,Yu J K.Nonrandom distribution and frequencies of genomic and EST-derived microsatellite markers in rice,wheat and barley[J].BMC Genomics,2005,6(2):23.
[30] Jung S,Abbott A,Jesudurai C,et al.Frequency type distribution and annotation of simple sequence repeats in Rosaceae ESTs[J].Funct Integr Genomics,2005,5(4):136-143.
[31] 范三红,郭蔼光,单丽伟,等.拟南芥基因密码子偏爱性分析[J].生物化学与生物物理进展,2003,30:221-225.
[32] Temnykh S,DeClerck G,Lukashova A,et al.Computational and experimental analysis of microsatellites in rice(Oryza sativa L.):frequency,length variation,transposon associations,and genetic marker potential[J].Genome Res,2001,11(7):1441-1452.
[33] Cho Y G,Ishii T,Temnykh S,et al.Diversity of microsatellites derived from genomic libraries and GenBank sequences in rice(Oryza sativa L.)[J].Theoretical and Applied Genetics,2000,100(5):713-722.
[34] Temnykh S,Park W D,Ayres N,et al.Mapping and genome organization of microsatellite sequences in rice(Oryza sativa L.)[J].Theoretical and Applied Genetics,2000,100(5):697 -712.
[35] 冯建明,郭绍贵,吕桂云,等.西瓜抗枯萎病相关EST-SSR的信息分析[J].华北农学报,2009,24(3):87-91.
[36] 詹少华,盛新颖,樊洪泓,等.大豆EST序列长度与SSR特性的关系[J].大豆科学,2009,28(2):204-209.
[37] Giraud T,Fournier E,Vautrin D,et al.Isolation of 44 polymorphic microsatellite loci in three host races of the phytopathogenic fungus Microbotryum violaceum[J].Molecular Ecology Notes,2002,31(2):142-146.
[38] Giraud T,Fournier E,Vautrin D,et al.Isolation of eight polymorphic microsatellite loci,using an enrichment protocol,in the phytopathogenic fungus Fusarium culmorum[J].Molecular Eology Notes,2002,31(2):121-123.
[39] Barve M P,Haware M P,Sainani M N,et al.Potential of microsatellites to distinguish four races of Fusarium oxysporum f.sp.Ciceri prevalent in India[J].Theoretical and Applie Genetics,2001,102(3):138-147.
[40] Field D,Wills C.Abundant microsatellite polymorphism in Saccharomyces cerevisiae,and the different distributions of microsatellites in eight prokaryotes and S.cerevisiae,result from strong mutation pressures and a variety of selective forces[J].Proc Natl Acad Sci,1998,95(1A):1647-1652.
[41] Wostemeyer J,Kreibich A.Repetitice DNA elements in fungi(Mycota):impact on genomic architecture and evolution[J].Curr Genet,2002,41:189-198.
