压缩感知可以加速医用1H核磁代谢成像过程
2012-07-24SairamGeethanathMSHyeonManBaekPhDSandeepGanjiBEYaoDingMSElizabethMaherMDRobertSimsMDChanghoChoiPhD2dMatthewLewisPhDVikramKodibagkarPhD
Sairam Geethanath, MSHyeon-Man Baek, PhDSandeep K. Ganji, BEYao Ding, MSElizabeth A. Maher, MDRobert D. Sims, MDChangho Choi, PhD,,2dMatthew A. Lewis, PhD,,Vikram D. Kodibagkar, PhD,,
压缩感知可以加速医用1H核磁代谢成像过程
Sairam Geethanath, MS1Hyeon-Man Baek, PhD2aSandeep K. Ganji, BE2bYao Ding, MS2bElizabeth A. Maher, MD2cRobert D. Sims, MD2aChangho Choi, PhD2a,2b,2dMatthew A. Lewis, PhD1,2a,2bVikram D. Kodibagkar, PhD1,2a,2b
1. 德州大学阿灵顿分校与德州大学西南医学院联合生物医学工程研究生计划项目,美国 德克萨斯;2. 德州大学西南医学院a.放射学系; b. 放射学研究生计划项目; c. 内科与神经学系; d. 高级成像研究中心,美国德克萨斯
目的回顾性评测使用压缩感知(compressed sensing)技术进行不同程度的核磁成像过程(MR)加速,对所得结果保真度的影响。材料和方法本文中的实验手段均由所在院校伦理审查委员会(Institutional Review Board, IRB)审查通过,采集MR成像数据前获得了患者书面知情同意。本研究遵守《健康保险携带和责任法案》 ( Health Insurance Portability and Accountability Act,HIPAA)。回顾性压缩感知技术被用于10名实验对象的核磁共振成像过程中,并获取了以下体素数据:600个来自6名健康对象的脑部;163个来自两名脑瘤患者的脑部;36个来自于两名前列腺癌患者。研究人员在加速常数分别为2,3,4,5,10的条件下进行核磁信号的重建,并通过均方根误差(RMSE)法,代谢物图谱(包括胆碱,肌酸,N乙酰天冬氨酸[NAA],柠檬酸)以及统计学分析(包括基于单个体素的配对t检验,代谢图谱上不同变量的单因素误差分析,以及加速重建后与原始变量的比值)进行评估。结果使用不超过10的加速常数,重建核磁信号的保真度很高,RMSE很低(<0.05)。在加速常数不超过5的情况下,重建信号的平均生化信号强度和生化热点位置与原信号非常接近,二者之间没有统计学意义上的显著差异。在加速常数不超过5的重建信号中,胆碱/NAA以及(胆碱+肌酸)/柠檬酸的比值与原始信号并无显著差异,在部分加速常数为10的重建信号中上述条件亦成立。结论本文阐明了一种最高能够将采样时间降低80%,同时保持信号信息量的医用1H核磁生化成像过程。
核磁成像(MR)技术正在越来越广泛地被应用于临床 诊疗过程中[1-3]。相关研究表明,恶性前列腺和脑部肿瘤均会表达高量的胆碱水平[4]。另有研究表明,脑部肿瘤会表达较低水平的N乙酰天冬氨酸和肌酸信号,因此可以使用胆碱 /NAA 比例作为肿瘤的生化指标[4-7]。与前者类似,对于前列腺肿瘤的诊断也可以使用(胆碱 +肌酸)/柠檬酸比例作为生化指标。研究表明,在恶性前列腺肿瘤中,柠檬酸水平会降低,而胆碱水平会升高[4]。
多体素(二维/三维)核磁共振成像相对于单体素核磁共振技术的优势已被广泛认知[7-8]。使用该技术,医护人员可以获取更优秀的肿瘤定位信息,并可以观察到形变病变区域以外的异常表象。但是多体素核磁技术的主要技术缺陷——增长的采样时间——是限制其临床应用的主要因素[7-9]。压缩感知技术[10-11]提供了一种创新性的利用在适当变换域(如小波变换域)下信号密度来对k-空间进行低密度取样的技术,利用该技术可以降低重建过程对核磁信号数据点的需求数量。此技术已经在脑部核磁共振成像、核磁共振血管造影[10]、径向成像[11]、心脏电影成像[12],以及其他动态对比高亮核磁共振成像手段[13]中得到广泛应用。压缩感知技术应用于核磁成像中,因为核磁信号数据在小波变换域的多个频率和空间维度上均比较稀疏,变化也较小。过去的研究中,基于小波的分析手段,包括信号量化波形修正和去噪技术,均被用于核磁共振成像[14-17]。研究人员通过使用小波技术手段,利用核磁数据在此变换域中频率和空间维度上的信号稀疏,已经实现了快速核磁共振成像[18-19]。本研究中,研究人员回顾性评测了压缩采样技术重建信号的保真程度。
1 材料与方法
本研究回顾性地展示了6位健康志愿者,2位脑瘤(恶性神经胶质瘤)患者及 2 位前列腺癌患者的二维1H 核磁成像数据,各项协议均经大学的伦理审查委员会批准,并获得了志愿者及患者的书面知情同意。本研究遵守 HIPAA 法案。所得磁共振参数详见表1。
1.1 核磁共振成像数据及采样
原始数据归一化至 0~1,在 k 空间中沿相位维度对该数据进行低密度采样,采样模式由不同的加速因子(2,3,4,5,10)以及采样密度决定。采样模式为二维平面分布函数加权的随机采样,使得k空间中心附近采样密度高于 边 缘 部位[11]。二 维 平 面分布函数 的 中 心抽样范围取决于加速因子的倒数(加速因子越大,充分抽样区域的面积越小)。
1.2 信号重建
将抽样 k空间全部设定为 0,并经过逆式傅立叶转换来获取对核磁共振数据的预估。核磁共振信号重建由对下述代价函数进行凸优化完成(Lustig 等人[11])。
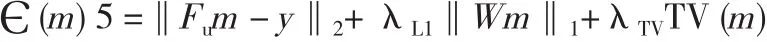
该 信 号 重 建 过 程 通 过 Matlab(MathWorks,Natick,Mass)提供的非线性共轭梯度算法完成,其中m代表核磁共振数据,Fu代表傅立叶转换运算符,y代表已测 k空间数据,W代表微波转换运算符,TV 代表总变差运算符,‖1和 ‖2为 L1 和 L2 各自的基准运算符 ;λL1和 λTV为基于L1和总变差各自的正规化参数。本研究中,研究人员使用Daubechies 小波变换技术[20]对 ky维度上所有像素点的 kx-t数据矩阵进行编译。λL1和 λTV的正规化值通过实验分别定为 0.001 和 0.005。通过对所获数据进行 8 次迭代操作,实验人员得以获取并比对代价函数的具体数值。
1.3 错误度量标准
信号重建误差由下列公式计算所得的均方根误差(RMSE)决定 :
其中N为一帧磁共振成像数据组中数据点的总和,y为完整的 k 空间的重建数据,y'为低密度取样 k 空间的重建数据。因为所用数据已经经过正态化至 0~1,所以上述方根误差等于通用的正态方根误差。
1.4 后期处理
磁共振成像数据组受基于 Java 的磁共振用户界面中的下列最少处理步骤的影响 (jMRUI; http://www.mrui.uab.es/mrui/ mrui_Overview.shtml)[21]: 用 衍 射 控 象 法 移 除 现 有 的 平 截 伪影, 基线校正, 时域的 Hankel-Lanczos 奇异值分解过滤的残余水峰和脂肪峰, 自动无序全相位校正, 使用量子估算算法(QUEST)[22]对相位核磁共振数据的实部进行分析,用以获取生化物质分布图,在该图中信号的强度代表 QUEST 算法结果中的谱图信号分布。本研究中,对于脑部磁共振成像的案例(健康志愿者与癌症患者),研究人员只分析了大脑内部区域的数据。进一步的分析中,关于健康大脑、脑瘤和前列腺癌患者的体素总数分别为600,163,36。计算相关比例时,原始数据或压缩传感重建数据中的分母为0的像素点的数值
(表示缺乏适合量子估值的适当应用手段)设定为0。

表1 磁共振数据采集参数
1.5 统计学分析
每组信号重建获取的生化组分分布图以及相对应的生化信号比值(胆碱 /NAA,健康大脑和脑瘤患者 ;胆碱 +肌酸 /柠檬酸,前列腺癌症患者)均通过配对t检验进行统计学分析(Excel; Microsoft,Redmond,Wash),并被用来与原始数据(加速因子为1)进行比对。此外,研究人员还通过单因子重复分析测试与 Bonferroni多组分比对测试来进一步评测各种加速比例对核磁信号重建质量的影响(GraphPad Prism ;GraphPad Software,La Jolla,Calif)。通过 Bonferroni检验获取的t 值数据被转化为P 值(Excel;Microsoft)。 如果P < 0.05,则认为该组比较中存在明显差异。
2 结果
图1和图2展示了典型健康大脑磁共振成像数据组的重建结果。图1展示了对解剖图中同一部位使用不同加速因子进行成像的核磁共振图谱。压缩感知的重建核磁信号与原始信号显示出了很高的相似度。用于评价成像质量的两个体素的位置由绿框和红框在解剖图中标出(图 2)。覆盖脑室的体素(绿框)数据与另一个体素(红框)相比,NAA,肌酸和胆碱(图 2右排)的浓度均有所降低,此结果与此前预期相符。图2中在不同加速因子下,重建的成像结果与原始数据相似。压缩感知重建数据可以保持原始数据的谱线形状,而且在高加速因子下,谱线更加平滑。图3展示了 NAA,肌酸和胆碱(谱峰覆盖面积)的代谢分布图谱和胆碱 /NAA 比值与加速因子之间的关系。原始和重建信号中的高亮区域出现在相似位置。而且在加速因子为10的时候,生化信号的强度比原始信号更强。
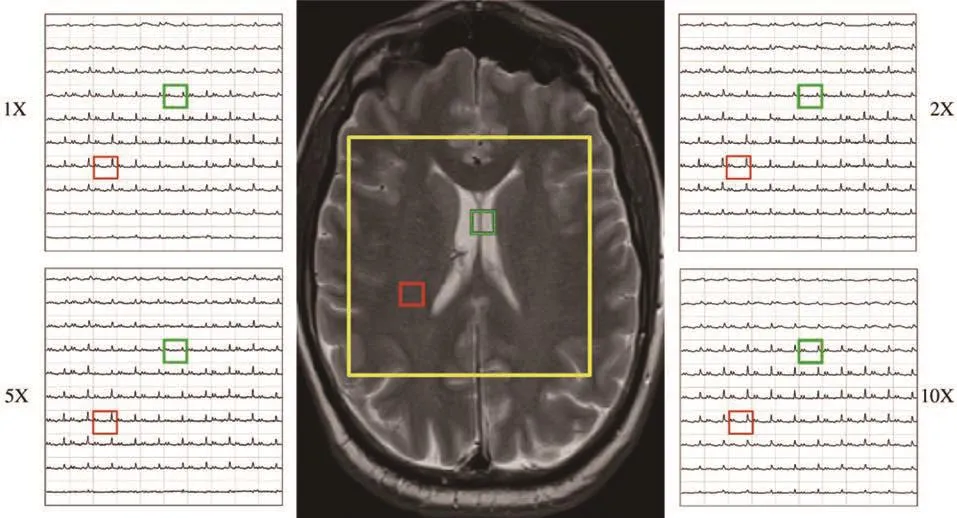
图1 1倍,2倍,5倍和10倍加速度下的典型大脑磁共振光谱成像数据组的数据网格的重建。中心部位:核磁共振成像显示值得注意的成像区域(黄框内)。2个位置(红框和绿框中的体素)的相关细节详见图2。
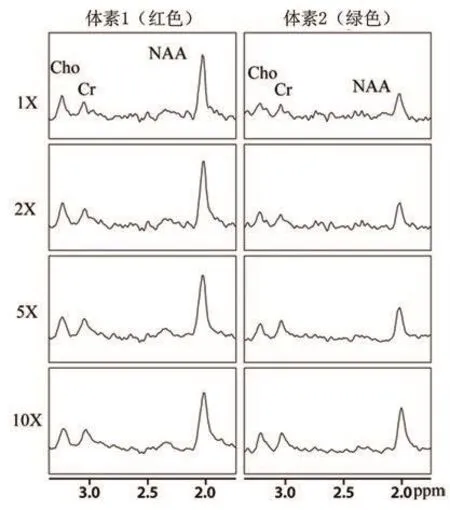
图2 在两个体素(图1中红色和绿色的框体范围)中采集到的加速因子为1,2,5和10的核磁共振光谱成像数据。y轴上下限为-0.25~1。Cho=胆碱,Cr=肌酸。
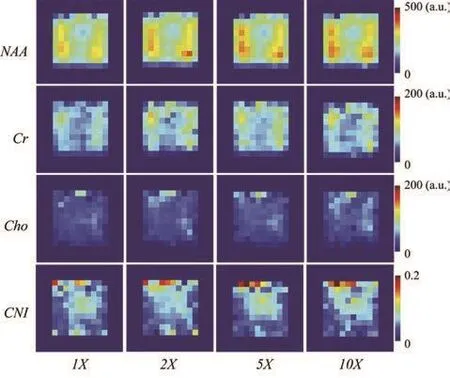
图3 加速因子为1,2,5和10的实验中健康大脑的NAA,肌酸,胆碱和胆碱/NAA指标的分布对照图。图1解剖图中黄框表明的区域为图谱上描绘的要注意的磁共振光谱成像区域。a.u.=arbitrary unit(任意单元)。
正常组织和恶性肿瘤组织的2个体素的原始数据和压缩感知重建信号的脑瘤核磁共振成像数据组详见图4。加速因子在 10以内的重建信号如实地保留了用来辨别正常脑组织与癌变组织的生化组分分布特点。图4为前列腺癌核磁共振成像数据组中代表正常组织与恶性肿瘤组织的2个体素的重建信号。压缩感知重建信号保持了正常前列腺与前列腺肿瘤病例的代谢物分布的特征。根据这些信号重建生成的生化组分分布图谱与加速因子之间的相关关系详见图 5a 和图 5b。对于脑瘤数据组,加速因子为 2 和 5 时,代谢产物分布图和胆碱 /NAA 指标图谱与原始信号拥有相同的特征,而加速因子为 10的压缩感知重建信号则相对于原始数据有较大的偏差。对于前列腺癌数据组,压缩感知重建信号中的生化组分分布图的信号强度有所降低,但(胆碱+肌酸)/柠檬酸指标图谱则保留了原始数据的所有特征,尤其是高亮区域的位置。

图4 加速因子为1,2,5和10的实验中下脑瘤(左)和前列腺癌(右)核磁共振光谱成像数据。在所有情况下,左列展示的是健康组织的数据,而右列展示的是肿瘤区域的数据。y轴上下限为-0.25~1。Cho=胆碱,Cit=柠檬酸,Cr=全部肌酸的甲基信号,Cr2=全部肌酸的亚甲基信号。
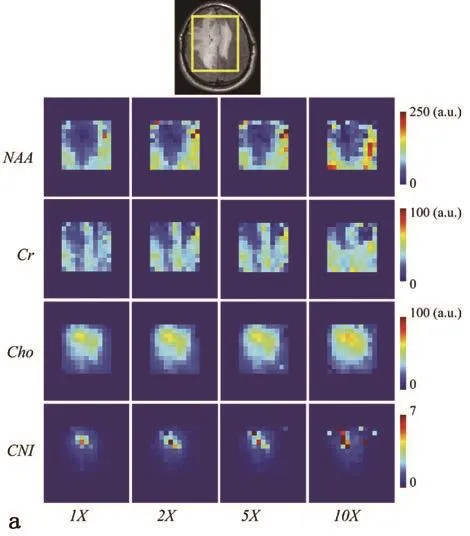
图5 a 加速因子为1,2,5和10的实验中脑瘤(NAA,肌酸,胆碱)和核磁共振光谱成像生化组分分布比较和比例图谱。
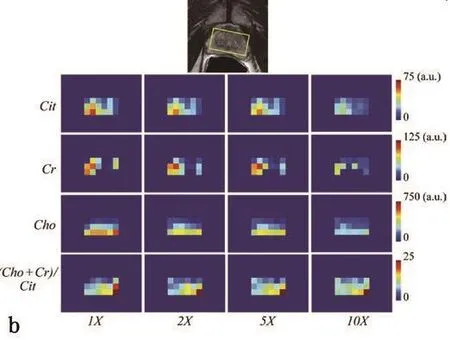
图5 b 前列腺癌(柠檬酸,肌酸,胆碱)比例图谱描绘了前列腺癌数据中脑瘤数据和(胆碱+肌酸)/柠檬酸的胆碱/NAA指标(CNI)。解剖图中的黄框指出感兴趣区域。a.u.=arbitrary unit(任意单元)。
加速因子 1,2,5,10 下的每项生化组分含量的平均值±标准差的计算结果详见表2。在加速因子为2和 5时,压缩感知重建信号的生化组分含量水平与原始信号比较一致,而加速因子为 10时,9种生化组分中有 6种较原始数据有着统计学上的显著差异。不同生化组分之间的比例指标分布图也与原始信号相似,这说明加速因子5以内的健康大脑/脑瘤数据和加速因子 10以内的前列腺癌的数据的关键生物指标值与原始信号相比没有显著差异。通过 Bonferroni检验分析单因子差异实验数据得出的P值详见表3。这些数值表明加速因子 1,2 和 5 之间联系紧密,而加速因子 10 和其它加速因子之间的关系显然没有那么紧密,从而导致P值的下降。在这 5 种加速度因子下的 3 种数据类型的 RMSE 量化信号重建误差详见图 6。与预期相同,RMSE 值随着加速因子的增大而增大,但是加速因子不超过 10的情况下,RMSE 维持在 0.05 以下。
3 讨论
本研究的目的是寻找一种可以在不丢失信息的情况下加速获取1H 磁共振图像的途径,以便将1H 核磁共振成像广泛应用于临床。通过使用压缩感知信号重建,我们证明了1H 核磁共振成像的采集时间可能节省 80% 甚至更多,且根据临床标准评估,几乎不损失任何重要信息。
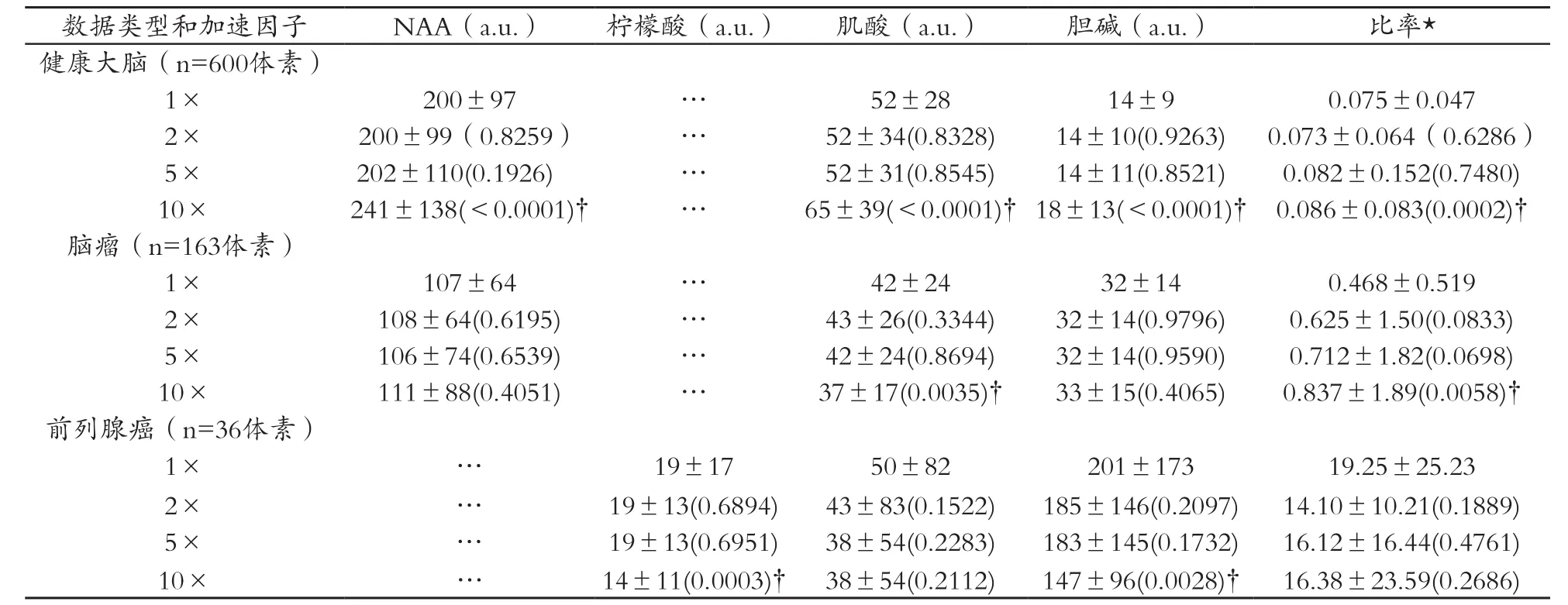
表2 健康大脑,脑瘤及前列腺癌代谢强度与体素数据比值作为加速函数
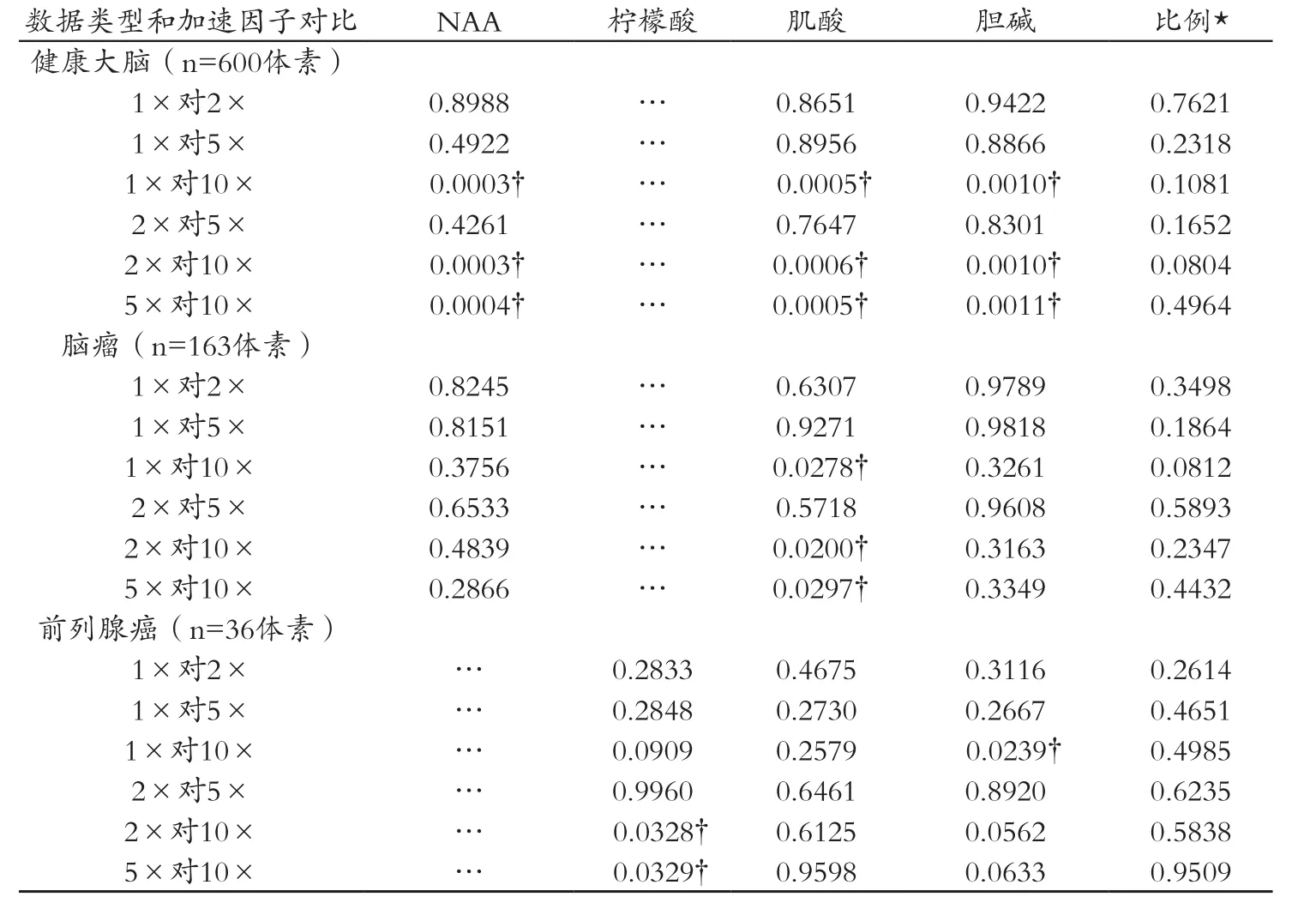
表3 Bonferroni对比试验后单因子差异分析产生的P值
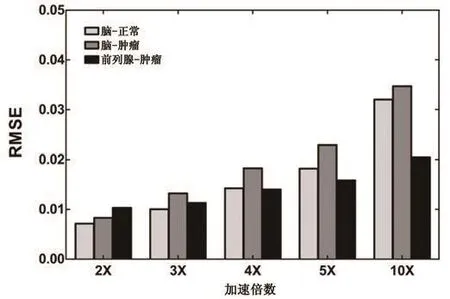
图6 加速倍数为2,3,4,5和10的实验中得出的核磁共振光谱成像数据RMSE值
重建光谱发出的噪音比原始光谱的小。这要归功于重建过程中的小波静噪功能和总变差因子的滤波效果。如上文所述,本研究中采用的信号重建方法能够产生相似于原始信号的生化组分分布图谱。核磁共振成像获取的生化组分分布信息对诊断和预后过程很重要,而本研究中的信号重建过程能够如实地保留这些信息。在原始信号中(加速因子 =1),即便是健康大脑的成像结果中,生化信号中均可观察到过高的标准差,而这一现象是源于头部线圈覆盖了整个头部,包括脑髓液部分(图 2~3)。除了对这些图谱的直接观测,配对t检验和单因子差异分析也可以被用来比较特定部位的原始信号和不同加速因子下压缩感知重建信号。当我们对加速度和原型之间的保真度和微小差异而非显著性差异进行测试的时候,将P值阈值设置于 0.05 时,比其更低的数据,如 0.01 更为严格,因为P 值小于 0.05 表明两种信号之间存在显著性差异,也就意味着信号重建算法的失败。本文的结果同样表明,对于各种生化指标,例如胆碱/NAA和胆碱/柠檬酸,信号重建并不导致其发生变化。因此,信号重建也同样维持了此类衍生参数的保真度。统计分析表明,加速因子为5的压缩感知重建信号与原始信号相似,而且没有统计学上的显著差异。然而在加速因子为 10 时,重建信号的误差及生物指标值的差异有所增加。另一方面,加速因子 10 以内的所有重建数据,其 RMSE 数值均在 0.05 以内。由于数据在处理之前已经正态化,这说明重建信号与原始信号之间的误差不超过5%。将核磁共振成像时域数据转换成生化组分分布是一个复杂的过程,涉及许多用户自定义的步骤,这些步骤会潜在地影响最终生化组分分布的图谱。研究人员可以通过比较原始数据和最少后处理的压缩感知重建信号数据来客观评估信号重建的保真度,但较高加速因子下的重建信号图谱中的差异可能是自动化整体位相校正中的差异造成的。大脑部位的生化组分分布图信号强度随加速因子的提高而增强,前列腺部位则反之。基于监控参数,网格尺寸以及k空间信噪比等参数的不同,上述两种趋势都可能出现,因此上述结果并无参考意义。对于给定的监控参数和加速因子并采用最优化程序的情况下,通过 RMSE 值来评价的试验结果误差随着加速因子的增加而增加。因此,对生化组分含量的估算数据将随加速因子的增加而逐渐偏离原始数据。从实验方法的实用性角度出发,文中所示方法测算所得的数据误差与加速因子成单增或单减关系(不管指标误差在越来越多的不充分采样中是增加还是减少),RMSE 值在最大为 10的加速因子下也被控制得很小。
其他几种快速化学位移成像和光谱学成像方法也已经发 展 成熟并付诸在 理 论[23]和 实 践[24]上进行了 相 关 对 比。上 述 技术中,回波平面 光 谱成像[25-26]是一 种在临床上[27]尚未被充分利用过的强大的光谱成像技术。本方法通过在一个频域和一个空间维度上同时进行回声平面采样来加速成像过程,从而达到相当于那个空间维度中像素点数的加速因子。例如,使用回波平面采样技术采集 1 个 16×16 的二维矩阵,所用时间会是传统化学位移成像手段的 1/16。然而,提高采样速度或许会以信噪比为代价,而且所得的成像结果也会受 Nyquist伪影[28]的影响。本文中使用的压缩感知技术中有选择地忽略了部分特定相位的数据,该步骤在二维和三维化学位移成像序列或回波平面成像技术中可以比较容易地完成。在进一步的研究中,可以通过设计快速成像序列,并将其与压缩感知技术进行联用,以获取更快的成像速度。最近的研究表明,在更多维度上使用压缩感知技术可以更有效地利用信号分布的稀疏性,并因此提高重建信号的质量[11]。对于核磁共振光谱成像数据,频域维度上的数据稀疏程度比空间维度上的更高。为了获取频域和时域上的随机采样数据,研究人员需要设计一组包含快速变换梯度的复杂激活信号序列。对超极化13C 核磁共振压缩感知数据的重建过程中已经用到此种技术。然而,通过在相位编译维度上进行压缩感知而减少的采样时间,要远远超出仅在临时维度上进行压缩感知能够达到的程度。而且与此同时,除了回声平面采样技术,使用其他采样手段的时候,信号收集加速比例和采样压缩比例之间并无直接的相关关系。在本研究中,我们致力于加快常规临床核磁共振成像的信号收集过程,而该结论的应用范围并不仅限于上述讨论的范畴。
在光谱成像领域,所有新型信号重建技术的一个重要考量是该技术对点扩散函数的影响,以及因此导致的相邻空间体素信息污染的问题。Lustig 等人[11]详细讨论过点扩散函数和用于核磁共振成像压缩感知的变换点扩散函数的性质。上述研究中涉及的使用三维傅立叶变换的案例中,点分布函数与本文中的二维核磁谱图非常类似。在本文中,该二维核磁谱图在三个维度上进行取样:两个相位编译的空间维度和一个采用完全模拟信号的临时维度(无延迟/回声)[11]。压缩感知技术的优势在于,该技术通过采用随机采样的方法,避免了伪影的干扰(在回声平面成像以及回声平面频域成像中常见),在整个成像区域中形成类似于噪音的信号。与之相反,伪影则是点分布函数的扩散导致的体素之间的信息干扰。
当前研究的局限在于它是在此前获得的临床数据的基础上回顾性完成的,而回顾性信号重建是决定我们研究方法可行性的重要的第一步,接下来的必要步骤是对健康的志愿者进行实时的信号压缩感知,重建,并对比不同加速因子下的结果。其他可能的改进包括将该技术扩展至三维核磁共振光谱成像,以及改进信号重建程序以更好地利用空间和频域的信号稀疏性,特别是三维核磁共振光谱成像过程。
总而言之,本研究表明,在加速因子不超过 5 的情况下,压缩感知重建信号对原始信号的损失是微不足道的,同时还可节省最高可达 80% 的采样时间。这将能够促进更多核磁共振光谱成像手段应用于临床。本文中设计的加速技术也可以用于其他组织的成像,如乳腺。此外,由于减少了每轮采样所用的时间,本技术也可以用于对更难以检测的生化组分,如甘氨酸。
出版前本文公布情况:
10.1148/radiol.11111098 Content code: MR
[1] Daly PF,Cohen JS.Magnetic resonance spectroscopy of tumors and potential in vivo clinical applications:a review[J].Cancer Res,1989,49(4):770-779.
[2] Gillies RJ,Bhujwalla ZM,Evelhoch J,et al.Applications of magnetic resonance in model systems: tumor biology and physiology[J].Neoplasia,2000,2(1-2):139-151.
[3] Pirzkall A,Li XJ,Oh JM,et al.3D MRSI for resected highgrade gliomas before RT:tumor extent according to metabolic activity in relation to MRI[J].Int J Radiat Oncol Biol Phys, 2004,59(1):126-137.
[4] Kurhanewicz J,Vigneron DB,Nelson SJ.Three-dimensional magnetic resonance spectroscopic imaging of brain and prostate cancer[J].Neoplasia,2000,2(1-2):166-189.
[5] Nelson SJ,Vigneron DB,Dillon WP.Serialevaluation of patients with brain tumors using volume MRI and 3D1H MRSI[J]. NMR Biomed,1999,12(3):123-138.
[6] Nelson SJ.Multivoxel magnetic resonance spectroscopy of brain tumors[J].Mol Cancer Ther,2003,2(5):497-507.
[7] Howe FA,Opstad KS.1H MR spectroscopy of brain tumours and masses[J].NMR Biomed,2003,16(3):123-131.
[8] Ebel A,Soher BJ,Maudsley AA.Assessment of 3D proton MR echo-planar spectroscopic imaging using automated spectral analysis[J].Magn Reson Med,2001,46(6):1072-1078.
[9] Zhu H,Barker PB.MR spectroscopy and spectroscopic imaging of the brain[J].Methods Mol Biol,2011,711:203-226.
[10] Candes EJ,Romberg J,Tao T.Robust uncertainty principles: exact signal reconstruction from highly incomplete frequency information[J].IEEE Trans Inf Theory,2006,52(2):489-509.
[11] Lustig M,Donoho D,Pauly JM.Sparse MRI: the application of compressed sensing for rapid MR imaging[J].Magn Reson Med,2007,58(6):1182-1195.
[12] Jung H,Sung K,Nayak KS,et al.k-t FOCUSS: a general compressed sensing framework for high resolution dynamic MRI[J].Magn Reson Med,2009,61(1):103-116.
[13] Jim J,Tao L.Dynamic MRI with compressed sensing imaging using temporal correlations[A].In: 5th IEEE International Symposium on Biomedical Imaging: From Nano to Macro[C]. Paris: Institute of Electrical and Electronics Engineers,2008: 1613-1616.
[14] Serrai H,Senhadji L,de Certaines JD,et al.Time-domain quanification of amplitude, chemical shift,apparent relaxation time T2,and phase by wavelet-transform analysis: application to biomedical magnetic resonance spectroscopy[J].J Magn Reson, 1997,124(1):20-34.
[15] Serrai H,Nadal-Desbarats L,Poptani H,et al.Lactate editing and lipid suppression by continuous wavelet transform analysis: application to simulated and (1)H MRS brain tumor timedomain data[J].Magn Reson Med,2000,43(5):649-656.
[16] Cancino-De-Greiff HF,Ramos-Garcia R,Lorenzo-Ginori JV.Signal de-noising in magnetic resonance spectroscopyusing wavelet transforms[J].Concepts Magn Reson,2002,14(6): 388-401.
[17] Zarei S,Alireazie J,Babyn P,et al.MRS feature extraction: time frequency and wavelet analysis[A].In:The 2nd International Conference on Bioinformatics and Biomedical Engineering[C].S hanghai,China:ICBBE,2008;1863-1866.
[18] Fu Y,Serrai H.Fast magnetic resonance spectroscopic imaging (MRSI) using wavelet encoding and parallel imaging:in vitro results[J].J Magn Reson,2011,211(1):45-51.
[19] Hu S,Lustig M,Chen AP,et al.Compressed sensing for resolution enhancement of hyperpolarized 13C flyback 3D-MRSI[J]. J Magn Reson,2008,192(2):258-264.
[20] Antonini M,Barlaud M,Mathieu P,et al.Image coding using wavelet transform[J].IEEE Trans Image Process,1992, 1(2): 205-220.
[21] Naressi A,Couturier C,Castang I,et al.Java-based graphical user interface for MRUI,a software package for quantitation of in vivo/medicalmagnetic resonance spectroscopy signals[J].Comput Biol Med,2001,31(4):269-286.
[22] Ratiney H,Coenradie Y,Cavassila S,et al.Time-domain quantitation of1H short echo-time signals:background accommodation[J].MAGMA,2004,16(6):284-296.
[23] Pohmann R,von Kienlin M,Haase A.Theoretical evaluation and comparison of fast chemical shift imaging methods[J].J Magn Reson,1997,129(2):145-160.
[24] Zierhut ML,Ozturk-Isik E,Chen AP,et al.(1)H spectroscopic imaging of human brain at 3 Tesla:comparison of fast threedimensional magnetic resonance spectroscopic imaging techniques[J].J Magn Reson Imaging,2009,30(3):473-480.
[25] Mansfield P.Spatial mapping of the chemical shift in NMR[J]. Magn Reson Med,1984,1(3):370-386.
[26] Posse S,DeCarli C,Le Bihan D.Threedimensional echo-planar MR spectroscopic imaging at short echo times in the human brain[J].Radiology,1994,192(3):733-738.
[27] Mulkern RV,Panych LP.Echo planar spectroscopic imaging[J]. Concepts Magn Reson,2001,13(4):213-237.
[28] Du W,Du YP,Fan X,et al.Reduction of spectral ghost artifacts in high-resolution echo-planar spectroscopic imaging of water and fat resonances[J].Magn Reson Med,2003,49(6):1113-1120.
[29] Choi C,Ganji SK,Deberardinis RJ,et al.Measurement of glycine in the human brain in vivo by1H-MRS at 3 T: application in brain tumors[J].Magn Reson Med,2011,66(3):609-618.
10.3969/j.issn.1674-1633.2012.07.001
1674-1633(2012)07-0001-08
本文英文原版出自Radiology杂志2012年第262卷3月刊第985~994页。翻译及转载均经过北美放射学会许可。北美放射学会对在翻译过程中出现的译文不准确现象概不负责。
2011-05-27
修回日期:2011-08-08
录用日期:2011-09-12
定稿日期:2011-10-18
美国国立卫生研究院基金项目支持(NCRR UL1RR024982, NCIR21CA132096-01A1)。
联系人:V.D.K. (e-mail: vikram.kodibagkar@asu.edu)
文章内容不代表美国联邦政府与资助机构的立场。
©RSNA 2012
