基于COI条形码序列的珍珠母及其混伪品的DNA分子鉴定△
2011-11-06杜鹤崔丽娜姚辉宋经元张辉
杜鹤,崔丽娜,姚辉,宋经元,张辉*
(1.长春中医药大学,吉林 长春 130117;2.中国医学科学院药用植物研究所,北京 100193)
资源
基于COI条形码序列的珍珠母及其混伪品的DNA分子鉴定△
杜鹤1,崔丽娜1,姚辉2,宋经元2,张辉1*
(1.长春中医药大学,吉林 长春 130117;2.中国医学科学院药用植物研究所,北京 100193)
目的:探讨应用COI条形码序列对珍珠母及其混伪品进行物种鉴定的可行性。方法:用MEGA 4.0等软件计算珍珠母及其混伪品种内及种间变异,构建珍珠母及其混伪品的NJ树。结果:珍珠母种内COI序列变异小,种间存在较多的变异位点,种间的遗传距离显著大于种内的遗传距离。通过构建的系统聚类树图可以看出,珍珠母不同来源个体均聚在一起,能够与其混伪品区分开。结论:基于COI序列的DNA条形码技术可以很好的鉴定珍珠母的正品来源及其混伪品。
珍珠母;DNA条形码;COI;鉴定
珍珠母Margaritifera concha为我国常用中药材,具有平肝潜阳,安神定惊,明目退翳的功效。用于头痛眩晕,惊悸失眠,目赤翳障,视物昏花[1]。据《中国药典》2010年版记载,珍珠母为蚌科动物三角帆蚌Hyriopsis cumingii(Lea)、褶纹冠蚌Cristaria plicata(Leach)或珍珠贝科动物马氏珍珠贝Pteria martensii(Dunker)的贝壳。去肉,洗净,干燥。现市场以其混伪品和同属做珍珠母药用的情况较为普遍,如蚬科动物河蚬Corbicula fluminea(Muller)、蚌科动物背瘤丽蚌Lamprotula leai(Gray)等[2]。由于珍珠母正品与其混伪品的形态学特征差别很细微,用传统的方法很难鉴定,所以为保正药材的质量及其临床疗效,需要更快速、准确地方法鉴定珍珠母的正品来源与其混伪品。
DNA条形码技术是近来发展起来的物种鉴定新方法,通过对基因组中一段短的、标准的DNA片段进行分析从而对物种进行准确鉴定[3-4],成为生物分类和鉴定的研究热点和方向[5-8]。本研究是应用DNA条形码技术,对珍珠母及其混伪品的DNA条形码COI序列进行比对分析,构建分子系统树,进而对珍珠母及其混伪品进行鉴别。
1 材料与方法
1.1 材料
褶纹冠蚌Cristaria plicata(Leach)药材购自安徽亳州药材市场,标本号AS13MT01、AS13MT02。两种混伪品COI序列来自于GenBank,珍珠母及其混伪品信息见表1。
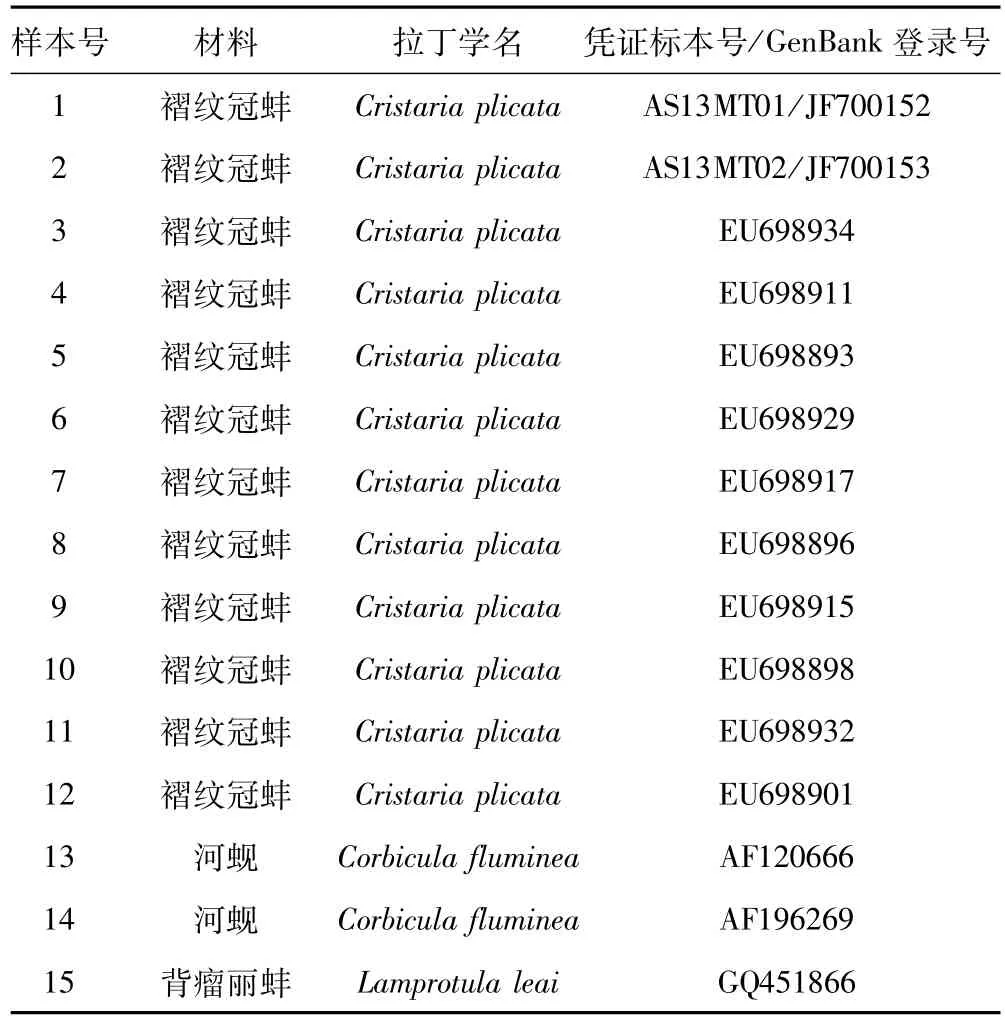
表1 材料来源
1.2 方法
1.2.1 DNA提取 取新鲜材料,去壳后用已灭菌的手术剪把样品剪成小块,取30 mg样品,用液氮研碎肌肉组织[9],再用 DNA提取研磨仪(Retsch MM400,Germany)研磨 2 min(30次·min-1)后,使用动物DNA提取试剂盒(北京天根生化公司)提取总DNA。
1.2.2 PCR扩增及测序 扩增引物COI序列通用引物:正向为 LCOI490 5′-GGTCAACAAATCATAAAGATATTGG-3′,反向为 HCO2198 5′-TAAACTTTCAGGGTGACCAAAAAATCA-3′[10]。PCR反应体积为 25μL,体系内含 MgCl22μL(25 mmol·L-1)、dNTP 2μL(2.5 mmol·L-1)、PCR buffer2.5μL(10×)、引物各1.0μL(2.5μmol·L-1)(北京生工生物技术公司合成)、聚合酶0.2μL(Biocolor BioScience&Technology Co.,China)、双蒸水13.3μL、总 DNA约1μL(~30 ng)。PCR反应条件为:94℃,1min;94℃,1 min,45℃,1.5 min,72 ℃,1.5 min,5cycles;94 ℃,1 min,50℃,1.5 min,72℃,1 min,35cycles;72℃,5 min。PCR扩增产物经纯化后,使用ABI 3730XL测序仪(Applied Biosystems Co.,USA)双向测序。
1.2.3 数据处理 测序峰图利用CodonCode Aligner V 2.06拼接并校正,去除引物区,获得长度为658bp的样品序列。对于从GenBank获得的COI序列,采用CodonCode Aligner V 2.06软件,与拼接好的样品序列比对后,去除与样品序列相对应的658 bp以外的区段,保留长度≥550 bp的网上序列。最后将序列用软件MEGA 4.0比对并进行遗传距离等分析,并构建NJ系统聚类树,同时利用bootstrap(1 000次重复)检验各分支的支持率。
2 结果与分析
2.1 种内序列变异
样品AS13MT01与AS13MT02序列长度为658 bp,含3个PolyT和1个PolyG结构,GC含量为39.6%。12个种内序列,比对后序列长度为658 bp,有15个碱基变异位点,详见表2。种内平均K2P距离为0.005 4,种内最大K2P距离为0.011 5。

表2 褶纹冠蚌种内碱基变异情况表
2.2 种间序列变异
褶纹冠蚌与其主要混伪品COI序列间存在较多的变异位点(见图1),种间平均K2P距离为0.356 3,最小K2P距离为0.193 4。

图1 珍珠母及其混伪品序列间的变异位点
2.3 珍珠母及其混伪品NJ树
基于COI序列,通过邻接法(NJ)所构建的系统聚类树图(图2)可以看出,珍珠母不同来源个体均聚在一起,支持率为100,同时也很容易与其余混伪品区别开来。因此COI条形码序列适用于药材珍珠母与其混伪品的鉴定。
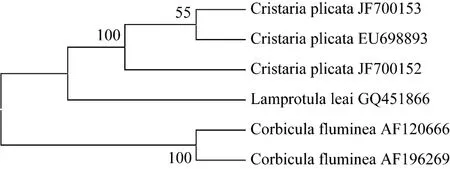
图2 基于COI序列构建的珍珠母及其混伪品的邻接(NJ)树
3 讨论
珍珠母种内COI序列有15个变异位点,种内平均K2P变异为0.005 4,种内最大K2P变异为0.011 5。说明珍珠母种内COI序列变异很小比较稳定。珍珠母及其混伪品COI序列种间K2P距离的平均值为0.356 3,最小 K2P距离为0.193 4。存在较多的变异位点,种间的遗传距离显著大于种内的遗传距离。同时,从基于COI序列构建的珍珠母及其混伪品NJ树可以看出,珍珠母不同来源个体均聚在一起,支持率较高,与其它混伪品能够很明显区分开。因此,基于COI序列的DNA条形码技术可以很好的鉴定珍珠母及其混伪品,为该药材的准确鉴定提供了新的方法。
[1]国家药典委员会.中国药典[S].一部.北京:中国医药科技出版社,2010:216.
[2]来复根,冯迈高.中药珍珠母及其混伪品的鉴别[J].中药材,1985,5:22-25.
[3]Schindel DE,Miller SE.DNA barcoding,a useful tool for taxonomists[J].Nature,2005,435:17.
[4]陈士林,宋经元,姚辉,等.药用植物DNA条形码鉴定策略及关键技术分析[J].中国天然药物,2009,7(5):322-327.
[5]陈士林,姚辉,宋经元,等.基于 DNAbarcoding(条形码)技术的中药材鉴定[J].世界科学技术—中医药现代化,2007,9(3):7-12.
[6]Hebert PD,Ratnasingham S,deWaard JR.Barcoding animal life:cytochrome c oxidase subunit1 divergences among closely related species[J].Proc Biol Sci,2003,270(S1):S96-S99.[7]Hebert PD,Cywinska A,Ball SL,et al.Biological identifications through DNA barcodes[J].Proc Biol Sci,2003,270(1512):313-321.
[8]Hebert PD,StoeckleM Y,Zemlak T S,et al.Identification of birds through DNA barcodes[J].Plos Biol,2004,2(10):1657-1663.
[9]徐巧情,刘俊.三角帆蚌不同组织基因组DNA提取效果比较[J].水生态学杂志,2010,3(2):121-124.
[10]Folmer O,Black M,Hoeh W,et al.DNA primers for amplification ofmitochondrial cytochrome c oxidase subunit I from diverse metazoan invertebrate[J].Mol Mar Biol Biotechnol,1994,3:294-299.
Molecular Identification of Margaritifera concha and its Adulterants Using COIBarcode Sequence
DU He1,CUILi-na1,YAO Hui2,SONG Jing-yuan2,ZHANG Hui1
(1.Changchun University of Chinese Medicine,Changchun130117,China;2.Institute of Medicinal Plant Development,Chinese Academy of Medical Sciences,Peking Union Medical College,Beijing100193,China)
Objective:This study explores the feasibility of using the COIbarcode sequence in identification of the Margaritifera concha and its adulterants.Methods:Intra-and inter-specific genetic distances ofMargaritifera concha and its adulterants have been analyzed by MEGA 4.0 software using COI sequences and the NJ tree has been constructed.Results:Margaritifera concha exhibited low intra-specific sequence variation and the inter-specific sequence have high variation between Margaritifera concha and its adulterants.The inter-specific genetic distanceswere obviously higher than the intra-specific ones.The sample of Margaritifera concha clustered together and can separated from its adulterants in the dendrogram constructed.Conclusion:As a DNA barcode,COI sequence can identify the Margaritifera concha with its adulterants.
Margaritifera concha;DNA barcoding;COI;identification
吉林省财政厅项目——药用动物DNA条形码鉴定技术研究
*张辉,E-mail:zhanghui_8080@163.com
2011-10-08)
