共代谢作用下芦苇根际细菌多样性与群落组成
2022-04-24洪志锋张旎晨仇荣亮林庆祺倪卓彪晁元卿
洪志锋,张旎晨,阿 丹,仇荣亮,,林庆祺,倪卓彪,晁元卿,5**
共代谢作用下芦苇根际细菌多样性与群落组成
洪志锋1,张旎晨1,阿 丹2*,仇荣亮1,3,4,林庆祺3,4,倪卓彪3,4,晁元卿1,5**
(1.中山大学环境科学与工程学院,广东 广州 510006;2.仲恺农业工程学院资源与环境学院,广东省普通高校农业产地污染综合防治工程技术研究中心,广东 广州 510225;3.岭南现代农业科学与技术广东省实验室,广东 广州 510642;4.华南农业大学资源环境学院,广东省农业农村污染治理与环境安全重点实验室,广东 广州 510642;5.中山大学环境科学与工程学院,广东省环境污染控制与修复技术重点实验室,广东 广州 510006.)
以对羟基苯甲酸(PHA)、对香豆酸(PCA)、咖啡酸(CA)和阿魏酸(FA)为代表性酚类根系分泌物(PREs),以对叔丁基苯酚(PTBP)作为典型烷基酚、以常见湿地植物芦苇为受试植物,结合高通量测序和生物信息学分析,研究PREs-PTBP交互作用下芦苇根际细菌多样性与群落组成的变化规律.结果表明,所有PREs均能有效提高根际细菌活性和有机碳降解,但只有单酚PREs(PHA、PCA、FA)可以促进PTBP的生物降解,而多酚PREs(CA)则不能.一方面,PREs作为碳源可以决定根际优势菌门,如变形菌门(Proteobacteria)和拟杆菌门(Bacteroidetes)等碳源利用能力较强的菌种在PREs组种丰度升高,平均占比分别增加了15.34%和4.73%;而放线菌门(Actinobacteria)和厚壁菌门(Firmicutes)等解毒能力或耐受性较强的菌种在空白组丰度升高,平均占比分别增加了15.92%和9.99%.另一方面,PREs功能结构还可以决定根际功能菌属,如单酚PREs(PHA、PCA、FA)可以富集PTBP等单酚降解相关的假单胞菌属(),占比可达1.45%~4.02%;而多酚PREs(CA)能够富集多酚降解相关的新鞘氨醇菌(),占比可达3.71%.此外,PREs的结构越简单越有利于扩增序列变体(ASVs)的富集,PREs间的结构越相近则ASVs的种类越相似.不同处理组植物根际细菌群落的存在不是随机发生的,而是植物根系通过释放分泌物对根际微生物组的定向选择.
酚类根系分泌物;对叔丁基苯酚;芦苇;根际细菌;多样性;群落组成
烷基酚是一种典型的环境内分泌干扰物,具有雌激素活性、生物富集性和生物毒性[1-2].烷基酚作为持久性有机污染物在环境中难以正常生物降解,其有效降解依赖于微生物共代谢[3].常用的共代谢底物主要有易被微生物利用的营养物质、以及与目标污染物化学结构相似的中间代谢产物.相较于特异性差、易二次污染的上述共代谢底物,根系分泌物是植物生成的天然成分,可以促进微生物生长、提高生物酶活性,从而增强有机污染物的分解与矿化[4-5].
酚类根系分泌物(PREs)可以有效促进烷基酚等芳香族化合物的生物降解[6-8].PREs具有与目标污染物相似的苯环结构易实现特异性降解,是植物天然成分,易被微生物直接利用.前期研究发现,湿地植物芦苇可以通过释放PREs来提高烷基酚降解菌的代谢活性[9],且不同功能结构PREs影响下烷基酚的降解速率也有所差异[10].然而,在该共代谢系统中,微生物群落面对烷基酚胁迫及PREs介导如何响应,微生物群落结构变化如何影响烷基酚的共代谢降解均尚不清晰.
针对上述问题,本文以不同功能结构PREs为共代谢底物[11],对叔丁基苯酚(PTBP)为典型烷基酚、以常见湿地植物芦苇()为受试植物,通过化学分析、高通量测序、生物信息学分析等方法,分析PREs-PTBP交互作用下芦苇根际微生物群落结构变化及响应机制,以期从分子水平上解释PREs功能结构、微生物群落演替、烷基酚降解途径三者间的内在联系.
1 材料与方法
1.1 实验设计
芦苇幼苗(重量为6.2~8.6g,高度为17.1~19.1cm)购自当地园圃,在使用前先用自来水冲洗干净附着土壤,再用超纯水润洗.实验过程中,将芦苇幼苗暴露在含有10mg/L PTBP的300mL Hoagland无机营养液中进行完全水培.本实验共设有5组处理,每组处理添加不同PREs,并设有3个平行.其中,处理①中添加50mg/L对羟基苯甲酸(PHA组),处理②中添加50mg/L对香豆酸(PCA组),处理③中添加50mg·L-1咖啡酸(CA组),处理④中添加50mg/L阿魏酸(FA组),处理⑤中不添加任何PREs作为空白对照(CK组).为提供适宜植物和微生物生长的环境,所有实验处理置于人工气候室中培养,温度为25 ℃、湿度为70%、光强为8000lx、光照时间为12h/d.
Hoagland无机营养液[14]的配方为: 36.1mg/L KNO3、293mg/L K2SO4、3.87mg/L NaH2PO4、103mg/L MgSO4·7H2O、147mg/L CaCl2·2H2O、3.33mg/L FeSO4·7H2O、0.95mg/L H3BO3、0.39mg/L MnCl2·4H2O、0.03mg/L CuSO4·5H2O、0.08mg/L ZuSO4·7H2O、0.254mg/L H2MoO4·H2O、pH 7.0.
1.2 采样与分析方法
实验开始后,分别在第0,2,4,6,8,10,12,14,16d连续采集各处理中芦苇根际水样,用于PTBP(LC-10A series,日本岛津)和总有机碳(TOC;TOC-VCPH,日本岛津)定量.实验结束后,采集各处理中芦苇根系样品,用于根际细菌的丰度、多样性及群落组成分析(16S rRNA基因测序).
1.3 DNA提取、16S rRNA扩增和高通量测序
采集不同处理组的样品共计15个,用Power SoilDNA Isolation Kit试剂盒(MoBio Laboratories, USA)提取水样中的DNA,用Nano Drop分光光度计(Nano-100,杭州奥盛)检测DNA的浓度和纯度.用引物341F(5'-CCTAYGGGRBGCASCAG-3')和806R (5'-GGACTACHVGGGTWTCTAAT-3')扩增细菌16S RNA序列的V3-V4区.PCR反应体系为50μL, 2xPremix Taq 25μL,341F/806R引物各1μL,DNA 3μL,Nuclease-free water 20μL,反应条件为:98℃预变性1min,98℃变性10s,50℃退火30s,72℃延伸30s,共30个循环,再在72℃下延伸5min,最后保存在4℃条件下.最后使用Illumina Hiseq 2500平台进行测序(北京百迈客生物科技有限公司).
1.4 序列预处理和生物信息学分析
使用FLASH v1.2.7软件,通过overlap将每个样品的双端序列数据拼成一条序列.生物信息学分析使用QIIME2(2019.07)平台,拼接好的序列通过DADA2(q2-dada2)降噪质控得到单核苷酸精度的扩增序列变体(ASVs),15个样品得到858649条ASV,平均每个样品得到的ASV为57243条.利用“q2- diversity”插件分析不同样品的Alpha多样性(observed_otus指数和Shannon指数)和β-多样性(基于加权UniFrac距离和非加权的UniFrac距离矩阵).利用主坐标分析法(PCoA)可视化分析考察样品β-多样性的聚类特征.使用“q2-feature-classifier”插件,比对Silva数据库进行物种分类注释,并去除掉注释为线粒体、叶绿体、真核生物域和未知域序列的序列.
1.5 数理分析
污染物去除速率常数(,d-1)和去除负荷(RL, mg/d)的计算公式如下.其中,t为时间水样中污染物的浓度,mg/L;为水样的体积,L.
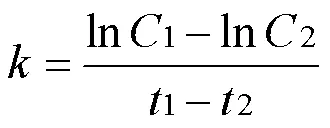

2 结果与讨论
2.1 PREs影响下PTBP与TOC的去除
如表1所示TOC的去除速率常数和去除负荷均在PCA组和CA组(0.37~0.50d-1和0.38~0.47mg/d)中明显高于CK组(0d-1和0.01mg/d) (<0.05),而在PHA组和FA组(0.25~0.27d-1和0.26~0.28mg/d)中略高于CK组(>0.05).各实验处理中的碳源主要来自外援添加的PREs和PTBP,虽然PREs的存在会提高TOC的初始浓度,但同时也为根际微生物提供了充足的碳源,从而促进微生物种群生长及改善微生物群落结构.
表1还显示了不同PREs处理下PTBP的去除速率常数和去除负荷. PTBP的去除速率常数和去除负荷均在PCA组(0.57d-1和0.46mg/L)中显著高于CK组和CA组(0.39~0.44d-1和0.31~0.35mg/d) (<0.05),而在PHA组和FA组(0.50~0.51d-1和0.41mg/d)中略高于CK组和CA组(>0.05).这表明单酚PREs(PHA、PCA、FA)有利于PTBP的生物降解,尤其是PCA,而多酚PREs(CA)则对PTBP的降解过程没有明显作用,可见与目标污染物相似的单酚结构是促进PTBP生物降解的关键功能结构.而这种关键功能结构很可能诱发了根际微生物群落结构的变化,从而介导了PTBP的降解速率.
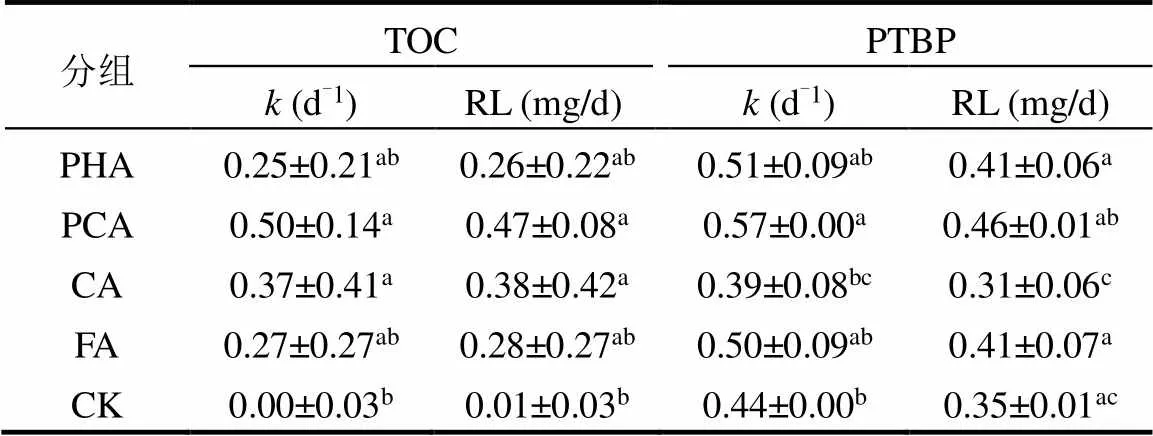
表1 TOC和PTBP的去除速率常数(k)和去除负荷(RL)
注:同一指标下不同字母表示不同PREs处理间存在显著性差异(<0.05).
2.2 PREs-PTBP交互作用下根际微生物群落的结构变化及影响机制
2.2.1 根际微生物群落结构的Alpha多样性分析 Alpha多样性采用observed_otus指数(图1a)和Shannon指数(图1b)衡量,数值越高表明微生物群落的丰富度和多样性越高.在PHA组、PCA组和CA组(917~1318)中observed_otus指数明显高于CK组(357);而在CA组(8.915)中Shannon指数显著高于CK组(7.032).可见,PREs不但提高了根际细菌群落的物种丰富度还增加了其物种多样性,这得益于外源添加PREs增加了能有效被微生物利用的碳源,从而促进了根际细菌的生长和活性,丰富了根际细菌的物种数量与种类.其他研究也有报道通过添加有机肥、生物炭等可利用碳源,提高了细菌群落的多样性[12-13].
上述现象还受到PREs功能结构的限制,例如CA能有效促进根际细菌的丰富度和多样性(图1),但在CA组中PTBP的降解速率和负荷却明显低于其他处理组(表1).由此可见,虽然多酚PREs(CA)可以有效提高根际细菌整体的数量与种类[14-15],但并不能增强PTBP相关降解菌的活性,因此不能促进PTBP的降解.相应的,单酚PREs(PHA、PCA、FA)虽然对微生物的富集效果与CA相比不显著,但可以强化根际细菌对单酚类化合物的利用能力,从而加快了同为单酚结构的PTBP的生物降解(表1).
2.2.2 根际微生物群落结构的Beta多样性分析 Beta多样性分析使用QIIME2(2019.07),先计算加权Unifrac距离和非加权Unifrac距离,并使用PCoA分析对矩阵进行可视化(图2),同时利用PERMANOVA多因素方差分析探究不同因素对Beta多样性的显著性.结果显示,不同PREs处理下芦苇根系样品在PCoA图中具有明显的成簇聚集现象(Weighted-Matrix距离矩:2=0.571,<0.01; Unweighted-Matrix距离矩:2= 0.381,<0.01).该结果表明不同PREs处理对样品中细菌群落的Beta多样性具有显著影响,且PREs组(PHA组、PCA组、CA组、FA组)与CK组之间的菌群结构差异相对较大;而在各PREs处理组之间,长链PREs组(PCA组、CA组、FA组)与短链PREs组(PHA组)的菌群结构差异较大.

图1 不同PREs处理下根际细菌群落的Alpha多样性分析
2.2.3 根际微生物群落物种分析 根际细菌门水平分析结果显示(图3),检测到的优势菌门有变形菌门(Proteobacteria)、拟杆菌门(Bacteroidetes)、疣微菌门(Verrucomicrobia),平均占比分别为35.82%~56.65%、14.19%~21.63%、6.06%~12.73%;而放线菌门(Actinobacteria)和厚壁菌门(Firmicutes)的平均占比也分别达到2.10%~21.74%和0.60%~12.80%.这些都是常见的湿地植物根际细菌优势种群[16-19].通过比较PREs组(PHA组、PCA组、CA组、FA组)和CK组的优势门组成,发现芦苇根际细菌群落在物种丰度分布上有所不同(图3).其中,PREs处理组中变形菌门、拟杆菌门、疣微菌门的平均占比分别增加了15.34%、4.73%、4.13%;而放线菌门、厚壁菌门的平均占比依次缩减了15.92%、9.99%;且这种趋势在长链PREs组(PCA组、CA组、FA组)中更为突出.变形菌门和拟杆菌门常被发现于高碳源土壤中,被认为是能够有效利用植物碳源的菌种[20],PREs的添加带来了更多的营养和能源,从而促进了它们的生长.与此相对应的是,CK组由于无外援PREs的添加缺乏充足的碳源,环境条件并不利于微生物的生长,为了抵御PTBP的胁迫,具有强大生物解毒能力的放线菌门大量繁殖并占据优势[21-22].周雅心等[13]在稻田土壤细菌群落的研究中也有相似的发现,由于施加炉渣和生物炭引起微生物生长环境的变化,一部分微生物的生长受到抑制,而另一部分微生物的生长受到促进,且在本研究中,有益微生物的促进作用更为显著.

图2 不同PREs处理下根际细菌群落的PCoA分析
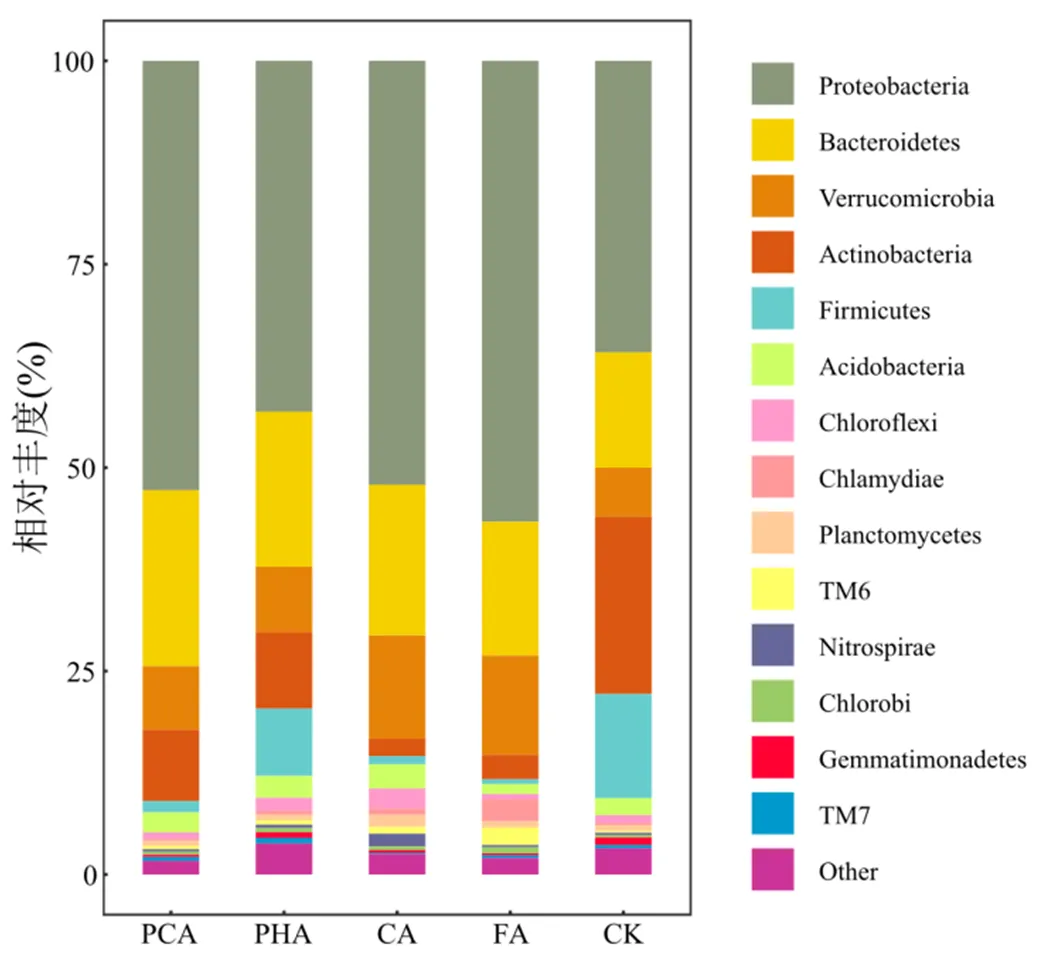
图3 根际细菌群落优势门分布
在属水平上对根际细菌的物种分布进行分析,选取每个样本中丰度前10的属进行分析,对数据进行均一化后制作热图,热图左侧为基于各物种的DNA序列构建的系统发育树,结果如图4所示.不同功能结构PREs可以促进对应降解功能菌种的生长.一方面,假单胞菌()已被证实是一种烷基酚降解菌[23],它在单酚PREs组(PHA组、PCA组、FA组;1.45%~4.02%)中的丰度高于多酚PREs组(CA组;0.96%).据此,推测单酚PREs首先需要通过羟基化反应进而开环,因此它们的存在可以催生单酚氧化降解相关菌群(如假单胞菌),而这种特性同样适用于单酚结构的PTBP的生物降解(表1),因而促进了单酚类化合物的PREs及PTBP的共代谢作用.潘声旺等[24]研究了苏丹草根系分泌物对有机氯农药降解的促进作用,也发现假单胞菌()对有机氯有降解作用,且根系分泌物的存在改变了细菌、真菌的种群数量及其群落结构,从而影响了有机氯农药的降解速率,这与本研究结果一致.另一方面,能够促进双酚A降解的新鞘氨醇菌[25]()的情况则刚好相反,它在多酚PREs组(CA组;3.71%)中的丰度高于单酚PREs组(PHA组、PCA组、FA组;1.24%~2.05%).可见,多酚PREs很可能有助于富集生成多酚氧化降解相关菌群(如新鞘氨醇菌),而这种特性并不适用于单酚结构的目标污染物PTBP,因此虽然CA可以提高根际细菌种群数(图1),但并不能促进PTBP的分解作用(表1).

图4 根际细菌群落优势属热图
2.2.4 根际微生物特异性富集菌群 为了探究不同PREs对细菌群落的影响,进一步明晰不同处理组之间核心菌群的差异,利用线性模型鉴定了相较于无添加空白组(CK组),不同PREs根际显著富集(<0.05)的细菌菌群.本研究将不同PREs水平下样品中富集的细菌菌群分为四类:对羟基苯甲酸富集的菌群(PHA_ASVs),对香豆酸富集的菌群(PCA_ ASVs),咖啡酸富集的菌群(CA_ASVs),阿魏酸富集的菌群(FA_ASVs),分析结果如图5所示.通过与CK组比较发现,短链且单取代基的PHA组中显著富集的ASVs最多(226个),其次是长链且单取代基的PCA组(81个),最后是长链且双取代基CA组和FA组(46~47个).可见,PREs的结构越简单,越有利于ASVs的富集.通过PREs组间比较发现,单取代基的PHA组和PCA组的共同富集菌群最多(140个),双取代基的CA组和FA组的共同富集菌群次之(16个),而CA组和PHA组或PCA组的共同富集菌群最少(4个).说明,PREs的结构越相近,ASVs的种类越相似.
通过图5还可以看出,PHA_ASVs和PCA_ ASVs的优势菌门分布相似,且种类最多,以变形菌门(Proteobacteria),拟杆菌门(Bacteroidetes),厚壁菌门(Firmicutes)为主要菌群.虽然FA_ASVs和FA_ ASVs的优势菌门分布也相似,以变形菌门(Proteobacteria),疣微菌门(Verrucomicrobia),拟杆菌门(Bacteroidetes)为主要菌群,但是FA_ASVs的种类更为丰富,例如FA_ASVs具有CA_ASVs没有的厚壁菌门(Firmicutes)和放线菌门(Actinobacteria).因此,不同处理组植物根际微生物群落的存在不是随机发生的,而是植物通过释放PREs等根系分泌物对根际微生物组的定向选择[24].

图5 不同处理组中特异性富集菌属
横坐标logFC用于衡量基因表达上下调,纵坐标logCPM用于衡量基因的表达量
综上可得,通过外源添加PREs可有效提高TOC的去除效率,PREs作为根际细菌的重要碳源来源,可以有效促进菌群生长、提高优势菌丰度,进而改善碳利用潜能.不同PREs化学结构影响下PTBP的降解速率出现差异,表明PREs功能结构是诱导PTBP降解相关功能菌群的关键,从而增强PTBP共代谢能力;相应的,不同PREs化学结构影响下根际细菌群落组成也发生变化,例如PREs的结构越简单则ASVs的富集越明显,PREs的结构越相近则ASVs的种类越相似.其中,具有多酚结构的PREs(CA)虽然可以显著促进根际细菌的生长和活性,同时提高多酚氧化相关降解菌(如新鞘氨醇菌)丰度,但多酚功能菌并不参与单酚污染物PTBP的代谢过程,因此不能促进PTBP的生物降解;而具有单酚结构的PREs(PHA、PCA、FA)虽然对根际细菌的活化能力不如CA,但可以提高单酚氧化降解相关菌(如假单胞菌)丰度,通过释放非特异性酶催化单酚化合物(单酚PREs和PTBP)分解,从而实现PTBP的共代谢降解.可见,正是根系分泌物的不同影响了根际细菌群落的定殖[26-27],而不同降解菌又选择了有机物的不同代谢方式,进而决定了污染物的不同降解行为.
3 结论
3.1 PREs的添加促进了根际细菌的丰度和多样性,并增强了TOC的去除效果,其中单酚PREs(PHA、PCA、FA)可以加速PTBP的生物降解,而多酚PREs(CA)则不能,这些现象与PREs-PTBP交互作用下根际细菌群落结构变化息息相关.
3.2 在门水平上,PREs作为碳源是决定根际优势菌的主要因素,在PREs处理组中变形菌、拟杆菌等碳源利用能力较强的菌种平均占比增加了15.34%和4.73%;而在CK组中放线菌、厚壁菌等耐受或解毒能力较强的菌种平均占比增加了15.92%和9.99%.
3.3 在属水平上,PREs功能结构是决定根际功能菌种的主要因素,单酚PREs组中PTBP等单酚降解相关的假单胞菌丰度较高,占比可达1.45%~4.02%;而多酚PREs组中多酚降解相关的新鞘氨醇菌丰度较高,占比可达3.71%.
3.4 PREs的结构越简单越有利于ASVs的富集,PREs间的结构越相近ASVs的种类越相似,不同处理组植物根际细菌群落的存在不是随机发生的,而是植物根系通过释放PREs等分泌物对根际微生物组的定向选择.
[1] Capaldo A, Gay F, Valiante S, et al. Endocrine-disrupting effects of nonylphenol in the newt,(Amphibia, Urodela) [J]. Comparative Biochemistry and Physiology Part C, 2012,155(2):352-358.
[2] Ismail N, Wee S Y, Aris A Z. Bisphenol A and alkylphenols concentrations in selected mariculture fish species from Pulau Kukup, Johor, Malaysia [J]. Marine Pollution Bulletin, 2018,127:536-540.
[3] Nsenga Kumwimba M, Meng F G. Roles of ammonia-oxidizing bacteria in improving metabolism and cometabolism of trace organic chemicals in biological wastewater treatment processes: a review [J]. Science of the Total Environment, 2019,659:419-441.
[4] Xie X M, Liao M, Jing Y, et al. Influence of root-exudates concentration on pyrene degradation and soil microbial characteristics in pyrene contaminated soil [J]. Chemosphere, 2012,88(10):1190-1195.
[5] Lakshmanan V, Selvaraj G, Bais H P. Functional soil microbiome: belowground solutions to an aboveground problem [J]. Plant Physiology, 2014,166(2):689-700.
[6] Leigh M B, Fletcher J S, Fu X, et al. Root turnover: an important source of microbial substrates in rhizosphere remediation of recalcitrant contaminants [J]. Environmental Science & Technology, 2002,36(7):1579-1583.
[7] Toyama T, Furukawa T, Maeda N, et al. Accelerated biodegradation of pyrene and benzo[a]pyrene in therhizosphere by bacteria-root exudate interactions [J]. Water Research, 2011,45(4): 1629-1638.
[8] Donnelly P K, Hegde R S, Fletcher J S. Growth of PCB-degrading bacteria on compounds from photosynthetic plants [J]. Chemosphere, 1994,28(5):981-988.
[9] A Dan, Fujii D, Soda S, et al. Removal of phenol, bisphenol A, and 4--butylphenol from synthetic landfill leachate by vertical flow constructed wetlands [J]. Science of the Total Environment, 2017,578: 566-576.
[10] A Dan, Zhang N C, Qiu R L, et al. Accelerated biodegradation of-butylphenol in therhizosphere by phenolic root exudates [J]. Environmental and Experimental Botany, 2020,169:103891-103896.
[11] Gao H X, Ding A W, Tang Y P, et al. Chemical constituents from the rhizomas of[J]. Chinese Journal of Natural Medicines, 2009,7:196-198.
[12] 张凯煜,谷 洁,王小娟,等.微生物有机肥对樱桃园土壤细菌群落的影响[J]. 中国环境科学, 2019,39(3):1245-1252.
Zang K Y, Gu J, Wang X J, et al. Effects of bio-organic fertilizer on the soil bacterial community in a cherry orchard [J]. China Environmental Science, 2019,39(3):1245-1252.
[13] 周雅心,王晓彤,王广磊,等.炉渣与生物炭施加对稻田土壤细菌多样性及群落组成的影响 [J]. 中国环境科学, 2020,40(3):1213-1223.
Zhou Y X, Wang X T, Wang G L, et al. Effect of the slag and biochar application on bacterial diversity and community composition of paddy field [J]. China Environmental Science, 2020,40(3):1213-1223.
[14] Khan S, Afzal M, Iqbal S, et al. Plant–bacteria partnerships for the remediation of hydrocarbon contaminated soils [J]. Chemosphere, 2013,90(4):1317-1332.
[15] Jitendra M, Rachna S, Arora N K. Alleviation of heavy metal stress in plants and remediation of soil by rhizosphere microorganisms [J]. Front Microbiol, 2017,241:124950-124957.
[16] Zeng Y X, Yang Z, Grebmeier J M, et al. Culture-independent and -dependent methods to investigate the diversity of planktonic bacteria in the northern Bering Sea [J]. Polar biology, 2012,35(1):117-129.
[17] Fu W G, Wang Y X, Wei W, et al. Species diversity and functional prediction of soil bacterial communities in constructed wetlands with different plant conditions [J]. Current Microbiology, 2019,76:338-345.
[18] Li T C, Fan Y C, Cun D S, et al. Treatment performance and microbial response to dibutyl phthalate contaminated wastewater in vertical flow constructed wetland mesocosms [J]. Chemosphere, 2019,246:125635- 125645.
[19] 滕泽栋,李 敏,朱 静,等.野鸭湖湿地芦苇根际微生物多样性与磷素形态关系[J]. 环境科学, 2017,38(11):4589-4597.
Teng Z D, Li M, Zhu J, et al. Effects of soil microbial diversity on the phosphate fraction in the rhizosphere ofcommunis in the Yeyahu wetland in Beijing, China [J]. Environmental Science, 2017,38(11):4589-4597.
[20] Chen Y M, Ding Q B, Chao Y Q, et al. Structural development and assembly patterns of the root-associated microbiomes during phytoremediation [J]. Science of the Total Environment, 2018,644: 1591-1601.
[21] Adegboye M F, Babalola O O. Isolation and identification of potential antibiotic producing rare actinomycetes from rhizospheric soils [J]. Journal of Human Ecology, 2016,56(1/2):31-41.
[22] A Álvarez, Yañez M L, Benimeli C S, et al. Maize plants () root exudates enhance lindane removal by nativestrains [J]. International Biodeterioration and Biodegradation, 2012,66(1): 14-18.
[23] Hai H, Daisuke I, Naonori M, et al. Characterization of novel 4--butylphenol-degradingstrains isolated from rhizosphere of giant duckweed,[J]. Japanese Journal of Water Treatment Biology, 2013,45(2):83-92.
[24] 潘声望,雷志华,吴云霄,等.苏丹草根分泌物在有机氯农药降解过程中的作用[J]. 中国环境科学, 2017,37(8):3072-3079.
Pan S W, Lei Z H, Wu Y X, et al. Effect of exudates fromgrass roots on degradation of organochlorine pesticides in soils [J]. China Environmental Science, 2017,37(8):3072-3079.
[25] Toyama T, Sato Y, Inoue D, et al. Biodegradation of bisphenol A and bisphenol F in the rhizosphere sediment of[J]. Journal of Bioscience and Bioengineering, 2009,108(2):147-150.
[26] Lu T, Ke M J, Jin Y J, et al. Rhizosphere microorganisms can influence the timing of plant flowering [J]. Microbiome, 2018,6:231- 242.
[27] Zhalnina K, Louie K, Hao Z, et al. Dynamic root exudate chemistry and microbial substrate preferences drive patterns in rhizosphere microbial community assembly [J]. Nature Microbiology, 2018,3:470-480.
Bacterial diversity and community composition in therhizosphere by cometabolism.
HONG Zhi-feng1, ZHANG Ni-chen1, A Dan2*, QIU Rong-liang1,3,4, LIN Qing-qi3,4, NI Zhuo-biao3,4, CHAO Yuan-qing1,5**
(1.School of Environmental Science and Engineering, Sun Yat-sen University, Guangzhou 510006,China;2.Engineering and Technology Research Center for Agricultural Land Pollution Integrated Prevention and Control of Guangdong Higher Education Institutes, College of Resources and Environment, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China;3.Guangdong Laboratory for Lingnan Modern Agriculture, Guangzhou 510642, China;4.Guangdong Provincial Key Laboratory of Agricultural & Rural Pollution Abatement and Environmental Safety, College of Natural Resources and Environment, South China Agricultural University, Guangzhou 510642, China;5. Guangdong Provincial Key Laboratory for Environmental Pollution Control and Remediation Technology, School of Environmental Science and Engineering, Sun Yat-sen University, Guangzhou 510006,China)., 2022,42(4):1812~1818
-hydroxybenzoic acid (PHA),-coumaric acid (PCA), caffeic acid (CA), and ferulic acid (FA) were selected as the representative phenolic root exudates (PREs),--butylphenol (PTBP) was chosen as the typical alkylphenol, and common reed () was used as the test plant. High-throughput sequencing and bioinformatics analyses were applied to investigate the variation of bacterial diversity and community by PRE-PTBP interaction inrhizosphere. PREs could effectively improve the rhizosphere bacterial activity and accelerate the organic carbon degradation. However, the PTBP biodegradation could be promoted by monophenol PREs (PHA, PCA and FA), but not by polyphenol PREs (CA). On the one hand, PREs could decide the dominant bacterial phyla as carbon source: Proteobacteria and Bacteroidetes with high carbon source utilization ability increased by 15.34% and 4.73% in +PRE treatments, respectively; whereas Actinobacteria and Firmicutes with better detoxification/tolerance capacity increased by 15.92% and 9.99% in -PRE treatments, respectively. On the other hand, the functional structure of PREs could determine the functional bacterial genus: monophenol PREs (PHA, PCA and FA) are conducive to enrich monophenol-degrading bacteria, like, accounting for 1.45%~4.02%; polyphenol PREs (CA) are favored to increase the abundance of polyphenol-degrading bacteria, like, accounting for 3.71%. Furthermore, simple structure of PREs is beneficial for the enrichment of ASVs. The closer the PRE structure are, the more similar the ASV species will be. The community composition of rhizosphere bacteria in different treatments is not a random phenomenon, but a directional adjustment of rhizosphere microbiome by exudates released from plants.
phenolic root exudate;--butylphenol;;rhizosphere bacteria;diversity;community structure
X172
A
1000-6923(2022)04-1812-07
洪志锋(1994-),男,福建漳州人,中山大学硕士研究生,主要研究方向为环境微生物学.发表论文1篇.
2021-09-23
国家自然科学基金资助项目(4207070035,41907293);广东省自然科学基金资助项目(2019A1515012217)
*责任作者, 副教授, adan@zhku.edu.cn; ** 副教授, chaoyuanq@mail.sysu.edu.cn
