山药块茎发育阶段基因共表达网络构建及阶段特异性分析
2022-03-16殷剑美张铅蒋璐张培通
殷剑美 张铅 蒋璐 张培通

摘要: 山药是一种药食同源作物,块茎是其主要产品器官,其发育机制对于山药产量和品质提升相关育种有着重要的指导作用。以不同生长阶段(块茎膨大第1 d、第11 d、第21 d、第31 d、第41 d、第51 d,T1~T6)的山药块茎为材料进行转录组测序,经从头(de novo)组装后获得42 042个unigenes,利用七大数据库进行注释,鉴定后发现山药块茎形成过程中涉及大量基因,包括激素合成、信号转导等相关基因。阶段特异性分析结果表明,块茎形成初始阶段为块茎发育的关键时期;加权基因共表达网络分析结果表明,在山药块茎形成初期及后期,激素相关基因的表达特别活跃,脱落酸(ABA)、茉莉酸(JA)是山药块茎生长发育的重要激素。研究结果为进一步研究山药块茎膨大分子机制奠定了基础。
关键词: 山药;转录组测序;阶段特异性基因;基因共表达网络
中图分类号: S632.101 文献标识码: A 文章编号: 1000-4440(2022)01-0030-09
Abstract: Chinese yam is a crop with hamology of medicine and food, and tuber is its main product organ. The development mechanism of tubers plays an important guiding role in the breeding related to the improvement of yield and quality of yam. Based on transcriptome sequencing of tubers at different growth stages (T1-T6), after de novo assembly, 42 042 unigenes were obtained and annotated using seven databases. After identification, it was found that a large number of genes were involved in the process of tuber formation, including hormone synthesis-related genes, signal transduction-related genes, and so on. The results of stage specificity analysis showed that the initial stage of tuber formation was a critical period. The results based on the weighted gene co-expression network analysis (WGCNA) showed that the expression of hormone-related genes was very active in the early and late stages of tuber growth. Abscisic acid (ABA) and jasmonic acid (JA) were important hormones for the growth and development of yam tubers. The results lay a foundation for further study on the molecular mechanism of tuber enlargement of yam.
Key words: yam;transcriptome sequencing;stage-specific gene;weighted gene co-expression network analysis
参薯山药(Dioscorea alata)是薯蓣科薯蓣属药食同源的作物,其地下块茎是参薯山药的主要产品器官[1],具有高钾、低纤维、高蛋白质、营养丰富等特点[2]。参薯山药块茎在发育过程中边膨大边积累养分,关于其块茎形成的分子机制研究较少。随着高通量测序技术的发展,人们可以从分子水平研究植物的动态发育[3-4],包括激素和次生代謝物形成的分子机制等[5]。目前,关于地下块茎发育的研究主要集中在马铃薯、甘薯等作物上,且块茎发育与激素密切相关。前人研究发现,ABR1在芥菜块茎中的表达量显著增加,脱落酸(ABA)介导的信号参与了块茎膨大[6];在马铃薯块茎膨大早期,调控赤霉素(GA)降解的基因表达量上调,GA含量迅速下降,从而促进块茎膨大[7]。Sarker等[8]认为,茉莉酸(JA)和茉莉酸甲酯(MeJA)在马铃薯块茎形成中起着重要作用。笔者之前对参薯山药块茎不同发育时期激素含量的研究发现,GA在块茎形成初期的含量较高,可以抑制块茎形成;ABA和JA对块茎膨大具有促进作用[9]。
本研究以不同生长时期参薯山药块茎为材料进行转录组测序,采用阶段特异性(Stage specific,SS)方法分析块茎发育过程中相关激素基因的表达,构建其基因共表达网络(Weighted gene co-expression network analysis, WGCNA),以期为揭示参薯山药块茎形成的分子调控机制提供理论依据。
1 材料与方法
1.1 试验材料
试验材料为米易山药(Dioscorea alata L.),引自四川省米易县,属于国家地理标志农产品,于2018年4月26日种植于江苏省农业科学院六合动物科学基地,当年10月24日收获。块茎于9月4日(播种后132 d)开始膨大,分别于块茎膨大的第11 d(9月14日)、21 d(9月24日)、31 d(10月4日)、41 d(10月14日)、51 d(10月24日)取地下块茎,每次取3株混合,用液氮速冻后保存于-70 ℃冰柜中备用。
1.2 RNA和cDNA文库的制备
采用TRIzol法(Invitrogen,USA)进行总RNA的提取,用1%琼脂糖凝胶电泳检测RNA是否降解及污染,用Nano光度计(IMPLEN,USA)检测RNA的纯度(OD260/280),用QubitRNA试剂盒(Life Technologies,USA)测定RNA的浓度,用RNA Nano 6000试剂盒(Agilent Technologies,USA)评估RNA的完整性。
取1.5 μg RNA,用poly-T寡聚物磁珠纯化mRNA。利用随机六聚体引物和M-MuLV逆转录酶(RNase H)合成第1链cDNA,用DNA聚合酶Ⅰ和RNase H合成第2链cDNA。用TruSeq PE Cluster Kit v3 cBot HS(Illumia)在cBot聚类生成系统上对索引编码样品进行聚类。在Illumina Hiseq平台上对文库进行测序,获得大小为125 bp/150 bp的双端reads。
1.3 测序数据质量的评估
对测序所得原始数据进行过滤,去除接头(未知碱基比例>10%,Qphred值≤20的碱基数>50%)。
1.4 拼接及功能注释
采用Grabherr[10]对clean reads进行拼接,获得unigene,随后进行七大数据库(包括NR、NT、PFAM、KOG、Swiss-Prot、KEGG、GO)功能的注释,同时基于斯皮尔曼相关系数(Spearman correlation coefficient,SCC)进行主成分分析(Principal component analysis,PCA)。
1.5 基因表达量的计算和差异基因的筛选
采用基因表达水平估算方法[11]对readcount(read数)进行每百万碱基对测序的每千碱基转录序列片段(fragments)数目(FPKM)估算,计算公式如下:
FPKM=total exon fragmentsmapped reads × exon length
式中,total exon fragments表示总外显子片段数,mapped reads表示定位read数(百万);exon length表示外显子长度(kB)。
以P值<0.05且 |log2FoldChange|>1为阈值筛选差异表达基因(Differentially expressed genes,DEGs)。
SS(i,j)=1-MaxEikEij,1≤k≤6,k≠j
1.6 阶段特异基因的筛选及共表达网络的构建
通过阶段特异性(Stage specific,SS)算法[12],筛选得分>0.5的unigene作为阶段特异基因。对基因变异系数>0.5的基因进行加权基因共表达网络分析(WGCNA)[13]。
1.7 实时荧光定量PCR验证
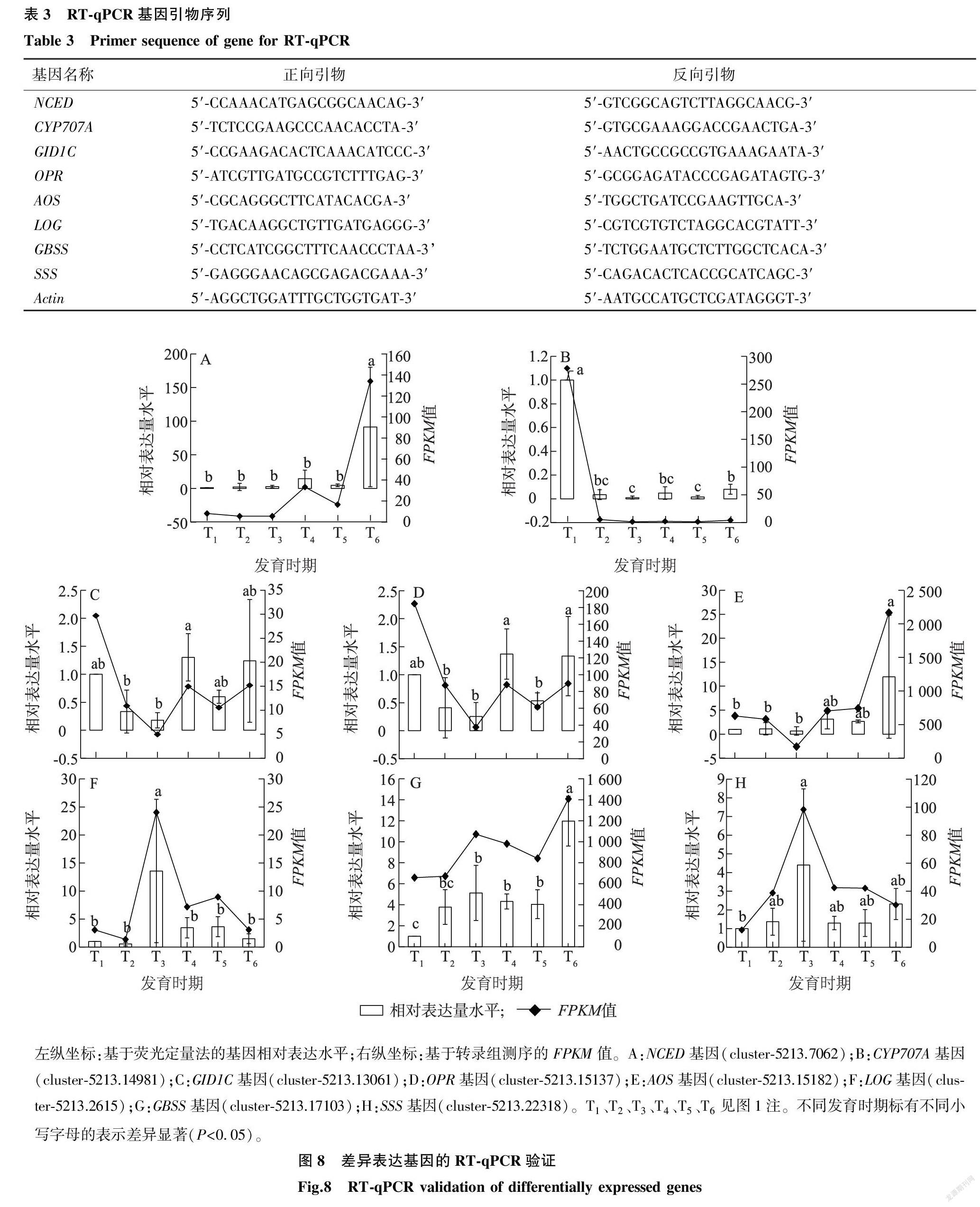
采用PrimeScript反转录试剂盒(TaKaRa,大连)合成cDNA,根据转录组表达量的差异,随机挑选8个基因,使用Primer 5.0(Premier BIosoft International,USA)设计引物,选择DaActin作为内参基因进行RT-qPCR。反应程序:95 ℃ 30 min;95 ℃ 5 s,58 ℃ 30 s,72 ℃ 30 s,45个循环。通过定量反应的熔解曲线,验证引物的特异性是否满足试验要求。RT-qRCR反应在Light Cycle 480荧光定量PCR检测系统(Roche, 瑞士)中进行,依据2-△△Ct算法计算基因的相对表达量[14]。
2 结果与分析
2.1 测序数据分析与基因注释
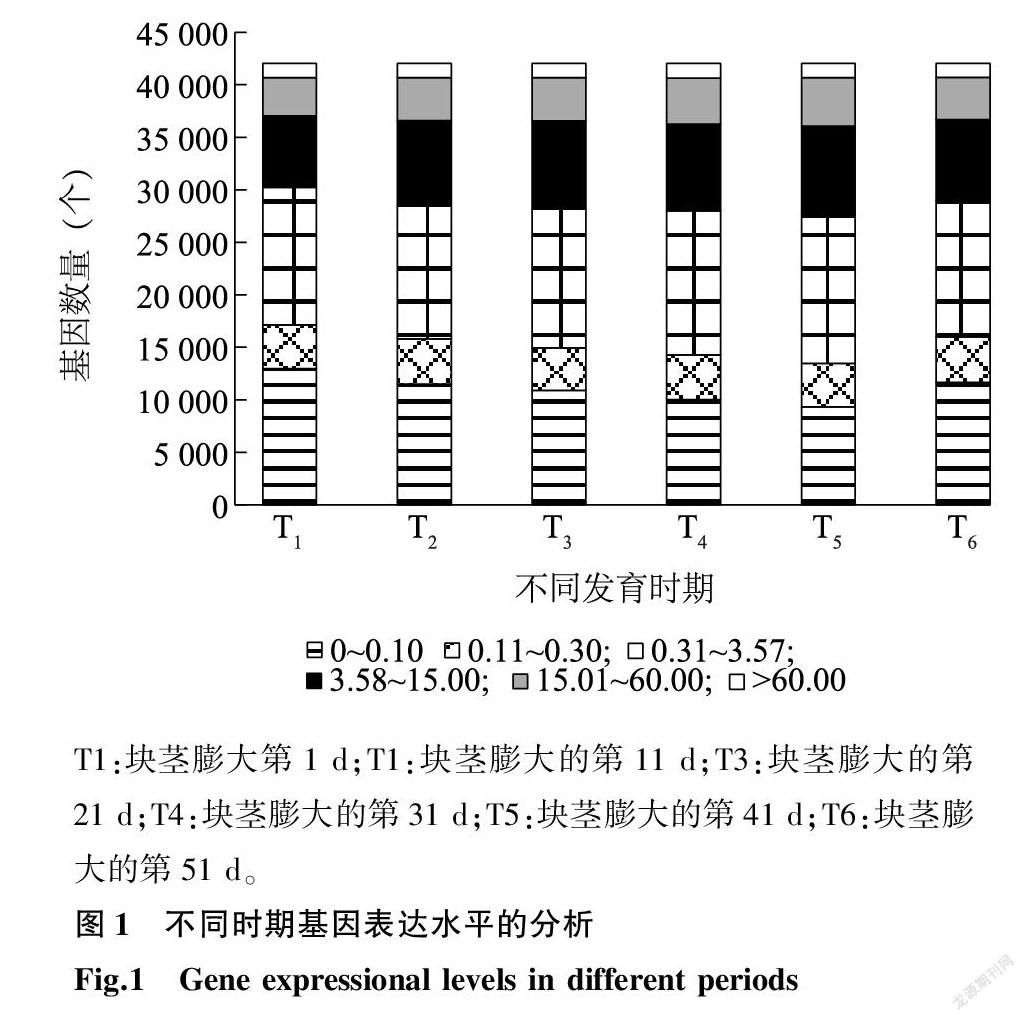
将测序的原始数据过滤后,平均每个文库含有50 266 758个clean reads,共获得186 793个transcripts和42 042个unigenes。不同FPKM区间的unigene数量如图1所示,可以看出,在不同时期,大部分基因的FPKM值为 0~0.10、0.31~3.57。
T1:块茎膨大第1 d;T1:块茎膨大的第11 d;T3:块茎膨大的第21 d;T4:块茎膨大的第31 d;T5:块茎膨大的第41 d;T6:块茎膨大的第51 d。
分别有46.48%、24.15%、14.62%、56.74%、35.30%、46.48%和44.01%的基因在GO、KEGG(KO)、KOG、NR、NT、PFAM和SwissProt数据库中得到注释。其中在NR数据库中的注释结果表明,与参薯山药比对率较高的植物是棕榈(23.6%)、海枣(18.7%)、菠萝(7.1%)、小果野蕉(6.8%)和芦笋(5.6%),它们与参薯山药均属于单子叶植物,具有较相似的基因信息。
由图2可以看出,在GO数据库中注释到55个富集条目,其中包括25个“生物学过程”、20个“细胞组成”和10个“分子功能”。“生物学过程”基因主要富集在“细胞过程”(GO:0009987)、“代谢过程”(GO:0008152)和“单一组织过程”(GO:0044699);“细胞组成”基因主要富集在“细胞”(GO:0005623)、“部分細胞”(GO:0044464)和“细胞器官”(GO:0043226);“分子功能”基因主要富集在“结合”(GO:0005488)和“催化活性”(GO:0003824)。
共有10 154个unigenes注释到130个KEGG通路中,可分为新陈代谢、遗传信息处理、环境信息处理、细胞过程和组织系统5类,其中大部分基因注释到新陈代谢的代谢通路中(图3)。
主成分分析结果显示,6个样本可明显分为T1时期、T2~T6时期,T1时期和其他各时期的相关性较低,T2~T6时期较为聚集;块茎在生长早期(T1时期)和后期(T2~T6时期)存在较大差异(图4)。
2.2 块茎发育过程中不同表达基因和阶段特异基因分析
分析差异表达基因发现,两两样品之间的DEGs数量为442~6 774个,其中与T5时期相比,T1时期的DEGs数量最多,有3 790个上调基因,2 984个下调基因(表1)。共获得8 222个阶段特异表达基因,数量范围为599~3 621个(图5)。GO分析发现,T1时期的大多数基因与蛋白质磷酸化、去磷酸化、细胞蛋白修饰过程、蛋白质修饰过程、生长素应答、脂质转运、激素应答等有关。
柱旁数据表示注释到相应基因的数量。
2.3 T1时期差异表达基因及特异基因分析
主成分分析和不同发育阶段特异基因分析结果显示,T1时期和T2~T6时期之间存在明显差异,在其有表达差异的基因中,共有211个基因编码转录因子,其中36个基因编码AP2/ERF,另外编码WRKY、C2H2、MYB、NAC、bHLH、bZIP的基因均表现为高表达。
通过比较T1时期外的其他时期与T1时期发现,差异表达的基因达9 701个,其中4 546个基因的表达量上调,5 181个基因的表达量下调。GO分析结果显示,上调表达的基因与细胞壁构建、细胞骨架构建、DNA复制、焦磷酸酶活性、染色体、DNA代谢过程调控有关;下调表达的基因与核糖核酸结合、蛋白激酶活性、蛋白激酶磷酸化活性、大分子代谢、初级代谢及氮化合物过程的调控、细胞大分子和大分子调节及细胞生物合成过程有关。KEGG分析结果表明,上调表达的基因主要与次生代谢物生物合成、氨基酸合成、代谢和降解、柠檬酸循环有关;下调表达的基因主要涉及植物激素信号转导、MAPK信号转导、苯丙氨酸生物合成、酪氨酸生物合成、脂肪酸降解、色氨酸生物合成、玉米素生物合成、生物合成酯类代谢。
2.4 激素相关基因转录谱分析
对所有时期的特异基因进行分析发现,与GA相关的3个关键基因在整个生长期的表达量均较低,0 2.5 基因共表达网络的构建 对20 988个基因进行共表达网络构建,可将其分为图6中的25个不同模块。进一步分析发现,模块1(绿黄模块,181个基因)与T1时期高度相关,其中有18个编码转录因子,包括转录因子BHLH148、WRKY与乙烯应答相关转录因子ERF036 like等。构建T1时期特征模块基因间联通性的共表达网络,详见图7。 T1-1、T1-2、T1-3:T1时期取样块茎的3个重复,其他处理以此类推。共分为25个不同模块,其中模块1(绿黄模块,181个基因)与T1时期高度相关;图例数据表示模块与样本间的相关性。 正方形节点:核心基因;菱形节点:转录因子及调控激酶基因;正六边形节点:合成和代谢相关基因;圆形节点:其他基因。 2.6 RT-qPCR验证 以Actin为内参基因,随机选取8个基因进行RT-qPCR,测定其表达量。引物序列见表3,基因表达量见图8。基因荧光定量表达趋势与转录组测序结果一致,说明转录组测序结果可靠。 左纵坐标:基于荧光定量法的基因相对表达水平;右纵坐标:基于转录组测序的FPKM值。A:NCED基因(cluster-5213.7062);B:CYP707A基因(cluster-5213.14981);C:GID1C基因(cluster-5213.13061);D:OPR基因(cluster-5213.15137);E:AOS基因(cluster-5213.15182);F:LOG基因(cluster-5213.2615);G:GBSS基因(cluster-5213.17103);H:SSS基因(cluster-5213.22318)。T1、T2、T3、T4、T5、T6见图1注。不同发育时期标有不同小写字母的表示差异显著(P<0.05)。 3 讨论 3.1 山药块茎发育关键时期分析 DEGs主要发生在块茎形成初期,T1时期的特异基因数量最多,推测块茎形成初期是最重要的阶段,这与马铃薯相关研究结果[15]一致。T1时期上调表达的DEGs在蛋白质磷酸化、蛋白激酶途径中显著富集,而蛋白质磷酸化作为细胞信号系统的重要组成部分,经常参与许多生物过程,如植物生长发育、细胞分化、刺激反应和抗应激调节[16]。T1时期的特异基因编码211个转录因子,揭示许多编码TFs的基因参与块茎形成。另外T1时期表达的大部分基因与激素有关,说明激素与块茎形成关系密切。 3.2 参与山药块茎发育过程的激素相关基因分析 植物激素的协同作用对块茎的形成和生长调节具有重要意义。GA和ABA是1对经典的植物激素组合,它们的前体都是甲戊龙酸,对种子休眠和萌发、根系发育和开花有拮抗作用,GA和ABA之间的平衡和相互调节在植物生长发育中起着重要作用[17]。关于GA对块茎生长的影响有不同观点,但一般认为,GA可以抑制或延缓块茎的发育。GA的信号转导通过环境和内部条件整合GA受体GID1和抑制因子DELLA,形成GA-GID1-DELLA三聚體复合体,从而调节植物的生长发育[18]。已发现的DELLA蛋白包括拟南芥GAI、RGA、RGL1、RGL2和RGL3[19]以及小麦RHT[20]、大麦SLN1[21]和水稻SLR1[22]等。本研究发现,编码DELLA蛋白的GAI在T1时期高表达,推测块茎开始形成时,GA就开始受到抑制,导致T2时期的GA含量迅速下降后,在以后生育期处于较低水平[9]。 研究发现,NCED3高表达显著增加拟南芥的ABA含量[23];ZEP参与拟南芥种子发育中ABA的合成[24];CYP707A[25]编码ABA8′羟化酶。本研究发现,1个NCED基因在T1~T5时期下调表达,但在T6期强烈上调表达;3个ZEP基因中有2个基因在早期高表达;3个CYP707A基因在T1时期上调表达,在T2~T6期的表达量显著降低。在块茎形成过程中,调控ABA的基因的表达一直较活跃,含量保持上升[9]。对水稻的研究发现,高ABA含量会诱导淀粉积累量增加[26],在山药块茎发育后期ABA含量持续增长,不仅有利于块茎生长,而且有利于淀粉积累。 丙二烯氧化物合酶(AOS)是JA合成途径中的第1个特异性酶,可以催化氧化丙二烯的合成,12-氧-植物二烯酸还原酶(OPR)是JA合成的关键酶,AOS基因的过度表达可增加马铃薯中JA含量[27]。在本研究中,AOS、OPR在整个生育期一直高表达,尤其在T1时期,推测在山药块茎的整个发育过程中,JA的合成和代谢相对较强,使得块茎中的JA含量较高[9]。 共表达网络分析结果显示,在T1时期的特异性模块中,乙烯应答转录因子ERF036 like、真核细胞翻译起始因子等基因的表达水平较高。ERF转录因子家族参与多种生物过程,包括激素信号转导、生长发育、代谢及对生物和非生物胁迫的响应[28-30],推测在参薯山药块茎发生初期涉及多种激素信号转导过程。在T6时期的特异性模块中,含有2个转录因子(WRKY71、WRKY79)编码基因。在水稻胚、糊粉层细胞中,WRKY71表达由ABA诱导并被GA抑制,α-淀粉酶amy32B和赤霉素诱导转录激活因子GAMYB协同表达[31];WRKY79在玉米中过度表达会增加茉莉酸和乙烯途徑、活性氧清除中相关基因及其他相关基因的表达量[32],推测在块茎发育后期,激素相关基因的表达特别活跃。 4 结论 本研究对山药转录组数据的分析发现,在山药块茎形成初期,尤其是T1时期为块茎发育的重要时期,阶段特异基因数目最多,上调的DEGs与细胞生长、激素均密切相关;块茎发育后期激素相关基因表达特别活跃,ABA和JA是块茎生长发育的重要激素。研究结果与之前关于块茎发育中激素含量的研究结果一致,转录组测序结合生理测定结果可以为进一步分析山药块茎膨大的分子机制奠定基础。 参考文献: [1] 韦本辉.中国淮山药栽培[M]. 北京:中国农业出版社,2013:2-4. [2] OSELEBE H O, OKPORIE E O. Evaluation of water yam (Dioscorea alata L.) genotypes for yield and yield components in Abakaliki agro-ecological zone of Nigeria[J]. Agro-Science, 2008, 7(3): 179-185. [3] LE B H, CHENG C, BUI A Q, et al. Global analysis of gene activity during Arabidopsis seed development and identification of seed specific transcription factors[J]. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(18): 8063-8070. [4] BELMONTE M F, KIRKBRIDE R C, STONE S L, et al. Comprehensive developmental profiles of gene activity in regions and subregions of the Arabidopsis seed[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(5): E435-E444. [5] ZHOU T, LUO X J, YU C N, et al. Transcriptome analyses provide insights into the expression pattern and sequence similarity of several taxol biosynthesis-related genes in three Taxus species[J]. BMC Plant Biology, 2019, 19(1): 33-42. [6] SUN Q, ZHOU G F, CAI Y F, et al. Transcriptome analysis of stem development in the tumourous stem mustard Brassica juncea var. tumida Tsen et Lee by RNA sequencing[J]. BMC Plant Biology, 2012, 12: 53. [7] ROUMELIOTIS E, VISSER R G F, BACHEM C W B. A crosstalk of auxin and GA during tuber development[J]. Plant Signaling & Behavior, 2012, 7(10): 1360-1363. [8] SARKER D, PANDEY S K, SHARMA S. Cytokinins antagonize the jasmonates action on the regulation of potato (Solanum tuberosum) tuber formation in vitro[J]. Plant Cell, Tissue and Organ Culture, 2006, 87(3): 285-295. [9] 張 铅,蒋 璐,张培通,等. 长江中下游地区参薯块茎发育动态特征分析[J]. 植物生理学报,2020,56(12):2736-2744. [10]GRABHERR M G, HAAS B J, YASSOUR M, et al. Full-length transcriptome assembly from RNA-Seq data without a reference genome[J]. Nature Biotechnology, 2011, 29: 644-652. [11]TRAPNELL C, ROBERTS A, GOFF L, et al. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks[J]. Nature Protocols, 2012, 7(3): 562-578. [12]ZHAN J P, THAKARE D, MA C, et al. RNA sequencing of laser-capture microdissected compartments of the maize kernel identifies regulatory modules associated with endosperm cell differentiation[J]. The Plant Cell, 2015, 27(3): 513-531. [13]LANGFELDER P, HORVATH S. WGCNA: an R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9(1): 559. [14]LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-△△Ctmethod[J]. Methods, 2001, 25(4): 402-408. [15]KLOOSTERMAN B, VORST O, HALL R D, et al. Tuber on a chip: differential gene expression during potato tuber development[J]. Plant Biotechnology Journal, 2005, 3(5): 505-519. [16]PAWSON T, SCOTT J D. Signaling through scaffold, anchoring, and adaptor proteins[J]. Science, 1997, 278(5346): 2075-2080. [17]SHU K, LIU X D, XIE Q, et al. Two faces of one seed: hormonal regulation of dormancy and germination[J]. Molecular Plant, 2016, 9(1): 34-45. [18]SUN T P. The molecular mechanism and evolution of the review GAGID1-DELLA signaling module in plants[J]. Current Biology, 2011, 21: 338-345. [19]TYLER L, THOMAS S G, HU J H, et al. DELLA proteins and gibberellin regulated seed germination and floral development in Arabidopsis[J]. Plant Physiology, 2004, 135: 1008-1019. [20]PENG J, RICHARDS D E, HARTLEY N M, et al. ‘Green Revolution genes encode mutant gibberellin response modu lators[J]. Nature, 1999, 400: 256-261. [21]FU X D, RICHARDS D E, AIT-AIT T, et al. Gibberellin-mediated proteasome-dependent degradation of the barley DELLA protein SLN1 repressor[J]. Plant Cell, 2002, 14(12): 3191-3200. [22]IKEDA A, UEGUCHI-TANAKA M, SONODA Y, et al. Slender rice, a constitutive gibberellin response mutant, is caused by a null mutation of the SLR1 gene, an ortholog of the height-regulating gene GAI/RGA/RHT/D8[J]. Plant Cell, 2001, 13: 999-1010. [23]IUCHI S, KOBAYASHI M, TAJI T, et al. Regulation of drought tolerance by gene manipulation of 9-cis-epoxycarotenoid dioxygenase, a key enzyme in abscisic acid biosynthesis in Arabidopsis[J]. Plant Journal for Cell & Molecular Biology, 2010, 27(4): 325-333. [24]SCHWARZ N, ARMBRUSTER U, LVEN T, et al. Tissue-specific accumula tion and regulation of zeaxanthin epoxidase in Arabidopsi reflect the multiple functions of the enzyme in plastids[J]. Plant Cell Physiology, 2015, 56(2): 346-357. [25]KROCHKO J E, ABRAMS G D, LOEWEN M K, et al. (+)-Abscisic acid 8-hydroxylase is a cytochrome P450 monooxygenase[J]. Plant Physiol, 1998, 118(3): 849-860. [26]WANG Z Q, XU Y J, CHEN T T, et al. Abscisic acid and the key enzymes and genes in sucrose-to-starch conversion in rice spikelets in response to soil drying during grain filling[J]. Planta, 2015, 241(5): 1091-1107. [27]PAN Z Q, CAMARA B, GARDNER H W, et al. Aspirin inhibition and acetylation of the plant cytochrome P450, allene oxide synthase, resembles that of animal prostaglandin endoperoxide H synthase[J]. The Journal of Biological Chemistry, 1998, 273(17): 18139-18145. [28]CAO F Y, DEFALCO T A, MOEDER W, et al. Arabidopsis ETHYLENE RESPONSE FACTOR 8(ERF8) has dual functions in ABA signaling and immunity[J]. BMC Plant Biol, 2018, 18 (1): 211. [29]LIU K, LI Y H, CHEN X N, et al. ERF72 interacts with ARF6 and BZR1 to regulate hypocotyl elongation in Arabidopsis[J]. Journal of Experimental Botany, 2018, 69 (16): 3933-3947. [30]JIN Y, PAN W Y, ZHENG X F, et al. OsERF101, an ERF family transcription factor, regulates drought stress response in reproductive tissues[J]. Plant Molecular Biology, 2018, 98(1/2): 51-65. [31]XIE Z, ZHANG Z L, ZOU X L, et al. Interactions of two abscisic-acid induced WRKY genes in repressing gibberellin signaling in aleurone cells[J]. The Plant Journal, 2006, 46(2): 231-242. [32]FU J Y, LIU Q, WANG C, et al. Zmwrky79 positively regulates maize phytoalexin biosynthetic gene expression and is involved in stress response[J]. Journal of Experimental Botany, 2018, 69(3): 497-510. (責任编辑:徐 艳)
