花青素调控植物花色的研究进展
2021-03-01刘国元方威余春梅连博琳陈艳红钟非张健
刘国元 方威 余春梅 连博琳 陈艳红 钟非 张健
摘要 花色是观赏植物最重要的特征之一,它的形成和调控受到多种内外部因素的影响。因此,了解观赏植物颜色产生的机理及其调控,为培育和改良观赏植物颜色新品种提供了重要的理论依据和前提。概述了常见花色素,尤其是花色苷的合成和种类;重点介绍了MYB转录因子调控花青素影响花色的最新研究进展,为观赏植物花色改良提供了广阔的背景。
关键词 花色;花青素;观赏植物;调控
中图分类号 Q943.2 文献标识码 A 文章编号 0517-6611(2021)03-0001-04
doi:10.3969/j.issn.0517-6611.2021.03.001
Abstract Flower color is one of the most important characteristics of ornamental plants. Its formation and regulation is affected by a series of internal and external factors. The understanding of mechanism and regulation of ornamental plant color production provides theoretical basis for breeding and improvement of new ornamental plant varieties.This article summarized the common pigment, especially the synthesis and types of anthocyanins. It focuses on the latest research progress of MYB transcription factors regulating anthocyanins to affect flower color, which provides a broad background for flower color improvement of ornamental plants.
Key words Flower color;Anthocyanidins;Ornamental plants;Regulation
植物花器官尤其是花瓣的颜色多种多样,它不仅可以吸引传粉者,同时也对保护植物花粉活性、提高授粉成功率有重要意义。植物开花后花器官暴露在阳光下,花瓣中类黄酮、类胡萝卜素等物质对维持紫外光线下花粉活性、防止花粉基因突变有重要作用。花色更是观赏植物的重要性状,对花色的研究一直是研究的热点。
研究者们已经对花瓣颜色的形成原因和调控机理进行了大量的研究。花色的形成与花瓣自身的结构、包含色素的种类及色素位置分布有关,受到外界环境因素以及自身遗传因素的调控。外界环境因素主要包括温度、光照、水等物理因素以及環境的pH、矿物离子、植物激素、糖类等化学因素。自身遗传因素主要包括植物本身产生的色素类种类以及其合成过程中的相关调控基因的差异。笔者介绍了一些常见色素种类,并重点描述了花青素合成和调控相关的最新进展,以利于更好地理解观赏植物花色形成过程。
1 花瓣中色素分布
花的颜色与花瓣的组织结构、花瓣细胞中色素的类型和数量有关,其中色素起主要作用。花瓣的组织结构可分为上表皮、栅栏组织、海绵组织和下表皮4部分。色素在花瓣的上表皮中分布较多,在有些颜色较深度花瓣中,栅栏组织和下表皮也有色素分布[1-2]。虽然花瓣的海绵组织中没有色素分布,海绵组织的厚度和密度会影响花色的亮度。海绵组织越厚越密,花瓣颜色越鲜艳[3]。
不同类型的色素在细胞中的亚细胞定位也不同。类胡萝卜素一般在细胞质的质体中积累,类黄酮主要存在于液泡中。在有些植物花瓣表皮细胞壁中也检测到黄酮类化合物的存在[4]。
2 色素的主要类型
迄今为止,根据色素的化学结构、细胞定位和生物化学合成途径,可以把色素大致上分为三大类:类胡萝卜素、类黄酮和生物碱。
2.1 类胡萝卜素
类胡萝卜素是自然界中分布最广泛的色素,它又能分为胡萝卜素和叶黄素两大类。除了花,它们还存在于高等植物的果实、叶和根中。类胡萝卜素一般表现出较为鲜艳的红色、橙色和黄色,因为其主要吸收短波长的光[5]。在不同物种的花瓣中类胡萝卜素的具体成分并不相同,在桂花中淡黄色花瓣含有少量β-胡萝卜素,金黄色花瓣中叶黄素含量高,橙红色花瓣中α-胡萝卜素和β-胡萝卜素含量较高[6];万寿菊和黄色菊花的花瓣中只含有叶黄素[7-8];莲花中含量较高的是紫黄质和玉米黄质[9];文心兰黄色花瓣中的主要成分是反式紫黄素和9-顺式紫黄素[10];藏红花花瓣中色素的主要成分是玉米黄素、β-胡萝卜素和ζ-胡萝卜素[11]。
2.2 类黄酮
黄酮类化合物是一大类次生代谢物,广泛存在于植物体内。一般来说,所有含有2-苯基色酮核结构的物质都可以称为黄酮类化合物,也简称为类黄酮。类黄酮是最重要的色素类群,它产生的颜色范围最广,从淡黄色到蓝紫色,是各种观赏植物花瓣中最重要的色素之一,在菊花[12]、大丽花[13]、月季[14]、紫罗兰[15]、芍药[16-19]等物种中都已被报道是主要色素。在黄酮类化合物中,主要有红色系、纯黄系2种颜色系列。其中红色系主要包括花青素,它能调控的颜色从粉红色到蓝紫色;其他黄酮类化合物主要属于纯黄系列,如深黄色的查尔酮和极光酮,淡黄色或近乎无色的黄酮和黄酮醇等。相同物种的不同颜色的花瓣中,黄酮类化合物的组成也可能有很大差异,如白色菊花中仅含有黄酮和黄酮醇,而粉红色菊花中主要含有花青素、黄酮和黄酮醇[20]。
2.3 生物碱
生物碱是一类含氮的碱性有机物,主要包括甜菜碱、罂粟碱和小檗碱等。其中,甜菜碱是一种水溶性氮化合物,主要存在于红色甜菜和一些植物的花、果、根、叶中。迄今为止,甜菜碱仅在石竹目植物中被检测到,而且甜菜碱和花青素是不共存的[21-22]。甜菜青素和甜菜黄素的含量和比例可以决定花色是红色、黄色还是中间色[22-23]。
3 花青素和花色
在三大类色素中,黄酮类化合物可以产生从淡黄色到蓝紫色的全光谱颜色。花青素也是黄酮类化合物的主要组成,表现出从粉红色到蓝紫色的广泛的颜色,在植物的花色形成过程中起着不可替代的作用。目前已知的花青素有100多种[24],其中有6种花青素在植物中比较常见,即天竺葵花青素、矢车菊花青素、飞燕草花青素、芍药花青素、牵牛花花青素和锦葵花青素。芍药花青素是由矢车菊花青素合成产生的,牵牛花花青素和锦葵花青素由飞燕草花青素合成而来的。因此,天葵花青素、矢车菊花青素和飞燕草花青素是3种最主要的花青素[25]。
由于花青素在植物中不能稳定的单独存在,它们可以与糖类以糖苷键结合,以花色苷的形式存在。可以结合形成花色苷的糖基主要有葡萄糖、鼠李糖、木糖、半乳糖和阿拉伯糖,以及由他们组成的二糖或三糖,最常见的是3-单葡萄糖苷、5-二葡萄糖苷、3,5-二糖苷和3,7-二糖苷[26]。
花色苷是一种糖苷,由花青素和各种糖自然形成。它们稳定地定位在植物器官中,已有研究表明,颜色差异与花色苷含量有关,Kazuma等[27]对白色、蓝色等蝶豆花花瓣中的花色苷含量进行了测定,发现花色苷的差异是各种颜色产生的重要原因。在银杏[28]、猪笼草[29]等植物的花中均有类似报道。相同花青素结合的糖苷类型不同也会导致花色的差异,在睡莲中,被检测到的飞草花青素3-半乳糖苷(Dp3Ga)呈红色,而飞草花青素3′-半乳糖苷(Dp3′Ga)呈蓝色[30]。
3.1 花青素的生物合成及关键基因
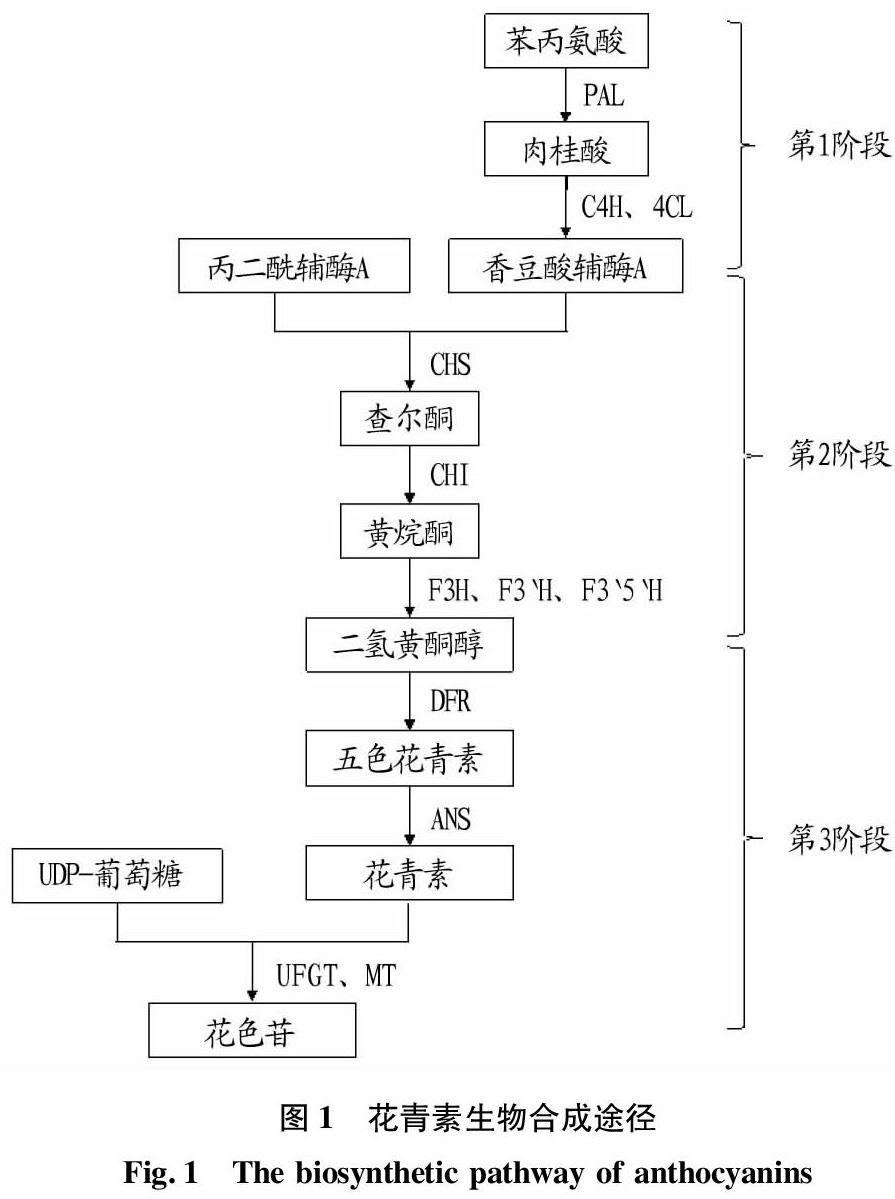
花青素在植物中的生物合成途径及关键基因已有过详细报道[31],其生物合成可以分为3个阶段,初始合成底物是苯丙氨酸(图1)。第1阶段是苯丙氨酸解氨酶(PAL)、肉桂酸-4-羟化酶(C4H)和4-香豆酸辅酶A连接酶(4CL)催化苯丙氨酸轉化为香豆酸辅酶A。第2阶段是1个香豆酸辅酶A分子和3个丙二酰辅酶A分子先后在查尔酮合酶(CHS)、查尔酮异构酶(CHI)、黄烷酮-3-羟化酶(F3H)、类黄酮3′-羟化酶(F3′H)和类黄酮3′5′-羟化酶(F3′5′H)的催化作用下生成二氢黄酮醇。第3阶段是二氢黄酮醇4-还原酶(DFR)和花青素合成酶(ANS)催化二氢黄酮醇合成各种类型的花青素,然后进一步在类黄酮葡萄糖基转移酶(UFGT)和甲基转移酶(MT)的作用下通过一系列的糖基化和甲基化步骤进行修饰,与UDP-葡萄糖结合形成稳定的花色苷。
CHS是植物花青素生物合成的第1个关键基因,在花色苷的合成和积累过程中起着重要作用。已有研究表明,CHS普遍存在于陆地植物、藻类植物中[32]。CHS在矮牵牛[33]、蝴蝶兰[34]和芍药[19]等观赏植物中都已被鉴定出来。该基因家族的蛋白质序列在不同的物种中高度保守,相似性高达80%~90%[35]。在矮牵牛中转入CHS1基因可以使其花色由白色变为粉红色[36],而在烟草中过表达海棠的CHS基因可以促进花青素的积累得到深红色花瓣[37]。
CHI是植物花青素生物合成过程中的第2个关键基因,它能催化查尔酮的异构化。查尔酮在CHI蛋白的作用下被修饰成黄烷酮。植物中的CHI蛋白根据其可催化的底物可以分为两类:①以6′-羟基查尔酮为底物,②6′-羟基查尔酮和6′-脱氧查尔酮为底物[38]。无论是哪种类型,CHI的表达会影响植物的类黄酮代谢水平,从而影响花色的发育。已有研究在康乃馨、紫锥花、仙客来和烟草等植物中发现CHI表达量的减少会导致花瓣中查尔酮的大量积累,使花瓣颜色变黄[39]。
F3H蛋白可以催化黄烷酮C3位羟基化生成二氢黄酮醇,它是类黄酮生物合成途径分支点上的关键酶。该基因经常与CHS和CHI共同调控并催化下游产物的形成[40]。该基因在肉桂[41]、雪莲[42]和芍药[19]等物种中已被克隆。
DFR是植物花青素生物合成途径中的另一个关键基因,在花色发育中起着重要作用。DFR蛋白能将二氢杨梅素类黄酮、二氢槲皮素和二氢山奈酚3种类型的二氢黄酮醇还原为相应的无色花青素,然后在下游基因修饰下合成各种花青素[43]。由于DFR表达的差异及其底物特异性会导致花色的变异,因此DFR对花色发育的调控机制一直是一个热点。在亚洲百合[44]、龙胆[45]、芍药[19]和雪莲[46]等观赏植物中均已鉴定到了DFR基因。在牡丹中发现DFR在花色苷积累较多的花瓣中表达量最高[19]。这些研究表明,DFR对花色发育的调控作用可能发生在转录水平。
ANS是花青素生物合成过程的另一个关键基因。该基因在Fe2+和2-氧化戊二酸作用下催化亮氨酸花青素转化为有色花青素[47]。前人研究表明,ANS在许多植物中由一个小的基因家族编码,该家族已在连翘、非洲菊[48]和芍药[19]等物种中被鉴定出来。ANS基因的缺失会导致连翘花瓣中花青素的减少[49],藤本花中也发现该基因能调控花瓣颜色变化[50]。
3.2 转录因子调控花青素合成
除了花青素生物合成途径中的结构基因外,许多转录因子已被报道可以通过调节结构基因的表达来调控花色的形成[51]。目前,已知的参与花色形成的转录因子主要有三大类:MYB、bHLH和WD40[52],三者主要通过形成MBW复合体来调控结构基因的表达,MYB转录因子是MBW复合体中的主要决定因子[53-54]。在MYB的3个亚型中,R2R3-MYB和R3-MYB型的转录因子通常被认为与花青素的代谢和调控密切相关[55-56]。
3.2.1 R2R3-MYB类型转录因子与花青素合成。
已有研究表明,有些R2R3-MYB转录因子对花青素合成途径的结构基因有促进作用,而有些R2R3-MYB转录因子抑制花青素合成途径基因的表达[57]。在非洲菊中,GhMYB10能促进花色苷的合成[58]。龙胆花GtMYB3基因的突变会引起花色漂白[59],百合中LhMYB6和LhMYB12能正调控花青素的生物合成[60]。金鱼草中首次鉴定到了抑制花青素合成的R2R3-MYB转录因子AmMYB308[61]。随后,草莓中FaMYB1[62]和FcMYB1[63]、矮牵牛中PhMYB27[56]、葡萄中VvMYBC2-L1/3和VvMYB4-like[57]、桃中PpMYB17-20、蘋果中MdMYB16和MdMYB15L、杨树中PtrMYB182和PtrMYB57、水仙中NtMYB2相继被鉴定到负调控花青素的合成。
然而,R2R3-MYB负调控花青素生物合成的分子机制目前仍未完全阐明,根据已有研究,主要有两类R2R3-MYB阻遏因子能抑制花青素的合成,一类是FaMYB1-like类型的转录因子,
这种类型的阻遏因子不能直接与靶基因的启动子结合,因此自身不能抑制转录,它作为一个辅阻遏蛋白结合到MBW复合体中,以抑制通常由MBW激活复合体靶向的基因。目前已知的该类转录因子主要有PhMYB27、FaMYB1、VvMYBC2、MdMYB15L、PtrMYB182和MtMYB2[56]。另一类是AtMYB4-like型阻遏因子,通过与花青素途径中许多结构基因的启动子中的MYB基序结合,作为直接阻遏因子,导致该途径的下调,它们通过其C末端结构域的基序来抑制转录。
3.2.2 R3-MYB转录因子与花青素合成。
除R2R3-MYB型转录因子外,拟南芥AtMYBL2和AtCPC、矮牵牛、龙胆GtMYB1R1和GtMYB1R9、杨树PtrRML1、番茄SlMYBATV和SlTRY等R3-MYB型转录因子也能对花青素的合成产生负调控作用[56,64]。在酸浆草中,通过QTL定位的方式找到了一个调控花瓣花青素浓度的主效基因ROI1,也是R3-MYB转录因子[65]。还有报道发现,ILMYBL1基因与曼陀罗中花色素的消失有关。BoMYBL2-1序列的缺失会导致紫色甘蓝变种的产生。所有这些研究表明,R3-MY型转录因子对花青素的生物合成也有调控作用。
与R2R3-MYB阻遏蛋白相比,R3-MYB阻遏蛋白在调节花青素生物合成中的作用机制更不明确[56]。目前仅知道CPC-like类型的R3-MYB抑制子可以与bHLH蛋白以竞争方式相互作用,阻止MBW复合体的形成,从而负向调节花青素的产生,拟南芥和矮牵牛等物种中已对此进行了证明[56]。
这些研究表明,正向和负向MYB调控因子之间表达水平的相互控制形成了一个复杂的调控环来调控花青素的生物合成[56]。转录激活因子会诱导抑制子的表达,抑制子的表达会抑制激活因子的表达,同时,抑制子还会对自身的表达产生抑制作用[64]。目前这种调控模式已在矮牵牛[56]、杨树、葡萄、拟南芥等物种中被报道。
4 总结
植物的花色是多种因素共同作用的结果,前人已对色素的种类、成分、含量、生物合成途径、关键基因以及相关调控基因进行了深入的研究,对许多观赏植物花色差异的形成有了较为深入的认识。这些研究对植物花色、叶色等颜色差异的调控机制以及相关色素的合成代谢和积累的研究有重要作用,为观赏植物花色发育与调控的研究、新品种的培育带来了新的机遇和挑战。
参考文献
[1] QI Y Y,LOU Q,LI H B,et al.Anatomical and biochemical studies of bicolored flower development in Muscari latifolium[J].Protoplasma,2013,250(6):1273-1281.
[2] SHOJI K,MIKI N,NAKAJIMA N,et al.Perianth bottomspecific blue color development in tulip cv.murasakizuisho requires ferric ions[J].Plant Cell Physiol,2007,48(2):243-251.
[3] 戴思兰,洪艳.基于花青素苷合成和呈色机理的观赏植物花色改良分子育种[J].中国农业科学,2016,49(3):529-542.
[4] MARKHAM K R,RYAN K G,GOULD K S,et al.Cell wall sited flavonoids in lisianthus flower petals[J].Phytochemistry,2000,54(7):681-687.
[5] CAMARA B.Carotenoids handbook[J].Plant science,2004,167(2):387.
[6] HAN Y J,WANG X H,CHEN W C,et al.Differential expression of carotenoidrelated genes determines diversified carotenoid coloration in flower petal of Osmanthus fragrans[J].Tree Genet Genomes,2014,10(2):329-338.
[7] MOEHS C P,TIAN L,OSTERYOUNG K W,et al.Analysis of carotenoid biosynthetic gene expression during marigold petal development[J].Plant Mol Biol,2001,45(3):281-293.
[8] OHMIYA A,KISHIMOTO S,AIDA R,et al.Carotenoid cleavage dioxygenase(CmCCD4a)contributes to white color formation in chrysanthemum petals[J].Plant Physiol,2006,142(3):1193-1201.
[9] SUZUKI S,NISHIHARA M,NAKATSUKA T,et al.Flower color alteration in Lotus japonicus by modification of the carotenoid biosynthetic pathway[J].Plant Cell Rep,2007,26(7):951-959.
[10] HIEBER A D,MUDALIGEJAYAWICKRAMA R G,KUEHNLE A R.Color genes in the orchid Oncidium Gower Ramsey:Identification,expression,and potential genetic instability in an interspecific cross[J].Planta,2006,223(3):521-531.
[11] CASTILLO R,FERNNDEZ J A,GMEZGMEZ L.Implications of carotenoid biosynthetic genes in apocarotenoid formation during the stigma development of Crocus sativus and its closer relatives[J].Plant Physiol,2005,139(2):674-689.
[12] CHEN S M,LI C H,ZHU X R,et al.The identification of flavonoids and the expression of genes of anthocyanin biosynthesis in the chrysanthemum flowers[J].Biol Plant,2012,56(3):458-464.
[13] THILL J,MIOSIC S,AHMED R,et al.Le Rouge et le Noir:A decline in flavone formation correlates with the rare color of black dahlia(Dahlia variabilis hort.)flowers[J].BMC Plant Biol,2012,12:1-13.
[14] SCHMITZER V,VEBERIC R,OSTERC G,et al.Color and phenolic content changes during flower development in groundcover rose[J].J Am Soc Hortic Sci,2010,135(3):195-202.
[15] TATSUZAWA F,SAITO N,TOKI K,et al.Flower colors and their anthocyanins in Matthiola incana cultivars(Brassicaceae)[J].J Jpn Soc Hortic Sci,2012,81(1):91-100.
[16] ZHAO D Q,HAO Z J,TAO J.Effects of shade on plant growth and flower quality in the herbaceous peony(Paeonia lactiflora Pall.)[J].Plant Physiol Biochem,2012,61:187-196.
[17] ZHAO D Q,HAO Z J,WANG J,et al.Effects of pH in irrigation water on plant growth and flower quality in herbaceous peony(Paeonia lactiflora Pall.)[J].Sci Hortic,2013,154:45-53.
[18] ZHAO D Q,JIANG Y,NING C L,et al.Transcriptome sequencing of a chimaera reveals coordinated expression of anthocyanin biosynthetic genes mediating yellow formation in herbaceous peony(Paeonia lactiflora Pall.)[J].BMC Genomics,2014,15:1-17.
[19] ZHAO D Q,TAO J,HAN C X,et al.Flower color diversity revealed by differential expression of flavonoid biosynthetic genes and flavonoid accumulation in herbaceous peony(Paeonia lactiflora Pall.)[J].Mol Biol Rep,2012,39(12):11263-11275.
[20] 陳建.非洲菊花瓣解剖结构及赤霉素处理对花色的影响研究[D].长沙:湖南农业大学,2010.
[21] GANDAHERRERO F,GARCACARMONA F.Biosynthesis of betalains:Yellow and violet plant pigments[J].Trends Plant Sci,2013,18(6):334-343.
[22] GANDAHERRERO F,GARCACARMONA F,ESCRIBANO J.Botany:Floral fluorescence effect[J].Nature,2005,437(7057):334.
[23] FELKER P,STINTZING F C,MSSIG E,et al.Colour inheritance in cactus pear(Opuntia ficusindica)fruits[J].Ann Appl Biol,2008,152(3):307-318.
[24] VEITCH N C,GRAYER R J.Flavonoids and their glycosides including anthocyanins[J].Nat Prod Rep,2008,25(3):555-611.
[25] TANAKA Y,BRUGLIERA F,CHANDLER S.Recent progress of flower colour modification by biotechnology[J].Int J Mol Sci,2009,10(12):5350-5369.
[26] 李巨秀,劉邻渭,王海滨.食品化学[M].2版.郑州:郑州大学出版社,2017.
[27] KAZUMA K,NODA N,SUZUKI M.Flavonoid composition related to petal color in different lines of Clitoria ternatea[J].Phytochemistry,2003,64(6):1133-1139.
[28] 孙卫,李崇晖,王亮生,等.花青苷成分对瓜叶菊花色的影响[J].园艺学报,2009,36(12):1775-1782.
[29] ZHENG Y J,TIAN L,LIU H T,et al.Sugars induce anthocyanin accumulation and flavanone 3hydroxylase expression in grape berries[J].Plant Growth Regul,2009,58(3):251-260.
[30] ZHU M L,ZHENG X C,SHU Q Y,et al.Relationship between the composition of flavonoids and flower colors variation in tropical water lily(Nymphaea)cultivars[J].PLoS One,2012,7(4):1-9.
[31] CHEYNIER V,COMTE G,DAVIES K M,et al.Plant phenolics:Recent advances on their biosynthesis,genetics,and ecophysiology[J].Plant Physiol Bioch,2013,72:1-20.
[32] SCHRDER J.A family of plantspecific polyketide synthases:Facts and predictions[J].Trends Plant Sci,1997,2(10):373-378.
[33] MORGRET M L,HUANG G H,HUANG J K.DNA sequence analysis of three clones containing chalcone synthase gene of petunia hybrida[J].FASEB J,2005,19:303.
[34] HAN Y Y,MING F,WANG J W,et al.Cloning and characterization of a novel chalcone synthase gene from Phalaenopsis hybrida orchid flowers [J].Russ J Plant Physiol,2006,53(2):223-230.
[35] BEERHUES L,ROBENEK H,WIERMANN R.Chalcone synthases from spinach(Spinacia oleracea L.)[J].Planta,1988,173(4):544-553.
[36] SUN W,MENG X Y,LIANG L J,et al.Molecular and biochemical analysis of chalcone synthase from Freesia hybrid in flavonoid biosynthetic pathway[J].PLoS One,2015,10:1-18.
[37] TAI D Q,TIAN J,ZHANG J,et al.A Malus crabapple chalcone synthase gene,McCHS,regulates red petal color and flavonoid biosynthesis[J].PLoS One,2014,9(10):1-15.
[38] CHMIEL E,STFELD R,WIERMANN R.Conversion of phloroglucinyltype chalcones by purified chalcone isomerase from Tulip anthers and from Cosmos petals[J].Biochem Physiol Pflanz,1983,178(2/3):139-146.
[39] NISHIHARA M,NAKATSUKA T,YAMAMURA S.Flavonoid components and flower color change in transgenic tobacco plants by suppression of chalcone isomerase gene[J].FEBS Lett,2005,579(27):6074-6078.
[40] OWENS D K,CROSBY K C,RUNAC J,et al.Biochemical and genetic characterization of Arabidopsis flavanone 3βhydroxylase[J].Plant Physiol Biochem,2008,46(10):833-843.
[41] HU K,MENG L,HAN K T,et al.Isolation and expression analysis of key genes involved in anthocyanin biosynthesis of cineraria[J].Acta Hortic Sin,2009,36:1013-1022.
[42] JIN Z P,GROTEWOLD E,QU W Q,et al.Cloning and characterization of a flavanone 3hydroxylase gene from Saussurea medusa[J].DNA Seq,2005,16(2):121-129.
[43] PETIT P,GRANIER T,DESTAINTOT B L,et al.Crystal structure of grape dihydroflavonol 4reductase,a key enzyme in flavonoid biosynthesis[J].J Mol Biol,2007,368(5):1345-1357.
[44] NAKATSUKA A,IZUMI Y,YAMAGISHI M.Spatial and temporal expression of chalcone synthase and dihydroflavonol 4reductase genes in the Asiatic hybrid lily[J].Plant Sci,2003,166(4):759-767.
[45] NAKATSUKA T,NISHIHARA M,MISHIBA K,et al.Temporal expression of flavonoid biosynthesisrelated genes regulates flower pigmentation in gentian plants[J].Plant Sci,2005,168:1309-1318.
[46] LI H H,QIU J,CHEN F D,et al.Molecular characterization and expression analysis of dihydroflavonol 4reductase(DFR)gene in Saussurea medusa[J].Mol Biol Rep,2012,39(3):2991-2999.
[47] HELLER W,FORKMANN G,BRITSCH L,et al.Enzymatic reduction of(+)dihydroflavonols to flavan3,4cisdiols with flower extracts from Matthiola incana and its role in anthocyanin biosynthesis[J].Planta,1985,165(2):284-287.
[48] WELLMANN F,GRIESSER M,SCHWAB W,et al.Anthocyanidin synthase from Gerbera hybrida catalyzes the conversion of(+)catechin to cyanidin and a novel procyanidin[J].FEBS Lett,2006,580(6):1642-1648.
[49] ROSATI C,CADIC A,DURON M,et al.Molecular characterization of the anthocyanidin synthase gene in Forsythia × intermedia reveals organspecific expression during flower development[J].Plant Sci,1999,149(1):73-79.
[50] SHIMIZU K,OHNISHI N,MORIKAWA N,et al.A 94bp deletion of anthocyanidin synthase gene in acyanic flower lines of lisianthus [Eustoma grandiflorum(Raf.)Shinn.] [J].Journal of the Japanese society for horticultural science,2011,80(4):434-442.
[51] XIE D Y,SHARMA S B,WRIGHT E,et al.Metabolic engineering of proanthocyanidins through coexpression of anthocyanidin reductase and the PAP1 MYB transcription factor[J].Plant J,2006,45(6):895-907.
[52] RAMSAY N A,GLOVER B J.MYBbHLHWD40 protein complex and the evolution of cellular diversity[J].Trends Plant Sci,2005,10(2):63-70.
[53] LAI B,LI X J,HU B,et al.LcMYB1 is a key determinant of differential anthocyanin accumulation among genotypes,tissues,developmental phases and ABA and light stimuli in Litchi chinensis[J].PLoS One,2014,9(1):1-12.
[54] JIN W M,WANG H,LI M F,et al.The R2R3 MYB transcription factor PavMYB10.1 involves in anthocyanin biosynthesis and determines fruit skin colour in sweet cherry(Prunus avium L.)[J].Plant biotechnology journal,2016,14(11):2120-2133.
[55] DAVIES K M,ALBERT N W,SCHWINN K E.From landing lights to mimicry:The molecular regulation of flower colouration and mechanisms for pigmentation patterning[J].Functional plant biology,2012,39(8):619-638.
[56] ALBERT N W,DAVIES K M,LEWIS D H,et al.A conserved network of transcriptional activators and repressors regulates anthocyanin pigmentation in eudicots[J].The plant cell,2014,26(3):962-980.
[57] PREZDAZ J R,PREZDAZ J,MADRIDESPINOZA J,et al.New member of the R2R3MYB transcription factors family in grapevine suppresses the anthocyanin accumulation in the flowers of transgenic tobacco[J].Plant molecular biology,2016,90(1/2):63-76.
[58] LAITINEN R A E,AINASOJA M,BROHOLM S K,et al.Identification of target genes for a MYBtype anthocyanin regulator in Gerbera hybrida[J].J Exp Bot,2008,59(13):3691-3703.
[59] NAKATSUKA T,HARUTA K S,PITAKSUTHEEPONG C,et al.Identification and characterization of R2R3MYB and bHLH transcription factors regulating anthocyanin biosynthesis in gentian flowers[J].Plant Cell Physiol,2008,49(12):1818-1829.
[60] YAMAGISHI M,SHIMOYAMADA Y,NAKATSUKA T,et al.Two R2R3MYB genes homologs of petunia AN2 regulate anthocyanin biosyntheses in flower tepals tepal spots and leaves of Asiatic hybrid lily[J].Plant Cell Physiol,2010,51(3):463-474.
[61] TAMAGNONE L,MERIDA A,PARR A,et al.The AmMYB308 and AmMYB330 transcription factors from Antirrhinum regulate phenylpropanoid and lignin biosynthesis in transgenic tobacco[J].The plant cell,1998,10(2):135-154.
[62] AHARONI A,DE VOS C H R,WEIN M,et al.The strawberry FaMYB1 transcription factor suppresses anthocyanin and flavonol accumulation in transgenic tobacco[J].The plant journal,2001,28(3):319-332.
[63] SALVATIERRA A,PIMENTEL P,MOYALEN M A,et al.Increased accumulation of anthocyanins in Fragaria chiloensis fruits by transient suppression of FcMYB1 gene[J].Phytochemistry,2013,90:25-36.
[64] TOMINAGAWADA R,NUKUMIZU Y,WADA T.Tomato(Solanum lycopersicum)homologs of TRIPTYCHON(SlTRY)and GLABRA3(SlGL3)are involved in anthocyanin accumulation[J].Plant signaling & behavior,2013,8(7):1-3.
[65] CAO X,QIU Z K,WANG X T,et al.A putative R3 MYB repressor is the candidate gene underlying atroviolacium,a locus for anthocyanin pigmentation in tomato fruit[J].Journal of experimental botany,2017,68(21/22):5745-5758.
