基于蛋白质互作网络挖掘自闭症谱系障碍的功能模块与核心基因
2020-07-15许逸聪胡婉雪赵洪波
许逸聪,胡婉雪,谢 芹,赵洪波*,唐 欣
(昆明医科大学a.分子临床医学研究院暨云南省干细胞和再生医学重点实验室;b.康复学院,中国云南昆明650500)
自闭症谱系障碍(autism spectrum disorder,ASD)是一类常见的神经发育疾病,诊断标准包括持续的社会交流与互动缺失、语言障碍、刻板重复行为及狭隘的兴趣。除上述症状外,患者可能同时伴随有癫痫、睡眠障碍、抑郁、焦虑以及胃肠道活动紊乱等症状,表现出明显的异质性和多样性[1],这些症状在儿童早期出现并严重影响患儿的日常生活[2]。作为一种具有高遗传性和生物复杂性的神经行为障碍类疾病,ASD是由遗传、环境等复杂因素相互作用形成的,其中遗传变异导致的发病率相对较高[3]。2016年发表的一项基于双胞胎研究的荟萃分析指出,64%~91%的ASD风险是由遗传因素引起的[4]。
尽管目前已发现上百种风险基因与ASD相关,表明ASD具有明显的遗传异质性,但遗传背景有差异的ASD个体往往表现出相似的行为特征[5]。越来越多研究也表明,与ASD密切相关的基因可能汇聚于一些共同的生物学过程[6~7],在ASD发生过程中起关键作用,但尚未有研究系统筛选这些功能模块和核心基因。因此,本研究利用生物信息学方法,通过疾病基因数据库中报道的ASD相关基因信息,整合蛋白质互作(protein-protein interaction,PPI)网络先验知识,挖掘ASD的功能模块和核心基因,并对每个模块进行通路富集分析,为ASD的遗传研究提供参考。
1 材料与方法
1.1 基因集数据获取
本研究从3个疾病数据库收集ASD相关基因。AutismKB 2.0(http://db.cbi.pku.edu.cn/autismkb_v2/index.php)是ASD遗传证据的知识库[8],当前版本包含ASD相关的1 379个基因(99个综合征自闭症相关基因和1 280个非综合征自闭症相关基因)、5 420个拷贝数变异(copy number variation,CNV)/结构变异(structural variation,SV)、11 669 个单核苷酸变异(single nucleotide variation,SNV)/插入和缺失(insertions and deletions,InDels)以及与ASD相关的172个连锁区域。SFARI Gene数据库(https://gene.sfari.org/)收录了1 089个ASD基因、2 290个CNV基因座和2 296个动物模型[9]。Dis-GeNET是一个综合性疾病基因数据库[10],整合了来自专家库、全基因组关联分析(genome-wide association study,GWAS)、动物模型和相关学术文献的数据,通过“Autism Spectrum Disorder”进行检索,得到571个相关基因。为获得稳健的基因集,筛选出在3个数据库中都出现的基因用于后续分析。
1.2 蛋白质互作网络构建
交互基因检索工具(Search Tool for the Retrieval of Interacting Genes,STRING)是一种用于评价蛋白质相互作用的工具[11]。将筛选出的ASD基因集导入STRING在线工具(http://string-db.org),选择物种为Homo sapiens,设置置信度阈值大于0.4,得到基因集表达的蛋白质互作网络(PPI network)。通过Cytoscape 3.7.2软件进行可视化和连通度分析,将筛选出的连通度大于25的节点作为PPI网络的重要基因。
1.3 功能模块及核心基因识别
利用MCODE(Molecular Complex Detection)算法对PPI网络中的节点进行密度聚类[12]。首先排除连通度小于3的节点,计算纳入节点的核聚类系数(core-clustering coefficient)并将其作为节点的权重,之后以当前节点权重百分率(vertex weight percentage,VWP)为阈值(本研究设VWP为0.2),筛选阈值范围内的相关节点构建模块,并将各模块中所包含的基因作为核心基因。上述过程通过Cytoscape软件中的MCODE插件进行,参数设置:degree cutoff≥3,K-score≥3,其余选择默认值。
1.4 通路富集分析
采用R软件包clusterProfiler[13]对得到的各功能模块进行KEGG(kyoto encyclopedia of genes and genomes)通路富集分析,通过超几何分布计算各通路富集结果的显著性,并使用Bonferroni方法进行多重假设检验校正,以错误发现率(false discovery rate,FDR)小于0.05为标准,对富集结果进行筛选,得到显著富集的KEGG通路。将富集到的KEGG通路输入KEGG数据库,识别其类别,进而评估功能模块之间的相互作用,利用Cytoscape 3.7.2软件构建功能模块与通路分类的关系。
2 结果
2.1 ASD相关基因筛选结果
为获得稳健的基因集,本研究共纳入了AutismKB数据库中的1 379个基因、SFARI Gene数据库中的1 089个基因以及DisGeNET数据库中的571个基因。通过取交集,最终筛选出共有的182个基因作为ASD相关基因集用于后续分析(图 1)。
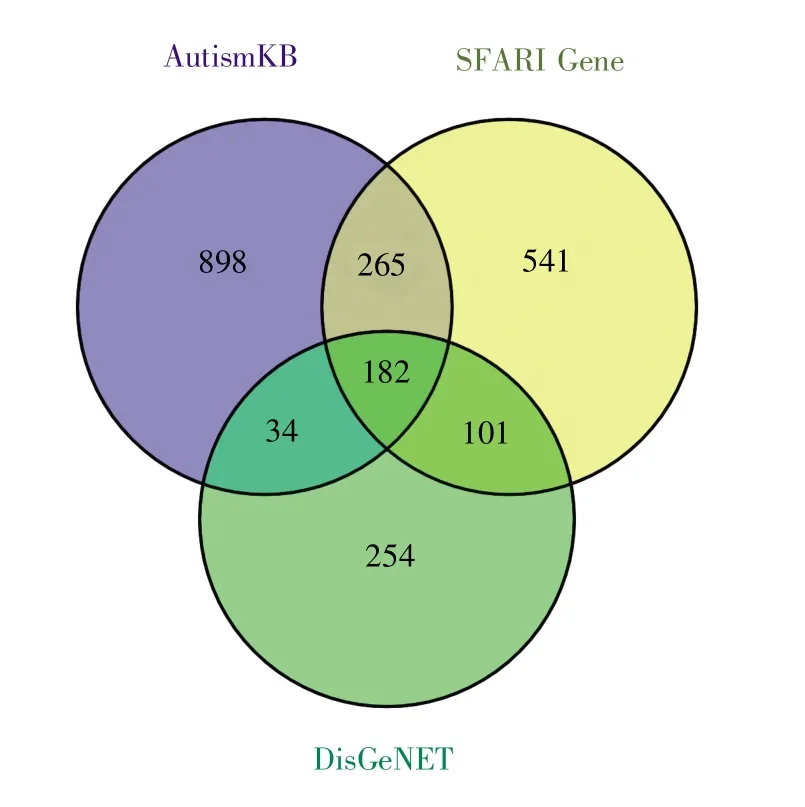
图1 基于3个数据库获得ASD共同基因的Venn图Fig.1 Venn diagram of common ASD genes from three databases
2.2 ASD基因集的蛋白质互作网络
将182个ASD相关基因导入STRING数据库,进行PPI网络分析。结果显示,整个网络包含171个节点和1 041条边,其中每个节点代表基因对应的蛋白质,每条边代表两个蛋白质之间的互作关系(图2)。根据PPI网络中每个节点的连通度,筛选得到PPI网络中核心蛋白质的编码基因23 个,分别为 NRXN1、GRIN2B、GRIN2A、DLG4、NLGN3、MECP2、CNTNAP2、BDNF、NLGN4X、FMR1、SHANK2、RELN、CHD8、SNAP25、CACNA1C、GAD1、PTEN、NRXN3、GABRB3、SYN1、SHANK1、SCN2A和UBE3A。
2.3 ASD疾病网络中的功能模块与核心基因
通过MCODE算法对PPI网络进行分析,最终获得5个功能模块,包含68个核心基因(表1和图3)。节点颜色越深表明该节点在其模块网络中的连通度越高,即其所代表的编码基因在模块网络中的重要性越高。评分最高的模块1(M1)包含20个基因及146条边,平均每个节点含有7.3条边,说明该模块中的基因之间关系比较密切,其中排名靠前的基因有 GRIN2B、GRIN2A、DLG4、MECP2、NLGN3、CNTNAP2、NLGN4X、FMR1、SHANK2等。模块2(M2)由20个基因和54条边组成,平均每个节点含有2.7条边,其中BDNF处于模块核心位置。模块3(M3)有16个核心基因,模块4(M4)含有的核心基因最少,仅4个。
2.4 各功能模块的通路富集分析
对筛选出的各个模块进行KEGG通路富集分析,结果显示:模块M1显著富集于谷氨酸能突触、细胞黏附分子和物质依赖通路;模块M2主要富集于突触囊泡循环、多巴胺能神经突触、神经活性的配体-受体相互作用等通路;模块M3富集的通路有促性腺激素分泌、昼夜节律、MAPK信号通路、钙离子通路等;模块M4主要富集在免疫相关通路;模块M5涉及到钙离子通路和神经活性的配体-受体相互作用(表2)。分析模块之间的共同通路发现,M1和M2都包括可卡因成瘾通路,M1和M4都包括细胞黏附分子通路,M2和M5都涉及神经活性的配体-受体相互作用通路,M3和M5都包括钙离子信号通路。
为了展示各模块之间的关系,根据各模块富集到的KEGG通路类别评估5个功能模块之间的相互作用,构建模块和通路分类的关系图(图4)。图中的功能类是KEGG通路的分类,包括信号分子和相互作用(signaling molecules and interaction)、神经系统(nervous system)、物质依赖(substance dependence)、内分泌代谢病(endocrine and metabolic disease)、信号转导(signal transduction)、转运和降解(transport and catabolism)、环境适应(environmental adaptation)、免疫系统(immune system)、免疫疾病(immune disease)、细胞生长和死亡(cell growth and death)、内分泌系统(endocrine system)、传染病(infectious disease)、循环系统(circulatory system)、抗药性(drug resistance)、心血管疾病(cardiovascular disease)和肿瘤(cancer)。其中信号分子和相互作用(M1、M2、M4 和 M5)与神经系统(M1、M2 和M3)这两个通路分类的连通度最高。

图2 ASD相关基因的PPI网络图每个节点代表各基因对应的蛋白质,连接各节点的边代表蛋白质之间的相互作用。节点连边越多表示其连通度越高,在网络中的位置越趋于中心,且颜色也越深。Fig.2 The PPI network of ASD related genesEach dot represents a protein corresponding to each gene,and the edge represents the interaction between the proteins.The center nodes with deeper color represent higher degree.

表1 各模块的基本信息Table 1 Basic information of each module
3 讨论
ASD是一种复杂的遗传性神经行为障碍类疾病,目前已有一些ASD相关的分子机制的研究[14~15],但各独立研究之间缺乏一个明确的共识。ASD的高异质性则表明,其发生发展涉及多种生物学过程的相互作用,而这些互作关系所涉及的具体机制尚不明确。本研究结合ASD疾病基因数据库与蛋白质互作网络先验知识,构建了ASD风险基因网络,通过网络分解,挖掘出紧密联系的功能模块和核心基因,进而分析了各模块的功能。为得到稳健的候选基因集,我们从3个数据库获取基因交集,增加了结果的可信度。分析结果显示筛选得到68个核心基因,并提取了ASD相关的5个功能模块。
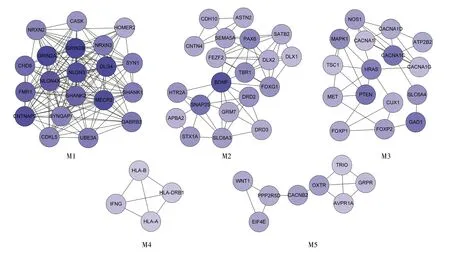
图3 各功能模块网络图各节点代表功能模块的核心基因,颜色越深表示该基因的连通度越大。Fig.3 Network of each functional moduleThe nodes represent the hub genes in functional module,and the deeper colors represent genes with higher degree.
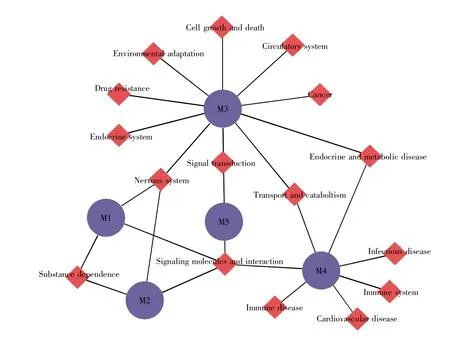
图4 功能模块与通路分类关系图蓝色表示模块,红色表示通路分类。Fig.4 Diagram of functional modules and pathway classificationBlue indicates the module and red indicates the pathway classification.
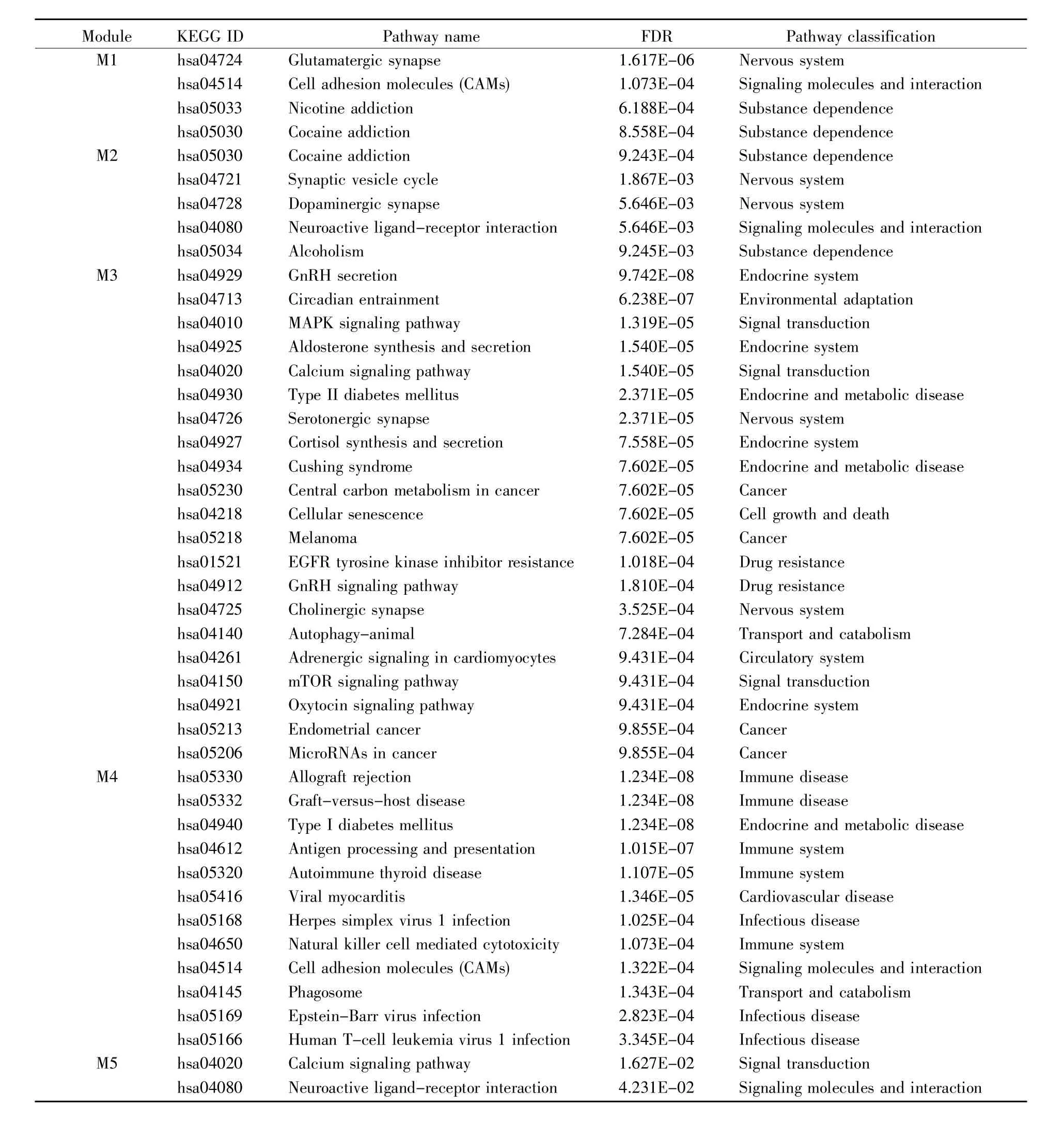
表2 各功能模块的KEGG通路富集分析结果Table 2 The results of KEGG pathway enrichment analysis in each functional module
为阐明模块的生物学功能,本研究对每个模块中的核心基因进行了基于KEGG信号通路的富集分析。评分最高的模块M1主要与谷氨酸能神经元突触形成及可塑性等过程相关。其中,NLGN3、NRXN3等基因编码的神经连接蛋白(neuroligins,NLGNs)家族和神经轴突蛋白(neurexins,NRXNs)家族均为单次跨膜Ⅰ型蛋白家族。在谷氨酸能或γ氨基丁酸(γ-aminobutyric acid,GABA)能神经元中,NLGNs蛋白家族位于突触后膜,而NRXNs蛋白家族则位于突触前膜。两者可以相互结合并与SHANK3蛋白绑定形成复合物,在突触的形成以及突触间的信息传递等过程中起到核心调控作用[16]。GRIN2A及GRIN2B基因分别编码N-甲基-D-天冬氨酸(N-methyl-D-aspartate,NMDA)受体的2a及2b亚基,其在神经发育早期阶段有着丰富的表达,并在发育后期的表达量逐渐降低,表明其主要参与神经系统的早期发育过程;此外,GRIN2B也在神经元的增殖迁移、轴突树突形成、学习记忆及突触可塑性等多种神经活动相关过程中起调控作用[17]。DLG4基因编码PSD-95蛋白,该蛋白质是突触后区域中一个特征明确的支架蛋白质,可与各种其他功能蛋白质相结合[18]。在突触后神经元中,SHANK蛋白与PSD-95构成复合物,并与NMDA受体交联,参与谷氨酸能神经活动调节的突触可塑性[19]。FMR1基因编码脆性X智力迟钝蛋白 (fragile X mental retardation protein,FMRP),FMRP定位于神经元的胞体和树突,通过与mRNA结合调节蛋白质合成,主要起转录抑制因子的作用。而SHANK蛋白家族则作为FMRP的下游靶标被调控[20]。CASK基因编码钙/钙调蛋白依赖性丝氨酸蛋白激酶,在突触前神经元中,通过与NRXNs结合构成复合物,从而连接细胞骨架,参与突触可塑性的相关调节[21]。通过文献挖掘和模块网络图可以发现,M1中各核心基因之间关系紧密,任何部分的改变均可能导致谷氨酸能神经元突触形成及可塑性过程的异常。比如:通过编辑小鼠的NLGNs蛋白家族相关基因发现,无论是这些基因的缺失还是过度表达,都可能导致小鼠出现ASD样的行为表现和中枢兴奋性与抑制性网络环路的异常[22];GRIN2B区域内的多个单核苷酸多态性 (single nucleotide polymorphism,SNP)与ASD的发病密切相关,不同的突变表型会影响Mg2+对Ca2+渗透性阳离子通道的电压依赖性阻滞作用,从而导致异常的神经电活动[23~24];CASK作为发育障碍相关的基因之一,其突变也在ASD患者中被发现[25]。此外,小鼠Fmr1的缺失导致功能性皮质内神经元连接的发育缺陷[26],兴奋与抑制的失衡[27],躯体感觉皮层神经元网络活动的同步性增加[28],以及视觉皮层中神经元电活动依赖的可塑性异常[29]。
模块M2中包括的核心基因则与神经递质的囊泡转运等过程相关。APBA2编码的蛋白质又称MINT2,是神经元衔接蛋白家族成员之一,其通过与CASK蛋白结合,进而与NRXNs家族蛋白交联,并构成一种转运蛋白复合体[30]。该转运蛋白复合体通过与CASK相连接的细胞骨架将Munc18-1等突触囊泡相关蛋白质募集到NRXNs家族蛋白周围,而Munc18-1是一种sec1样蛋白,参与胞吐作用等神经递质释放相关的过程[31]。SNAP25与STX1A编码的蛋白质共同参与构成可溶性N-乙基马来酰亚胺敏感因子附着蛋白受体(soluble N-ethylmaleimide-sensitive factor attachment protein receptor,SNARE)复合物[32],这种蛋白质复合物在Munc18-1的拉链作用促进下黏附谷氨酸或GABA递质囊泡,并通过囊泡的依赖性膜融合开始突触囊泡循环过程[33]。模块M3的核心基因主要涉及突触后膜电活性调节等相关过程。其中NOS1编码神经元型一氧化氮合酶(neuronal nitric oxide synthase,nNOS),nNOS通过PSD-95与NMDA受体交联,调节NMDA受体相关下游过程的激活,包括CACNA1C、CACNA1D等编码的电压依赖性钙通道等,从而对突触后区域的活性及可塑性进行调节[34]。
通过对功能模块内核心基因的分析,我们发现NRXNs家族、PSD-95、CASK等以功能蛋白质复合物的形式参与ASD相关生物学过程。这些核心基因的异常将会影响模块的功能,进而导致疾病的发生发展。根据富集到的KEGG通路类别,我们得到了更为深入的功能联系,说明了模块与模块之间不是孤立的,而是相互作用,共同参与ASD演化的核心病理机制。
综上所述,本研究通过数据库挖掘及蛋白质互作网络分析,筛选出了ASD相关的5个功能模块,得到68个核心基因,它们汇聚于一些共同的生物学过程,如突触传递、细胞间相互作用及细胞内信号传递的分子通路等。这些模块和基因异常均会导致整体网络的失衡,使得神经发育中的某些过程发生延迟或中断,从而导致感知觉与学习记忆能力异常、昼夜节律障碍等ASD相关症状的发生。本研究对ASD基因信息的挖掘和整合,有助于进一步地了解ASD的分子机制,可为ASD的基础研究提供新的参考。
