基于高通量测序技术的藏系绵羊瘤胃与粪便微生物群落结构差异分析
2020-06-04王循刚徐田伟刘宏金胡林勇张晓玲耿远月徐世晓
王循刚,徐田伟,刘宏金,胡林勇,张晓玲,耿远月,赵 娜,徐世晓
(1.中国科学院西北高原生物研究所,西宁 810001;2.中国科学院大学,北京 100049)
反刍动物的消化道尤其是瘤胃中栖息着丰富的微生物,包括纤维素降解菌、淀粉降解菌等细菌,产甲烷菌等古菌,以及大量的原虫和真菌等[1-3]。这些微生物通过相互作用,共同保持瘤胃内环境的相对稳定,在宿主对营养物质的消化吸收、能量代谢和生理健康等方面起重要作用[4-6]。作为反刍动物消化道中重要的发酵器官,瘤胃在大量微生物的协同作用下,可以高效率地对宿主所摄入的营养物质进行降解,产生大量能源物质对机体的生命活动进行供能[7-8]。研究报道,随着宿主的种类、年龄、饲粮组成、饲养方式等的改变,瘤胃中微生物的组成和丰度也会发生相应变化[9-12]。除了瘤胃,反刍动物的肠道微生物群落同样值得关注,尤其是粪便微生物,往往认为与宿主的营养和健康密切相关[13]。许强等[14]通过限制性片段长度多态性技术(Restricted fragment length polymorphisms,RFLP)分别分析了腹泻和健康状态下荷斯坦犊牛直肠粪便中的微生物群落,发现直肠形成了独特且复杂多样的微生物群落环境,且腹泻时其中的肠球菌属、乳杆菌属等显著增加。Li等[15]通过对不同程度腹泻林麝的粪便微生物进行检测,发现与健康状态下的微生物区系存在显著差异。现有研究认为,对粪便样品化学成分的测定,有助于了解宿主对所摄入营养物质的表观消化情况[16-17],而对其中微生物群落结构的分析,则可以了解宿主的生理健康状况。藏系绵羊(Ovisaries)作为青藏高原地区特有的绵羊品种,长期生活在海拔3 000 m以上,对青藏高原严酷的高寒环境具有很强的适应性,是藏区牧民重要的生产和生活资料[18]。瘤胃和直肠粪便这两大微生物区系特征以及两者之间的群落结构差异研究对于了解藏系绵羊独特的高原适应性具有重要意义。目前,关于藏系绵羊消化道微生物的研究仅围绕瘤胃开展[19-20],而对直肠粪便微生物的研究鲜有报道。因此,本研究以16S rRNA基因的V3-V4高变区作为分子标记,利用IlluminaHiSeq 2500测序平台分别对藏系绵羊瘤胃和直肠粪便的微生物群落进行高通量测序,探究藏系绵羊瘤胃和直肠粪便微生物菌群的结构和组成,并对两者之间的群落结构差异进行分析,旨在为藏系绵羊的健康养殖、疫病防疫及相关营养学研究提供一定的数据积累。
1 材料与方法
1.1 试验动物
2016年4月至7月在青海省海北高原现代生态畜牧业科技示范园放牧样地对藏系绵羊进行天然放牧,牧草中含粗蛋白质11.45%、粗脂肪 1.53%、中性洗涤纤维50.43%、酸性洗涤纤维 31.57%,地上生物量为103.71 g/m2。于7月挑选5只平均月龄为6月龄,平均体质量为 (21.50±0.59)kg的天然放牧藏系公绵羊进行样品 采集。
1.2 样品的采集与处理
分别采集每只绵羊的瘤胃液和粪便样品,每组样品5个重复。采集瘤胃液时,打开羊的口腔,使用瘤胃插管法将瘤胃管从羊口腔缓慢插入,抽取瘤胃内容物约50 mL,然后用4层纱布过滤,收集滤液;粪便采集时,于直肠处采集新鲜粪便约 5 g,装入冻存管,编号后立即投入液氮中保存。所有样品带回实验室,于-80 ℃保存,待测。
1.3 DNA提取及高通量测序
取200 mg瘤胃液或粪便样品,使用TIANGEN DNA提取试剂盒提取样品的总DNA,具体操作步骤参照试剂盒说明书进行。以样品提取的总DNA为模板对细菌16S rRNA的V3~V4区域进行PCR扩增,扩增引物为:338F(5′-ACTCCTACGGGAGGCAGCA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)[21],反应体系为25 μL:Q5 high-fidelity DNA polymerase 0.25 μL,5×Reaction Buffer 5 μL,5×High GC Buffer 5 μL,dNTP(1×104μmol/L)0.5 μL,模板DNA 1 μL,正向引物(10 μmol/L)1 μL,反向引物(10 μmol/L)1 μL,H2O 11.25 μL。反应参数设置为:98 ℃预变性2 min;循环30次(98 ℃30 s; 50 ℃ 30 s;72 ℃1 min);72 ℃延伸5 min,4 ℃冷却,PCR扩增产物用20 g/L琼脂糖凝胶电泳检测,检测合格后使用凝胶回收试剂盒回收目的片段。通过IlluminaHiSeq 2500测序平台对DNA片段进行高通量测序,获得原始数据后再进行后续生物信息学分析。
1.4 微生物多样性的生物信息学分析
使用FLASH[22]和QIIME[23]对原始数据进行拼接过滤,得到优化序列,根据相似度为97%的原则对序列进行操作分类单元(Operational taxonomic unit,OTU)聚类,并挑选出相对丰度最高的序列作为每个OTU的代表性序列。利用RDP classifier (http://rdp.cme.msu.edu/)对每条代表性序列进行物种注释并构建OTU分类信息表,绘制在门水平和属水平上的物种分布柱状图。计算每个试验样本的α多样性和β多样性。α多样性由反映群落丰富度的ACE指数、Chao1指数及反映群落多样性的Simpson指数、Shannon指数进行评估。β多样性选择利用加权遗传距离矩阵(Weighted uniFracdistance)计算,并进行PCoA分析。使用PICRUSt[24]进行KEGG微生物基因功能预测分析。数据均以“平均值±标准差”的形式表示,组间差异利用SPSS 20.0软件进行独立样本t检验,P<0.05为显著性差异,P<0.01为极显著性差异。
2 结果与分析
2.1 各样品测序结果
经测序,藏系绵羊瘤胃液和粪便10个样品共获得729 326条原始序列(Raw tags),原始序列经过滤后获得优化序列(Clean tags)651 449条,平均每个样品含有(65 145±320)条,每条序列平均长度为(435.20±2.78)bp。样品的稀释曲线趋于平缓(图1-A),说明本试验测序所得数据量已经覆盖样本中的绝大多数物种。基于相似度大于97%的原则,将测序所得有效序列进行OTU聚类,共得到1 207个OTU(图1-B),其中瘤胃中独有的有479个,粪便中独有的有309个,两组间共享的OTU有419个。
2.2 微生物群落多样性分析
2.2.1 α多样性分析 为了研究藏系绵羊瘤胃和粪便微生物群落的丰富度和多样性,对两组的α多样性指数进行统计计算和差异显著性检验,结果如表1所示。瘤胃微生物的Chao 1指数和ACE指数显著高于粪便微生物(P<0.05);而Simpson指数和Shannon指数在两组之间差异均不显著(P>0.05)。

表1 瘤胃和粪便微生物群落α多样性分析Table 1 α diversity analysis between rumen and feces
2.2.2 β多样性分析 为了探究藏系绵羊瘤胃和粪便微生物群落组成的差异性,基于样本的OTU信息,计算样本之间的加权遗传距离矩阵(Weighted uniFrac distance),并进行PCoA分析,结果如图2所示。瘤胃微生物群落聚为一类,粪便微生物群落聚为一类,且两组样品在第一轴均明显分开,表明样品组内相似程度要高于组间,且粪便样品组内的相似程度要高于瘤胃样品。

图2 基于加权遗传距离矩阵计算的PCoA分析Fig.2 PCoA analysis based on matrix calculation by weighted genetic distance
2.3 不同分类水平微生物群落结构差异分析
在不同分类水平上对测序所得有效序列进行物种注释,共得到14个门,21个纲,27个目,44个科,107个属。根据物种注释信息,分别选择样品微生物在门分类水平和属分类水平上相对丰度排名前10位的物种进行柱状图比较(图3),结果发现藏系绵羊瘤胃和粪便微生物在门分类水平上组成相似,其中拟杆菌门和厚壁菌门是优势菌门;在属水平上,细菌、理研菌科_RC9_gut_group和瘤胃球菌科_ UCG_010等是优势菌属。
2.3.1 门水平微生物群落结构差异 在门分类水平上对两组微生物群落中相对丰度大于1%的物种进行统计(表2),其中瘤胃微生物群落中相对丰度大于1%的门有5个,分别为拟杆菌门、厚壁菌门、互养菌门、变形菌门和软壁菌门,占门水平总丰度的98.74%;粪便微生物群落中相对丰度大于1%的门有8个,分别为厚壁菌门、拟杆菌门、疣微菌门、黏胶球形菌门、螺旋体门、变形菌门、蓝藻菌门和软壁菌门,占门水平总丰度的 98.81%。其中,瘤胃中厚壁菌门、疣微菌门、黏胶球形菌门、螺旋体门和蓝藻菌门5个门的相对丰度极显著低于粪便(P<0.01);而拟杆菌门和互养菌门显著高于粪便(P<0.05)。

图3 门水平(A)和属水平(B)上菌群组成(相对丰度前10)Fig.3 Composition of microbial community at phylum(A)and genus level(B)(top 10 of relative abundances)
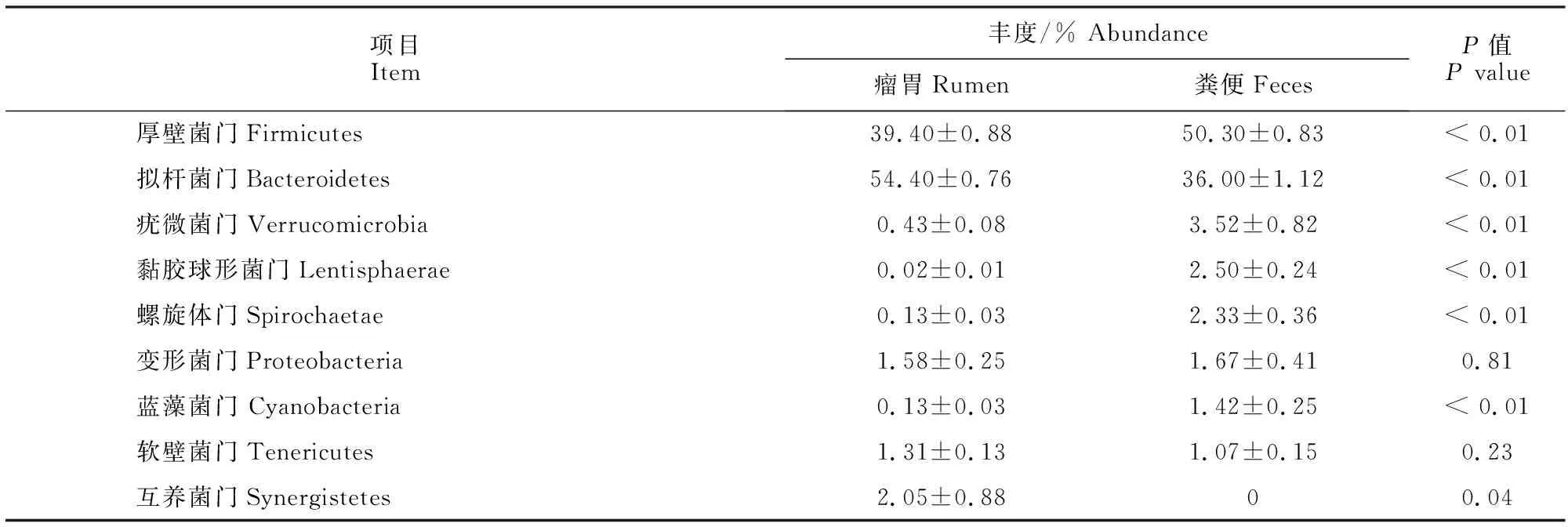
表2 微生物相对丰度在门分类水平上>1%的物种Table 2 Relative abundance >1% of microbial species at phylum level
2.3.2 属水平微生物群落结构差异 在属分类水平上对两组微生物群落中相对丰度大于2%的物种进行统计(表3),结果发现瘤胃中相对丰度大于2%的属有9个,相对丰度最高的为细菌,其次为普雷沃氏菌属_1、瘤胃菌属和理研菌科_RC9_gut_group;粪便中相对丰度大于2%的属有12个,其中丰度最高的为瘤胃球菌科_UCG_010,其次为细菌、理研菌科_RC9_gut_group和瘤胃球菌科_UCG_005。其中,瘤胃中瘤胃球菌科_UCG_010、瘤胃球菌科_UCG_005和拟杆菌属等9个属的相对丰度极显著低于粪便(P<0.01);而理研菌科_RC9_gut_group、克里斯滕森菌科_R-7_group和普雷沃氏菌属_1等6个属显著高于粪便(P<0.05)。
2.4 微生物基因功能预测分析
利用PICRUSt软件对瘤胃和粪便微生物群落进行功能预测分析。共匹配到43个KEGG二级代谢通路,其中有显著性差异的代谢通路有34个(P<0.05)。选取相对丰度大于1%的代谢通路共18个进行热图比较(图4),丰度总和分别占总丰度的94.15%和94.16%。代谢通路主要富集在代谢、遗传信息处理、环境信息处理和细胞过程四大类。对18个代谢通路的相对丰度进行两组间比较,发现仅碳水化合物代谢在两组间差异不显著(P>0.05);全局和概述地图、氨基酸代谢、膜转运、脂代谢、信号转导、折叠分拣和降解、萜类化合物和多酮类化合物的代谢和外源物质的生物降解与代谢,这8个代谢通路在粪便微生物中丰度较高(P<0.05);能量代谢、辅因子和维生素的代谢、核苷酸代谢、转录、复制与修复、糖生物合成与代谢、其他氨基酸代谢及次生代谢产物的生物合成和细胞运动,这9个代谢通路在瘤胃微生物中丰度较高(P<0.05)。

表3 微生物相对丰度在属分类水平上>2%的物种Table 3 Relative abundance >2% of microbial species at genus level

横坐标为组名,纵坐标为功能基因的相对丰度。越偏蓝色表示相对丰度越高,越偏黄色表示相对丰度越低。ns表示相对丰度值在两组之间差异不显著(P>0.05);*表示差异显著(P<0.05);**表示差异极显著(P<0.01);***表示差异极显著(P<0.001)
Abscissa is group name and ordinate is relative abundance of functional genes. The closer to blue the color is, the higher relative abundanceis , while the closer to yellow the color is,the lower relative abundance is.ns=No significant difference(P>0.05); * Significant difference(P<0.05); ** Significant difference(P<0.01); *** Significant difference(P<0.001)
图4 功能基因预测热图
Fig.4 Heat map of predicted function of microbiota
3 讨 论
3.1 瘤胃和粪便的微生物群落多样性
微生物群落的多样性往往由α多样性和β多样性进行评估。α多样性反映了样品中物种的多样性和丰富度,具体评价指标包括Chao1、ACE、Shannon、Simpson指数等。其中Chao1和ACE指数用于衡量物种丰富度即物种数量的多少;Shannon和Simpson指数用于衡量物种多样性。Shannon指数越大,Simpson指数越小,说明样品的物种多样性越高[25]。本研究为了解藏系绵羊瘤胃和粪便微生物的多样性水平,对Chao 1、ACE、Simpson和Shannon指数进行计算评估。结果发现,藏系绵羊瘤胃微生物的Chao 1指数和ACE指数显著高于粪便微生物,说明瘤胃中的微生物群落丰富度较高,高丰富度的微生物群落可以使宿主更高效地对所摄入食物进行分解代谢;而Simpson指数和Shannon指数在两组之间无显著差异,说明两组间菌群多样性水平相似。王继文等[26]研究表明,波尔山羊粪便的微生物多样性要略高于瘤胃,这与本研究结果相悖,分析产生差异的原因可能是家畜品种和饲养方式不同所造成的,有研究表明在不同精粗比或不同营养水平日粮饲喂下,瘤胃微生物和粪便微生物多样性水平都会发生显著改变[27-28]。微生物群落的β多样性主要是用以评估样品间物种多样性差异,本研究中利用加权遗传距离矩阵进行PCoA分析,结果发现藏系绵羊瘤胃微生物菌群能够很清晰地和粪便菌群区分开,说明这两者的菌群分别具有较高的稳定性且在微生物结构上具有较大差异。
3.2 瘤胃和粪便微生物在不同分类水平上的群落结构差异
Liu等[13]和张雪娇等[29]研究均表明反刍动物瘤胃微生物中相对丰度较高的优势菌门依次为拟杆菌门和厚壁菌门,这与本研究结果相同,即瘤胃中拟杆菌门丰度(54.40%±0.80%)显著高于厚壁菌门(39.40%±0.90%),但在粪便中却呈现相反的趋势,丰度最高的为厚壁菌门(50.30%±0.80%),其次才为拟杆菌门(36.00%±1.10%),这也与以往的研究结果相一致[30]。这种相似性的出现显示反刍动物胃肠道微生物菌群的共性特征。进一步探究发现,在属分类水平上藏系绵羊瘤胃液中丰度较高的菌群为细菌(18.20%± 1.51%)、普雷沃氏菌属1(12.10%±1.43%)和瘤胃菌属(12.00%±1.10%)。其中普雷沃氏菌属1在分类学上隶属于拟杆菌门,在瘤胃内可以降解淀粉、蛋白质、木聚糖等多类营养物质,用以能量供给和调节机体免疫等[31],多项研究均证明普雷沃氏菌属为反刍动物瘤胃内的优势菌群[32-33]。而在粪便中瘤胃球菌科UCG_010 (17.10%± 1.65%)、细菌(16.30%±0.52%)和理研菌科RC9_gut_group(8.63%±0.54%)丰度排名位于前三位。瘤胃球菌属在分类学上隶属于厚壁菌门,Wang等[34]对小尾寒羊胃肠道不同区段微生物的动态变化进行比较分析,结果发现在大肠中瘤胃球菌属的相对丰度要显著高于其他胃肠道部位。这与反刍动物后肠所行使的生理功能相关,后肠中的瘤胃球菌属能够产生淀粉酶和纤维素酶等碳水化合物降解酶,用以破坏植物细胞壁,从而实现对碳水化合物的分解发酵[35]。但从本研究结果来看,在属水平仍存在许多未成功注释到的基因序列,以及相对应的基因功能,这就对总体结果的分析产生了一定影响,后续可以加入宏转录组等新兴微生物学分析技术[36],以实现对反刍动物胃肠道微生态环境的更准确认知。
3.3 瘤胃和粪便微生物功能预测
使用PICRUSt软件对藏系绵羊瘤胃和粪便微生物群落进行功能预测分析,发现基因功能主要富集在代谢、遗传信息处理、环境信息处理和细胞过程四大功能类群上。其中碳水化合物代谢在两组中均富集程度最高且差异不显著;瘤胃微生物在能量代谢、核苷酸代谢、糖代谢和其他氨基酸代谢等机体代谢功能上显著富集;粪便微生物在氨基酸代谢、膜转运、脂代谢、信号转导等功能上显著富集,这与已报道的奶牛胃肠道微生物功能研究结果相类似[37],说明瘤胃微生物和后肠中的微生物均参与营养物质的消化代谢。
4 结 论
本研究结果表明,藏系绵羊瘤胃中微生物的多样性高于粪便中,瘤胃微生物和粪便微生物在结构组成上相对稳定且存在差异。
