新疆驴盲肠、腹结肠、背结肠固相食糜细菌多样性研究
2019-08-20方雷陈根元刘利林周小玲
方雷 陈根元 刘利林 周小玲
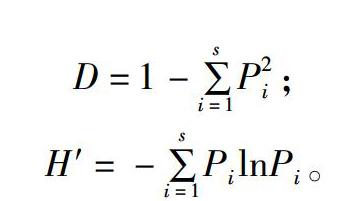

摘要:运用PCR-DGGE技术研究新疆驴盲肠、腹结肠、背结肠中固相食糜细菌组成多样性。选用3头成年、健康新疆驴,自由采食90%稻草+10%精料混合日粮,舍饲30 d后屠宰、分段取样备用。经过微生物基因组提取、目标片段扩增、变性梯度凝胶电泳、图像分析和数据处理,结果表明,不同肠道固相食糜细菌条带组成上有相似区域,也有各自的特异条带,而同一肠道段的个体样本间的细菌条带具有较高相似性,且盲肠固相食糜细菌的Shannon-Wiener指数、Simpson指数最大,与腹结肠差异不显著,但都分别显著高于背结肠。新疆驴盲肠固相食糜细菌多样性最为丰富,腹结肠次之,背结肠相对最弱。
关键词:新疆驴;盲肠;腹结肠;背结肠;固相食糜;细菌多样性
中图分类号: S822.1文献标志码: A
文章编号:1002-1302(2019)08-0176-03
驴属于单胃草食动物,与猪禽等单胃动物不同,具有发达的盲肠和结肠,这是驴重要的消化器官。肠道内长期寄居着种类丰富、数量庞大的微生物,彼此间协同作用发酵、降解消化道食糜,产生挥发性脂肪酸,为宿主提供养分。有关研究结果表明,驴对稻草、麦秸、玉米秸秆日粮中的中性洗涤纤维(NDF)消化率为40%~50%,与牛羊等反刍动物对这3种秸秆日粮的NDF消化能力相当,说明驴的盲肠、结肠的肠道微生物对秸秆粗饲料有较强的降解能力[1],其功能类似于反刍动物的瘤胃。反刍动物的瘤胃微生物主要黏附在饲料颗粒上、存在于瘤胃液中和附着于瘤胃内壁上[2],且瘤胃固相食糜中的微生物数量高于液相,是瘤胃中的主要功能微生物[3-5]。瘤胃微生物的多样性受饲料组成[6]、宿主遗传背景[7]和环境等因素影响[8];反刍动物的瘤胃、网胃、瓣胃和皱胃4个胃室中菌群多样性也不一样,呈现先高后低再升高的趋势[9]。驴的盲肠、腹结肠、背结肠是日粮中纤维素、半纤维素等结构性碳水化合物降解的重要场所,不同消化部位的固相食糜细菌多样性如何变化是本研究的切入点。
1材料与方法
1.1试验动物、时间及地点
试验于2017年6月5日至7月5日在塔里木大学动物科学学院试验养殖基地进行。试验选用3头成年、健康、3~4岁新疆驴,舍饲喂以含有90%粉碎稻草和10%精料(70%玉米粉+30%棉粕)的混合日粮,自由采食、饮水,饲养30 d。
1.2样品处理
饲养结束后,屠宰前1 d晚上禁食、供水,屠宰前先用钝器打击头部致晕,立即颈动脉放血、开膛、暴露内脏;找到“逗号”状盲肠,随后向后继续找到膨大腹结肠、膨大背结肠,用绳子扎紧各段间狭小接口,水平放置,从各肠道段中部取样;分别采集盲肠、腹结肠、背结肠食糜,并用4层灭菌纱布过滤,将过滤后的固相食糜迅速收集于数个2 mL灭菌指形管中,立即投入液氮罐于实验室,分类保存于-80 ℃冰箱备用。
1.3食糜微生物总DNA提取
参照Kang等[10]和Laure等[11]的方法洗脱固相食糜上附着的微生物,用天根生物技术有限公司微生物基因组DNA提取试剂盒(DP328)提取微生物基因组总DNA,具体步骤按说明书操作。
1.4目标片段扩增及检测
试验采用细菌16S rDNA的V3区通用引物对目标片段进行扩增,上游引物HDA1-GC序列为:5′-[JP9]CGCCCGGGGCGCGCCCCGGGCGGGGCGGGGGCACGGGGGGACTCCTACGGGAGGCAGCAGT-3′,下游引物HDA2序列为:5′-[JP9]GTATTACCGCGGCTGCTGGCAC-3′。以上引物由上海生工生物技術有限公司合成,可扩增长约200 bp的目标片段。
PCR反应条件如下:95 ℃预变性5 min;95 ℃变性30 s,56 ℃退火30 s,72 ℃延伸50 s,共30个循环。PCR产物由 1.0% 琼脂糖凝胶电泳检测。
1.5变性梯度凝胶电泳
试验中DGGE采用10%聚丙烯酰胺凝胶,丙烯酰胺和甲叉双丙烯酰胺比例为37.5 ∶1,变性剂浓度梯度为45%~60%。电泳采用Dcode DGGE系统(Bio-Rad,美国),在 0.5×TAE缓冲液中、60 ℃恒温下进行,200 V预电泳10 min,85 V电泳16 h。电泳结束后,用0.5 μg/mL的EB溶液染色30 min,用凝胶成像系统(Gel DOCTM XR+,Bio-Rad美国)检测、拍照。
1.6图谱处理与数据分析
用Quantity One 4.62软件对DGGE图谱进行分析,将各泳道条带光密度值计算、输出用于计算Simpson指数和Shannon-Wiener指数[12-13],采用SAS 8.0软件对细菌多样性指数进行Duncans检验和统计分析,P<0.05表示差异显著,P<0.01表示差异极显著。用NTSYSpc 2.10S对图谱条带聚类分析。
D=1-∑si=1P2i;
H′=-∑si=1PilnPi。
式中:Pi为某样品中某1条带光密度值占该样品中总光密度值的比率,D为Simpson指数,H′为Shannon-Wiener指数。
2结果与分析
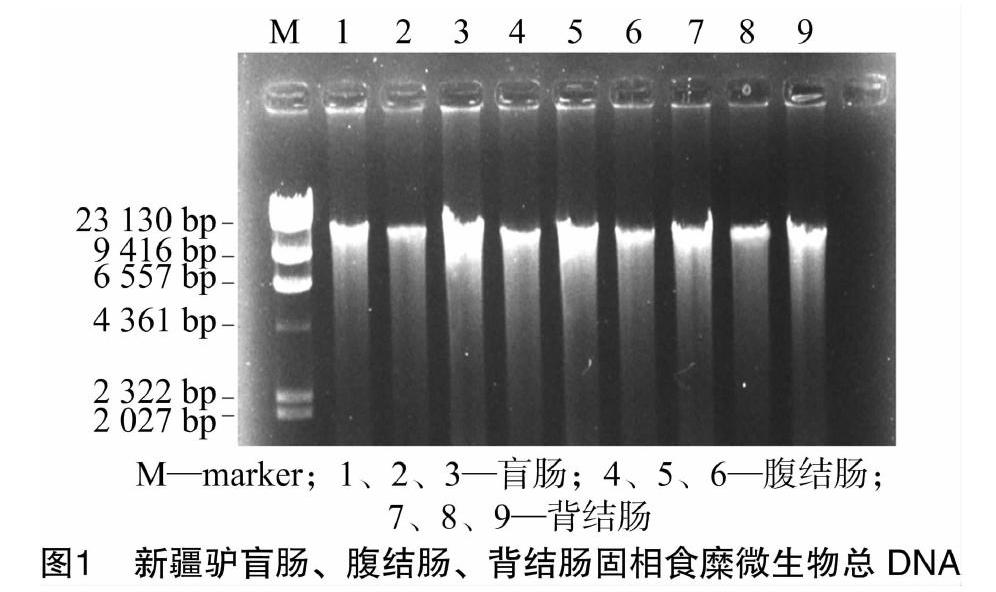
2.1新疆驴盲肠、腹结肠、背结肠固相食糜微生物总DNA
新疆驴不同段肠道固相食糜微生物总DNA提取结果见图1。由图1可见,盲肠、腹结肠、背结肠各代表样品分别在 1~9 泳道均有明亮条带,与marker相比,条带大小在2.3 ku左右,有稍许拖尾,比较完整,D260 nm/D280 nm比值为1.71,可以用作后续PCR模板。
2.2新疆驴盲肠、腹结肠、背结肠固相食糜细菌16S rDNA V3区PCR扩增产物
以新疆驴盲肠、腹结肠、背结肠固相食糜微生物总DNA为模板,细菌16S rDNA V3区通用引物进行PCR扩增,经 1.0% 琼脂糖凝胶电泳、染色、拍照,结果表明,不同肠道固相食糜各样本均获得整齐、明亮的目标PCR产物,大小为 200 bp(图2),符合引物扩增预期产物结果,可以用于后续变性梯度凝胶电泳。
2.3新疆驴盲肠、腹结肠、背结肠固相食糜细菌16S rDNA V3区PCR-DGGE指纹图谱
新疆驴盲肠、腹结肠、背结肠固相食糜细菌V3区PCR扩增产物经45%~60%变性梯度凝胶电泳、染色、照相,结果表明,各肠道段各样本在各自泳道均出现丰富、明亮、清晰条带,同一肠道段样本间有大量相似条带,不同肠道间既有部分相似条带,也有所属肠道段的特异条带(图3)。
2.4新疆驴盲肠、腹结肠、背结肠固相食糜细菌DGGE指纹图谱聚类分析
新疆驴盲肠、腹结肠、背结肠固相食糜细菌PCR-DGGE图谱聚类分析结果见图4。各段肠道固相食糜细菌条带都以高于0.93的相似性分别聚合成盲肠(1、2、3)、腹结肠(4、5、6)和背结肠(7、8、9)三大类群,腹结肠和背结肠又以0.67左右的相似性聚合成大结肠类群,最后再以0.56相似性和盲肠细菌条带聚合成一大簇。
2.5新疆驴盲肠、腹结肠、背结肠固相食糜细菌多样性指数分析
新疆驴盲肠、腹结肠、背结肠固相食糜细菌PCR-DGGE凝胶图片经Quantity One软件对各泳道条带光密度值输出、多样性指数计算和SPSS统计分析后,结果(表1)显示,新疆驴盲肠固相食糜细菌的Shannon-Wiener指数和Simpson指数均为最大,分别为3.209和0.947,与腹结肠差异不显著,但均分别极显著高于背结肠(P<0.01)。
3讨论与结论
反刍动物瘤胃微生物的存在方式,主要以黏附固相饲料颗粒、游离于瘤胃液中和附着于瘤胃内壁上,其中固相食糜黏附微生物占瘤胃微生物总数的50%~70%[14],以大量纤维降解菌为主,纤维素降解也是瘤胃代谢的限速步骤[15]。通过表1 细菌多样性指数来看,盲肠固相食糜的Shannon-Wiener和Simpson指数值都是最大,分别为3.209和0.957,与腹结肠的3.168和0.927相比差异不显著,但都分别极显著高于背结肠的2.950和0.915(P<0.01)。也就是说,盲肠固相食糜上附着的细菌种类更加丰富,腹结肠次之,背结肠相对最弱;瘤胃微生物在降解日粮过程中,主要包括附着和酶解2个过程,其中附着过程是瘤胃微生物降解日粮的前提[16-17],靠附着微生物间协同作用降解食糜;推测盲肠在对固相食糜颗粒降解的作用要强于腹结肠,强于背结肠;可能一方面盲肠是驴大肠微生物发酵降解食糜重要的起始段,胃、小肠等消化液消化后,食糜中不易被消化或逃脱的易消化养分首先来到盲肠进行微生物降解,食糜中的养分组成和含量有利于盲肠微生物的生长繁殖,使其食糜附着细菌多样性更好;另一方面,随着盲肠、腹结肠和背结肠微生物对食糜的逐级降解,食糜中易被利用和能被利用的养分也越来越少,也导致了固相食糜上附着的细菌多样性逐渐降低。
从新疆驴盲肠、腹结肠和背结肠固相食糜指纹图谱(图3)可以看出,在相同试验条件下,不同消化道段都获得了较丰富的细菌条带,有共同、特异和很亮的优势细菌条带,但试验中均未回收、克隆和测序,无法获知其分类信息;从聚类信息(圖4)上看,各不同肠道处理内的细菌条带组成都有0.9以上的相似系数,而驴的不同肠道处理间只有0.56的相似系数。本试验中,我们对新疆驴盲肠、腹结肠、背结肠固相食糜附着细菌多样性有了初步认识,这些不同消化道微生物差异表现在微生物什么分类水平上,是什么原因造成的仍需进一步研究和探讨。
新疆驴的相同肠道段固相食糜细菌组成具有较高的相似性,不同肠道段间细菌组成有所差异;盲肠固相食糜的细菌多样性最为丰富,腹结肠次之,背结肠相对最弱。
参考文献:
[1]方雷,矿理扬,牛志涛. 新疆驴对4种秸秆日粮采食与消化的研究[J]. 新疆农业大学学报,2009,32(3):45-48.
[2]Mcallister T A,Bae H D,Jones G A,et al. Microbial attachment and feed digestion in the rumen[J]. J Ainm Sci,1994,72:3004-3018.
[3]冯薇,王加启,刘开朗,等. 运用PCR-DGGE分析比较瘤胃中不同饲料固相黏附微生物区系[J]. 畜牧兽医学报,2010,41(12):1556-1562.
[4]Peter R M,Forster R J,Yang W,et al. Characterization of rumen bacterial diversity and fermentation parameters in concentrate fed cattle with and without forage[J]. Journal of Applied Microbiology,2012,112(6):1152-1162.
[5]Huws S A,Lee M R,Muetzel S M,et al. Forage type and fish oil cause shifts in rumen bacterial diversity[J]. FEMS Microbiology Ecology,2010,73(2):396-407.
[6]吴小燕,彭全辉,王之盛,等. 不同蛋白质饲料对宣汉黄牛瘤胃固相黏附纤维降解微生物数量的影响[J]. 动物营养学报,2014,26(11):3298-3306.
[7]Benson A K,Kelly S A,Legge R,et al. Individuality in gut microbiota com position is a complex polygenic trait shaped by multiple environmental and host genetic factors[J]. Proceedings of the National Academy of Sciences of the United States of America,2010,107(44):18933-18938.
[8]Uyeno Y,Sekiguchi Y,Tajima K,et al. An r-RNA-based analysis for evaluating the effect of heat stress on the rumen microbial com position of Holstein heifers[J]. Anaerobe,2010,16(1):27-33.
[9]曾燕,曾東,倪学勤,等. 应用PCR-DGGE技术比较绵羊瘤胃、网胃、瓣胃和皱胃菌群的多样性[J]. 动物营养学报,2015,27(1):298-304.
[10]Kang S H,Denman S E,Morrison M I. An efficient RNA extraction method for estimating gut microbial diversity by polymerase chain reaction[J]. Current Microbiology,2009,58(5):464-471.
[11]Larue R,Yu Z T,Parisi V A,et al. Novel microbial diversity adherent to plant biomass in the herbivore gastrointestinal tract,as revealed by ribosomal intergenic spacer analysis and rrs gene sequencing[J]. Environmental Microbiology,2005,7(4):530-543.
[12]崔振亮,孟庆翔,吴浩,等. PCR-DGGE技术研究青贮桑叶对肉牛瘤胃细菌区系的影响[J]. 饲料研究,2011(1):1-4.
[13]许晴,张放,许中旗,等. Simpson指数和Shannon-Wiener指数若干特征的分析及“稀释效应”[J]. 草业科学,2011,28(4):527-531.
[14]Yang W,Beauchemin K,Rode L. Effect of dietary factors on distribution and chemical composition of liquid-or solid-associated bacterial populations in the rumen of dairy cow[J]. Journal of Animal Science,2001,79(10):2736-2746.
[15]Koike S,Yoshitani S,Kobayashi Y,et al. Phylogenetic analysis of fiber-associated rumen bacterial community and PCR detection of uncultured bacterial[J]. FEEMS Microbiol Lett,2003,229(1):23-30.
[16]Michalet-Doreau B,Fernandez I,Fonty G. A comparison of enzymatic and molecular approaches to characterize the cellulolytic microbial ecosystems of the rumen and the cecum[J]. Journal of Animal Science,2002,80(3):790-796.
[17]Merry R,Mcallan A. A comparison of the chemical composition of mixed bacteria harvested from the liquid and solid fractions of rumen digest[J]. British Journal of Nutrition,1983,50(03):701-709.
