沙塘鳢线粒体基因组全序列的测定和分析
2016-10-21张燕萍郑红梅邵芳韩晓磊徐建荣顾志良
张燕萍,郑红梅,邵芳,韩晓磊,徐建荣,顾志良
(1.常熟理工学院生物与食品工程学院,江苏常熟215500;
2.南京医科大学附属常州第二人民医院肿瘤研究所,江苏常州213003)
沙塘鳢线粒体基因组全序列的测定和分析
张燕萍1,郑红梅1,邵芳2,韩晓磊1,徐建荣1,顾志良1
(1.常熟理工学院生物与食品工程学院,江苏常熟215500;
2.南京医科大学附属常州第二人民医院肿瘤研究所,江苏常州213003)
为揭示沙塘鳢(Odontobutis)线粒体基因组特征,采用PCR扩增和测序的方法获得沙塘鳢(Odontobutis)线粒体基因组全序列.通过生物信息学软件(tRNAscan-SE、RNAfold)定位沙塘鳢线粒体基因组基因结构及预测tRNA和rRNA的二级结构.沙塘鳢线粒体基因组全长16 930 bp,结构组成和其他硬骨鱼基本一致,包括13个蛋白质编码基因,22个tRNA,2个rRNA和1个D-loop区. 13个蛋白质编码基因中,除了CO1是以GTG作为起始密码子,ND6以TTA作为起始密码子以外,其余都以ATG起始.沙塘鳢线粒体基因的终止密码子有3种类型,分别为完全密码子TAA、不完全密码子TA和T.除了tRNASer(AGN)和tRNACys外,其余20个tRNA基因的二级结构都是典型的三叶草结构.识别了沙塘鳢终止序列区、中央保守区D、E、F和保守序列区CSB-1、CSB-2和CSB-3.比较沙塘鳢cytb基因序列与其他物种同源性发现,沙塘鳢与平头沙塘鳢的同源性最高(87.03%),河川沙塘鳢次之(83.57%),与人的同源性最低(70.64%).该研究结果可为深入探讨沙塘鳢属及塘鳢科鱼类的系统分类提供依据,更真实地反映了沙塘鳢的分类地位和进化关系.
沙塘鳢;线粒体DNA序列;D-Loop区;系统进化
鱼类是脊椎动物中最原始而在种属数量上又最占优势的类群,其起源复杂,分布广泛,拥有丰富的遗传多样性[1].在分子生物学技术应用于鱼类分类之前,人们主要从鱼类化石资源、形态结构和生理特征等方面探求鱼类的遗传进化历程,但由于多种客观条件限制,使得传统的分类学方法难以彻底解决鱼类遗传结构和遗传分化等问题.近年来,随着鱼类线粒体DNA(mitohondrial DNA,mtDNA)研究技术的不断发展成熟,使其成为鱼类学相关研究的重要标记,并在鱼类进化遗传学、种群遗传结构、分子生态学和保护生物学等方面取得了很多有意义的成果[2].mtDNA同其他脊椎动物一样,是细胞核外具自主复制、转录和翻译能力的共价闭合环状双链DNA.与核DNA相比,鱼类mtDNA具有分子较小、结构简单、进化速度快、遗传相对独立性和母系遗传等特点,是一个相对独立的复制单位[3-4].
目前已经报道了上百种鱼类线粒体DNA的全序列,发现鱼类mtDNA分子大小在15~19 kbp之间,主要包括37个编码基因13个蛋白基因、2个rRNA基因、22个tRNA编码基因)、1个负责复制和转录起始的控制区(D-loop)和1个轻链复制起始区[5].蛋白质和tRNA基因的进化速度适中,适合于从种间到种内遗传差异分析,作为线粒体上唯一的结构和功能被了解得较为清楚的蛋白质编码基因,Cytb基因容易用一些通用引物扩增和测序,因而Cytb基因被广泛应用于系统进化研究[6].
沙塘鳢属鲈形目(Perciformes)、虾虎鱼亚目(Gobioidei)塘鳢科(Eleotridae)、沙塘鳢属(Eleotris),是分布于我国长江中下游以及钱塘江、闽江水系的中小型溪流经济鱼类.沙塘鳢肉质鲜美,经济价值高,由于自然环境的改变,其数量急剧衰减.目前对沙塘鳢的养殖技术研究广泛,但其基因水平的研究甚少,基因库中线粒体基因序列公布很少.本实验采用PCR扩增和测序的方法,获得沙塘鳢线粒体DNA的全序列,确定了沙塘鳢线粒体基因组编码的所有基因,还分析了沙塘鳢线粒体基因组的碱基组成、基因排列和密码子使用情况.本研究扩充了鱼类线粒体基因组数据库,为其系统分类提供分子水平证据,同时为沙塘鳢群体遗传学和生物资源的合理利用等提供有价值的资料.
1 材料与方法
1.1实验材料和总DNA的提取
沙塘鳢由苏州市长江特色水产繁殖工程中心提供,活鱼宰杀后,采集肌肉组织放于-80℃冰箱保存备用.采用经典的酚-氯仿法提取沙塘鳢肌肉总DNA,用NanoDrop2000和琼脂糖凝胶电泳检测其浓度及完整性,于-20℃保存备用.
1.2引物设计与PCR扩增、测序
根据GenBank数据库中已公布塘鳢科线粒体部分序列以及其他相关物种的同源性保守序列设计引物.引物由上海生工生物工程有限公司合成(表1).PCR反应体系为10×Buffer 2.5 μL,dNTP 2.0 μL,DNA模板1.0 μL,上、下游引物各1.0 μL,Taq酶0.2 μL,然后加去离子水17.3 μL至终体积25 μL.PCR反应程序:94℃预变性5 min,94℃变性30s,60℃退火30s,72℃延伸90s,运行35个循环,最后72℃延伸10 min,4℃保存.另外,依据引物的长短对退火温度、延伸时间和循环次数做了相应的调整.将所有PCR产物均用1%的琼脂糖凝胶电泳检测,使用DNA凝胶回收试剂盒进行纯化.将PCR得到的回收产物进行直接测序,测序均由上海生工生物工程有限公司完成.

表1 PCR扩增引物序列
1.3序列拼接、注释和分析
使用DNAMAN软件对测序结果进行序列比对、拼接,再经测序峰图结果分析软件Chromas仔细校正后,得到沙塘鳢线粒体全序列.使用Gene Runner软件统计全序列的各碱基百分比,蛋白质编码基因的碱基使用情况.使用软件tRNAscan-SE(http://lowelab.ucsc.edu/tRNAscan-SE/)定位tRNA基因、蛋白质编码基因、rRNA基因和D-loop区,以确定沙塘鳢线粒体基因组的基因结构.用RNAfold软件(http://rna.tbi.univie.ac.at/cgi-bin/ RNAfold.cgi)预测rRNA基因的空间二级结构和自由能值.
1.4进化分析
将获得的沙塘鳢线粒体Cytb基因DNA序列与GenBank已经公布的中华乌塘鳢等17个物种线粒体全基因组的Cytb基因序列进行比对,利用MEGA软件中的邻接法(Neighbor-Joining,NJ)构建分子进化树.
2 结果与分析
2.1线粒体基因组结构
沙塘鳢线粒体基因组全长为16930 bp,共编码37个基因包括13个蛋白质编码基因,22个tRNA基因,2个rRNA基因及一个介于tRNAPro和 tRNAPhe之间的D-loop区,沙塘鳢线粒体基因组结构如图1所示.这37个编码基因在沙塘鳢线粒体基因组中的分布呈不均一性,L链上编码9个基因,包括8个tRNA基因(tRNAGln、tRNAAla、tRNAAsn、tRNACys、tRNATyr、tRNASer(UCN)、tRNAGlu、tRNAPro)和一个编码蛋白的基因ND6;其余的28个基因则由H链编码.
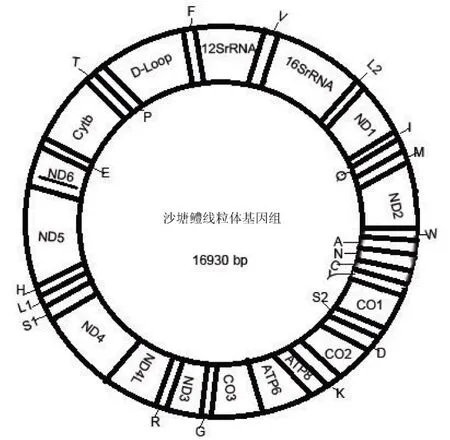
图1 沙塘鳢线粒体基因组结构组成
沙塘鳢线粒体基因组有15处基因间隔区域,长度范围在1~336 bp之间,共518 bp,最大的基因间隔出现在tRNALeu(CUN)和tRNAHis这两个基因之间.基因组中还存在5处重叠,长度范围在1~7 bp之间,共计20 bp,最大基因重叠出现在ATPase8和ATPase6及ND4L和ND4之间.既没重叠,也没有间隔的紧密排列基因对共计18处.沙塘鳢线粒体基因各个基因大小及分布见表2.
加下划线“_”的基因在L链上编码;tRNA基因用氨基酸单字母表示;L1、L2和S1、S2分别表示tRNALeu(CUN)、tRNALeu(UUR)和tRNASer(AGN)、tRNASer(UCN)
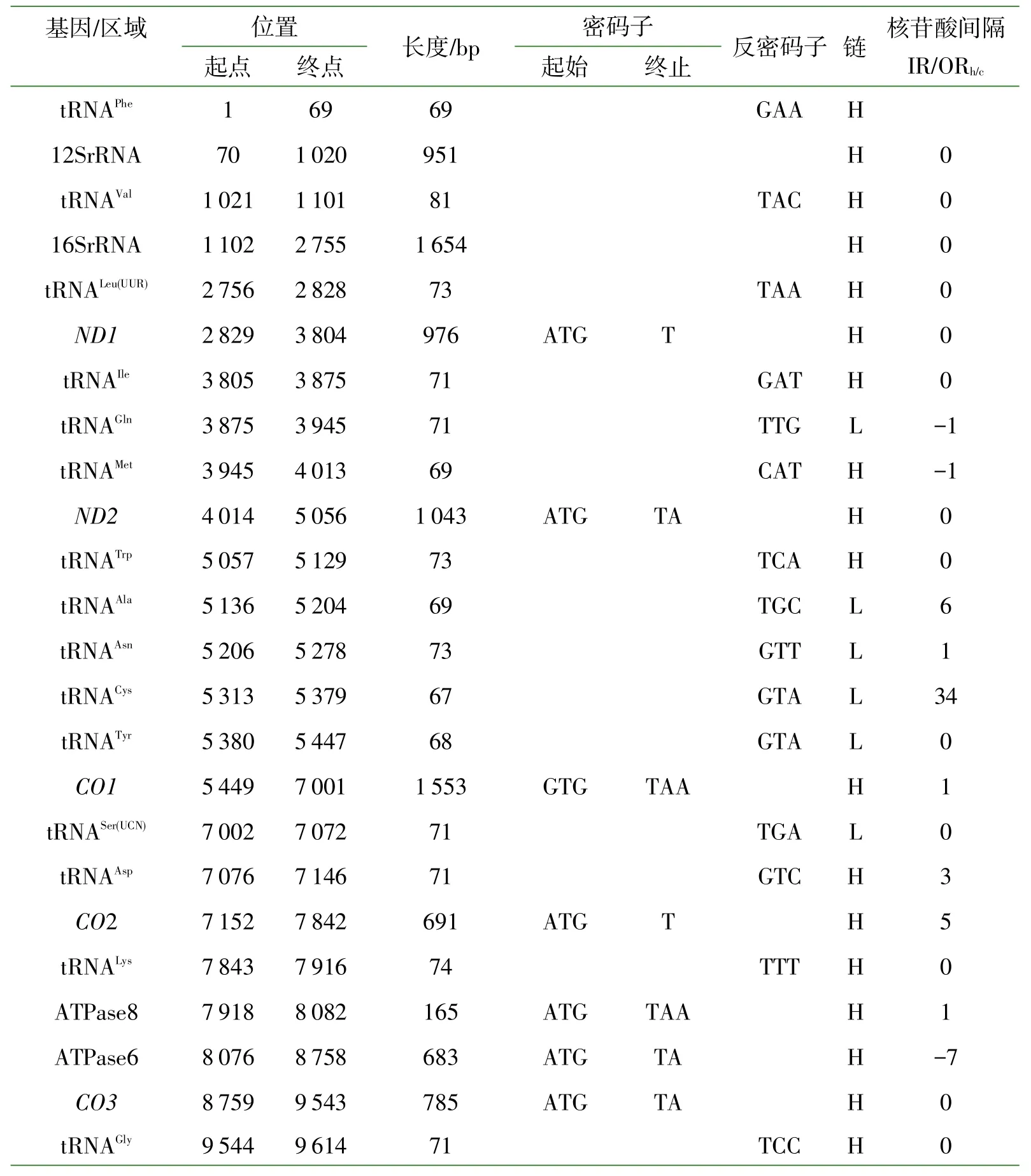
表2 沙塘鳢线粒体基因组的基因组成
2.2线粒体碱基组成
沙塘鳢线粒体基因组碱基百分含量见表3.蛋白编码基因4种碱基百分含量与整个线粒体基因组最为相似.在不同密码子碱基含量的问题上,密码子第一位点的4种碱基含量较为接近,没有碱基使用偏好;第二位点T的含量最高,为36.5%,G含量最低,为12.5%,显示出T偏倚和反G偏倚;第三位点反G偏倚最明显,平均含量仅为9.2%.
2.3蛋白编码基因及密码子使用
沙塘鳢的13个蛋白编码基因总长度为11 419 bp,占线粒体全长的67.4%.13个蛋白编码在碱基组成上都有明显的反G偏倚.所用的起始密码子除CO1基因为GTG,ND6基因为TTA外,其余均为ATG.常用的终止密码子有3种类型,包括完全密码子TAA和不完全密码子TA以及T,如:CO1、ATPase8、ND4L、ND5是以TAA为终止密码子;ND2、ATPase6、CO3是以TA为终止密码子,其余都是以T终止的.
2.4tRNA二级结构
沙塘鳢线粒体基因组有22个tRNA基因,全长1 565 bp,单个基因长度为67~81 bp,其中14个由L链编码,8个由H链编码.通过tRNAscan-SE软件预测并确定了沙塘鳢线粒体22个tRNA基因的位置及二级结构.除tRNASer(AGN)和tRNACys基因的结构比较特殊外,其余均能形成典型的三叶草结构.其中氨基酸臂最保守,长度都为7 bp,TψC臂和反密码子臂为4~5 bp,DHU臂为2~4 bp.TψC环为7 bp(除Lys为9 bp,Phe为8 bp),反密码子环为7 bp(除Thr、Val为9 bp),而DHU环的碱基数差别很大,在4~11 bp之间.沙塘鳢的20个tRNA基因中共有16对不匹配的碱基配对.其中位于氨基酸臂上的有10对,分别为tRNAAsn、tRNAAsp、tRNAHis、tRNAIle、tRNALeu(CUN)、tRNAPhe、tRNASer(UCN)、tRNAThr、tRNATrp、tRNAVal;4对在TψC臂上,分别为tRNAHis、tRNAMet(有2对)、tRNATrp;2对在反密码子臂上,分别为tRNAAla、tRNAVal.其中6个AC错配,3个UC错配,3个UU错配,2个AA错配,2个 CC错配.
2.5rRNA二级结构
沙塘鳢rRNA基因由12S rRNA和16S rRNA两种组成,分别位于tRNAPhe和tRNAVal、tRNAVal和tRNALeu(UUR)之间,其长度分别为951 bp和1 654 bp.rRNA基因的二级结构很保守,形成多个大小不一的茎环结构,其进化速度在线粒体各基因组分中最慢.通过RNAfold在线软件预测出了沙塘鳢两种rRNA基因的二级结构,其中,12S rRNA基因的自由能值为-234.90 kcal/ mol,16S rRNA基因的自由能值为-422.30 kcal/mol.一般而言,自由能值越低则代表其分子结构越稳定,表明沙塘鳢12S rRNA基因比16S rRNA基因更保守.
2.6D-loop控制区
沙塘鳢D-loop控制区长度为844 bp.A+ T含量(64.34%)显著高于基因组全序列的A+ T含量(55.5%).这与其他鱼类A、T含量高,G、C含量低的特点很一致.沙塘鳢线粒体D-loop区分为终止序列区(termination associatedsequence,TAS)、中央保守区(central conserved dormain,CD)和保守序列区(conservedsequence block,CSB)3个区段.以CSB-F和CSB-1的起点分别作为终止序列区,中央保守区和保守序列区的分界线见图2.
接表1
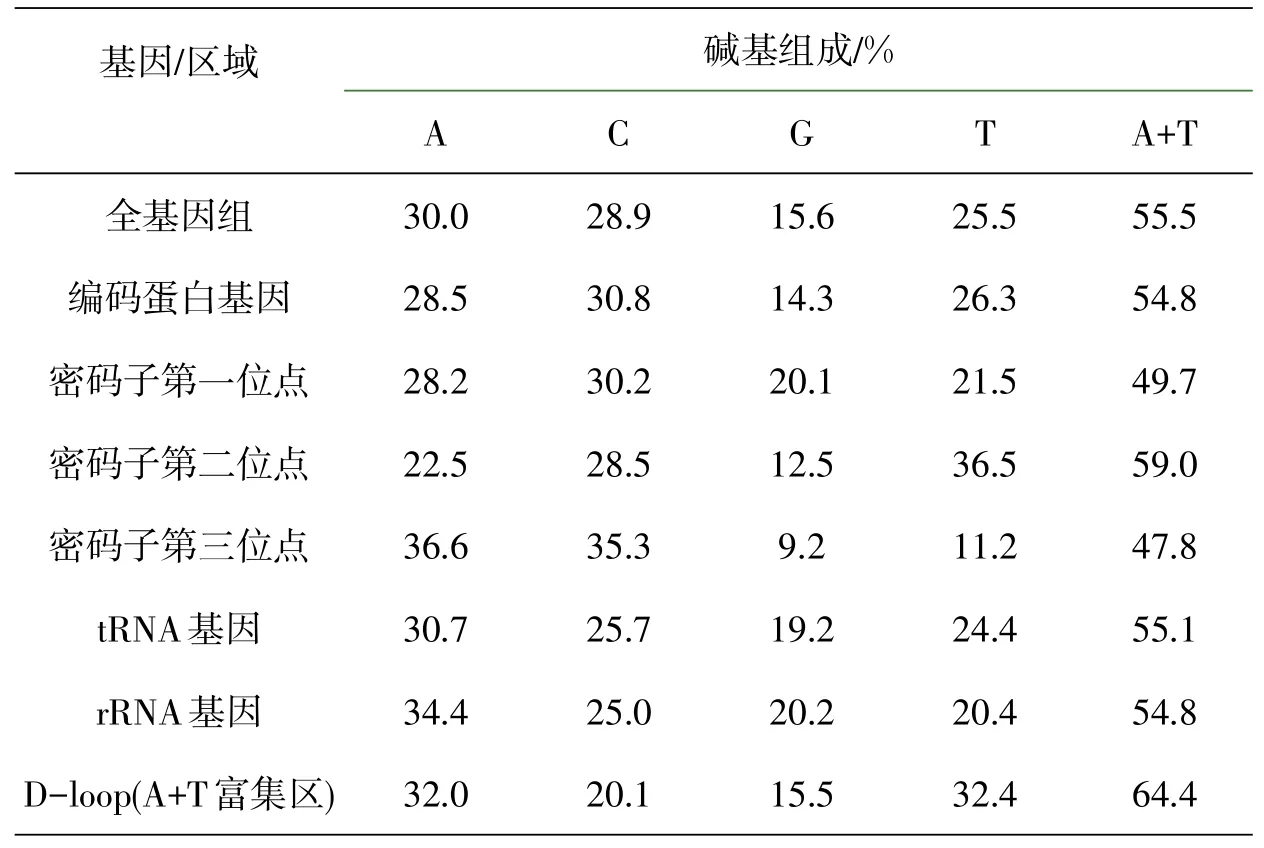
表3 沙塘鳢线粒体不同基因的碱基组成
终止区是控制区变异最大的区域,沙塘鳢的终止结合序列区长度为280 bp,在此区段中识别了2个终止相关序列(TAS),其中包括核心序列TACAT及其反向互补序列ATGTA,其二级结构可形成稳定的发夹结构,这一现象在许多类群中存在.经比对得到沙塘鳢的ETAS序列为:TACAT-TATGTATTA-CCATATAT-ATATAACCATA(“-”表示发生变异的碱基,即转换、颠换或缺失).
沙塘鳢中央保守区为355 bp,分为CSB-F、E、D.其中CSB-F是分开终止序列区和中央保守区的标志,关键形式为:ATGCAGATAAG-A-ACATTC,但存在变异.紧跟其后是CSB-E,关键序列为:AGGGACAA-TTGTGGGGGTTTCAC,含有GTGGG-box .CSB-D,序列为:GTGAACTATTCCTGGCATTTGGTTCCTATTTCAGGGCCATT-,CSB-D和CSB-E保守性都要高于CSB-F.
沙塘鳢的保守序列区总长为209 bp,包括CSB-1、CSB-2和CSB-3.研究表明CSB-1与mtDNA的复制起始相关,3段CSB序列都被确定,其中CSB-1的关键序列为:-TTGCATAACTGATATCAAGAGCATA-;CSB-2的关键序列为:AAACCCCCCTACCCCCCACTC;CSB-3的关键序列为:TCCTGAAACCCCCCGGAAACAGGAA.


图2 沙塘鳢线粒体基因组控制区序列与特征
中央保守序列(CSB-F,CSB-E,CSB-D)、保守序列(CSB-1,CSB-2,CSB-3)用下划线表示;终止相关序列(TAS)用阴影表示,它的反向互补序列(ATGTA)用双下划线表示.
2.7基于Cytb基因的系统进化分析
2.7.1沙塘鳢与其他物种的同源性
利用DNAMAN软件,将沙塘鳢Cytb基因序列分别与其他参照物种的Cytb基因进行比对,得到沙塘鳢与其他物种的同源性系数(见表4).可见沙塘鳢与平头沙塘鳢的同源性最高(87.03%),河川沙塘鳢次之(83.57%),与人的同源性最低(70.64%).
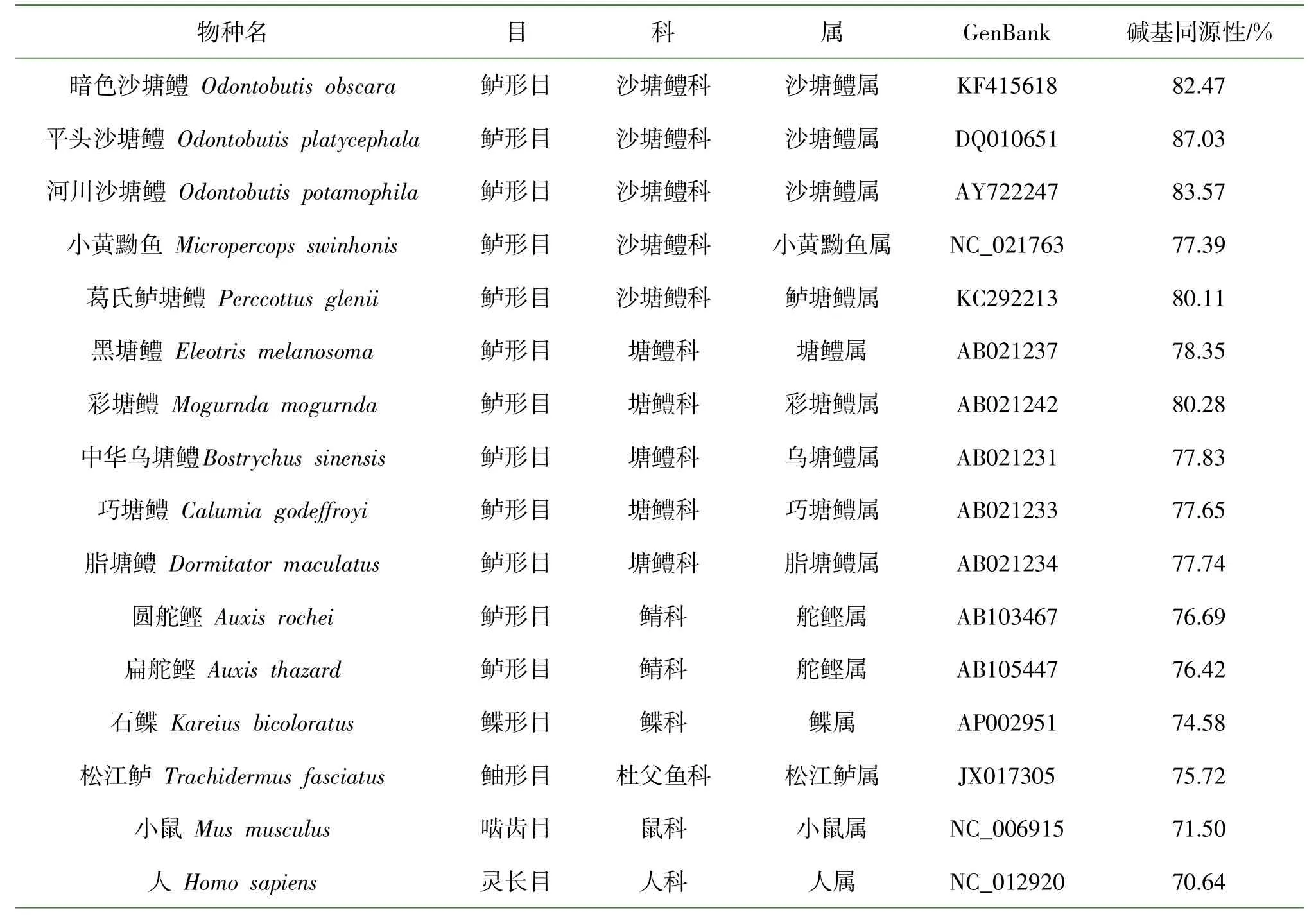
表4 沙塘鳢与对照物种的同源性系数
2.7.2基于Cytb基因序列的系统进化树
利用MEGA 4.1软件构建表4中17个物种的分子进化树(图3).结果表明,所有硬骨鱼形成一个大的分支,哺乳类形成另一个分支.硬骨鱼又分为4个独立的小分支,沙塘鳢首先与平头沙塘鳢聚成一支,接着与河川沙塘鳢、暗色沙塘鳢聚类,这4种鱼类都属于鲈形目沙塘鳢科沙塘鳢属,接着这4种沙塘鳢属鱼类与葛氏鲈塘鳢(鲈塘鳢属)聚类,再与小黄黝鱼(小黄黝鱼属)聚类,这6种沙塘鳢科鱼类形成独立的分支;黑塘鳢、彩塘鳢、中华乌塘鳢、巧塘鳢和脂塘鳢则单独形成另一个小的分支,这5种鱼类同属于鲈形目的塘鳢科;鲈形目鲭科鱼类形成另一个分支;而属鲽形目的石鲽和属鲉形目的松江鲈则形成其他分支.结果从另一个方面证实沙塘鳢在鱼类分类系统中的位置,此外还说明鲈形目不同科的鱼类亲缘关系有远近之分,沙塘鳢科鱼类与鲭科鱼类亲缘关系较远.这与动物形态学分类基本一致.

图3 基于沙塘鳢cytb基因序列构建的NJ法系统树
3 讨论与结论
3.1沙塘鳢线粒体基因组分析
本研究测定了沙塘鳢的线粒体基因组全序列,全长为16 930 bp,共编码37个基因.沙塘鳢线粒体基因组的基因排列顺序与已公布的其他鱼类线粒体基因组的基因排列顺序完全一致,各基因长度也大致相等[4,7-8],其mtDNA的分子大小在种间存在差异主要是由于串联重复序列的存在以及长度不等的碱基插入和缺失,如沙塘鳢在基因tRNATrp和tRNAAla之间间隔6 bp,岩原鲤(Procypris rabaudi)只有2 bp的重复[9].沙塘鳢线粒体基因组A、T、C、G这4种碱基的含量都非常接近,且A+T含量高于C+G含量,这与已报道的绝大多数鱼类线粒体基因组碱基含量分布一致;蛋白编码基因4种碱基百分含量与整个线粒体基因组最为相似[8,10-11].13个蛋白编码基因分3种类型终止密码子,除了CO1、ATPase8、ND4L、ND5是以完全密码子TAA为终止密码子外,其余都是以不完全终止密码子TA或T终止翻译.这种以不完整的密码子终止翻译的现象在后生动物的线粒体基因组中较为常见,这些不完整的终止密码子可以通过转录为mRNA后由3′端的多聚腺苷酸化作用将其补全变成完整的终止密码子(TAA)[12].
沙塘鳢的22种tRNA除了tRNACys以及tRNASer(AGN)DHU臂发生缺失外,其余20种tRNA均呈典型的三叶草结构.沙塘鳢12S rRNA基因比16S rRNA基因自由能值低,说明12S rRNA比16S rRNA更保守,这和大多数鱼类的研究结果一致.鱼类二级结构总体差异较大,没有明显规律,因此不利于对物种间的rRNA二级结构做详细比较[13].
线粒体DNA控制区又称D-环区(D-loop),位于tRNAPro和tRNAPhe基因之间,是整个线粒体DNA序列中长度变异最大的区域,也是线粒体基因组上进化最快的部分.鱼类线粒体D-loop区分为H-链复制起始区OH、TAS区、CD、CSB区、L-链启动子和H-链启动子.D-loop区是mtDNA中替换率最高和碱基数量最易发生变化的区域.目前识别了D-loop的中央保守区的保守序列B、C、D、E、F .Randi等发现保守序列C、D、E、F在鸟类中存在[14].Lee等对众多鱼类序列进行比较时,仅识别了CSB-D的存在[15],随后相继有人识别出了鱼类的CSB-E和CSB-F[16].本实验识别出了沙塘鳢控制区的终止序列区,这是控制区最易发生变异的区域,还识别了中央保守区序列CSB-F、CSB-E、CSB-D和保守区序列CSB-1、CSB-2、CSB-3并给出关键序列.然而金逍逍等人发现包括刺盖塘鳢、中华乌塘鳢在内的26种虾虎鱼存在不同的缺失现象,未能识别到CSB-F[16].多数鱼类识别到CBS-E的核心序列(GTGGG),CSB-D序列则相对较为保守,这可能与其参与线粒体代谢及重链复制有关联[15].
3.2沙塘鳢进化分析
沙塘鳢科鱼类隶属鲈形目虾虎鱼亚目,而虾虎鱼类是鲈形目鱼类中最大的一个类群.塘鳢科产于中国有5个属,其中包括沙塘鳢属(Odontobutis)和鲈塘鳢属(Perccottus)等,这两个属常见的经济种类有河川沙塘鳢(Odontobutis potamophila)、鸭绿沙塘鳢(Odontobutis yaluensis)、中华沙塘鳢(Odontobutissinensis)、葛氏鲈塘鳢(Perccottus glenii)等[17].伍汉霖等从形态学上将中国沙塘鳢属鱼类分为河川沙塘鳢、鸭绿沙塘鳢、中华沙塘鳢、海丰沙塘鳢(Odontobutis haifengensis)4个种[18].但由于虾虎鱼类形态上的多样性以及不同程度的退化与特化,导致单纯依据形态学对其鉴定显得较为困难,很多分类结果一直存在争议[19-20].
鉴于以上种种原因,线粒体基因作为分子标记应用到鱼类的物种鉴别、分类及系统发育等领域,相比于形态学研究,分子生物学分析方法能很好地克服鱼类形态特征多样性、特性特征不明显等困难,能更真实有效地反映其分类地位和进化关系.谢楠等[21]基于cytb基因序列分析了虾虎鱼亚目9个属的进化关系,塘鳢属与黄黝鱼属2个属构成1支;沙塘鳢属与鲈塘鳢属2个属构成1支;乌塘鳢属、嵴塘鳢属、尖塘鳢属、头孔塘鳢属4个属构成1支;鳍塘鳢属单独构成1支,未能在亚科水平上聚类.同样我们基于沙塘鳢的cytb基因序列构建了类似的系统进化树,沙塘鳢属4种鱼类聚类,接着与葛氏鲈塘鳢(鲈塘鳢属)聚类,再与小黄黝鱼(小黄黝鱼属)聚类;塘鳢科同样也发生聚类,这一结果能够在科及属水平上达到聚类.侯新远等[22]基于线粒体D-loop序列研究我国5种虾虎鱼类的进化关系发现,河川沙塘鳢、鸭绿沙塘鳢、中华沙塘鳢、平头沙塘鳢聚为一大支,刺盖塘鳢、黑体塘鳢、褐塘鳢、尖头塘鳢、溪鳢、葛氏鲈塘鳢聚为另一支,这与我们得出葛氏鲈塘鳢与沙塘鳢科鱼类聚类却未与塘鳢科鱼类聚类的结果不同,这说明选择不同的基因在系统发育研究中存在差异.该研究结果可为深入探讨沙塘鳢属及塘鳢科鱼类的系统分类提供依据.
[1]NELSON Js.Fishes of the World[M].3th ed.New York:Wiley,1994:402-412.
[2]袁娟,张其中,罗芬.鱼类线粒体DNA及其在分子群体遗传研究中的应用[J].生态科学,2008(4):272-276.
[3]CUROLE J P,KOCHER T D.Mitogenomics:digging deeper with complete mitochondrial genomes[J].Trends Ecology Evolution,1999,14(10):394-398.
[4]KAWAHARA R,MIYA M,MABUCHI K,et al.Interrelationships of the 11 gasterosteiform families(sticklebacks,pipefishes,and their relatives):a new perspective based on whole mitogenomesequences from 75 higher teleosts[J].Molecular Phylogenetics Evolution,2008,46(1):224-236.
[5]BOORE J L.Animal mitochondrial genomes[J].Nucleic Acids Research,1999,27(8):1767-1780.
[6]WANG H Y,TSAI M P,DEAN J,et al.Molecular phylogeny of gobioid fishes(Perciformes:Gobioidei)based on mitochondrial 12S rRNAsequences[J].Molecular Phylogenetics Evolution,2001,20(3):390-408.
[7]MIYA M,TAKESHIMA H,ENDO H,et al.Major patterns of higher teleostean phylogenies:a new perspective based on 100 complete mitochondrial DNAsequences[J].Molecular Phylogenetics Evolution,2003,26(1):121-138.
[8]CHENG J,MA G Q,SONG N,et al.Complete mitochondrial genomesequence of bighead croaker Collichthys niveatus(Perciformes,Sciaenidae):a mitogenomic perspective on the phylogenetic relationships of Pseudosciaeniae[J].Gene,2012,491(2):210-223.
[9]ZHANG X,YUE B,JIANG W,et al.The complete mitochondrial genome of rock carp Procypris rabaudi(Cypriniformes:Cyprinidae)and phylogenetic implications[J].Molecular Biology Report,2009,36(5):981-991.
[10]LIU Y,CUI Z.The complete mitochondrial genomesequence of the cutlassfish Trichiurus japonicus(Perciformes:Trichiuridae): Genome characterization and phylogenetic considerations[J].Marine Genomics,2009,2(2):133-142.
[11]PENG Z,WANG J,HEs.The complete mitochondrial genome of the helmet catfish Cranoglanis bouderius(Siluriformes:Cranoglanididae)and the phylogeny of otophysan fishes[J].Gene,2006,376(2):290-297.
[12]CAOs Y,WU X B,YAN P,et al.Complete nucleotidesequences and gene organization of mitochondrial genome of Bufo gargarizans[J].Mitochondrion,2006,6(4):186-193.
[13]MIYA M,NISHIDA M.Molecular phylogeny and evolution of the deep-sea fish genussternoptyx[J].Molecular Phylogenetics Evolution,1998,10(1):11-22.
[14]RANDI E,LUCCHINI V.Organization and evolution of the mitochondrial DNA control region in the avian genus Alectoris[J].J Mol Evol,1998,47(4):449-462.
[15]LEE W J,CONROY J,HOWELL W H,et al.Structure and evolution of teleost mitochondrial control regions[J].J ournal of Molecular Evolution,1995,41(1):54-66.
[16]金逍逍,孙悦娜,王日昕,等.虾虎鱼类线粒体全基因组序列结构特征分析及系统发育关系探讨[J].遗传,2013,35(12):1391-1402.
[17]李明德,罗阳,郭斌.中国鱼类名录Ⅻ:鳚亚目、(鱼+衔)亚目、喉盘鱼亚目、鰕虎鱼亚目、微体鱼亚目、刺尾鱼亚目、带鱼亚目、鲭亚目、鲳亚目、金枪鱼亚目、攀鲈亚目、刺鳅亚目[J].海洋通报,2003(5):64-70.
[18]伍汉霖,陈义雄,庄棣华.中国沙塘鳢属(Odontobutis)鱼类之一新种(鲈形目:沙塘鳢科)[J].上海水产大学学报,2002(1):6-13.
[19]THACKER C E.Molecular phylogeny of the gobioid fishes(Teleostei:Perciformes:Gobioidei)[J].Molecular Phylogenetics Evolution,2003,26(3):354-368.
[20]THACKER C E,HARDMAN M A.Molecular phylogeny of basal gobioid fishes:Rhyacichthyidae,Odontobutidae,Xenisthmidae,Eleotridae(Teleostei:Perciformes:Gobioidei)[J].Molecular Phylogenetics Evolution,2005,37(3):858-871.
[21]谢楠,冯晓宇,郭水荣,等.河川沙塘鳢线粒体细胞色素b(Cytb)基因序列分析[J].现代农业科技,2010(19):288-290.
[22]侯新远,祝斐,张丽娟,等.基于线粒体D-loop基因序列研究我国5种虾虎鱼类的系统进化关系[J].海洋渔业,2013(1):1-7.
Sequencing and Analysis of the Complete Mitochondrial Genome of Odontobutis
ZHANG Yanping1,ZHENG Hongmei1,SHAO Fang2,HAN Xiaolei1,XU Jianrong1,GU Zhiliang1
(1.School of Biology and Food Engineering,Changshu Institute of Technology,Changshu 215500;2.Cancer Institute,Changzhousecond People's Hospital Affiliated to Nanjing Medical University,Changzhou 213003,China)
The entire mitochondrial genome of Odontobutis was obtained by PCR based on conservative primers. It wasshowed that the mitochondrial genomesequence was 16,930 bp long,including 13 protein genes,22 tRNA genes,2 rRNA genes and a noncoding region.The base composition of the genome was A 30.0%,C 28.9%,G 15.6%and T 25.5%.Protein-coding genes in Odontobutis mitochondrial genome used ATG as the initiation codons with the exception of CO1 which used GTG and ND6 used TAA.It was found thatstop codons in Odontobutis included TAA,uncomplement codons TA and T.Most tRNA genes could form typicalsecondarystructures except tRNASer(AGN)and tRNACys.In addition,the D-loop region of Odontobutis mtDNA was 844 bp in length.Compared with the mitochondrial DNA control region from otherspecies,the extended terminal associatedsequences,central conservedsequence blocks,conservedsequence blocks and putative promoters of the mitochondrial DNA control region in Odontobutis were observed.Sequence alignment of Cyt b genes of Odontobutis and other 10species from GenBankshowed that Odontobutis obscara and Odontobutis had the closest genetic relationship in the 11species.A phylogenetic tree constructed using the Neighbor-Joining method indicated that the relationship amongsleeper Branch was close,the next weresculpin Branch andsciaenidae,and Cyprinid was distantly related.
Odontobutis;mitochondrial DNAsequence;D-Loop region;phylogeny
Q493;Q75
A
1008-2794(2015)04-0097-05
2016-04-12
教育部留学回国人员科研启动基金(第42批)“鳡鱼和赤眼鳟线粒体基因组及进化关系研究”;苏州市科技基础设施建设项目“农业动物(畜禽、水产)种质研究重点实验室”(SZZD201001)
顾志良,教授,博士,研究方向:动物分子遗传学,E-mail:zhilianggu88@hotmail.com.
