贵州地方黄牛品种遗传分化的分子评估
2016-08-22杨红文韩勇刘镜肖礼华粟朝芝
杨红文,韩勇,刘镜,肖礼华,粟朝芝
(1.贵州省畜禽遗传资源管理站,贵州 贵阳 550001;2.贵州省畜牧兽医研究所,贵州 贵阳 550005;3.贵州省遵义市畜牧技术推广站,贵州 遵义 563100)
贵州地方黄牛品种遗传分化的分子评估
杨红文1,韩勇2,刘镜2,肖礼华3,粟朝芝2
(1.贵州省畜禽遗传资源管理站,贵州 贵阳 550001;2.贵州省畜牧兽医研究所,贵州 贵阳 550005;3.贵州省遵义市畜牧技术推广站,贵州 遵义 563100)
选用联合国粮农组织(FAO)和国际动物遗传学会(ISAG)推荐的23对微卫星引物,采用荧光标记-多重PCR技术检测了4个贵州地方黄牛品种的遗传分化。结果显示,思南牛、关岭牛、威宁牛、黎平牛平均杂合度分别为0.71、0.69、0.69、0.65,23个位点多态信息含量在0.38~0.84之间,高度多态位点21个,中度多态位点2个。检测结果为贵州省本地黄牛品种分类提供了分子水平的科学依据。
地方黄牛品种;微卫星;遗传分化
开展畜禽品种遗传分化的分子评估,为畜禽的品种分类、保护和开发利用提供科学依据,有利于保护畜禽的优良特性和选育出能适应环境、疾病和市场需求的新品种,对促进畜牧业可持续发展具有重要的意义[1~8]。目前,关于贵州黄牛品种遗传分化在分子水平的评估研究仅有少量文献报道。本研究选用联合国粮农组织(FAO)和国际动物遗传学会(ISAG)推荐的23对微卫星引物,采用荧光标记-多重PCR技术,分析了4个贵州地方黄牛品种的遗传分化。
1 材料和方法
1.1 材料本研究中4个牛品种血样的采集地及采集数量见表1。每个牛品种随机采集约60头,其中公牛不少于10头。要求采样个体具备品种的典型特征,并且在3代或2代内没有血缘关系。采用前腔静脉采血,每头牛采血量10 mL。新鲜血液用1︰1的裂解液(100 mmol/L Tris-HCL,100 mmol/LEDTA,2%SDS)裂解,常温下即可运输,-20℃长期保存。
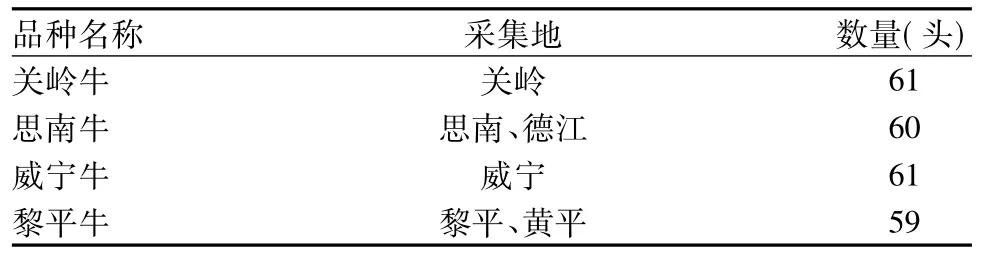
表14 个牛品种名称和采样情况
1.2 实验方法
1.2.1 基因组DNA提取:血液DNA提取采用常规酚氯仿抽提法。Taq酶、10×buffer均购自宝生物工程(大连)有限公司,dNTPs购自上海生工生物工程技术服务有限公司,POP4胶、甲酰胺等电泳检测试剂均使用美国应用生物系统公司进口试剂。提取出的基因组DNA首先经1.5%琼脂糖凝胶电泳检测合格,然后用生物分光光度计将其浓度调整为20~50 ng/μL后即可进行PCR扩增。
1.2.2 多重PCR扩增:根据联合国粮农组织(FAO)推荐的引物,选取了其中12对微卫星引物:CSSM014、CSSM016、CSSM022、ETH3、ETH10、ETH225、BM1818、BM1824、BM2113、TGLA122、TGLA126、TGLA227、MGTG7、TGLA48、TGLA53、TGLA263、SPS113、SPS115、GSSM036、MGTG4B、TGLA57、TGLA73、AGLA293,引物信息见FAO网址(http://www.fao.org)。上游引物5端标记荧光,根据各引物的序列、复性温度、目的片段大小,将引物有机组合进行扩增,分别对引物浓度、Mg2+浓度、复性温度、循环次数进行优化,取得最佳扩增效果。
PCR反应体系为:2.5 μL MgCl2,2.5 μL dNTP(2 mM),2.5 μL 10×缓冲液,上下游引物各1 μL,1 μL TaqE(1 U/μL),1 μL DNA,13.5 μL dH2O,总体系为25 μL。PCR扩增程序为:94℃预变性4 min;94℃变性50 s,退火50 s(退火温度视引物而定),72℃延伸50 s,共35个循环;最后72℃延伸10 min。
1.2.3 PCR产物检测:PCR产物扩增成功后,采用ABI3100-Avant全自动基因分析仪进行检测,微卫星基因型用仪器自带软件Gene Mapper Vers ion 3.2进行分析。
1.2.4 统计分析:用POPGENE1.32软件(http://www.ualberta.ca/~fyeh/)进行统计分析。
2 结果
2.1 遗传距离通过3种途径估算的遗传距离见表2。Nei’s最小遗传距离在0.017 9~0.036 4之间,Nei’s标准遗传距离在0.117 8~0.061 8之间,Nei’s最大遗传距离在0.068 6~0.078 1之间。3种不同遗传距离估算方法获得的结果均显示,关岭牛和思南牛之间遗传距离最小,黎平牛和思南牛之间遗传距离最大。

表24 个品种两两间的遗传距离
2.2 基因型一致概率我省4个地方黄牛品种两两间基因型一致的概率见表3,关岭牛和思南牛之间的概率最大,为2.892×10-23;黎平牛和思南牛之间的概率最小,为1.001×10-24。
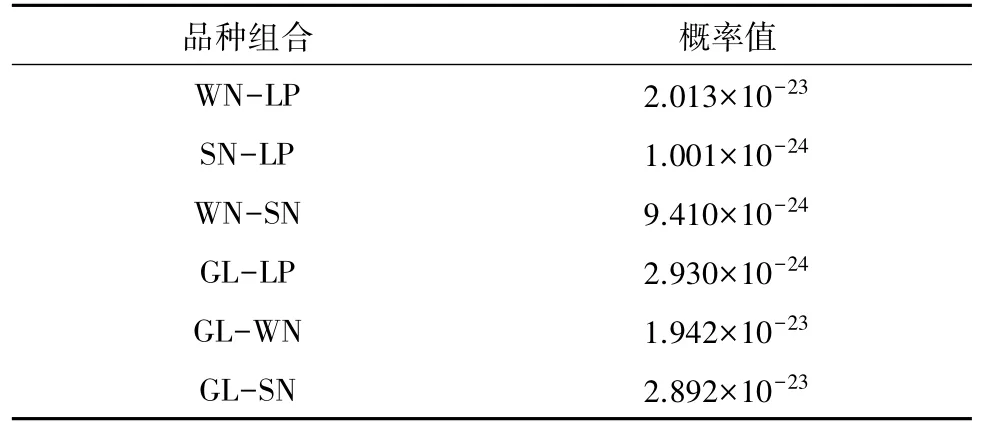
表34 个地品种两两间基因型一致的概率
2.3近郊系数和分化系数我省4个地方牛品种在23个微卫星位点上的F-statistics分析结果见表4。从表4可以看出,4个地方牛品种在各位点上Fit为-0.956~0.992,平均值0.242;Fis为-0.949~0.973,平均值0.333。Fst值在位点TGLA263最大,为0.798;在位点TGLA53最小,为0.510。Fst平均值为0.713,表明28.7%的遗传变异发生在品种内,71.3%的遗传变异发生在品种间,表明各品种的遗传变异主要发生在品种间。
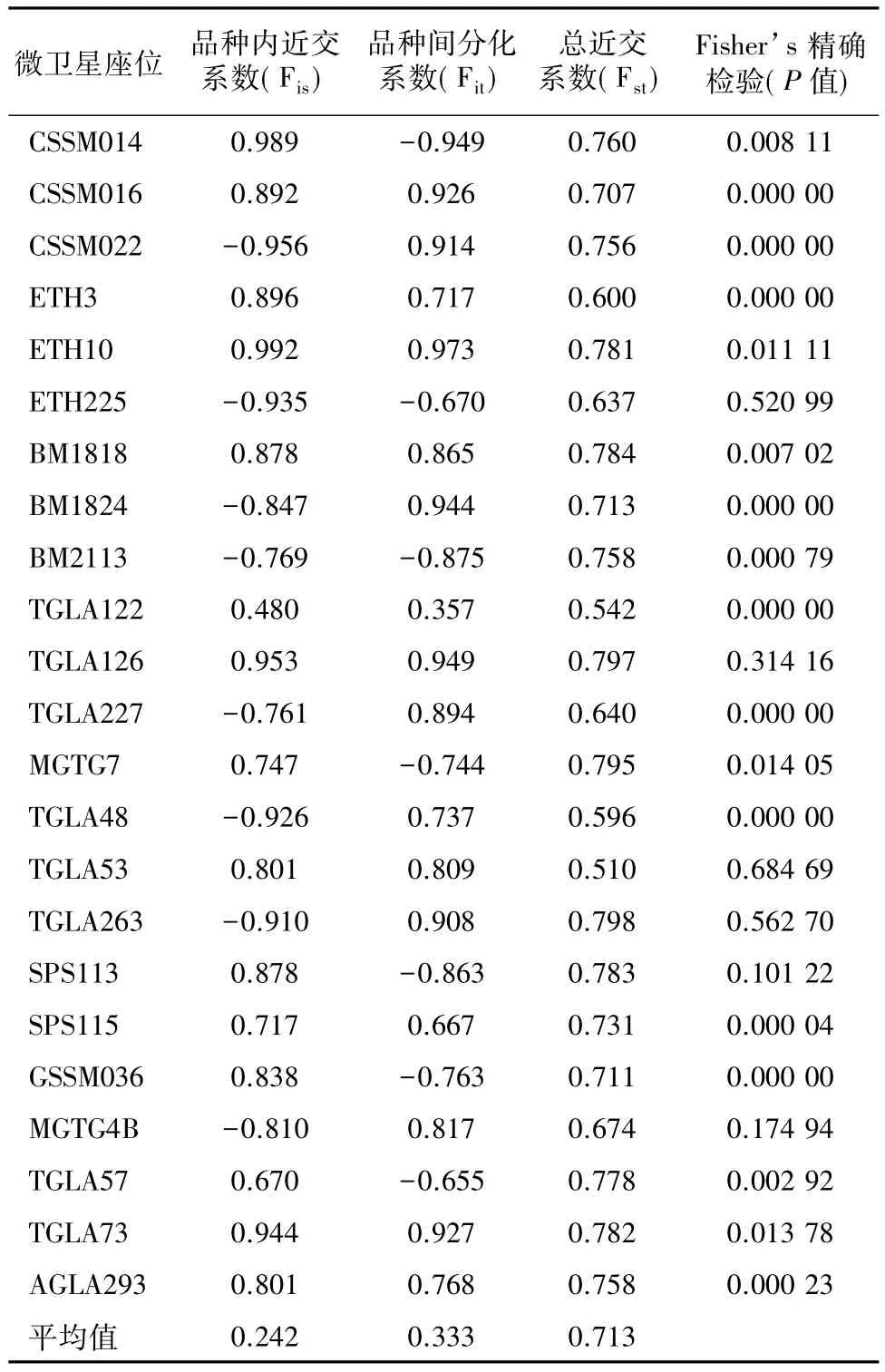
表4 群体分化的F统计和Fisher’s精确检验
3 讨论
3.1 关于遗传距离Nei等通过计算机模拟对各种遗传距离进行研究,证明在分析物种内群体间的遗传变异时,通过最小遗传距离、标准遗传距离、最大遗传距离3种途径估算的遗传距离最能反映出群体间变异实际情况[11~15]。本研究3种不同遗传距离估算方法获得的结果均显示,关岭牛和思南牛之间遗传距离最小,黎平牛和思南牛之间遗传距离最大。
3.2关于近郊系数和分化系数品种内近交系数(Fit)、总近交系数(Fis)、品种间分化系数(Fst)均是反应品种间近交程度或遗传分化的指标。Fis表示的是亚群内个体间由于非随机交配从而导致的亚群内杂合度的缺乏,Fit则反映总品种杂合度的缺乏程度;Fst反映的是各亚群内平均杂合度与总品种平均杂合度的差异程度。目前,基于微卫星DNA分子标记对贵州黄牛品种遗传分化研究较少,能提供的遗传学数据不多。我省地方黄牛品种71.3%的遗传分化发生在品种间,28.7%的遗传变异发生在品种内。此结果为我省本地黄牛品种分类提供了分子水平的科学依据,应用前景广阔。
[1]谷继承,刘丑生,刘刚,等.牛遗传资源分子遗传多样性的研究进展[J].中国畜牧兽医,2010,37(9):127-130.
[2]何正权,张亚平,筒承松,等.贵州黄牛品种间mtDNA的限制性片段长度多态性研究[J].动物学研究,1999,20(1):7-11.
[3]李荣岭,张桂香,王志刚,等.微卫星标记对12个中外牛品种群体遗传分化的研究[J].遗传,2007,29(12):1463-1470.
[4]林瑞意,杨胜林,徐龙鑫.贵州务川黑牛mtDNA Cytb基因遗传多样性研究[J].云南农业大学学报,2010,25(5):622-625.
[5]刘若余,夏先林,雷初朝,等.贵州黄牛mtDNA D-loop遗传多样性研究[J].遗传,2006,28(3):279-284.
[6]马月辉,曹红鹤,陈幼春,等.部分黄牛品种(群体)遗传多样性分析[J].中国农业科学,2003,36(6):696-699.
[7]王均辉,昝林森,张桂香,等.中国9个南方黄牛品种和3个引进品种的遗传多样性分析[J].西北农林科技大学学报(自然科学版),2008,36(3):1-7.
[8]张桂香,郑有民,王志刚,等.我国部分黄牛品种线粒体D-loop区遗传多样性与起源分化[J].遗传,2009,31(2):160-168.
[9]Yu Y,Nie L,He ZQ,et al.Mitichondrial DNA variation in cattle of south China:origin and introgression[J].Animal Geneties,1999(30):1-6.
[10]DE-XING ZHANG,GODFREY M.HEWITT.Nuclear DNA analyses in genetic studies of populations:practice,problems and prospects[J].Molecular Ecology,2003(12):563-584.
[11]Ginja C,Penedo MC,Melucci L,et al.Origins and genetic diversity of New World Creole cattle:inferences from mitochondrial and Y chromosome polymorphisms[J].Anim Genet,2010(41):128-141(a).
[12]Ginja C,Telo Da Gama L,Penedo MC.Analysis of STR markers reveals high genetic structure in Portuguese native cattle[J].J Hered,2010,101(2):201-210(b).
[13]M.Sodhi,M.Mukesh,S.P.S.Ahlawat,et al.Genetic diversity and structure of two prominent zebu cattle breeds adapted to the arid region of india inferred from microsatellite polymorphism[J].Biochem Genet,2008(46):124-136.
[14]Susana DUNNER,Nuno FERRAN,David GARCIA,et al.Genetic diversity measures of local European beef cattle breeds for conservation purposes[J].Genet.Sel.Evol.2001(33):311-332.
[15]Catarina Ginja,Maria CT Penedo,Maria F Sobral,et al.Meseoarlceh.Molecular genetic analysis of a cattle population to reconstitute the extinct Algarvia breed[J].Genetics Selection Evolution,2010(42):18.
Molecular Evaluation on Genetics Differentiation of Native Yellow Cattle Breeds from Guizhou Province
Yang Hongwen1,Han Yong2,Liu Jing2,Xiao Lihua3,Su Chaozhi2
(1.Guizhou Management Station of Animal Genetic Resources,Guiyang Guizhou 550001,China;2.Guizhou Institute of Animal Husbandry and Veterinary Science,Guiyang Guizhou 550005,China;3.Zunyi Committee for Agriculture,Zunyi Guizhou 563100,China)
Four native yellow cattle breed from Guizhou province were studiedthe genetics differentiation by using the multiple PCR with fluorescence labeling-method.A total of 23 pair primers recommended by FAO and ISAG were used to amplify.The results showed the mean heterozygosity of Sinan cattle,Guangling cattle,Weining cattle,and Lipingcattle are 0.71,0.69,0.69 and 0.65 respectively.Thepolymorphism information contents(PIC)of 23 loci ranged from 0.38 to 0.84,including 21 highly polymorphic loci and 2 moderately polymorphic loci.These results provided the molecular proof for the varieties classificationof native yellow cattle breed from Guizhou province.
Nativeyellow Cattle Breed;Microsatellite;Genetics Differentiation
S823.2:Q75
A
1007-1474(2016)06-0005-03
2016-09-05
贵州省自然科学基金(黔科合J字[2011]2224号)
杨红文(1979—),男,高级畜牧师,研究方向:分子遗传学与动物育种。
