大肠杆菌酶基因缺失菌株碳中心代谢系统的代谢通量分析
2014-03-28金杰军范亚东
芮 斌,金杰军,范亚东,文 汉
(安徽农业大学生命科学学院,合肥230036)
大肠杆菌E.coli是进行微生物遗传研究的重要材料,如局限性转导就是1954年在大肠杆菌K12菌株中发现的.莱德伯格(Lederberg)采用两株大肠杆菌的营养缺陷型进行实验,奠定了研究细菌接合方法学以及基因工程研究的基础.大肠杆菌作为外源基因表达的宿主,遗传背景清楚,技术操作简单,培养条件简单,大规模发酵经济,所以倍受遗传工程专家的重视.目前大肠杆菌是应用最广泛,最成功的表达体系,常做高效表达的首选体系.
系统生物学着重以系统的思路和方法定量研究生物过程.由于生物系统的组成关系主要用网络来表示,代谢网络研究就自然成为热门领域[1-2].最初的网络理论往往只涉及一个底物和一个产物,故而与实际网络不符.FELL等学者致力于线性代数的方法来更便利地计算代谢途径,其主要思想是先假设代谢网络的途径是代谢网络计量系数矩阵的线性无关解,然后通过对这些途径的线性组合或者枚举来产生存在的(但未必是热力学可行的)代谢途径.最近,凸分析被运用到代谢途径分析中,代谢网络理论于是得到了更进一步的发展,渐渐形成了普遍认可的两种方法:基元模式分析(elementary flux mode analysis)和极端途径分析(extreme pathway analysis).同时在另一个层面上,对代谢网络的研究进一步逐渐演化出单独的静态分析和动态分析.静态分析主要包括Schilling等学者提出的极端途径(EPS),基元模式(EFMS),FBA流量平衡分析和jeong等学者提出的图论法(graph theory)[3-6].而动态分析方法主要是基于动力学信息的,它包括代谢控制分析(metabolic control analysis),控制论模拟方法(cybernetic modeling).目前对基因组规模的代谢网络来说,许多反应的动力学参数和反应物浓度参数都是不清楚的,故静态的研究方法占据了目前代谢网络研究的主流位置[7-8].
本文在matlab cobra工具箱的基础上对大肠杆菌代谢系统进行FBA和FVA分析[9-12],将分析结果进行汇总后,为显示在敲除和不敲除各种酶基因的结果变化,对数据进行绘图,通过直观的图形对比,显示出敲除各种酶基因对大肠杆菌代谢系统中每一个反应的影响,以及在敲除大肠杆菌某个酶基因后代谢系统的全部反应的FBA和FVA的变化之间是否存在共性,以此为依据,大致判断各个酶在大肠杆菌代谢系统中所起的作用.大肠杆菌代谢系统中有很多的酶基因参与,本研究以25个酶为例,分别单独将每一个酶相应的酶基因从代谢系统中敲除后,对代谢系统进行FBA和FVA分析[13-15],将结果与未敲除任何酶基因的代谢系统的FVA和FBA结果进行对比,以确定所敲除的酶在代谢系统中所起的作用,这提供了一个全新的酶分析方法.这样的结果虽说只是猜测,但可对以后的实验起到指导性作用.
1 材料与方法
1.1 所需操作软件以及文件
Matlab版本7.0以上.cobra toolbox版本2.0以上.libsbml版本4.0.1以上.Sbml toolbox版本3.1.1以上.gurobi 5.0以上.
大肠杆菌小型中心代谢模型‘ecoli_core_model.xml’,此模型中包含大肠杆菌中心代谢的全部信息[6].
1.2 方法
1.2.1 代谢模型读取 在matlab命令窗口中执行指令:model=readCb Model(‘文件名’)其中文件名为模型保存目录[11,15].
1.2.2 模型的修饰 执行指令:
model=changeRxnBounds(model,'EX_glc(e)',-16.7,'l');其中'EX_glc(e)'代表了底物摄取速率,‘l’是‘lower’的缩写,表示模型的下限[2].1.2.3酶基因的敲除 执行指令:
model=removeRxns(model,'rxnRemove List');'rxn Remove List'代表的是含有所敲除反应的反应信息的单元矩阵,酶基因的敲除中,rxnRemove List可直接用所需敲除酶的英文缩写代替.如敲除烯醇化酶即ENO酶,直接在命令窗口中输入指令model=removeRxns(model,'ENO')即可,执行命令后表示该酶所直接参与的反应的电子信息从模型中去除,该反应在模型中不再起作用.
1.2.4 FBA最优化值的获取 执行指令:change-CobraSolver('gurobi5','LP');Fluxdistribution=optimizeCb Model(model);flux=geometricFBA(model),
其中第一步指令是修改cobra solver;第二步是执行标准FBA命令.
1.2.5 FVA(Flux Variability Analysis)值的获取 执行命令:
[minFlux,max Flux]=flux Variability(model,90);其中‘90’代表一个百分比,为模型所能达到的目标函数相对于最优值的比例.FVA命令的结果有min Flux和max Flux,分别代表模型中95个反应的通量分布情况.
1.3 作图说明
作图从两个方向进行,分别为横向和纵向,横向作图在于显示模型中每个单独反应在不同酶基因被敲除后受到的影响,作图时横坐标代表被敲除的酶基因,为更好的显示结果,分别用数字代表,对应情况为:1.没有敲除任何酶基因.2.EDD.3.GLUUPT.4.GDH.5.GND.6.TK2.7.PFK.8.HX I.9.TAL.10.TK1.11.SER.12.SHMT.13.GEV.14.ENO.15.PYK.16.PDH.17.ACEX.18.GLTA.19.ICD.20.AKD.21.FUM.22.MDH.23.GS1.24.GS2.25.PPC.26.MEZ[4].横向作图时FBA的结果使用折线图显示,FVA的结果选用柱形图来显示区间的变化.纵向作图在于显示敲除某个酶基因对整个代谢系统的影响,作图时横坐标的数字分别代表大肠杆菌代谢模型中的95个反应.纵向作图时由于反应有95个,在进行FVA分析时,用柱形图难以显示结果的变化,所以将结果进行拆分,分别对minflux和maxflux作图.
2 结果与分析
2.1 横向分析
文中只将反应ATP synthase,Biomass,glutamate transport的流量变化图附上(图1~图14),其他反应可以同理进行分析.
2.2 纵向分析
横向分析后,会发现在酶基因14(ENO),15(PYK),16(PDH),21(FUM),25(PPC)等被敲除后,反应受到的影响最大,本文只将酶ENO即烯醇化酶,酶PDH即丙酮酸脱氢酶,酶FUM即延胡索酸酶,酶PPC即磷酸烯醇式丙酮酸羧化酶的纵向分析图附上,其它酶可以用同种方法进行分析.

图1 反应ATP synthase在敲除不同的酶基因后最优流量值的变化Fig.1 Changes in reaction ATP synthase after the knockout of different enzyme gene

图2 反应ATP synthase在敲除不同的酶基因后流量范围的变化Fig.2 Changes in flux range of reaction ATP synthase after the knock out of different enzyme gene

图3 反应Biomass在敲除不同的酶基因后最优流量值的变化Fig.3 Changes in reaction Biomass after the knockout of different enzyme gene

图4 反应Biomass在敲除不同的酶基因后流量范围的变化Fig.4 Changes in the flux range of reaction Biomass after the knockout of different enzyme gene

图5 反应glutamate transport在敲除不同的酶基因后最优流量值的变化Fig.5 Changes in reaction glutamate transport after the knockout of different enzyme gene

图6 反应glutamate transport在敲除不同的酶基因后流量范围的变化Fig.6 Changes in flux range of reaction glutamate transport after the knockout of different enzyme gene

图7 敲除酶基因ENO的代谢系统中全部反应的FBA结果变化Fig.7 The FBA results change of all reaction in ENO knockout metabolic system

图8 敲除酶ENO的代谢系统中全部反应FVA结果的变化Fig.8 The FVA results change of all reaction in ENO knockout metabolic system

图9 敲除酶PDH的代谢系统中全部反应的FBA结果变化Fig.9 The FBA results change of all reaction in PDH knockout metabolic system
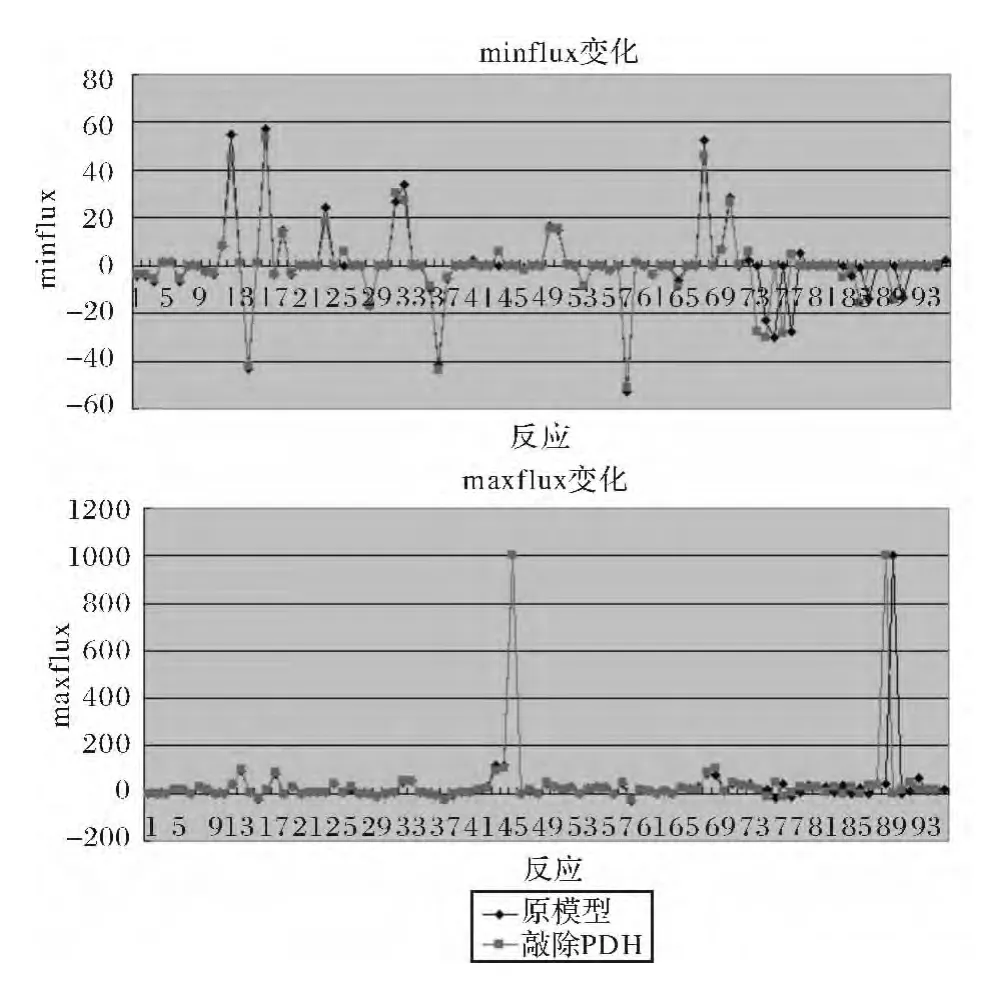
图10 敲除酶PDH的代谢系统中全部反应的FVA结果变化Fig.10 The FVA results change of all reaction in ENO knockout metabolic system

图11 敲除酶FUM的代谢系统中全部反应的FBA结果变化Fig.11 The FBA results change of all reaction in FUM knockout metabolic system
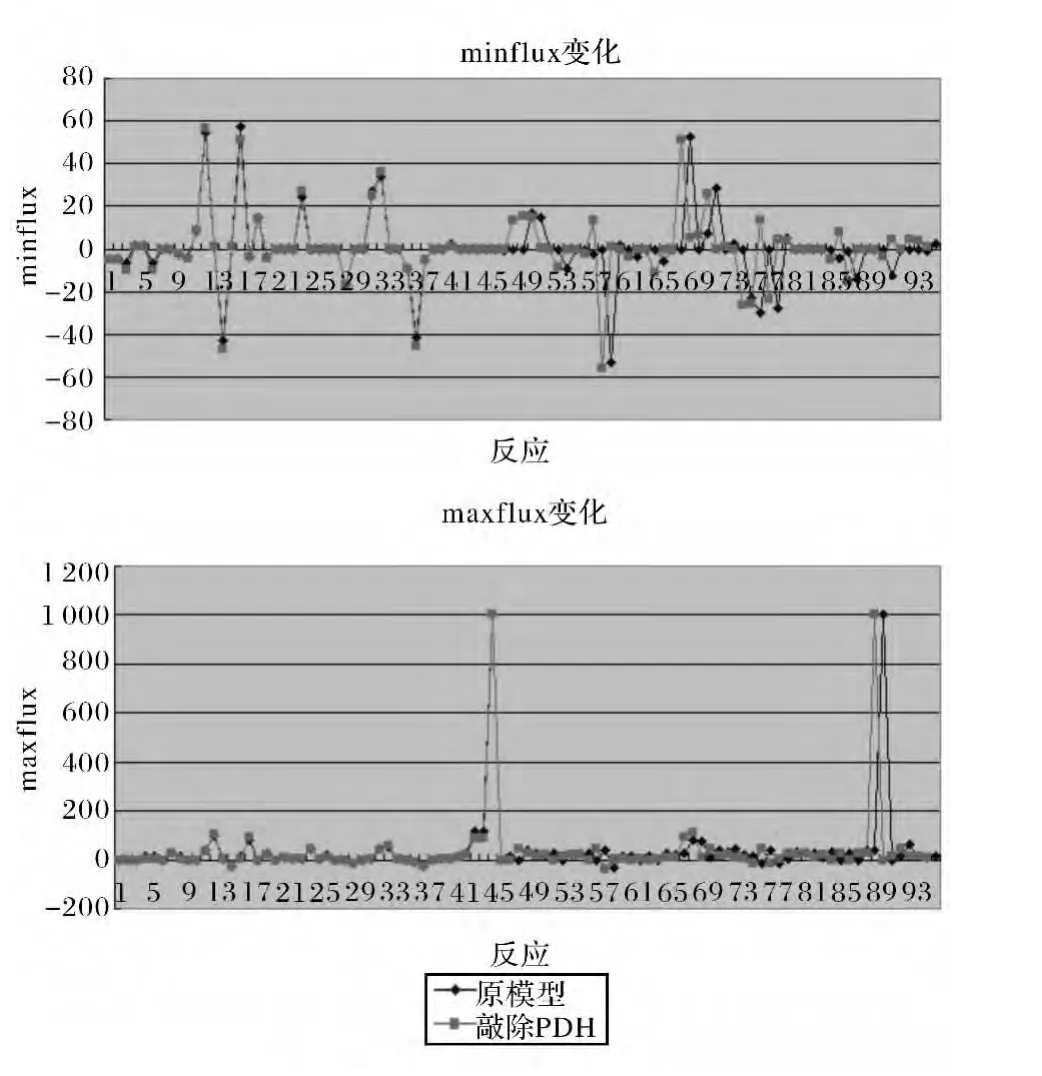
图12 敲除酶FUM的代谢系统中全部反应的FVA结果变化Fig.12 The FVA results change of all reaction in FUM knockout metabolic system
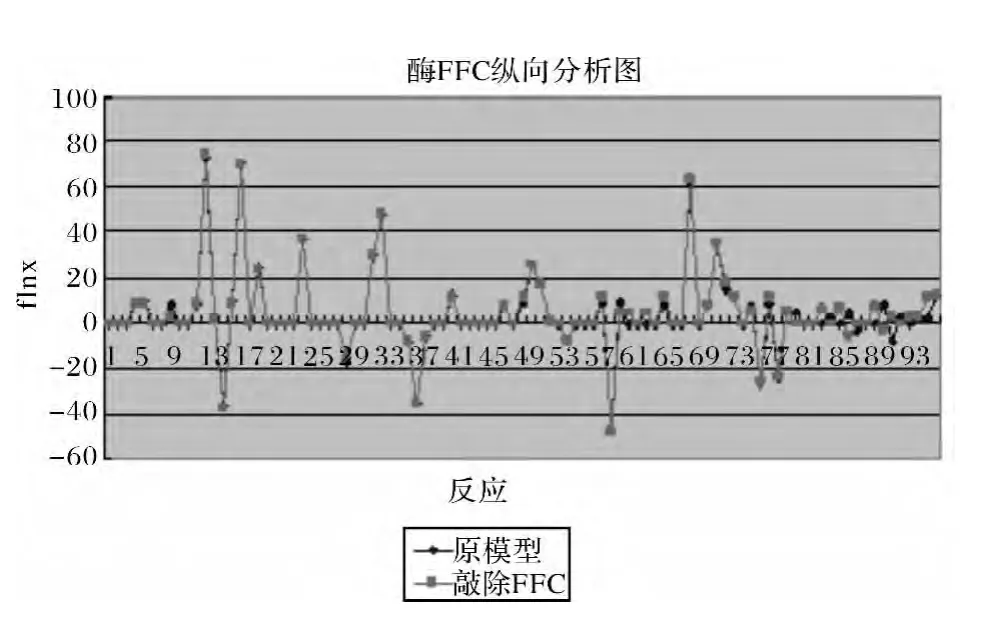
图13 敲除酶PPC的代谢系统中全部反应的FBA结果变化Fig.13 The FBA results change of all reaction in PPC knockout metabolic system
3 结论与讨论
综合横向分析和纵向分析,可以确定酶ENO即烯醇化酶,酶FUM即延胡索酸酶,酶PDH即丙酮酸脱氢酶等对整个代谢系统的影响较大,所以以酶ENO、FUM和PDH为例进行讨论.
3.1 关于ENO
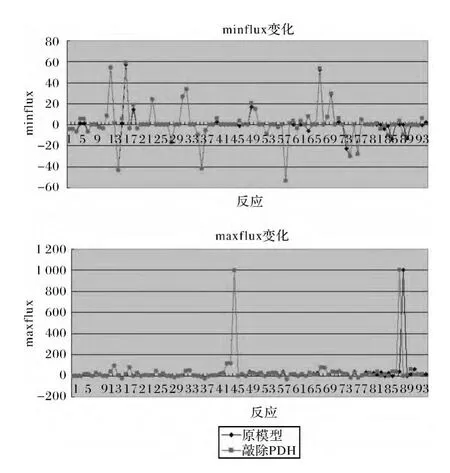
图14 敲除酶PPC的代谢系统中全部反应的FVA结果变化Fig.14 The FVA results change of all reaction in PPC knockout metabolic system
通过纵向分析,发现ENO敲除后,在大肠杆菌代谢系统中有相当多的反应都受到了影响,而酶的催化作用具有专一性,即只催化一种或一类底物,因此我们可以推测出酶ENO所参与的反应应该是位于大肠杆菌代谢系统比较靠前的位置,可以为代谢系统之后的反应提供底物等.再返回汇总数据,可以发现,酶ENO被敲除后反应代谢系统中反应就会少一个,说明在这些酶所直接参与的反应中,它们的作用是不可替代的,即大肠杆菌代谢系统中ENO酶无同工酶,由此可以基本判定酶ENO所催化的反应.再观察横向分析图与汇总的数据,会发现酶ENO对反应的影响可以分为两类:
(1)ENO酶敲除后,反应被抑制,可分为两类:A.ENO酶敲出后该反应被抑制,但是并未停止,如反应ATP synthease;B.ENO酶被敲出后该反应完全停止,如反应Biomass.
这说明代谢系统中反应ATP synthease和反应Biomass在酶ENO直接参与的反应后面进行.而且反应Biomass离ENO酶直接参与的反应可能更近一点,这样有利于我们对大肠杆菌代谢网络结构的理解.
(2)ENO酶被敲除后,反应被促进,如反应glutamate transport.考虑到大肠杆菌是兼性厌氧性微生物,因此猜测:酶ENO可抑制反应glutamate transport的进行或者是抑制反应glutamate transport底物的合成,也可能是酶ENO的产物对反应有抑制作用,即反应glutamate transport可能是大肠杆菌在另一种不同的环境才能进行的.
3.2 关于FUM
(1)FUM酶敲除后,会有小部分反应被抑制.可分为两类:A.FUM酶敲除后该反应被抑制,但是并未停止,如enolase催化的反应;B.FUM酶被敲出后该反应几乎停止,如由aconitase催化citrate生成isocitrate的反应.这说明代谢系统中FUM反应生成物对enolase和aconitase催化的反应产生了约束,当FUM被敲除后,这二者都受到影响.
(2)在FUM被敲除后,还有一部分反应是发生了大程度的提升.比如pyruvate reversible transport和Fumarate transport发生明显的上调.这个结果是由于fumarate的消除途径被敲除后,fumarate累积之后外流到胞外.同时,整个TCA cycle的循环受到阻遏,使得pyruvate也发生累积并外流到胞外.这代表细菌代谢系统发生了非常明显的转化.
(3)还有部分反应在FUM敲除后,只发生了非常微弱的上调.这些反应是在野生型菌株中就有着很高流量的反应.这说明FUM的敲除对于代谢网络的物质分配影响不大.
3.3 关于PDH
PDH敲除后对整体代谢有一定的影响,但是其影响程度并没有前两种酶基因敲除后的影响大.这表明,在氧气充分的条件下,PDH通路在大肠杆菌体内有其替代途径,存在冗余性.
同理可以应用于大肠杆菌代谢系统中的其他酶和反应的分析,这将有利于我们对大肠杆菌代谢系统的认识.在此所用的大肠杆菌小型中心代谢系统模型,是比较简单的的代谢系统,这样的分析方案不仅适用于这样的小型代谢系统,也可用于对比较复杂的代谢网络分析.
[1] 花 强,杨 琛.代谢流量比率分析及其在代谢工程中的应用[J].生物工程学报,2009,25(9):1303-1311.
[2] 史晋辉.细胞代谢过程分析及模型优化[J].生命的化学,2001,21(1):71-73.
[3] Tie Shen,Bin Rui,Hong Zhou,et al.Metabolic flux ratio analysis and multi-objective optimization revealed a globally conserved and coordinated metabolic response of E.coli to paraquat-induced oxidative stress[J].Molecular BioSystems,2012,9(1):121-132.
[4] 杨志杰.基于代谢流量分析的微生物细胞代谢特性研究[D].上海:华东理工大学,2011.
[5] 陈 琦,王 卓,魏冬青.代谢网络流分析进展及应用[J].中国科学,2010,55(14):1302-1309
[6] 丁德武,何小青,陆克中,等.用约束法研究大肠杆菌代谢网络核心模型[J].计算机与应用化学,2010,27(6):835-838.
[7] 李 毅,郑浩然,钮俊清,等.VFA:一种可视化代谢网络建模工具[J].北京生物医学工程,2008,27(5):490-494.
[8] 谭青乔,陈 宁,王东洋.代谢工程发展及应用[J].生物技术通讯,2001,12(2):123-126.
[9] 魏 春,陈 宁.代谢网络定量分析研究进展[J].生物技术通讯,2002,13(3):234-237.
[10] 马红武,赵学明,迟万忠.应用Excel处理生化过程数据(Ⅰ)代谢通量分析及代谢网络优化[J].计算机与应用化学,1998,15(6):274-278.
[11] 蒋 达.代谢网络通量和结构分析[M].大连:大连理工大学出版,2006.
[12] 赵 静.细胞代谢网络的结构、功能与进化研究[D].上海:上海交通大学,2008.
[13] Tassoula E A.Growth possibilities of E.Coli in natural waters[J].International Journal of Environmental Studies,1997,52(1-4):67-73.
[14] Kostromins Andrejs,Stalidzans Egils,Paint Net.COBRA Toolbox extension for visualization of stoichiometric models of metabolism[J].BioSystems,2012,109(2):233-241.
[15] Jan Schellenberger,Richard Que,Ronan M T Fleming,et al.Quantitative prediction of cellular metabolism with constraint-based models:the COBRA Toolbox v2.0[J].Nature,2011,21(6):1290-1307.
