棕颈钩嘴鹛分子系统发育及分类关系初探
2011-12-25刘鲁明邹发生雷富民李寿先杨晓君
宋 蕊, 董 锋, 刘鲁明 吴 飞 王 凯 邹发生, 雷富民, 李寿先, 杨晓君
(1. 中国科学院昆明动物研究所, 云南 昆明 650223; 2. 华南濒危动物研究所, 广东 广州 510260;3. 中国科学院动物研究所, 北京 100101; 4. 台湾师范大学 生命科学系, 台湾 台北 116; 5. 中国科学院研究生院, 北京 100049)
棕颈钩嘴鹛分子系统发育及分类关系初探
宋 蕊1,5, 董 锋1,5, 刘鲁明1, 吴 飞1, 王 凯1, 邹发生2, 雷富民3, 李寿先4,*, 杨晓君1,*
(1. 中国科学院昆明动物研究所, 云南 昆明 650223; 2. 华南濒危动物研究所, 广东 广州 510260;3. 中国科学院动物研究所, 北京 100101; 4. 台湾师范大学 生命科学系, 台湾 台北 116; 5. 中国科学院研究生院, 北京 100049)
棕颈钩嘴鹛(Pomatorhinus ruficollis)各亚种间系统发育关系一直存在分歧。该文测定了棕颈钩嘴鹛 10个亚种20个样本的完整线粒体Cyt b和ND2基因序列(共2 184 bp), 以棕颈钩嘴鹛(P. ruficollis)和灰头钩嘴鹛(P.schisticeps)为内群; 栗背钩嘴鹛(P. montanus)、棕头钩嘴鹛(P. ochraceiceps)、红嘴钩嘴鹛(P. ferruginosus)和剑嘴鹛(Xiphirhynchus superciliaris)为外群, 通过最大简约法、最大似然法以及贝叶斯推断法, 构建了棕颈钩嘴鹛亚种间的系统发育树。结果显示:棕颈钩嘴鹛是并系群, 灰头钩嘴鹛聚合在其内部; 棕颈钩嘴鹛可分为3个地理支序, 即中国西南部支序、中国东南部支序和中国中部支序;棕颈钩嘴鹛亚种间系统发育关系与形态特征不完全一致, 并澄清了一些分类问题。
棕颈钩嘴鹛;Cyt b;ND2;种下分类
棕颈钩嘴鹛(Pomatorhinus ruficollis)隶属雀形目(Passeriformes)鹟科(Muscicapidae)画鹛亚科(Timaliinae)钩嘴鹛属(Pomatorhinus), 主要分布于东洋区中印亚区中北部以及喜玛拉雅山南部(Sibley& Monroe, 1990), 分布海拔为200~3 000 m (Collar& Robson, 2007), 属于林下层活动的鸟类, 其典型特征为具栗色的颈圈、白色的长眉纹和黑色的贯眼纹, 胸部具有纵纹或无纹。通过检查中国科学院昆明动物研究所的馆藏标本和结合现有文献(Ramsay,1878; Cheng, 1962; Cheng et al, 1987; Yang & Yang,2004; Collar, 2006; Collar & Robson, 2007), 发现棕颈钩嘴鹛种下的形态变化非常大, 仅依据胸纹就至少可以分为3个类群, 即胸部具黑褐色点斑的有P. r. musicus和P. r. nigrostellatus;胸部几乎为白色的有P. r.bhamoensis、P. r. beaulieui和P. r.albipectus;胸部具橄榄褐色至深栗色纵纹的有P. r.bakeri、P. r. laurentei、P. r. styani、P. r. similes、P. r.godwini、P. r. eidos、P. r. stridulus、P. r. hunanensis和P. r. reconditus。对棕颈钩嘴鹛种下形态殊异的亚种, 存在几种不同分类意见:Ramsay(1878)曾将其划分为3个种, 即P. ruficollis、P. nigrostellatus和P. musicus; 后来的分类处理均将其归为P. ruficollis,最常见的是将其分为 14个亚种(Deignan, 1964;Clements, 2000; Dickinson, 2003)。Collar (2006)及Collar & Robson(2007)认为P. musicus较之P.ruficollis来说体型更大, 上嘴全黑且较长, 头部深灰色, 后颈的栗色更宽更多, 胸纹为清晰的黑褐色的卵圆状, 腹部和两胁为明显栗色, 可以作为一个独立种; Collar (2006)认为虽然P. r. nigrostellatus的胸纹与P. musicus相似, 但其胸羽还保持有稀薄的白色边缘,背侧也与其它亚种相似, 因此,不提倡将P. r. nigrostellatus提升为种,从而将棕颈钩嘴鹛分为两个种:一个是含13个亚种的P. ruficollis; 一个是单型种P. musicus。然而, Dong et al (2010a)和Reddy & Moyle(2011)的分子研究结果均表明, 灰头钩嘴鹛(P. schisticeps)个体与棕颈钩嘴鹛个体呈现嵌套关系并共同构成一个单系。目前的形态学和分子生物学的研究均说明,棕颈钩嘴鹛各亚种的系统发育关系有进一步探讨的必要。
本研究以线粒体DNA数据重构棕颈钩嘴鹛的种下系统发育关系, 以检验Ramsay (1878)、Collar(2006)、Collar & Robson (2007)对本种的分类意见,了解该物种的演化历史并完善其分类处理。同时,探讨鸟类形态和环境的适应过程, 促进东南亚众多形态存在高度差异物种的研究工作。
1 材料与方法
根据 Collar & Robson (2007)和 Cheng (1962)的分类建议, 在取样时暂将P. r. laurentei、P. r. styani、P. r. musicus和P. r. nigrostellatus作为独立亚种, 并根据Dong et al (2010a)的研究结果, 以棕颈钩嘴鹛和灰头钩嘴鹛为内群; 以栗背钩嘴鹛(P. montanus)、棕头钩嘴鹛(P. ochraceiceps)、红嘴钩嘴鹛(P.ferruginosus)和剑嘴鹛(Xiphirhynchus superciliaris)为外群, 构建线粒体细胞色素b(Cyt b)与NADH脱氢酶第二亚基(ND2)两个基因合并数据集的系统发育树。
1.1 样本采集及总DNA的提取
除P. r. godwini外, 采集到在国内分布的棕颈钩嘴鹛有10个亚种, 20个样本。样本保存于99%的酒精分析纯溶液中, 并存放于−80 ℃冰箱。总DNA的提取采用经典的异丙醇沉淀法, 提取后的 DNA经1.5%琼脂糖凝胶电泳检测。灰头钩嘴鹛、栗背钩嘴鹛、棕头钩嘴鹛、红嘴钩嘴鹛和剑嘴鹛的序列均下载自GenBank, 所有样本的标本号及采集地见表1。KIZ:中国科学院昆明动物研究所; SCIEA:华南濒危动物研究所;NTNU:台湾师范大学; IOZ:中国科学院动物研究所; GenBanka~d来自Dong et al (2010a); GenBanke来自 Cibois (2003)和 Pasquet et al (2006)。KIZ: Kunming Institute of Zoology, the Chinese Academy of Sciences;SCIEA: South China Institute of Endangered Animals; NTNU, National Taiwan Normal University; IOZ: Institute of Zoology, Chinese Academy of Sciences; GenBanka−dcame from Dong et al (2010a); GenBankecame from Cibois (2003) and Pasquet et al (2006).
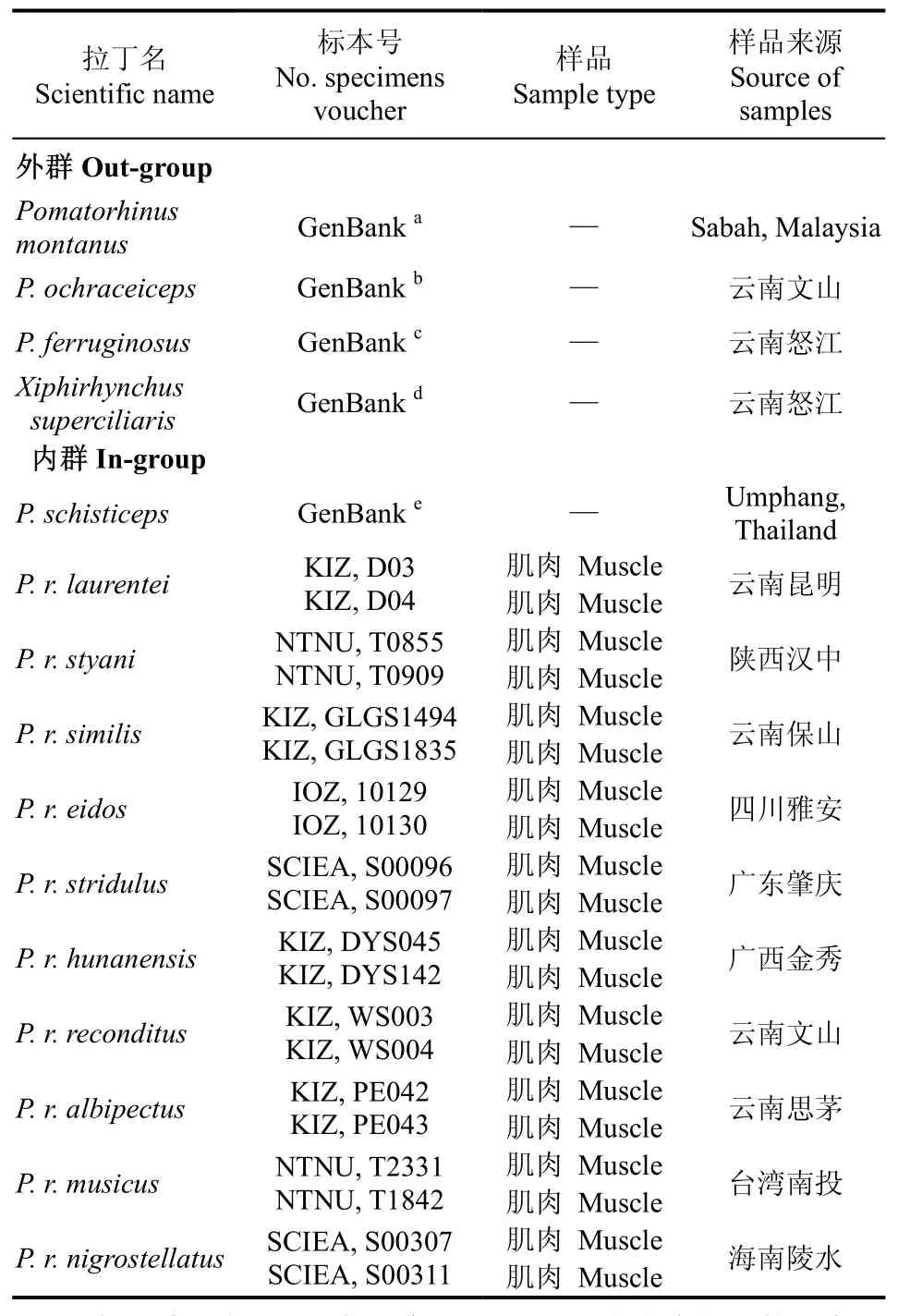
表1 本研究中的样品及其来源Tab. 1 Sample species and source in this study
1.2 目的基因的扩增及序列测定
分别使用两对引物(表2)对线粒体Cyt b和ND2基因进行扩增, PCR扩增产物测序时, 使用同样的引物。PCR反应体系(50 µL体系)为:双蒸水38.5 µL,10×PCR Buffer 5 µL, dNTP 3 µL(2.5 mmol/L), 小牛血清蛋白(bovine serum albumin, BSA)2 µL, 正反引物各1 µL(10 m mol/L), Taq DNA聚合酶0.25 µL,模板DNA 1 µL。PCR反应条件为:94 ℃预变性5 min; 以94 ℃变性30s 、55 ℃退火40 s、72 ℃延伸1 min为1个循环, 共进行35个循环; 循环结束后72 ℃延伸10 min。PCR产物用1.5%琼脂糖凝胶电泳检测后送交测序公司进行双向测序。

表2 本研究使用的引物列表Tab. 2 Primers used in this study
1.3 数据分析
测序得到的序列片段使用 DNASTAR软件包(DNASTAR Inc, Madison, WI)中的SeqMan程序进行正反向拼接并校正。拼接后的序列在 Clustal X 1.83(Thompson et al, 1997)中使用默认参数进行对位排列。遗传距离在MEGA 4.0(Tamura et al, 2007)中计算。
使用PAUP* 4.10b (Swofford, 1999)、Phyml 3.0(Guindon & Gascuel, 2003)和MrBayes 3.1.2(Huelsenbeck & Ronquist, 2001)分别构建最大简约树(MP树)、最大似然树(ML树)和贝叶斯推断树(BI树)。MP树是采用启发式搜索(heuristic research), 分支交换算法是使用树二等分再连接法(treebisection-reconnection), 各分支节点的可靠性使用1 000次重复的自展重抽样值——Bootstrap support,BS (Felsenstein, 1985)进行评估; 构建ML和BI树之前, 在 Modeltest 3.7 (Posada & Crandall, 1998)中进行合并数据集的模型检测, 根据赤池信息量准则(Akaike Information Criterion, AIC)选择最适核酸置换模型为 GTR+G (-lnL=6705.8291,K=9, AIC=13429.6582)。ML树在Phyml 3.0中计算, 碱基频率在最大似然框架(the maximum likelihood framework)下估计, 分支交换算法设为 SPR(subtree pruningregrafting), 树的搜索完成后也采用 1 000次的自展重抽样值——Bootstrap support, BS (Felsenstein,1985)评估各分支节点的可靠性, 其余选项选用系统默认参数; BI分析采用与 ML相同的模型, 在MrBayes 3.1.2中同时运行2个独立的由4条链组成的Metropolis-coupled Markov Chain Monte Carlo程序, 每个程序运行500万 代, 每隔100代进行一次抽样, 分支置信度用后验概率(Posterior probability,PP) 表示。
2 结 果
测序获得全部20个样本的完整Cyt b和ND2基因(Cyt b:1 143 bp;ND2: 1 041 bp)。在SeqMan程序中使用脊椎动物线粒体密码子将基因序列翻译成氨基酸序列, 在氨基酸序列中未发现终止密码子和空位。因此, 得到的Cyt b和ND2基因全长序列均应来自线粒体DNA, 而非核假基因。
2.1 系统发育分析
使用MP法、ML法和BI法三种方法构建的系统发育树的拓扑结构完全一致(图 1), 且大部分节点都得到了较高的支持率(BS>70%, PP>0.95)。
MP树、ML树和BI树均显示棕颈钩嘴鹛不是单系群, 灰头钩嘴鹛位于其系统发育树的内部, 并以较高的支持率(MP BS=75, ML BS=86, BI PP=0.98)与云南地区的棕颈钩嘴鹛聚合为一支。同时, 系统树也显示棕颈钩嘴鹛各个亚种可稳定地分为3个支序, 即支序A(Clade A)、支序B(Clade B)和支序C(Clade C)。

图1 基于线粒体DNA序列合并数据集构建的50% MP一致树Fig. 1 50% MP tree based on the concatenated dataset of mitochondrial DNA sequence
Clade A (MP BS=85, ML BS=81, BI PP=1.00)包括P. r. albipectus、P. r. similis和P. r. laurentei, 这3个亚种在国内均分布在云南。值得注意的是P. r.laurentei的两个样本并没有聚为一支, 其中一个样本聚合在P. r. similis内部(MP BS=96, ML BS=97, BI PP=1.00), 再共同与P. r. albipectus聚为姐妹群(MP BS=94, ML BS=95, BI PP=1.00)。
Clade B (MP BS=62, ML BS=75, BI PP=0.81)由P. r. musicua、P. r. nigrostellatus、P. r. stridulus和一个P. r. reconditus样本构成, 主要分布于广东、福建、江西、浙江、台湾、海南等地。比较意外的是分布于云南东南部的P. r. reconditus的一个样本也位于Clade B中, 并与P. r. stridulus聚为姐妹群, 但是支持率不高(MP BS=61, ML BS=62, BI PP=0.74)。
Clade C (MP BS=99, ML BS=94, BI PP=0.97)包括P. r. eidos,P. r. hunanensis,P. r. styani和P. r.reconditus的另一个样本, 分布范围集中在四川、湖南、湖北、甘肃、陕西、河南、江苏、安徽、广西、贵州等地。同Clade B一样, Clade C中也聚合了P. r.reconditus的一个样本, 但因支持率较低, 未能解决其在Clade C中的具体位置。
2.2 遗传距离
在MEGA 4.0中采用K2P(Kimula 2- Parameter)模型获得未经校正的遗传距离。计算内外群间的平均遗传距离时以最近外群P. montanus为代表, 所得平均遗传距离为9.0%; 内群中3个支序之间的平均遗传距离分别为 Clade A/Clade B=3.5%、Clade A/Clade C=2.4%和Clade B/Clade C=3.6%; 各支序内部样本间的遗传距离分别为0.8%、1.9%和0.5%;灰头钩嘴鹛与各支序(Clade A至C)之间的平均遗传距离分别为1.8%、3.5%和2.3%。
3 讨 论
研究结果显示,棕颈钩嘴鹛不是单系群, 灰头钩嘴鹛位于其系统发育树的内部; 棕颈钩嘴鹛可分为明显的3个支序, 且各支序内部的遗传距离(0.8%,1.9%, 0.5%)较之各支序之间的遗传距离(3.5%,2.4%, 3.6%)要小得多; 同时P. r. musicus和P. r.nigrostellatus也均位于一个支序的内部。以上结果既不同于Ramsay (1878)将棕颈钩嘴鹛划分为3个种, 也不同于Collar (2006)和Collar & Robson (2007)划分为两个种的建议。
3.1 灰头钩嘴鹛与棕颈钩嘴鹛的关系
灰头钩嘴鹛位于系统发育树的内部, 与棕颈钩嘴鹛P. r. albipectus、P. r. similis和P. r. laurentei的亲缘关系非常近(遗传距离为 1.8%), 比棕颈钩嘴鹛3个支序之间2.4%~3.6%的差异程度还要低。从形态上看, 灰头钩嘴鹛与棕颈钩嘴鹛也非常相似, 两者仅以胸部有无纵纹相区别。已有研究表明, 灰头钩嘴鹛与棕颈钩嘴鹛在老挝中、北部存在杂交现象(Delacour & Greenway, 1940; Duckworth et al,1998)。本文中的灰头钩嘴鹛样本来自临近泰缅边界的翁旁(Umphang), 与已知的杂交带相距较远, 在形态特征上亦为典型的灰头钩嘴鹛, 但仍然与云南的棕颈钩嘴鹛表现出较近的亲缘关系。因此, 两者的系统发育关系, 尚需扩大取样进行进一步的研究。
3.2 P. r. musicus和P. r. nigrostellatus的分类学地位
P. r. musicus与P. r. nigrostellatus均位于Clade B中, 并没有分别构成与其它亚种相对独立的支序。P. r. musicus同CladeA/B/C之间的遗传差异分别为 3.3%、3.5%和 3.5%, 远小于内外群的种间差异 9.0%。依据系统发育种的定义, 如果承认P. r.musicus为独立种, 则应将棕颈钩嘴鹛重新分割为至少4个种; 如果承认P. r. nigrostellatus为独立种,则更应将棕颈钩嘴鹛细分为 5个以上的种, 而非Ramsay(1878)所划分的3个种, 也非Collar (2006)、Collar & Robson (2007)所提议的2个种。因此, 本文不完全支持Ramsay (1878)、Collar (2006)和Collar& Robson (2007)对棕颈钩嘴鹛的分类意见。
3.3 P. r. laurentei和P. r. styani的分类地位
Cheng et al (1987)认为P. r. laurentei是P. r.reconditus的同物异名, 应将P. r. laurentei归为P. r.reconditus; Yang & Yang (2004)更是将其归为P. r.reconditus、P. r. albipectus和P. r. reconditus3亚种的居间类群。本文结果显示,P. r. laurentei与P. r.reconditus分别位于不同的支序中(Clade A与Clade B、C), 两者的遗传距离为 2.7%, 亲缘关系较远。通过查看中国科学院昆明动物研究所的馆藏标本,发现P. r. laurentei的背部和胸纹均呈橄榄褐色, 上嘴几乎为黄色, 形态特征与P. r. similis比较接近;而P. r. reconditus的背部和胸纹均呈栗色, 上嘴基部为黑色, 与P. r. laurentei有明显区别。因此, 不支持Cheng et al (1987)的观点。
从发育树上还可以看出,P. r. laurentei的2个样本并未单独聚为一支, 一个样本位于Clade A的根部(MP BS=85, ML BS=81, BI PP=1.00); 而另一个样本则位于Clade A的内部并与P. r. similis聚合(MP BS=96, ML BS=97, BI PP=1.00), 显示其有一定的分化, 或者是否如Yang & Yang (2004)所推测的为杂交类群, 需进一步的研究证明。
P. r. styani长期以来一直作为P. r. stridulus的同物异名(Diegnan, 1964; Dickinson, 2003), 但Cheng et al (1987)和Collar & Robson (2007)认为两者区别明显, 应当作为独立的亚种。本文的结果显示,P. r.styani和P. r. stridulus分别位于不同的支序中, 两者间的遗传差异为 4.0%, 与各支序之间的差异程度相当; 从形态上来看,P. r. styani的背部及胸纹均呈橄榄褐色, 而P. r. stridulus的背部及胸纹均呈栗色,区别比较明显; 另外,P. r. styani主要分布在长江流域, 而P. r. stridulus集中在东南沿海地区, 两者分布区基本无重叠。因此, 本文支持Cheng et al(1987)和Collar & Robson(2007)的观点, 认为P. r. styani是独立的亚种。
3.4 棕颈钩嘴鹛亚种形态与发育关系
从构建的系统发育树看, 棕颈钩嘴鹛的3个支序在地理分布上明显可分为3个地理类群, 即中国西南部类群Clade A、中国东南部类群Clade B和中国中部类群Clade C, 该结果与灰眶雀鹛(Zou et al,2007; Song et al, 2009)的地理类群分布基本一致,揭示这3个地区在生物分布与系统发生中可能存在更深入的关系。
Ramsay (1878)曾指出, David & Oustalet (1877)已发现棕颈钩嘴鹛在羽色上有相当大的变异。经比对馆藏标本, 发现棕颈钩嘴鹛的3个支序在形态上有一定的重叠, 不具有明显的可区分特征, 同一个支序中亚种间的羽色变化也很大, 尤其以胸纹为甚,如在Clade A中,P. r. albipectus的胸纹几乎为白色,有时仅具细微淡黄色, 但P. r. similis却为较浅的橄榄褐色胸纹; 在Clade B中,P. r. stridulus的胸纹为深栗色, 但P. r. nigrostellatus和P. r. musicus却为黑色斑块状胸纹; Clade C中,P. r. hunanensis的胸纹是栗色, 而P. r. styani的胸纹则呈橄榄褐色。由此可见,对棕颈钩嘴鹛来说, 亚种间的形态特征与其地理分布和发育关系并不相对应。这与在美国布莱克沃特国家野生动物保护区(Blackwater National Wildlife Refuge, NWR)越冬的来自不同繁殖地的两群雪雁(Chen caerulescens caerulescens)一样, 虽然它们形态上有明显的区别, 却不存在遗传差异(Humphries et al, 2009); 同样, 山椒鸟属(Pericrocotus)鸟类羽色和体型相近的种,并不意味着具有更近的亲缘关系(Jønsson et al, 2010)的研究结果也与本文相似。这暗示着鸟类的羽色变化较其遗传结构变化更为容易,邻近区域中的种群可能由于环境压力或性选择差异而分化出不同的羽色, 而不同的地理类群又可能因趋同进化形成近似的羽色。因此, 不能仅依据体色的变化对其进行分类或推测其亲缘关系。
在鸟类中, 大多数姐妹种的Cyt b基因的遗传差异在 2%以上(Johns & Avise, 1998); Hebert et al(2004)的研究也表明, 鸟类中同种个体间COI基因的平均差异是0.29%, 而在同属的种间为7.05%。从本文构建的系统发育结果来看, 棕颈钩嘴鹛已经开始逐渐分化为几个稳定的支序, 但支序之间的遗传差异在 2.4%~3.6%之间, 远小于同外群的平均种间差异(9.0%)。同时,3种方法构建的系统发育树均稳定地将灰头钩嘴鹛聚合在棕颈钩嘴鹛的内部, 亦将P. r. reconditus划分到了不同的类群中(CladeB和C)。中国科学院昆明动物研究所馆藏标本中, 云南昭通和文山采自同一地点的P. r. reconditus标本在胸纹上都存在橄榄褐色与栗色两种形态, 这一结果与Zink (2004)指出的传统亚种划分并不能完全体现物种内部正确的遗传结构和地理格局分化的观点相符。本研究的结果暗示棕颈钩嘴鹛各亚种间的系统发育关系是非常复杂的, 有必要进行更加深入和系统的分析。
Cheng TH. 1962. A systematic review ofPomatorhinusheretofore recorded from China [J].Acta Zool Sin, 14(2): 197-218. [郑作新. 1962. 中国钩嘴鹛属的系统发育研究. 动物学报, 14(2):197-218.]
Cheng TH, Long ZX, Zheng BL. 1987. Fauna sinica: AVES Vol. 11:Passeriformes, Muscicapidae Ⅱ, Timaliinae [M]. Beijing: Science Press.[郑作新, 龙泽虞, 郑宝赉. 1987. 中国动物志鸟纲第十一卷雀形目鹟科: Ⅱ 画眉亚科[M]. 北京: 科学出版社.]
Cibois A. 2003. Mitochondrial DNA phylogeny of Babblers (Timaliidae)[J].Auk, 120(1): 35-54.
Cibois A, Pasquet E, Schulenberg TS. 1999. Molecular systematics of the Malagasy babblers (Passeriformes: Timaliidae) and Warblers(Passeriformes: Sylviidae), based on cytochromeband 16S rRNA sequences [J].Mol Phylogenet Evol, 13(3): 581-595.
Cicero C, Johnson NK. 2001. Higher-level phylogeny of new world vireos(Aves: Vireonidae) based on sequences of multiple mitochondrial DNA genes [J].Mol Phylogenet Evol, 20(1): 27-40.
Clements JF. 2000. Birds of the World: A Checklist [M]. Robertbridge, East Sussex: Pica Press.
Collar NJ. 2006. A partial revision of the Asian babblers (Timaliidae)[J].Forktail, 22: 85-112.
Collar NJ, Robson C. 2007. Family Timaliidae (Babblers) [M]// Del Hoyo J, Elliott A, Christie DA. Handbook of the Birds of the World, Vol.12: Picathartes to Tits and Chickadees. Barcelona: Lynx Edicions,70-291.
Deignan HG. 1964. Timaliinae, Panurinae, Picathartinae [M]//Mayr E,Paynter RA. A Check-list of Birds of the World: A Continuation of the Works of James L Peters, 10(2): Order Passeriformes. Cambridge.Massachusetts: Museum of Comparative Zoology.
Delacour J, Greenway JC. 1940. Notes critiques sur certain oiseaux indochinois [J].L’oiseau RFO, 10: 60-77.
Dickinson EC. 2003. The Howard and Moore Complete Checklist of the Birds of the World [M]. 3rd ed. Princeton: Princeton University.
Dong F, Li SH, Yang XJ. 2010a. Molecular systematics and diversification of the Asian scimitar babblers (Timaliidae, Aves) based on mitochondrial and nuclear DNA sequences [J].Mol Phylogenet Evol,57: 1268-1275.
Dong F, Wu F, Liu LM, Yang XJ. 2010b. Molecular phylogeny of the Barwings (Aves: Timaliidae:Actinodura), a paraphyletic group, and its taxonomic implications [J].Zool Stud, 49(5): 703-709.
Duckworth JW, Tizard RJ, Timmins RJ, Thewlis RM, Robichaud WG,Evans TD. 1998. Birds records from Laos, October 1994 - August 1995[J].Forktail, 13: 33-68.
Felsenstein J. 1985. Confidence limits on phylogenies: an approach using the bootstrap [J].Evolution, 39: 783-791.
Guindon S, Gascuel O. 2003. A simple, fast and accurate algorithm to estimate large phylogenies by maximum likelihood [J].Syst Biol, 52(5):696-704.
Hebert PDN, Stoeckle MY, Zemlak TS, Francis CM. 2004. Identification of birds through DNA barcodes [J].PLoS Biol, 2(10): 1657-1663.
Huelsenbeck JP, Ronquist F. 2001. MRBAYES: Bayesian inference of phylogenetic trees [J].Bioinformatics, 17: 754-755.
Humphries EM, Peters JL, Jónsson JE, Stone R, Afton AD, Omland KE.2009. Genetic differentiation between sympatric and allopatric wintering populations of snow geese [J].Wilson J Ornith, 121(4): 730-738.
Johns GC, Avise JC. 1998. A comparative summary of genetic distances in the vertebrates from the mitochondrial cytochromebGene [J].Mol Biol Evol, 15(11): 1481-1490.
Jønsson KA, Irestedt M, Ericson PGP, Fjeldså J. 2010. A molecular phylogeny of minivets (Passeriformes: Campephagidae:Pericrocotus):implications for biogeography and convergent plumage evolution [J].Zool Scripta, 39: 1-8.
Pasquet E, Bourdon E, Kalyakin MV, Cibois A. 2006. The fulvettas (Alcippe,Timaliidae, Aves): a polyphyletic group [J].Zool Scripta, 35: 559-566.
Posada D, Crandall KA. 1998. Modeltest: testing the model of DNA substitution [J].Bioinformatics, 14: 817-818.
Ramsay LW. 1878. A synopsis of the genusPomatorhinus[J].IBIS,20(2):129-145.
Reddy S, Moyle RG. 2011. Systematics of the scimitar babblers(Pomatorhinus: Timaliidae): phylogeny, biogeography, and specieslimits of four species complexes [J].Biol J Linn Soc, 102: 846-869.
Sibley CG, Monroe BL. 1990. Distribution and Taxonomy of Birds of the World [M]. New Haven: Yale University.
Song G, Qu YH, Yin ZH, Li SH, Liu NF, Lei FM. 2009. Phylogeography of theAlcippe morrisonia(Aves: Timaliidae): long population history beyond late pleistocene glaciations [J].BMC Evol Biol, 9: 143: 1-11.
Swofford DL. 1999. PAUP*: Phylogenetic Analysis Using Parsimony (*and other methods), Version 4.0 [M]. Sunderland, MA: Sinauer Associates.
Tamura K, Dudley J, Nei M, Kumar S. 2007. MEGA 4: Molecular evolutionary genetics analysis (MEGA) software version 4.0 [J].MolBiol Evol, 24(8): 1596-1599.
Thompson JD, Gibson TJ, Plewniak F, Jeanmougin F, Higgins DG. 1997.The clustal x windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools [J].Nucleic Acids Res, 25(24): 4876-4882.
Yang L, Yang XJ. 2004. The avifauna of Yunnan China. Vol. II:Passeriformes [M]. Kunming: Yunnan Science and Technology Press.[杨岚, 杨晓君. 2004. 云南鸟类志下卷: 雀形目. 昆明: 云南科技出版社.]
Zhang SX, Yang L, Yang XJ, Yang JX. 2007. Molecular phylogeny of the yuhinas (Sylviidae:Yuhina): a paraphyletic group of babblers includingZosteropsand PhilippineStachyris[J].J Ornithol, 148: 417-426.
Zink RM. 2004. The role of subspecies in obscuring avian biological diversity and misleading conservation policy [J].Proc R Soc Lond B,271: 561-564.
Zou FS, Lim HC, Marks BD, Moyle RG, Sheldon FH. 2007. Molecular phylogenetic analysis of the Grey-cheeked Fulvetta (Alcippe morrisonia) of China and Indochina: A case of remarkable genetic divergence in a “species” [J].Mol Phylogenet Evol, 44: 165-174.
Preliminary discussion on the phylogenetic and taxonomic relationship ofPomatorhinus ruficollis
SONG Rui1,5, DONG Feng1,5, LIU Lu-Ming1, WU Fei1, WANG Kai1, ZOU Fa-Sheng2,LEI Fu-Min3, LI Shou-Hsien4,*, YANG Xiao-Jun1,*
(1.Kunming Institute of Zoology, the Chinese Academy of Sciences, Kunming Yunnan650223,China; 2.South China Institute of Endangered Animals,Guangzhou510260,China; 3.Institute of Zoology, Chinese Academy of Sciences, Beijing100101,China; 4.Department of Life Science, National
Taiwan Normal University, Taipei116,China;5.Graduate University of the Chinese Academy of Sciences,Beijing100049,China)
The phylogenetic relationships among subspecies ofPomatorhinus ruficollisare not clear. In this paper we sequenced two mitochondrial genes (Cyt bandND2, in total 2 184 bp) and examined twenty individuals representing ten of the eleven subspecies ofP. ruficollisin China. Molecular phylogenetic trees were reconstructed using maximum parsimony, maximum likelihood and Bayesian inference methods, withP. ruficollisandP. schisticepsas in-groups,P.montanus,P. ochraceiceps,P. ferruginosusandXiphirhynchus superciliarisas out-groups. Results indicated thatP.ruficolliswas paraphyletic, in whichP. schisticepswas embedded, and was divided into three phylogeographic groups:southwestern China group, southeastern China group and central China group. Our results also suggested discordances between phylogenetic relationships and morphological characteristics, and clarified some intraspecific taxonomic debates.
Pomatorhinus ruficollis;Cyt b;ND2; Phylogeny
Q959.739; Q959.739.09
A
0254-5853-(2011)03-0241-07
10.3724/SP.J.1141.2011.03241
2010-12-06;接受日期:2011-02-28
国家自然科学基金 (30870279); 国家自然科学基金国际(地区)合作与交流项目 (31010103901); 澜沧江中下游与大香格里拉地区科学考察项目 (2008FY110300); 中国科学院对外合作重点项目 (GJHZ200822)
∗通讯作者(Corresponding author),E-mail: t43028@gmail.com, yangxj@mail.kiz.ac.cn
女, 硕士研究生, 主要从事鸟类分子系统发育研究
猜你喜欢
杂志排行
Zoological Research的其它文章
- 白介素1β对大鼠皮层神经元钠电流的急性作用
- 尼罗罗非鱼Orexin前体基因的克隆、组织分布及其在摄食调控中的表达
- Effects of urethane on the response properties of visual cortical neurons in young adult and old cats
- Phylogenetic evaluation of the taxonomic status of Papilio maackii and P. syfanius (Lepidoptera: Papilionidae)
- Genetic differentiations between randomly and selectively bred pig populations in Yunnan, China
- 阿魏酸对脂多糖诱导的小鼠小胶质细胞炎性反应的抑制作用
