基于环境DNA 技术的珠江中下游鱼类多样性初步研究
2024-03-06朱书礼陈蔚涛夏雨果杨计平李跃飞
朱书礼,陈蔚涛,武 智,夏雨果,杨计平,李跃飞,李 捷
中国水产科学研究院珠江水产研究所/农业农村部珠江中下游渔业资源环境科学观测实验站/国家渔业资源环境广州观测实验站,广东 广州 510380
鱼类是水生态系统的重要组成部分,不仅为人类提供了丰富的食物蛋白,也维护着水生态系统的稳定[1-2]。然而,受栖息地退化、过度捕捞、水污染、气候变化和外来物种入侵等因素的影响[1,3],鱼类生物多样性和渔业资源锐减,进而威胁水生态系统功能、渔业经济和人类生存[4-5]。对鱼类物种分布和种群动态的准确、及时地监测与评估显得尤为迫切,同时也可为渔业资源相关保护和管理决策的制定提供科学指导。利用传统鱼类监测方法,如电捕、网捕、陷阱诱捕等[6],虽可获得鱼类种类和种群的关键信息,但存在成本高、费时费力、调查范围有限[7]、破坏性大、稀有物种捕获率低[7-8]、形态鉴别困难[9]等问题。传统调查方法已无法满足及时、全面和准确的鱼类监测和评估要求。因此,迫切需要更准确、有效和经济的调查方法,以弥补渔业资源监测和管理方面的不足。
近年来,环境DNA (Environmental DNA,eDNA) 技术作为一种新兴的检测评估方法,被认为是生物多样性研究中发展和普及最快的方法之一[10-11]。eDNA 是指存在于任何环境样品 (如水、冰、土壤、沉积物、生物膜和空气) 中的DNA 的总和[7]。对于鱼类来说,eDNA 通常是鱼类体细胞释放到水体中的胞内遗传物质和细胞结构裂解或死亡后释放到水体中的胞外DNA[12-13]。eDNA 技术通过直接提取环境样品中的DNA,利用特异性引物进行PCR 扩增和高通量测序,对环境样本中存在的多个目标物种进行识别[14]。基于eDNA 技术的调查方法具有灵敏度高、分辨率高、效率高、成本低和无损伤等优点,是解决渔业资源调查和管理难题的有效方法[15-16]。eDNA 技术首次在鱼类研究中的应用是关于特定物种的监测[17]和鱼类多样性研究[18]。目前,eDNA 技术在渔业领域的应用发展迅速,已广泛应用于渔业管理、鱼类种类及多样性监测、资源量评估和种群分布等[8,19-21]。
珠江是中国南方第一大河,全长2 214 km,流域面积45.37 万km2,年径流量约3 300 亿m3[22]。珠江地处热带、亚热带季风气候区,气候温和多雨,地形地貌复杂多样[23]。独特的生境孕育了珠江丰富的鱼类资源,鱼类物种多样性在全球大河流域中排名第四,特有种类数位居第二[24]。据统计,珠江水系记录鱼类达682 种,其中特有种242 种(亚种),种类数和特有种数均居中国各流域之首。近几十年来,由于人类活动影响加剧,导致珠江生态环境退化、鱼类适宜栖息地丧失和水生生物入侵等,造成鱼类种群衰减、物种丧失和鱼类群落结构与功能改变[25-27]。为此,政府和管理部门实施了多项渔业资源养护措施,包括建立禁渔期制度、开展增殖放流活动和划定水产种质资源保护区等,以期改善珠江流域鱼类生物多样性和渔业资源现状[1,28-29]。
珠江鱼类生物多样性和渔业资源保护亟需构建快速有效且环境友好的监测体系,为渔业保护和管理决策的制定及实施提供科学依据。本研究首次利用eDNA 技术对珠江中下游鱼类多样性进行研究,旨在探讨该技术对检测珠江中下游鱼类生物多样性的适用性,为珠江鱼类生物多样性的监测及保护提供新的技术支持。
1 材料与方法
1.1 样品采集
本次调查于2023 年2 月8—12 日进行,在珠江中下游选取了桂平G P (1 1 0°0 7'3 4.4 5''E,23°25'11.92''N)、藤县TX (110°50'5.06''E,23°26'47.19''N)、封开FK (111°30'41.25''E,23°23'35.73''N)、德庆DQ (111°49'53.35''E,23°08'24.83''N)、肇庆ZQ (112°24'48.24''E,23°07'58.23''N) 和九江JJ (112°56'40.33''E,22°51'22.53''N) 6 个采样点 (图1)。每个采样点采集3 个重复样品,每个样品用采水器从表层水体采集2 L 水样置于聚乙烯瓶中。采样人员佩戴一次性手套,采水器和采样瓶在使用前均经10% (w)的次氯酸钠消毒。所有样品均在24 h 内使用0.45 μm 混合纤维素滤膜 (Whatman,英国) 进行真空抽滤。为评估是否存在外源DNA 污染,每次过滤设置1 个阴性对照。每份样品过滤后,将滤膜置于4.5 mL无酶冻存管中于液氮中冷冻保存,并对过滤器材进行消毒和冲洗,避免样品间交叉污染。
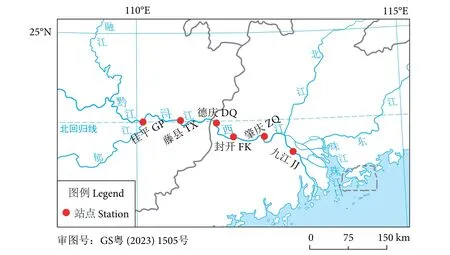
图1 调查站点示意图Fig. 1 Sampling stations in this study
1.2 eDNA 提取
使用PowerWater DNA Isolation Kits (Qiagen 公司) 按照试剂盒说明书提取滤膜中的DNA,用1%(w) 琼脂糖凝胶电泳检测提取DNA 的质量。每份样品独立提取,并使用空白滤膜设置为阴性对照。将提取的DNA 样品快速置于−20 ℃冷冻保存,直至PCR 扩增。
1.3 遗传标记扩增及高通量测序
本研究使用鱼类eDNA 宏条形码分析常用引物mlCOlintF (5'-GGWACWGGWTGAACWGTWTAYCCYCC-3') 和jgHCO219 (5'-TAIACYTCIGGRTGICCRAARAAYCA-3') 扩增线粒体COI基因,扩增序列长度约313 bp[30]。20 μL PCR 扩增体系包括:2~5 μL 模板DNA (10 ng·μL−1)、正反向引物各0.8 μL (5 μmol·L−1)、2 μL dNTPs、4 μL 5×FastPfu 缓冲液、0.4 μL FastPfu 聚合酶,最后用ddH2O 将体系补至20 μL。PCR 反应程序为:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸45 s,35 个循环;72 ℃延伸10 min。每个样品每次PCR 反应均使用ddH2O 为模板作阴性对照。每个样品进行3 次重复扩增,并将同一样品的扩增产物混合,使用2% (w) 琼脂糖凝胶电泳检测PCR 产物。所有阴性对照均无目的条带,表明无外源鱼类DNA 污染;6 个采样点的样品均得到可检测的PCR 产物。PCR 产物胶回收后送至上海凌恩生物科技有限公司通过Illumina NovaSeq 6000 测序平台进行高通量测序。
1.4 数据库构建
2013—2019 年,在珠江中下游采集78 种共384 尾鱼类样本,利用鱼类通用引物扩增603 bp 线粒体COI基因序列[31]。此外,从NCBI (https://www.ncbi.nlm.nih.gov/) 数据库下载12 种鱼类的COI基因序列进行补充,分别是叉尾斗鱼 (Macropodusopercularis)、大鳞副泥鳅 (Paramisgurnus dabryanus)、福建纹胸鮡 (Glyptothoraxfukiensis)、虹彩光唇鱼 (Acrossocheilusiridescens)、黑鳍鳈 (Sarcocheilichthysnigripinnis)、黄鳝 (Monopterus albus)、横纹南鳅 (Schisturafasciolata)、金黄舌鰕虎鱼 (Glossogobiusaureus)、美丽沙鳅 (Botia pulchra)、攀鲈 (Anabastestudineus)、三线舌鳎(Cynoglossustrigrammus) 和云斑尖塘鳢 (Oxyeleotris marmoratus)。本研究利用这90 种鱼类的COI基因序列构建珠江中下游鱼类DNA 条形码数据库。利用MEGA-X 软件,基于Kimura2-parameter (K2P)模型对种间遗传距离 (Interspecific distance) 和种内遗传距离 (Intraspecific distance) 进行计算,并分析条形码间隙 (Barcode gap),以证实条形码数据库的可用性。此外,为了更直观地理解物种聚类模式,在MEGA-X 软件中,基于K2P 模型,自展值(Bootstrap) 设置为1 000,构建Neighbor-Joining(NJ) 系统发育树。
1.5 数据分析
测序序列经过质控过滤、拼接后获得各样品的优质序列。序列按照相似性≥97%进行OTU (Operational taxonomic unit) 聚类分析,然后将OTU代表序列与自建的数据库进行比对、分类注释,并得到相应的OTU 丰度表[32]。本研究以OTU 聚类与注释结果为基础,进行物种组成和Alpha 多样性分析。将OTU 序列与数据库比对确定鱼类物种信息,阈值条件为Identity 值≥97%,E-value<10−5。鱼类分类学信息参考《珠江鱼类志》[33]《中国内陆鱼类物种与分布》[34]和中国生物物种名录(2022 版)。Alpha 多样性分析选取Chao1 指数(Chao1 index)、香农指数 (Shannon index)、辛普森指数 (Simpson index) 及覆盖度 (Coverage) 指数进行群落丰富度、多样性及覆盖度分析[32]。利用R 软件vegan 包,基于Bray-Curtis 距离矩阵进行主坐标分析 (Principal coordinates analysis, PCoA)[35],探讨不同采样点样本间群落组成空间分布差异。
2 结果
2.1 条形码数据库
本研究构建的条形码数据库共463 条COI序列,包括90 种鱼类,隶属于17 目32 科75 属。K2P 遗传距离模型计算结果显示,基于数据库COI序列的种内遗传距离为0%~24.94%,平均值为0.95%;种间遗传距离为1.27%~34.93%,平均值为2 2.4 7%,种间平均遗传距离是种内的23.65 倍。条形码间隙分析显示,除了黄尾鲴,数据库中其他物种的最小种间距离均大于最大种内距离,具有条形码间隙 (图2)。基于COI序列构建的NJ 系统发育树见图3。

图2 数据库鱼类最大种内遗传距离与最小种间遗传距离比较Fig. 2 Maximum intraspecific distance compared with minimum interspecific distance for fishes in barcode library
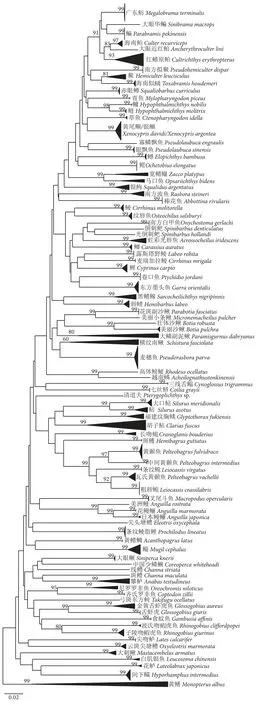
图3 基于COI 序列构建的NJ 系统发育树Fig. 3 Neighbor-joining phylogenetic tree based on COI sequence
2.2 鱼类物种组成
从6 个采样点共获得13 025 个OTU,其中九江的OTU 数量最多 (6 476 个),肇庆最少 (3 829个),其他采样点分别为德庆5 076 个,封开5 012 个,桂平4 244 个,藤县3 889 个;6 个采样点共有的OTU 为724 个。对OTU 进行注释,共获得已知鱼类物种序列2 6 1 3 条,检出鱼类30 种,隶属于4 目10 科27 属,其中土著鱼类26 种,外来种4 种,各采样点的物种及序列数见表1。检出鱼类种类最多的为肇庆 (18 种),其他分别为桂平13 种、藤县11 种、封开10 种、德庆8 种和九江14 种。鱼类检测结果中鲤形目鲤科鱼类最多 (17 种),占鱼类物种总数的56.67%;鲈形目和鲇形目分别为7 和3 种。基于各采样点的序列丰度,物种种类和相对序列丰度组成情况见图4。由图4 可知,eDNA 所检测序列数最多的7 个物种依次为子陵吻鰕虎鱼 (Rhinogobiusgiurinus,27.75%)、瓦氏黄颡鱼 (Pelteobagrusvachellii,17.53%)、鲢 (Hypophthalmichthysmolitrix,11.71%)、尼罗罗非鱼 (Oceochromisnilotica,8.50%)、齐氏罗非鱼 (O.zillii, 7.27%)、南方波鱼(Rasborasteineri, 5.82%) 和鲤 (Cyprinuscarpio,3.41%);且除齐氏罗非鱼外,其他几种鱼类在各采样点均被检测到并显示出相对较高的序列丰度。
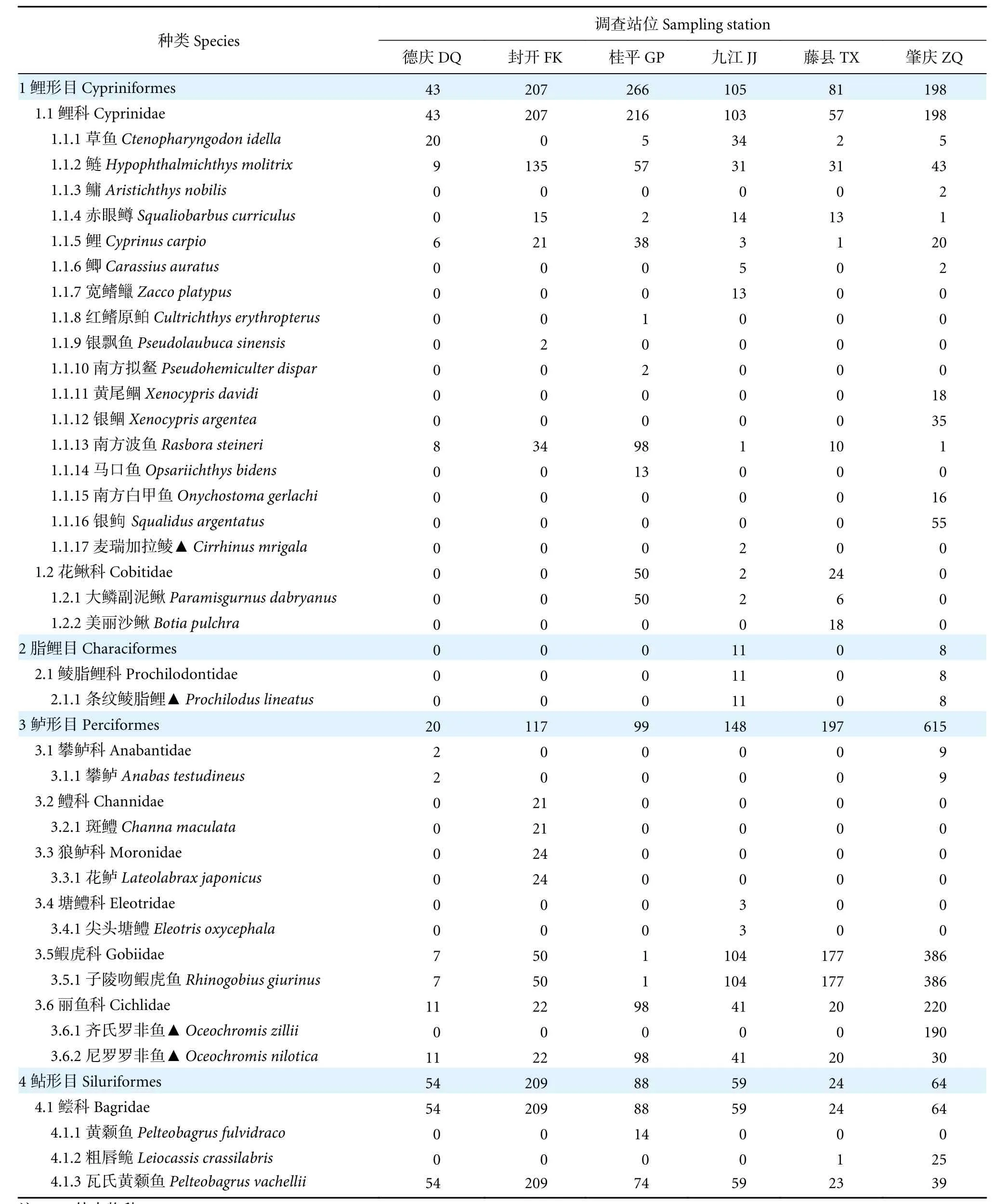
表1 各采样点检测物种序列数Table 1 Number of reads detected for each species at each sampling station

图4 各采样点鱼类物种组成Fig. 4 Composition of fish species at each sampling site
2.3 历史调查数据比较
根据珠江中下游鱼类调查文献[23,25,27,36-37],统计得出近十多年传统调查共捕获鱼类132 种 (未统计未鉴定种),隶属于13 目30 科 (附录A,详见http://dx.doi.org/10.12131/20230111 的资源附件)。本研究较传统调查资料新检出美丽沙鳅和齐氏罗非鱼2 种鱼类。
2.4 多样性分析
对珠江中下游鱼类群落进行Alpha 多样性分析,各样本的Chao1、Shannon 和Simpson 多样性指数及覆盖度Coverage 计算值见表2。各采样点中,Shannon 指数最高的为九江 (2.90),其次为桂平 (2.89),最低的为藤县 (2.15);Simpson 指数最高的为桂平 (0.84),其次为九江 (0.82),最低的为藤县 (0.63)。各个样本的Coverage 值范围为0.993 4~1.000 0,表明检测结果基本覆盖到了全部鱼类物种。
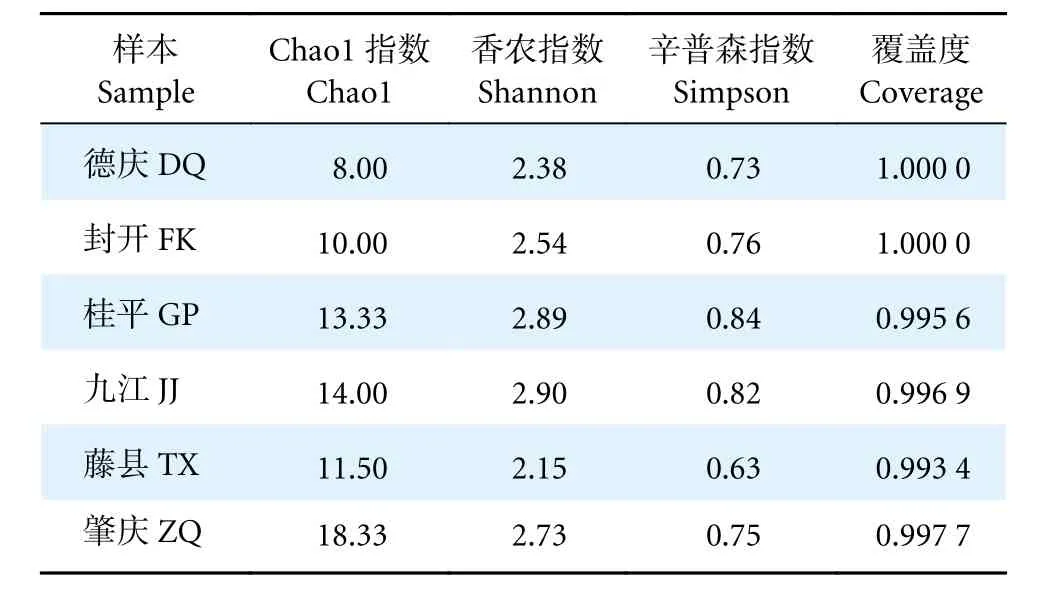
表2 各样本Alpha 多样性指数Table 2 Alpha diversity index of each sample
基于eDNA 检测的各采样点的序列丰度,采用Bray-Curtis 距离矩阵进行PCoA 分析 (图5),由图可知,桂平和封开站点距离较近,九江、藤县和肇庆站点距离较近,德庆站点与其他站点距离较远。表明桂平和封开站点具有相似的鱼类组成,九江、藤县和肇庆站点具有相似的鱼类组成,德庆站点具有与其他站点不同的鱼类组成。
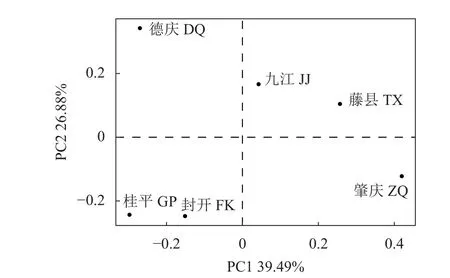
图5 eDNA 所检测鱼类的基于Bray-Curtis 距离矩阵的主坐标分析 (PCoA)Fig. 5 Principal coordinates based on Bray-Curtis distance matrix analysis of fish detected by environmental DNA
3 讨论
3.1 条形码数据库的适用性
目前COI 条形码已广泛应用于鱼类种类鉴定中。Hebert 等[38]提出了“10 倍规则”的标准用于区分物种,即满足种间平均遗传距离大于种内平均遗传距离的10 倍。蒋佩文等[39]在构建珠江河口鱼类COI 条形码数据库时,其种间与种内平均遗传距离比值为127.7,符合“10 倍规则”的标准,可实现珠江口鱼类物种的鉴定。郜星晨和姜伟[40]建立三峡鱼类COI 条形码数据库,其种间平均遗传距离为种内的9.38 倍,表明COI基因可以作为三峡库区常见鱼类鉴定的有效条形码基因。本研究中,C O I 序列种间平均遗传距离是种内的23.65 倍,也符合“10 倍规则”标准。此外,条形码间隙分析显示,除黄尾鲴外,其他种类均具有明显的条形码间隙。因此,COI 序列在本研究中可满足鱼类物种的鉴别需求。
3.2 基于eDNA 检测的鱼类组成
本研究首次使用eDNA 技术分析了珠江中下游鱼类多样性,共检测出鱼类30 种,其中鲤形目鲤科鱼类最多,占鱼类物种总数的56.67%,相较于传统调查方法[23,27],检出鱼类种类组成结构一致,均以鲤科鱼类为主,符合珠江水系鱼类区系组成特点[41]。传统调查方法研究显示,珠江中下游鱼类优势种包括广东鲂 (Megalobramaterminalis)、赤眼鳟 (Squaliobarbuscurriculus)、鲮 (Cirrhinus molitorella)、鲤、子陵吻鰕虎鱼、鲢、草鱼 (Ctenopharyngodonidella)、瓦氏黄颡鱼和尼罗罗非鱼等[23,27,36]。本研究的鱼类优势种为子陵吻鰕虎鱼、瓦氏黄颡鱼、鲢、尼罗罗非鱼、齐氏罗非鱼、南方波鱼和鲤,优势种类与传统调查研究结果基本一致。但部分传统调查中的优势种类在本研究中未检测到,如广东鲂和鲮。这可能是由于本研究所用COI基因通用引物对广东鲂和鲮的扩增效率低,在低浓度DNA 模板的条件下无法对目标基因序列进行扩增。其次,在设置调查站位时,综合考虑地理环境和有关鱼类生活习性,能够提高eDNA 的物种检测效率[32]。本研究设置站位数量有限,未能充分考虑站位环境和鱼类生活习性。广东鲂为江河洄游性鱼类,4—8 月为其产卵繁殖期,在西江的青皮塘和罗旁江段形成渔汛,珠江三角洲是其主要育肥和生长场所[42];珠江中下游鱼类优势种组成具有一定的季节差异,鲮主要是在秋季作为优势种[23],而本研究采样时间为春季。
已有研究表明,eDNA 方法检测到的鱼类物种丰富度与基于传统的调查方法相似或更高[43-44]。例如,利用eDNA 技术检测的鱼类种类数与使用刺网和电捕法在静态和激流水体中进行长期调查所检测的物种数相当[45]。但本研究检测到的鱼类物种数少于传统调查方法,李捷等[25]2005—2008 年调查鱼类97 种,与本研究检出相同鱼类24 种;李捷等[36]2006—2008 年调查鱼类81 种,与本研究检出相同鱼类24 种;李跃飞等[37]2006—2007 年调查鱼类75 种,与本研究检出相同鱼类18 种;帅方敏等[27]2015 年调查鱼类62 种 (不包括未鉴定种),与本研究检出相同鱼类20 种;张迎秋等[23]2016—2018 年调查鱼类96 种 (不包括未鉴定种),与本研究检出相同鱼类26 种。这可能由于本研究使用的COI 通用引物没有足够的特异性,在原核生物和非鱼类真核生物DNA 存在的情况下,引物对样品中的鱼类DNA 扩增效率不高。已有研究表明,对于水体中低浓度的环境DNA 模板,COI 引物对鱼类DNA 的扩增效率不高[46]。刘军等[47]研究发现,COI 引物对鱼类样品和环境DNA 样品均有较好的扩增效果,但测序结果显示COI 引物从环境DNA中扩增的基因片段属于细菌微生物的基因片段,而未扩增出鱼类基因片段。此外,物种多样性调查结果与采样频次和eDNA 数据库物种信息完整性有关。在生态环境的生物多样性调查中,特别是水生生态系统中,由于检测概率[48]、物种的稀有性[49]和采集强度[50]的影响,对物种多样性的评估可能受到限制,通过增加采样点和采样次数可以提高鱼类物种的检测效果[15]。基于eDNA 技术的生物评价的有效性依赖于序列参考数据库的完整性,数据库缺少目标物种序列时会导致假阴性检测[51]。
相较于珠江河口水域eDNA 研究,Zou 等[52]在珠江河口水域的南沙湿地利用eDNA 检测出35 种淡水鱼类,与本研究相同的鱼类有7 种:草鱼、鲢、赤眼鳟、鲤、南方白甲鱼 (Onychostomagerlachi)、斑鳢 (Channamaculata)和尼罗罗非鱼。这可能与研究的区域、选用的引物和比对数据库的不同有关。Zou 等[52]选用12S rRNA 基因区的通用引物,利用GenBank 数据库进行比对;本研究使用COI基因区的通用引物,比对自行构建的数据库。已有研究表明不同DNA 区域 (如COI和12S rRNA基因) 元条形码引物在鱼类多样性和物种分辨率方面存在差异[53-54]。
3.3 鱼类多样性分析
本研究中Chao1 指数范围为8.00~18.33,其中肇庆站点的值最高,表明该站点的群落丰富度最高。Shannon 和Simpson 指数可反映鱼类的多样性水平[55],本研究中Shannon 和Simpson 指数表明九江和桂平站点鱼类多样性最高。桂平站点位于郁江和黔江的交汇处,该江段为桂平东塔产卵场,是珠江流域最大的产卵场[56],水域环境复杂,是珠江众多珍稀特有鱼类的重要栖息地,因此具有较高的鱼类多样性。九江站点靠近珠江河口,受径流和潮汐相互作用,该水域咸淡水混合、物质交汇频繁、生产力丰富,是鱼类栖息、觅食和繁育的重要区域[57],因此鱼类生物多样性也较高。
鱼类群落结构的空间差异对于维持水生生态系统功能具有重要作用。在本研究中,基于环境DNA 检测的鱼类物种序列丰度,通过PCoA 分析显示桂平和封开站点具有相似的鱼类组成,藤县和九江站点具有相似的鱼类组成,但在地理位置上桂平和封开、藤县和九江站点相距较远。相反,地理位置较近的肇庆和德庆站点具有不同的鱼类组成。当然,仅基于本研究6 个站点的数据不能充分展示珠江中下游鱼类群落空间分布格局,如需充分展示该区域的鱼类空间分布,需要设置更加密集的站位和更高频次的调查研究。
3.4 入侵物种和特有鱼类
防止有害物种入侵、保护稀有濒危物种是物种和生态系统监管的重要内容;eDNA 技术由于其敏感性、快速性和特异性,因而在外来物种防控和珍稀濒危物种保护方面具有巨大的优势[20]。本研究中检测到4 种外来入侵鱼类,分别为麦瑞加拉鲮(Cirrhinusmrigala)、条纹鲮脂鲤 (Prochiloduslineatus)、齐氏罗非鱼和尼罗罗非鱼,且尼罗罗非鱼和齐氏罗非鱼已成为优势种类。罗非鱼主要是从养殖水体逃逸后,在珠江水系扩散并建群,目前已遍布整个流域并建立了自然群体[27,58-59]。珠江流域外来水生生物安全面临严重威胁,控制形势十分严峻。外来物种入侵是生物多样性丧失和全球同质化的主要原因之一[60],入侵物种会与土著种通过竞争成为优势种,从而对本地物种的种类和数量产生影响[61]。一旦外来物种入侵成功,不但控制成本高且根除的可能性很小[62]。eDNA 技术增加了对入侵物种早期监测的可能性,降低了物种入侵的控制成本和生态影响。
eDNA 技术检测灵敏度高,相较于传统调查方法显示出更高的检测效率和成本效益[63-64]。南方波鱼是珠江水系特有鱼类[36],属于山溪小型鱼类[65],喜栖息于溪流和水沟。由于调查所用网具和调查范围的限制,在近些年的传统调查研究中均未采集到该种鱼类;而在本研究的6 个采样点中均可检出该种鱼类,说明珠江中下游存在一定种群数量的南方波鱼,也表明eDNA 技术在特有鱼类的监测中具有一定优势。
4 小结
eDNA 技术具有成本低、检测灵敏度高、检测效率高和非破坏性采样等特点,为鱼类多样性监测和保护工作提供了新的选择。本研究首次利用eDNA 技术检测了珠江中下游鱼类多样性,证明其对检测珠江中下游鱼类生物多样性及分布情况的可行性。理论上eDNA 技术对鱼类多样性检测的高效性取决于一系列的生物和技术因素[13],无法完全替代传统鱼类调查方法。eDNA 和传统鱼类调查方法各有其优势,可以提供重叠和互补信息,因此两者应该共同使用,以提供对鱼类生物多样性更全面的了解[66-67],也为制定旨在保护和增强水生态系统功能和恢复力的保护策略提供数据支撑。
