直立型半野生大豆叶绿体基因组分析
2023-12-04郭冉昊赵淑文米福贵候伟峰赵力兴
郭冉昊, 赵淑文*, 米福贵, 候伟峰, 赵力兴
(1.内蒙古农业大学草原与资源环境学院, 内蒙古 呼和浩特 010000; 2.内蒙古农业大学植物学国家级实验教学示范中心,内蒙古 呼和浩特 010000; 3.兴安盟农牧科学研究所, 内蒙古 兴安盟 137400)
叶绿体属于一种半自主性细胞器,一般为共价闭合的环状四分体结构,是高等植物细胞中光合作用的主要场所。叶绿体具有独立的遗传物质,即cpDNA,主要编码与光合作用相关的上百种基因,大小介于120~180 kbp,包括长单拷贝区(Large single copy,LSC)、短单拷贝区(Small single copy,SSC)及2个反向重复序列(Inverted repeat sequence,IRS)。cpDNA在物种分类、系统发育、遗传多样性、物种形成、适应性进化等诸多研究中具有重要作用[1-4]。随着DNA测序技术进步、成本降低,植物基因组学的研究也越来越深入。而相较于大豆核基因组结构和遗传多样性的广泛研究报道,大豆属细胞质基因组研究相对滞后[5]。
大豆属(Glycine)属于豆科(Leguminosae),蝶形花亚科(Papilionatae),含一年生Soja和多年生野生Glycine两个亚属[6]。栽培大豆(G.max)是我国重要的经济作物之一,与野大豆(G.soja)同属Soja亚属。野大豆具有蛋白含量高、抗逆性强、生物量较高等特点,是重要的育种材料和优质饲草,但由于其茎缠绕,利用上有一定难度。除一年生栽培大豆和野生大豆外,还存在一种特殊类群,即性状介于野生大豆与栽培大豆之间,被前苏联学者Skvortzow称为的半野生大豆(G.gracilis)[7],其种子百粒重通常在3 g乃至10 g以上,形态上有黑、褐、黄、绿、双色种皮及各种中间种皮颜色,茎呈缠绕、弱缠绕、葡匐、蔓生、半蔓生、半直立、甚至直立类型,形态丰富[8]。半野生大豆兼具栽培大豆和野生大豆性状等特征,不仅是栽培大豆驯化研究的重要遗传材料[5],与栽培大豆杂交,也可以比野大豆更便捷有地效拓宽大豆遗传基础,创造优异种质。由于半野生大豆的百粒重与栽培大豆有重叠,而且其茎形态与野生大豆和栽培大豆也有重叠,因此半野生大豆的分类地位一直存在争议。有学者认为半野生大豆是野生大豆向栽培大豆驯化后的中间产物,而另一些学者提出半野生大豆是栽培大豆和野生大豆杂交后的产物[9-12]。
直立型半野生大豆既有半野生大豆高蛋白、多花荚、生育繁茂等优良特性,也克服了野大豆茎缠绕,种植利用困难等问题,具有较高的研究价值。本研究以兴安盟农牧科学研究所提供的一份直立型半野生大豆为试材,采用高通量测序技术对其cpDNA序列、密码子偏好性、SSR位点及亲缘关系等进行分析,以期为半野生大豆细胞质多样性、系统分类地位、分子育种及种质利用提供依据。
1 材料与方法
1.1 试验材料
试验材料为采自内蒙古自治区兴安盟科尔沁右翼前旗索伦镇草根台嘎查(120°54′E,46°45′N)的直立型半野生大豆。2021年秋季采收的种子于2022年种植在呼和浩特市内蒙古农业大学试验园区。
1.2 基因组DNA提取和测序
在田间采集健康新鲜植株叶片,置于液氮带回实验室于-80℃冰箱存储,委托北京百迈客生物科技有限公司对样品进行高通量测序、DNA文库构建等工作。
1.3 叶绿体全基因组组装和注释
原始测序数据(raw data)经Fastqc程序质检后,用trim_galore除去低质量序列得到clean data,然后用Getorganelle[13]对cpDNA进行组装。进一步借助在线工具GeSeq[14](https://chlorobox.mpimp-golm.mpg.de/geseq.html)对组装结果进行注释(注释参考基因组:NC_0229868.1)。注释过程中,先依据注释后IR区的位置手动调整基因组起始位置,并将基因组按照LSC,IRb,SSC和IRa顺序排列,最后将排列完成的cpDNA用在线软CPGAVAS2[15](http://47.96.249.172:16019/analyzer/home)进行cpDNA注释。cpDNA物理图谱由Chloroplot[16]R软件包绘制。
1.4 cpDNA基本特征
用tRNAscan-SE software软件确定编码基因总数、编码tRNA和rRNA的基因数目[17]。
1.5 密码子偏好性和SSR分析
运用CodonW 1.4.2软件[18](http://mobyle.pas-teur/fr/cgi-bin/portal.py?from=codonw)分析材料cpDNACDS序列,获得试验材料同义密码子的相对使用度(relative synon-ymous codon usage,RSCU);采用在线网站(https://webblast. ipk-gatersleben. de/misa/index.php?action=1)扫描分析直立型半野生大豆cpDNA序列进行微卫星扫描分析,单核苷酸、双核苷酸、三核苷酸、四核苷酸、五核苷酸和六核苷酸设置参数重复数分别不少于10,5,4,4,4,4。
1.6 野大豆及其近缘种系统发育树构建
在NCBI数据库中下载所有已公布大豆属植物cpDNA序列,包括Glycine和Soja两个亚属共8个材料同时以模式植物拟南芥[(Arabidopsisthaliana)(MZ323108.1)]、烟草[(Nicotianatabacum)(MZ707522.1)]作为外类群,将所有参试物种的cpDNA序列选择MAFFT进行多序列比对,手动校正,使用PhyloSuite软件[19]中的RAxML,bootstrap为1 000,构建系统发育树。
2 结果与分析
2.1 cpDNA结构、分类、功能及特征
供试直立型半野生大豆的cpDNA全长为148 320 bp,属典型环状四分体结构(图1)。共编码108个基因,包括66个蛋白编码基因、29个tRNA基因和4个rRNA基因。按基因功能可将供试直立型半野生大豆cpDNA编码基因分为3大类:57个基因表达相关基因、42个光合作用相关基因、7个开放阅读和其他蛋白编码基因以及2个未知功能基因。其中基因表达相关基因包含5个小类,数量最多的为转运RNA基因,RNA聚合酶基因与核糖体RNA基因数量较少,仅为4个(表1)。
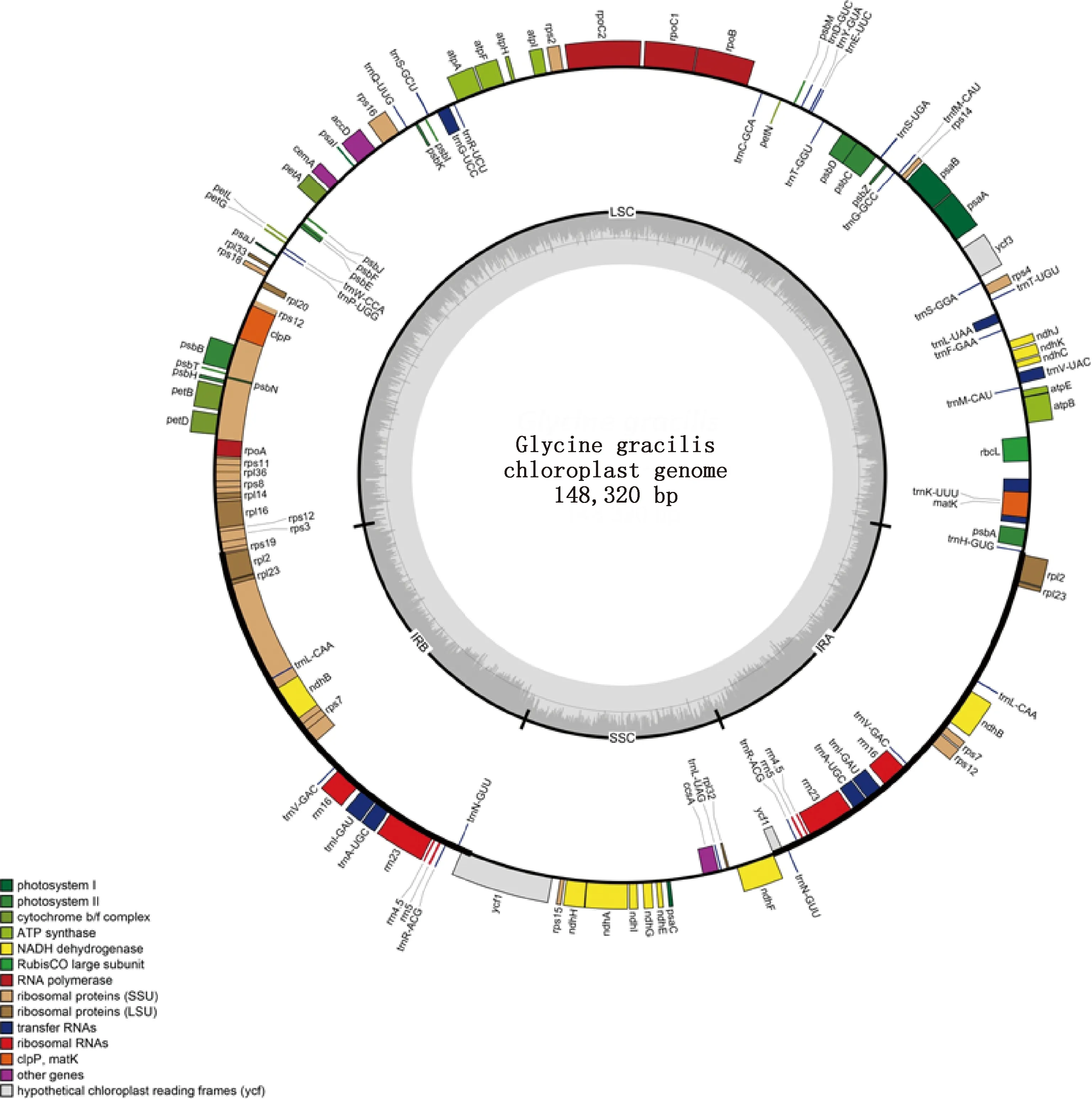
图1 直立型半野生大豆(G.gracilis)cpDNA环形图谱Fig.1 Circularized map of the chloroplast genome of the erect semi-wild soybean (Glycine gracilis)
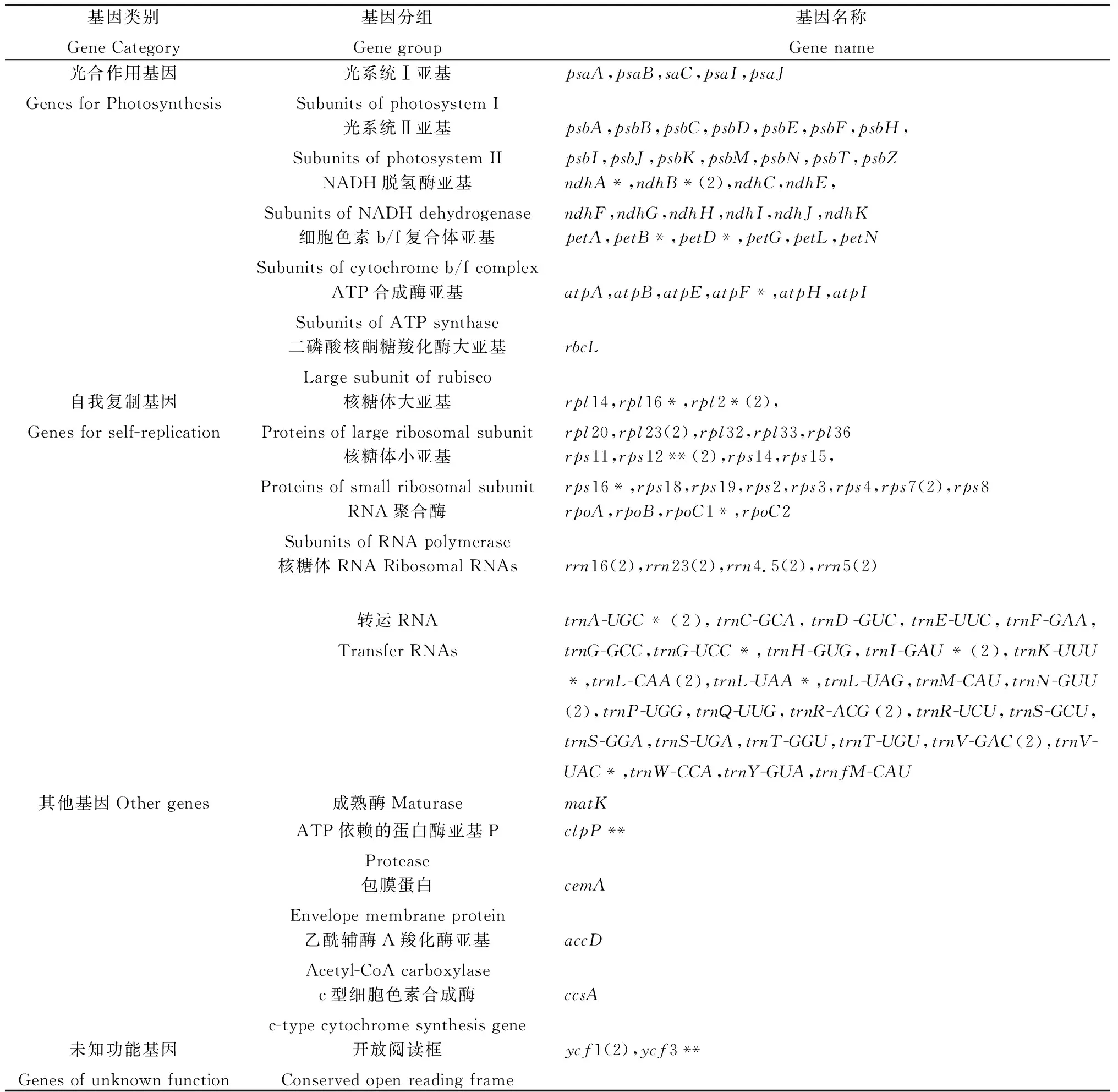
表1 直立型半野生大豆(G. gracilis)cpDNA注释信息Table 1 Gene annotation of the chloroplast genome of the erect semi-wild soybean (Glycine gracilis)
2.2 密码子偏好性
材料cpDNA密码子偏好性等分析结果如图2所示,可见编码亮氨酸(L)、精氨酸(R)、丝氨酸(S)的密码子数量较多,编码色氨酸(W)的密码子数量较少。RSCU值大于1的密码子共有31个,其中29个以A/U结尾,仅有2个以G/C结尾。
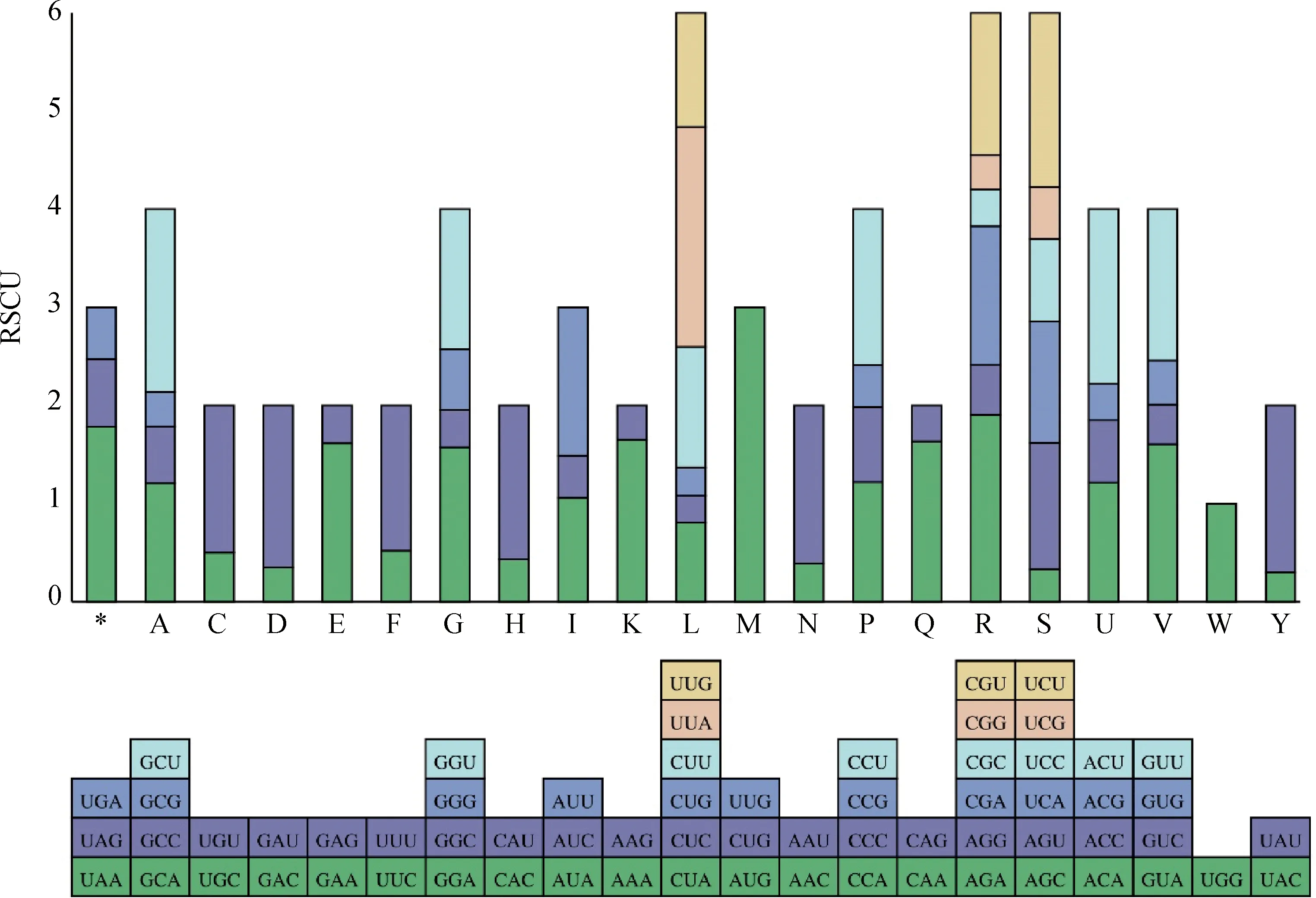
图2 直立型半野生大豆(G.gracilis)叶绿体基因组各氨基酸的RSCU分析Fig.2 RSCU analysis of each amino acid in the erect semi-wild soybean (G. gracilis)
2.3 简单重复序列分析
从材料cpDNA中共鉴定出5种不同类型的SSR位点87个(表2)。其中单核苷酸重复序列最多,共55个,分为A(31SSRs),T(23SSRs),C(1SSRs)三种类型;其次是双核苷酸重复序列19个,包含AT/TA两种类型。此外,鉴定出三核苷酸重复序列4个、四核苷酸重复序列7个、五核苷酸重复序列2个。绝大多数SSR分布于大单拷贝区,其余分布于小单拷贝区和反向重复序列a(表2)。
2.4 野生大豆及其近缘种系统发育分析
根据已公布的所有大豆属材料cpDNA构建系统发育树。结果表明,大豆属9个物种构成一个单系类群,靴带支持率(bootstrap)高达100%(见图3)。该类群进一步又可划分为两类,即一年生的半野生大豆G.gracilis.、栽培大豆G.max和野大豆G.soja与被测直立型半野生大豆组成一类,多年生大豆G.falcata,G.canescens,G.syndetika,G.dolichocarpa等组成一类。该结果与目前系统分类学对大豆属植物的分类相符。
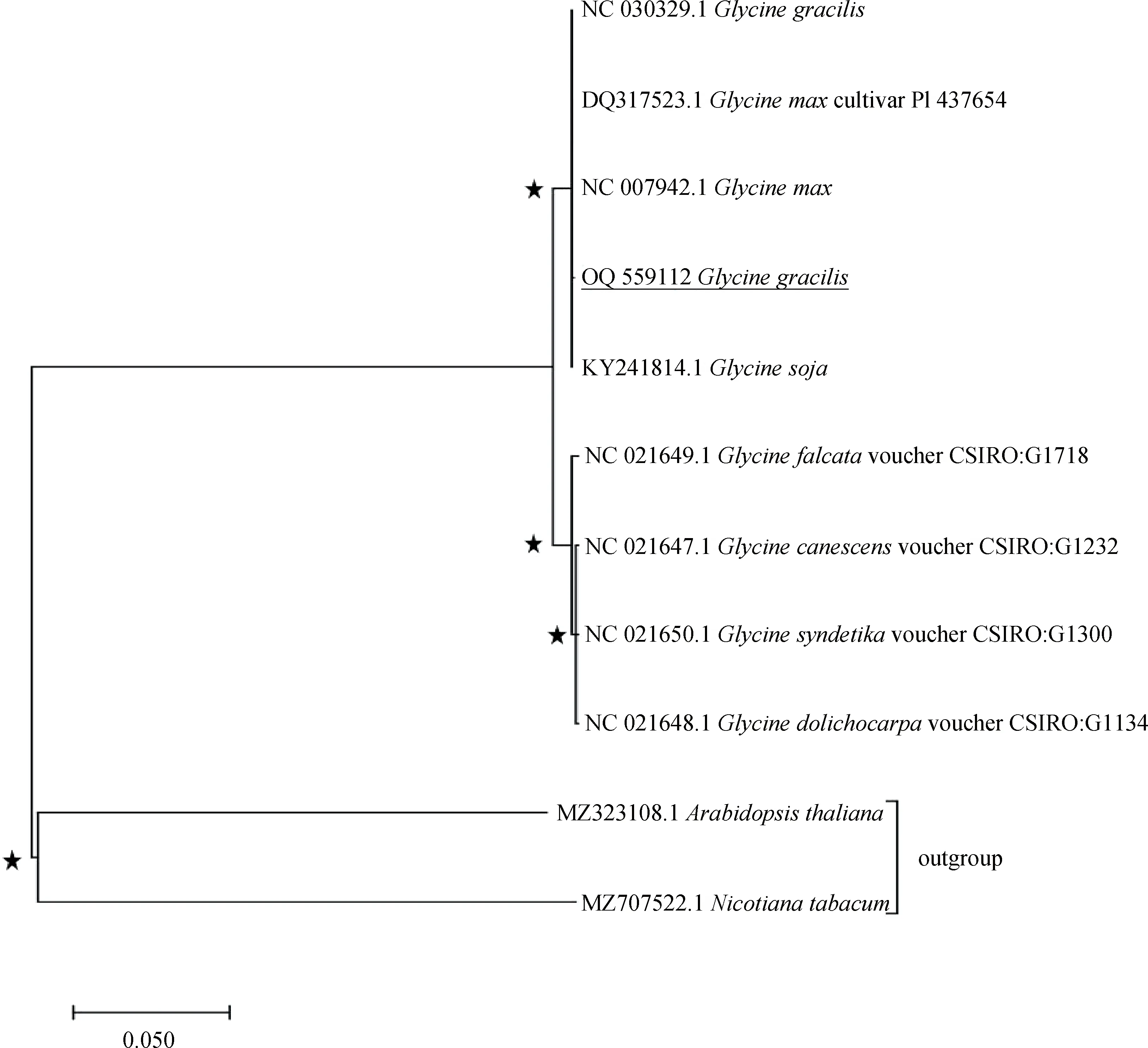
图3 基于cpDNA序列构建的大豆属系统发育树Fig.3 Phylogenetic tree constructed based on the DNA sequence in Glycine chloroplast genomes注:“★”代表分支节点的靴带支持率为100%Note:“★”represents the bootstrap support rate of 100% on the branch
3 讨论
叶绿体DNA与线粒体DNA一起被称为细胞质基因(Cytoplasmic genome),有遗传多样性和单亲遗传限制,异交重组等特点,是植物系统发育研究、作物驯化追溯、作物改良和基因工程研究的重要对象[9,11,20-24]。随着测序技术水平提高,测序成本不断降低,植物cpDNA研究开始兴起。据报道,植物cpDNA长度通常在120~180 kb,编码基因一般在 100~130 个,其中蛋白编码基因多至 80 个、tRNA编码基因30~32个,rRNA编码基因数稳定,常有4种[25]。本文试验材料cpDNA大小、编码基因数目和种类等与前人研究结果一致,符合cpDNA高度保守性的特性。
植物翻译蛋白时并不平均地使用同义密码子,某一或几种特定密码子使用频率高于其他同义密码子,这种现象即为密码子偏好性。RSCU常被用作衡量密码子偏好性,可在一定程度上反映基因乃至物种起源及进化方式,并对基因功能及其编码蛋白表达有一定影响[26]。当某一密码子RSCU值大于1,则表明其使用频率相对较高。本研究中密码子偏好性分析结果表明,RSCU值大于1的同义密码子共有31个,其中以A/T结尾的有29个,与扁蓿豆、紫花苜蓿等豆科植物密码子偏好性一致[27-28]。简单重复序列(SSR)又称微卫星,是整个基因组中1~6 bp的重复序列,多态性高、分布广泛,是高等真核生物基因组的重要组成部分。又因为cpDNA结构简单、相对保守,cpSSR成为在遗传分析、物种鉴定、群体遗传多态性等研究中的重要工具[29]。在本研究中共鉴定出单核苷酸、双核苷酸、三核苷酸、四核苷酸、五核苷酸共5种不同类型的SSR位点87个,重复序列数及类型与已报道的其他被子植物cpDNA的SSRs序列构成基本一致[30],同时印证了cpSSRs主要由短的poly A、poly T而非C或G的重复串联构成[31]。cpDNA尤其对近缘物种,能有效进行鉴定和系统亲缘关系分析[32]。本文基于cpDNA序列对大豆属9个物种构建系统发育树,结果与目前系统分类学对大豆属植物的分类相符合。但半野生大豆在soja亚属中的系统分类地位尚不能完全说明。现已公布soja亚属cpDNA数据仅有9个,或许随着数据量的增加可以从cpDNA角度解释半野生大豆在soja亚属中的分类地位。
4 结论
直立型半野生大豆cpDNA全长148 320 bp,典型四分体结构,编码基因108个,其中蛋白编码基因66个、tRNA基因29个、rRNA基因4个。基因组序列上共检测出单核苷酸、双核苷酸、三核苷酸、四核苷酸、五核苷酸,五种类型SSR位点87个。在系统发育上,直立型半野生大豆与已公布的普通型一年生半野生大豆(G.gracilis)、大豆(G.max)及野大豆(G.soja)亲缘关系较近,但其在soja亚属中的分类地位还有待进一步研究。本研究对直立型野大豆cpDNA进行了初步分析,为半野生大豆细胞质多样性、半野生大豆系统分类、分子育种及种植利用提供依据,也为soja亚属系统进化和遗传研究提供了重要的数据资源。
