基于GBS测序和连锁分析的藜麦单株粒重QTL定位
2023-11-22吴文强代越琪陈天青隋建枢罗永露何庆才
吴文强, 代越琪, 陈天青, 隋建枢, 罗永露, 王 伟, 何庆才
(1.贵州省农业科学院旱粮研究所, 贵阳 550006; 2.贵州金农科技有限责任公司, 贵阳 550006)
藜麦(ChenopodiumquinoaWilld.)属藜科藜属,原产于南美洲安第斯山脉,作为印第安人的传统食物,被联合国粮食及农业组织(FAO)认定为唯一一种单体植物即可满足人体基本营养需求的食物[1-2],且具有耐旱、耐寒、耐盐碱、耐瘠薄等特性[3-5]。近年来,藜麦在世界各国的需求量大幅度上升,仅靠南美洲进口藜麦品种已经不能满足藜麦市场需求[6]。因此,欧洲、亚洲和非洲各国开始了一系列引种试验和藜麦栽培研究[7-8],为藜麦产业发展奠定了一定的基础。同时,藜麦相关基因组学研究也取得了一定的成果,如Maldonado等[9]利用PI614889 和CHEN-109 构建F2和F3群体,定位到生育期、株型、千粒重以及皂苷含量等发育相关QTL 15个;以及Cervantes等[10]利用Red Carina 和Atlas 为亲本,构建94个F3个体与1 076个SNP定位到穗色、开花时间、成熟时间、株高以及千粒重等22个农艺性状相关QTL,其中pleio7.1与藜麦单株粒重相关。这些研究为解析藜麦单株粒重遗传基础提供了参考。然而,藜麦单株粒重作为复杂数量性状,是大量微效等位基因共同作用的结果[11-12],基于少数藜麦种质材料尤其是国外的种质,以及低密度标记所揭示的遗传基础,对于深度揭示认识藜麦单株粒重变异的遗传基础所提供的信息较为有限。近年来,随着高通量的基因型鉴定技术的大量应用,结合连锁分析和高通量基因型鉴定技术,深入解析重要性状变异的遗传研究,能够有效缩小目标QTL,提高定位结果的分辨率,该方法已被大量应用在主要粮食作物重要性状的遗传区段鉴定上[13-14],为藜麦单株粒重的遗传基础解析提供了很好的借鉴。因此,选用国内选育,且拥有较大种植面积的陇藜1号高代选系和与其在单株粒重上有较大差异的ZL00114高代选系为亲本构建作图群体,并结合基因测测序(GBS),解析藜麦单株粒重变异的遗传基础,挖掘相关性状的遗传区段,对于充分利用国内藜麦种质的遗传优势及改良其单株粒重具有重要的科学理论意义和实践利用价值。
1 材料与方法
1.1 材 料
本研究以单株粒重较高的藜麦品种陇藜1号高代选系和单株粒重低的高代自育选系ZL00114为亲本构建的120个单株的F2分离群体以及120个F2∶3分离家系为材料。
1.2 田间试验设计及表型评价
试验采用随机区组设计,单行区,行长3 m,株距0.3 m,行距0.6 m,并于2021年冬季在海南南繁基地(九所)种植120个F2单株。于2022年夏季在贵州省农业科学院试验基地种植120个F2∶3家系, 田间管理同大田生产,并于收获后进行单株粒重表型鉴定,每个F2∶3家系鉴定10株。
1.3 DNA提取和基因型鉴定
藜麦6叶期,取F2单株幼嫩叶片,利用改良CTAB法提取基因组DNA,并将合格的DNA样品送北京康普森生物技术公司进行DNA质量评估和简化GBS,鉴定基因型。
1.4 数据分析
利用SAS9.0、IciMapping等软件对数据进行方差分析和QTL定位,鉴定出控制藜麦单株粒重的遗传区段。
2 结果与分析
2.1 高密度遗传图谱构建
利用34 484个SNP构建了高质量的遗传图谱。结果(图1)显示,所有SNP分布于18条染色体上。其中第1条染色体上SNP分布最多,为5 493个;第9条染色体分布最少,为234个;其余各染色体上SNP分布数量分别是:第2条1 704个、第3条2 113个、第4条1 787个、第5条2 446个、第6条3 611个、第7条1 642个、第8条1 780个、第10条1 212个、第11条1 348个、第12条2 083个、第13条505个、第14条1 850个、第15条2 190个、第16条1 542个、第17条1 907个以及第18条1 037个。

注:每行代表一个SNP标记,不同的颜色显示不同的标记密度;浅色表示标记的密度低,而深色表示标记的密度高。
2.2 控制藜麦单株粒重QTL
利用完备区间作图法,定位到7个控制藜麦单株粒重相关QTL,结果列于表1。分析发现,这些QTL分布于第3,4,5,7,12条染色体上,其中,第3、第7染色体上分别分布了2个相关QTL。结果显示,单个位点可解释6.09%~10.17%的单株粒重变异,单个QTL大小在10.54~401.86 kb之间。

表1 控制藜麦单株粒重相关QTL
定位于第3条染色体控制藜麦单株粒重的QTL有2个 (表1,图2),分别是qSPW3-1和qSPW3-2,qSPW3-1大小为265.76 kb,LOD值为2.59,可解释藜麦单株粒重表型变异6.34%;qSPW3-2大小为401.86 kb,LOD值为2.99,可解释藜麦单株粒重表型变异10.17%;其中qSPW3-2表现出较高的遗传效应。

注:x轴表示每个染色体上SNP标记的物理位置;y轴表示LOD值,虚线表示阈值。下同。
定位于第4条染色体控制藜麦单株粒重的QTL是qSPW4,大小为10.54 kb,LOD值为2.55,可解释藜麦单株产量表型变异7.58%(表1、图3)。
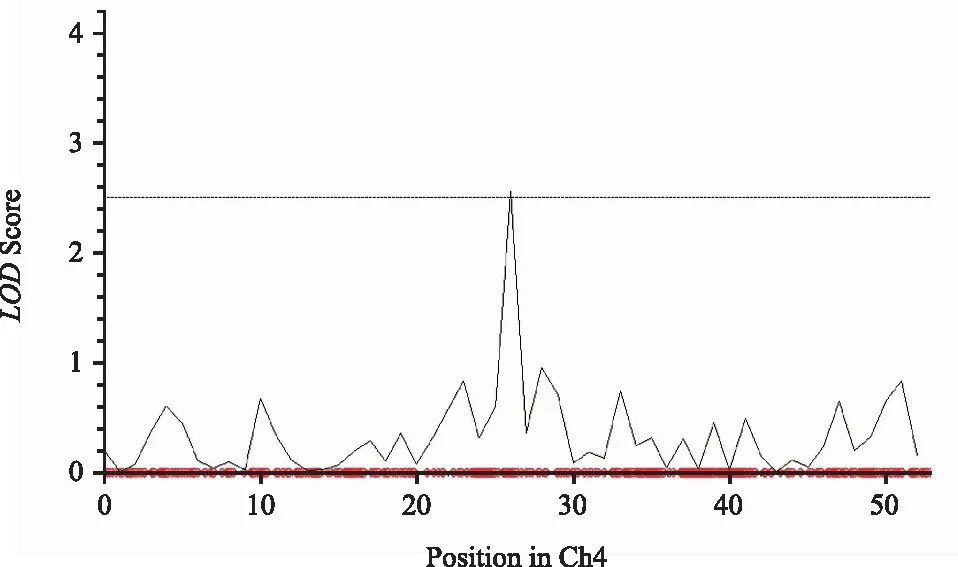
图3 定位于第4条染色体控制藜麦单株粒重QTL
定位于第5条染色体控制藜麦单株粒重的QTL是qSPW5,大小为194.87 kb,LOD值为3.56,可解释藜麦单株产量表型变异6.09%(表1、图4)。

图4 定位于第5条染色体控制藜麦单株粒重的QTL
定位于第5条染色体控制藜麦单株粒重的QTL 有2个,分别是qSPW7-1和qSPW7-2(表1、图5)。其中,qSPW7-1大小为199.07 kb,LOD值为3.13,可解释藜麦单株粒重表型变异的7.70%(表1);qSPW7-2大小为63.04 kb,LOD值为4.07,可解释藜麦单株粒重表型变异9.33%(表1),表现出了较高的遗传效应。
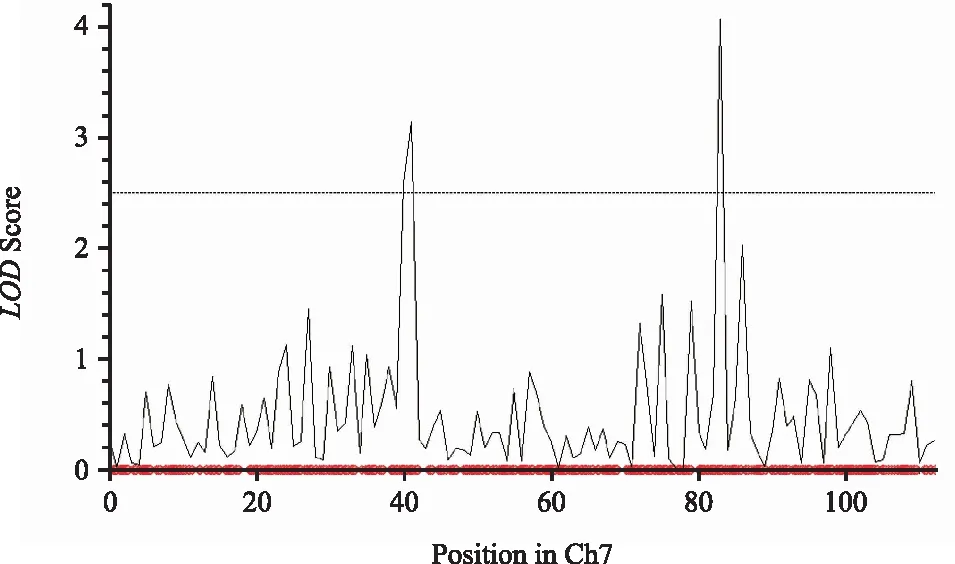
图5 定位于第7条染色体控制藜麦单株粒重的QTL
定位于第12条染色体的QTL是qSPW12,大小为102.09 kb,LOD值为2.98,可解释藜麦单株粒重表型变异的6.63%(表1、图6)。

图6 定位于第12条染色体控制藜麦单株粒重的QTL
3 讨 论
目前,藜麦QTL定位相关研究报道较少,尤其是单株粒重相关QTL定位,只有Cervantes等[10]利用Red Carina和Atlas为亲本构建94个F3个体与1 076个SNP定位到与藜麦单株粒重相关的QTL pleio7.1,为解析藜麦单株粒重遗传基础,提供的理论依据有限。本研究以单株粒重差异较大的两个亲本构建的双亲分离群体为材料,基于GBS测序和表型鉴定结果,利用完备区间作图法定位到7个控制藜麦单株粒重的QTL(表1),均为新定位到的控制藜麦单株粒重相关QTL,分别是位于第3染色体的qSPW3-1、qSPW3-2;位于第4染色体的qSPW4;位于第5染色体的qSPW5,位于第7染色体的qSPW7-1、qSPW7-2;以及位于第12染色体的qSPW12;单个QTL表型变异贡献率为6.09%~10.17%,可累计解释藜麦单株粒重变异的58.34%;单个QTL变幅为10.54~401.86 kb。这些研究将为初步揭示藜麦产量相关性状变异的遗传基础提供分子遗传学依据,为基于分子标记辅助选择的藜麦高产新种质创制提供一定支撑。下一步,本研究将结合连锁分析和关联分析,针对双亲分离群体重组事件相对较小以及自然群体定位结果假阳性较高的特点,提高定位精度,为进一步的精细定位和基因筛选提供参考。
