流形学习在红松籽仁蛋白质含量近红外检测中的应用
2023-06-08仇逊超张春越张怡卓曹军
仇逊超 张春越 张怡卓 曹军


摘要: 为研究检测红松籽仁蛋白质含量的近红外光谱分析技术,在用变量标准化校正+一阶导数+小波变换对原始光谱进行预处理的基础上,分别运用主成分分析、改进型局部线性嵌入、局部切空间对齐、黑塞特征映射进行光谱数据的降维处理,分别构建偏最小二乘、岭回归、支持向量回归、极度梯度提升数学模型。结果表明,改进型局部线性嵌入+支持向量回归法建立的参数优化模型质量最佳。其降维方法优化参数为:维度取4,邻域数取50;验证集均方差均值为0.568 1,验证集皮尔逊相关系数均值达0.940 8。可见,模型的预测结果是可靠的,能够实现对红松籽仁蛋白质含量的无损、准确检测。
关键词: 红松籽仁;蛋白质;流形学习;近红外光谱
中图分类号: TS255.6 文献标识码: A 文章编号: 1000-4440(2023)01-0246-09
Application of manifold learning in quantitative detection of protein in Korean pine seed kernels using near-infrared quantitative detection
QIU Xun-chao1,2, ZHANG Chun-yue1, ZHANG Yi-zhuo2, CAO Jun2
(1.Department of Computer Engineering, Harbin Finance University, Harbin 150030, China;2.College of Mechanical and Electrical Engineering, Northeast Forestry University, Harbin 150040, China)
Abstract: To study the near-infrared spectroscopy for protein content detection in Korean pine seed kernels, principal components analysis (PCA), modified locally linear embedding (MLLE), local tangent space alignment (LTSA) and Hessian based locally linear embedding (HLLE) were used separately to reduce dimensions of the spectroscopic data, based on pretreatment of the original spectrum by standard normalized variate (SNV)+first derivative (1st-Der)+Symlet4 (SNV+1st-Der+Sym4) method. Partial least square (PLS), ridge regression (Ridge), support vector regression (SVR) and extreme gradient boosting (XGBoost) were adopted separately to establish mathematical models. The results showed that, the quality of the parameter optimization model established by MLLE+SVR method was the best. The optimized parameters for dimension reducing were as follows: the dimension (n-components) was four, the neighborhood number (n-neighbors) was 50, the mean value of mean squared error of validation (mean-MSEV) was 0.568 1, and the mean value of Pearson correlation coefficient of validation (mean-PCCV) was 0.940 8. Therefore, the prediction results of the model is reliable, and non-destructive, accurate and quantitative detection of protein in Korean pine seed kernels can be realized.
Key words: Korean pine seed kernel;protein;manifold learning;near-infrared spectroscopy
紅松籽是红松的种子,主要产于中国北方地区,红松籽市场需求旺盛,供不应求,红松籽产业是促进农林增收、拉动东北三省地区经济快速发展的重要产业之一。红松籽仁味道鲜香,蛋白质含量高,其总氨基酸中必需氨基酸占1/4,是优质的植物蛋白,能为人体提供丰富的营养,红松籽仁中蛋白质的定量研究是植物种子营养成分和新旧判别的重要指标。传统的凯式定氮蛋白质定量法,是将样品与硫酸铜和硫酸钾混合溶液及浓硫酸挥发性溶剂进行融合的破坏性化学分析方法,其测试过程繁琐、耗时长、会产生刺激气体,危害检测人员身体健康的同时,也无法满足大规模测试和生产的需要。因此,非破坏性、快速、简便、准确、绿色的近红外光谱分析技术近年来被应用到坚果中蛋白质的定量检测研究中[1-3]。
在红松籽仁蛋白质近红外检测方面,前人开展的研究较少。蒋大鹏等[4]通过构建的支持向量机模型,对红松籽仁的蛋白质品质进行了分类。仇逊超等[5]前期运用无信息变量消除法、反向间隔偏最小二乘法,通过波段筛选建立了红松籽仁蛋白质偏最小二乘近红外模型。全波段范围内包含的数据信息量大,且存在冗余信息,除采用波段筛选方法外,还可以采用降维方法来提高建模的效率和准确性。传统的降维方法主要是通过主成分分析的线性变化来实现,线性降维由于受到技术限制,在映射到低维空间的过程中无法很好地反映高维空间中的非线性信息[6]。非线性降维方法分支中的流形学习,其核心思想是高维欧式复杂空间的模型是由其内在的低维流行模型生成的,因而降维为低维数据模型后,可以更好地反映映射关系,发掘低维特征,保证非线性信息的保留。
本研究在對原始光谱数据进行变量标准化校正+一阶导数+小波变换的预处理基础上,进一步利用主成分分析、改进型局部线性嵌入、局部切空间对齐、黑塞特征映射进行降维处理,以近红外技术中最为广泛采用的偏最小二乘为定标模型[7],比对岭回归、支持向量回归、极度梯度提升的建模结果,探索不同降维、不同建模方法对红松籽仁蛋白质定量检测精度的影响,以期找到最优的降维和建模方法,构建质量较优的近红外模型,实现对红松籽仁蛋白质的准确、无损定量检测。
1 材料与方法
1.1 材料
红松籽样品购买于凉水国家级自然保护区,于当年采摘。对红松籽进行手工去壳脱红衣,并随机选取完整的120粒作为样品,将每个红松籽仁样品分别放入贴有1~120编号标签的密封袋中。另随机选取完整的20粒红松籽仁样品,用于建模后对模型的测试。将上述样品置于恒湿恒温(相对湿度和温度分别为50%~60%、-1~2 ℃)的阴凉处保存。
1.2 方法
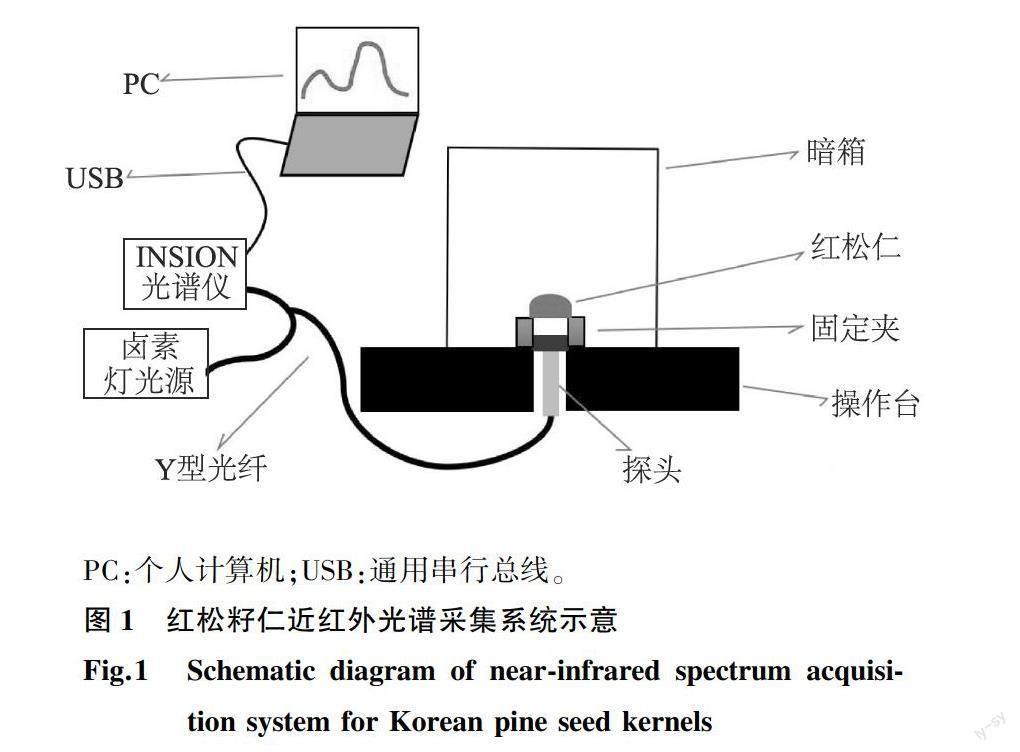
1.2.1 近红外光谱数据的采集 近红外光谱采集系统如图1所示。经过查阅相关文献发现,光谱波长范围为950~1 700 nm时,包含的信息可以较理想地满足本研究需求[8-9]。本研究采用德国INSION公司的NIR-NT-spectrometer-OEM-system微型近红外光纤光谱仪,光谱适用波长范围为900~1 700 nm,光谱分辨率在16 nm以下,具有抗震性和高集成性。卤素灯光源工作电压为24 V。在进行红松籽仁近红外光谱数据采集前,保持环境温度在26 ℃左右,将样品静置在该环境下24 h以上。为使近红外光谱仪处于稳定的工作状态,将其打开预热15 min左右。设定仪器参数,其中,光谱仪积分时间设置为30 ms,平均扫描次数设置为3次。将探头放入操作台底端的孔洞内,保持探头与样品距离在3 mm左右,固定光纤。扫描红松籽仁光谱数据时,将倒卵状三角形的红松籽仁平滑腹部置于探头上,以实现光源的完全遮挡。
1.2.2 传统蛋白质的定量测定 红松籽仁蛋白质的定量测定参考GB 5009.5-2010《食品安全国家标准 食品中蛋白质的测定》中的凯式定氮法。
2 结果与分析
2.1 红松籽仁漫反射近红外光谱分析
图2为获取到的红松籽仁近红外原始光谱图像,实际采集的光谱波长范围为906.90~1 699.18 nm,扫描间隔为6.83 nm。
蛋白质是由氨基酸以“脱水缩合”的方式组成的多肽链,主要由碳(C,50%)、氢(H,7%)、氧(O,23%)、氮(N,16%)元素组成,具有一级、二级、三级、四级结构,分子中有O-H、C-H、N-H含氢基团。图2中1 400 nm附近和1 550 nm附近的明显吸收峰为一级胺基(-NH2)组合频吸收峰和一级胺基与亚氨基(-NH)的倍频吸收峰[10],1 100~1 200 nm附近的强烈吸收峰为C-H基团二级倍频吸收峰[11],1 690 nm附近的微弱吸收峰为C-H基团一级伸缩振动吸收峰[12]。蛋白质N-H标志性基团的一倍频和二倍频吸收峰分别分布在1 428~1 700 nm、1 000~1 428 nm[13]。由此可知,本研究选定的光谱范围可以表征红松籽仁的蛋白质特征。
2.2 训练集与验证集的切分
红松籽仁样品蛋白质含量分布情况如图3所示,中位数为16.06%,标准差为2.46%,虚线内样品数占总样品数的72.50%,蛋白质含量为12.79%~24.98%,分散差异较大,且基本覆盖了红松籽仁蛋白质含量常规分布范围,表明试验样品合理,符合后续的模型建立要求。
为了测试本研究构建模型的可靠性和稳定性,对训练集与验证集按照4∶1的比例进行10次不同切分。为了保证每次切分结果的可重复性,通过为随机种子分配10个固定取值,使得10次切分结果与该10个固定取值分别对应,进而保证每次切分结果是可重复的。分别在不同的训练集上,进行10次近红外红松籽仁蛋白质定量模型的建立,以10次模型的平均评价指标来评价模型。10次切分结果如表1所示。
观察切分结果,发现10次切分的结果均不相同,并且每次切分后训练集蛋白质含量覆盖范围均大于验证集,说明10个红松籽仁训练集样品所建立的模型可以较好地适用于相应的验证集样品。
2.3 光谱预处理
原始光谱由于受到采样环境、采集方式等影响,存在信噪比低、发生散射变化等现象,此外还发现原始光谱图像存在较大方差、吸收宽度分散的现象,所以需要进行光谱预处理[14]。
在漫反射式光谱数据采集的过程中,由于红松籽仁颗粒度不均匀,会使得光谱数据因散射影响而产生差异,采用变量标准化校正(SNV)可以进行有效校正[15]。光谱信息中吸收宽度存在重叠现象,会互相干扰,影响模型的稳健性,因此在SNV预处理的结果上进行一阶导数(1st-Der)处理[16]。光谱求导后会提高噪声水平,降低信噪比,因此进一步进行小波变换平滑处理。近似对称的紧支集正交小波(SymN)被实践证明在近红外滤波方面十分有效,SymN具备较好的正则性,作为一种对称小波,在对信号进行分析和重构时能够减少相位失真[17]。因此,采用Sym4小波基函数进行2尺度分解来进行平滑处理。经过SNV+1st-Der+Sym4预处理后的光谱图像如图4所示,随机选取1条滤波前后的光谱曲线,并将滤波后的光谱曲线向上平移一段距离,进行直观的对比观察。由图5可知,经Sym4小波变换处理后,光谱曲线去掉了一些毛躁噪声,变得较为平滑。
2.4 光谱数据的降维与建模
全光谱波长范围内的信息量大,含有与需求无关的信息,会降低建模效率,还会影响模型精准度。对数据进行降维处理后,可以保留有用信息,降低构建定量模型的学习复杂程度。采用经典的线性主成分分析(PCA)[18]及非线性流形学习中的改进型局部线性嵌入(MLLE)、局部切空间对齐(LTSA)、黑塞特征映射(HLLE)降维方法,对经过预处理的光谱数据进行降维处理。为了研究不同建模方法对红松籽仁蛋白质定量预测模型的影响,进一步分别运用岭回归(Ridge)[19]、支持向量回归(SVR)[20]、极度梯度提升(XGBoost)[21]方法构建红松籽仁蛋白质定量模型,并以偏最小二乘法(PLS)建立的模型為定标,根据模型的评价指标确定最佳的降维和建模方法。
局部线性嵌入(LLE)[22]的中心思想是,找到每个数据点的原始高维领域线性关系,即假设高维空间数据点Xi的邻域线性关系表达式为:
Xi=ωihXh+ωikXk+ωilXl(1)
其中,ωih、ωik、ωil为权重系数,在经过LLE降维处理后,在低维空间这种线性关系表达得到同样的保持。权重系数(ωij)可以通过式(2)求取:
其中,Q(i)表示数据点Xi的k个邻域数据点集合,m表示样本个数。
低维空间数据点yi通过式(3)求取:
LLE只需确定邻域数,即可完成降维操作,但其存在当邻域数大于输入数据的维度时,权重系数矩阵不是满秩的情况,为了解决类似问题,衍生出了MLLE、LTSA和HLLE方法。MLLE方法不仅寻找最近距离的邻域数,还对邻域的分布权重进行度量,以期使邻域的分布权重尽量在样本的各个方向。LTSA方法用样本点的近邻区域的切空间来表示局部几何结构,然后对局部切空间进行重新排列,得到非线性流形的、用自然参数刻画的低维线性关系。HLLE方法不是通过线性关系来构建邻域内的样本,而是依据黑森矩阵的二次型关系展开构建。
降维方法对建模效果的影响会因其参数的不同取值而有所差别,因此需要优化降维方法的参数,进而建立高质量的红松籽仁蛋白质定量数学模型。
PCA需要确定方差累计贡献率(n-contribution)的最优取值,一般要求累积贡献率达到85%以上,因而其参数取值情况为:n-contribution=[0.86,0.88,0.90,0.92,0.94,0.96,0.98,0.99]。MLLE、LTSA和HLLE方法需要对邻域数(n-neighbors)和维度(n-components)进行最优值的选取,n-neighbors越大,算法的复杂度会增加,建立样本局部关系的时间会越长,但降维后样本的局部关系会保持得更好。此外n-neighbors最大取值不能超过红松籽仁训练集样品个数。MLLE方法要求n-neighbors>n-components,HLLE方法要求n-neighbors>n-components×(n-components+3)/2,因此将MLLE、LTSA方法参数取值情况设置为:n-neighbors=[20,30,40,50,60,70,80,90]、n-components=[3,4,6,8,10,12,14,16,18];HLLE参数的设定分为以下几种情况,当n-components=[3,4]时,n-neighbors=[20,30,40,50,60,70,80,90];当n-components=6时,n-neighbors=[30,40,50,60,70,80,90];当n-components=8时,n-neighbors=[50,60,70,80,90];当n-components=10时,n-neighbors=[70,80,90]。
为了构建出一个高质量的PLS定标模型,需要对PLS主成分数(n-components)进行确定,根据方差累计贡献率为86%~99%的需求,主成分数取值范围为[3,4,5,6,7,8,9,10,11,12,13,14,15]。根据比对10次不同切分出的10个验证集均方差(MSEV)的均值(mean-MSEV),确定最优的主成分数,比对结果如图6所示。
需要说明的是,mean-MSEV越小,拟合效果越好。由图6可知,当主成分数为5个时,PLS模型质量最优,其mean-MSEV为1.150 7,验证集皮尔逊相关系数(PCCV)均值(mean-PCCV)为0.889 8,mean-PCCV越接近1越好。由定标模型可知,采用近红外光谱技术对红松籽仁蛋白质进行定量分析是可行的,结果是可靠的。
分别在全波段、光谱降维范围下构建红松籽仁蛋白质的Ridge、SVR、XGBoost、PCA+Ridge、PCA+SVR、PCA+XGBoost、MLLE+Ridge、MLLE+SVR、MLLE+XGBoost、LTSA+Ridge、LTSA+SVR、LTSA+XGBoost、HLLE+Ridge、HLLE+SVR、HLLE+XGBoost数学模型,并对降维方法进行参数优化。为了测试模型的稳定性,每个模型在10次不同切分出的10个训练集上进行模型构建,通过对比10次建模的mean-MSEV,进而确定降维、建模的选取方法,并找到相应降维方法的最优参数。10次建模的mean-MSEV对比情况如图7、图8所示。
由图7可知,不同建模方法构建出的模型质量不同,在全波段范围内XGBoost模型质量最佳,其mean-MSEV为2.952 5,SVR模型质量次佳,SVR与XGBoost均为非线性模型,而Ridge属于线性模型,这说明红松籽仁光谱数据中,包含了对蛋白质定量分析建模有用的非线性信息。此外,由于PLS在建模过程中进行了PCA降维处理,去除了冗余信息,因此其模型质量与全波段范围下Ridge、SVR、XGBoost模型相比更佳。
由图8可知,与全波段构建的模型相比(图7),经过降维处理后模型的质量均有所提升。其中,4种降维方法对XGBoost模型质量的提升效果没有其他2种建模方法明显,这是由于XGBoost对数据维度的敏感度相对较弱。进一步以表格(表2)形式更清晰地比较各最优参数模型。
需要说明的是,由于10次切分出的训练集不同,因而依照方差累计贡献率的PCA降维方法,在不同训练集上降维后的维度不同。
由表2可知,与PLS最优参数定标模型相比,其余最优参数模型的质量均更佳。此外,整体上非线性降维方法对模型质量的提升效果优于线性降维方法。这是因为,一方面经典的PCA线性降维方法,当数据具有较复杂的非线性结构时,只是简单地将数据投射到低维空间中,会导致非线性信息的丢失;另一方面,PCA是关注数据方差的降维方法,而MLLE、LTSA、HLLE是关注数据局部线性关系的非线性降维方法,在降维时保持了数据的局部线性特征。
相同建模方法采用MLLE、LTSA、HLLE不同降维方法后,构建的模型质量相近但又略有不同。这是由于MLLE、LTSA、HLLE三种降维方法的原理均基于LLE降维方法,只是在低维数据进行恢复时遵循的优化原理不同。其中,SVR建模方法经MLLE最优参数降维、XGBoost建模方法经LTSA最优参数降维后,构建的模型质量最佳,mean-MSEV分别为0.568 1、0.943 1;Ridge建模方法经LTSA、HLLE最优参数降维后,构建出了质量相当的最优预测模型,mean-MSEV均为1.049 4。
采用经过参数优化的PCA、MLLE、LTSA、HLLE降维方法后,SVR法构建的模型质量均优于其他建模方法,其中MLLE+SVR模型质量最佳,其10个驗证集上的MSEV分别为0.798 6、0.512 8、0.415 9、0.550 5、0.673 1、0.327 4、0.550 9、0.511 2、0.778 5、0.562 3,mean-MSEV为0.568 1,mean-PCCV达0.940 8,最优参数的取值为:n-components=4,n-neighbors=50。
2.5 MLLE数据降维可视化
为了比较直观地观察MLLE降维处理后对红松籽仁光谱特征性峰与形态提取等的影响,同时为了测试降维结果的可靠性和稳定性,在10次不同训练集与验证集切分结果的基础上,随机选取2个训练集,并对往年53粒红松籽仁样品进行光谱信息的获取,在预处理的基础上,将光谱数据降至二维,以散点图的形式进行可视化展示。图9为随机选取的2个训练集与往年红松籽仁样品降维数据的散点图。
观察图9发现,红松籽仁近红外光谱数据经过MLLE降维处理后,试验当年与往年的样品数据点形成了较明显的两簇,且簇间几乎无交叉现象。
2.6 MLLE+SVR模型的测试
采用最优参数的MLLE+SVR模型,对用于测试的20粒红松籽仁样品蛋白质含量进行定量预测。此外,为了测试MLLE+SVR模型的适用性,进一步对往年30粒红松籽仁样品进行蛋白质含量的定量预测,其中往年MLLE降维方法的最优参数为:n-components=4,n-neighbors=50。
由图10可知,实测值与预测值均比较均匀地分布在45°线两侧。进一步计算实测值与预测值间的平均绝对误差(MAE),来评估预测值和实测值间的接近程度,从而对预测结果的准确程度进行描述。MAE的计算公式为:MAE=1n∑ni=1xi-yi。其中,xi、yi分别表示第i粒红松籽仁样品蛋白质含量的实测值和预测值。整体上,红松籽仁样品的实测值与预测值MAE=0.50%,表明预测结果具有可靠性和准确性,而试验当年与往年红松籽仁样品的MAE分别为0.37%、0.59%。往年预测结果的准确性略低,这是由于最佳降维及建模方法是在当年红松籽仁样品的基础上确定的,但仍可很理想地实现对往年红松籽仁样品蛋白质含量的预测,这在一定程度上表明该MLLE+SVR模型的适用性。
3 讨论
本研究利用NIR-NT便携式近红外光谱仪,通过在900~1 700 nm波长范围内获取的红松籽仁光谱数据,开展了红松籽仁蛋白质定量无损检测试验。在训练集与验证集划分的过程中,进行了10次不同的切分,分别在10个不同的训练集上进行模型的建立,通过10个模型的平均评定指标来评价模型,保障了所建模型的稳定性和可靠性。采用降维方法对光谱数据进行降维处理,可以提升模型的评价指标,表明光谱降维在模型训练中具有重要作用,并且非线性降维方法由于可以更好地保留非线性信息,与线性降维方法相比,可以更好地优化模型质量。运用不同建模方法构建预测模型,结果会存在很大差异,只有选取合适的建模方法才能构建出高质量的近红外模型。降维方法参数的取值也会影响模型的预测结果,参数优化可以有效地提高模型预测的准确性。试验结果表明:(1)经过SNV+1st-Der+Sym4预处理后的光谱数据分散程度得到降低,同时也抑制了部分噪声信息,光谱数据变得较为平滑。(2)经过参数优化的MLLE+SVR模型,构建的红松籽仁蛋白质定量预测模型质量最优,降维方法优化参数取值为:n-components=4,n-neighbors=50,mean-MSEV为0.568 1,mean-PCCV达0.940 8。(3)运用最优参数模型,对选取的试验当年20粒红松籽仁样品进行蛋白质定量预测,比较预测结果与化学实测结果,得到MAE=0.43%;进一步用MLLE+SVR模型,对往年30粒红松籽仁样品进行蛋白质定量预测,其中往年MLLE降维方法的优化参数为:n-components=4,n-neighbors=50,得到MAE=0.43%。由此可见,采用本研究方法对红松籽仁蛋白质进行定量分析是可行的,该MLLE+SVR模型具有一定的适用性,并且预测结果是可靠的、稳定的。
参考文献:
[1] 马文强,张 漫,李忠新,等. 基于近红外光谱的核桃仁蛋白质含量检测分析[J].农业机械学报,2017,48(S1):407-411.
[2] 刘 洁,李小昱,王 为,等. 基于近红外光谱的板栗蛋白质检测方法研究[C]//汪慰华,朱 明,傅泽田,等. 中国农业工程学会2011年学术年会论文集. 重庆:中国农业工程学会,2011:1653-1656.
[3] 汪庆平,黎其万,董宝生,等. 近红外光谱法快速测定山核桃品质性状的研究[J].西南农业学报,2009,22(3):873-875.
[4] 蒋大鹏,张冬妍,李丹丹,等. 基于近红外的松子蛋白质品质分类处理[J].计算技术与自动化,2018,37(3):180-184.
[5] 仇逊超,曹 军. 近红外光谱波段优化在东北松子蛋白质定量检测中的应用[J].现代食品科技,2016,32(11):303-309.
[6] 刘丽娜,马世伟,芮 玲. 基于可信赖性和连续性的流形降维效果评价方法[J].计算机应用研究,2018,35(6):1707-1711.
[7] 黄建军,李雪梅,滕宏泉. 基于偏最小二乘法的黄土湿陷性评价模型[J].灾害学,2021,36(2):60-64.
[8] 赵思梦,于宏威,高冠勇,等. 花生蛋白组分及其亚基含量近红外分析检测方法[J].光谱学与光谱分析,2021,41(3):912-917.
[9] 方 彦,王汉宁. 近紅外光谱法在玉米粗蛋白含量测定研究中的应用[J].甘肃农业大学学报,2004,39(1):32-35.
[10]邵学广,宁 宇,刘凤霞,等. 近红外光谱在无机微量成分分析中的应用[J].化学学报,2012,70(20):2190-2114.
[11]王培培,张德全,陈 丽,等. 近红外光谱法预测羊肉化学成分的研究[J].核农学报,2012,26(3):500-504.
[12]TSENKOVA R, KOVACS Z, KUBOTA Y. Aquaphotomics: near infrared spectroscopy and water states in biological systems[J]. Subcell Biochem,2015,71:189-210.
[13]曹 璞,潘 涛,陈星旦. 小型近红外玉米蛋白质成分分析仪器设计的波段选择[J].光学精密工程,2007,15(12):1952-1958.
[14]TSUCHIKAWA S, KOBORI H. A review of recent application of near infrared spectroscopy to wood science and technology[J]. Journal of Wood Science,2015,61(3):213-220.
[15]张怡卓,苏耀文,李 超,等. 蒙古栎抗弯弹性模量多模型共识的近红外检测方法[J].林业工程学报,2016,1(6):17-22.
[16]张 银,周孟然. 然近红外光谱分析技术的数据处理方法[J].红外技术,2007,29(6):345-348.
[17]TIAN H, LI M, WANG Y, et al. Optical wavelength selection for portable hemoglobin determination by near-infrared spectroscopy method[J]. Infrared Physics and Technology, 2017,86:98-102.
[18]CORTES V, RODRIGUEZ A, BLASCO J, et al.Prediction of the level of astringency in persimmon using visible and near-infrared spectroscopy[J]. Journal of Food Engineering,2017,204(7):27-37.
[19]张素兰,黄金龙,秦 林,等. 基于高光谱特征的松材线虫岭回归估测模型研究[J].农业机械学报,2019,50(4):196-202.
[20]沈广辉,曹瑶瑶,刘 馨,等. 近红外高光谱成像结合特征波长筛选识别小麦赤霉病瘪粒[J].江苏农业学报,2021,37(2):509-516.
[21]曹立源,范勤勤,黄敬英. 基于特征选择和XGBoost优化的术中低体温预测[J].数据采集与处理,2022,37(1):134-146.
[22]LOPEZ E, GONZALEZ D, AGUADO J V, et al. A manifold learning approach for integrated computational materials engineering[J]. Archives of Computational Methods in Engineering, 2018,25(1):59-68.
(责任编辑:张震林)
