食管鳞癌淋巴结转移的影像组学与预后的关联
2023-05-13陈小琪徐华霞王小玥李锐进邱明链邱模良吴凯明胡志坚
陈小琪, 徐华霞, 王小玥, 李锐进, 邱明链, 邱模良, 吴凯明, 胡志坚
我国是食管癌(esophageal carcinoma, EC)的高发地区,发病率为11.130/100 000,居我国肿瘤发病率第六位;其死亡率为8.280/100 000,居我国肿瘤死亡率第五位[1]。EC的病理类型包括鳞状细胞癌和腺癌,我国约90% EC为食管鳞癌(esophageal squamous cell carcinoma, ESCC)[2]。淋巴结转移(lymph node metastasis, LNM)是ESCC的主要转移方式,是TNM分期中重要的病理特征之一,也是影响ESCC患者总生存期的关键性预后指标[3]。由于独特的淋巴引流方式,EC极易在食管黏膜及黏膜下层丰富的淋巴管网内发生广泛或跳跃性转移[4],具有较高的LNM发生率[5],导致患者预后不佳。因此,识别ESCC患者是否发生LNM并评估患者预后显得尤为重要。
影像组学作为一种非侵入性方法,能够从医学影像中挖掘出定量特征,并结合统计学方法筛选出最有价值的影像特征,用于疾病的诊断、疗效评估及预后预测,包括乳腺癌、肺腺癌、结直肠癌等恶性肿瘤的诊断及预后评估[5-6],特别是用于识别原发性肿瘤是否伴有LNM[7]。已有研究[8-9]证实,影像组学在预测ESCC LNM和预后方面的潜在价值。然而,这些研究仅仅关注原发性肿瘤的影像特征,未探讨淋巴结影像特征与LNM的关联。同时,这些研究大多使用人工手动法对影像特征进行分割与提取[10-12]。人工手动分割不仅耗时,且受经验与主观因素影响比较大,难以适用于大规模的病例研究[12]。与人工手动分割相比,用卷积神经网络(convolutional neural networks, CNN)获取到的特征可以让复杂的内部信息得到更好的描述,能够发现更深层次的特征[13],适用于大数据研究。本研究基于CNN分割提取到的淋巴结影像组学特征,探讨LNM相关的影像组学特征与ESCC LNM及预后的关联,为ESCC淋巴结的识别及预后判断提供理论依据。
1 对象与方法
1.1 对象 收集2014年6月—2021年9月福建医科大学附属第一医院308例新发ESCC患者,其中淋巴结未转移者154例,LNM者154例。将研究对象按照2∶1随机分为训练集(n=206)和验证集(n=102)。纳入标准:(1)经手术病理组织学确诊的原发性ESCC;(2)术前未接受放、化疗;(3)在福建省居住10 a以上。排除标准:(1)经病理诊断确诊为非原发性ESCC、复发性ESCC患者;(2)伴有肝肾功能不全或急慢性感染;(3)病情危重不能清晰回答问题者。本研究通过福建医科大学伦理委员会批准(审批号:201495),患者及家属均知情同意。
1.2 影像资料收集 影像资料来源于福建医科大学附属第一医院胸外科。采用Toshiba Aquilion One 320排CT机,从胸廓入口到肺底,进行胸部螺旋扫描。扫描参数:根据医院统一标准执行。所有患者数据格式均保持一致,采用digital imaging and communications in medicine (DICOM)格式。
1.3 影像组学特征提取与筛选
1.3.1 影像资料标记 根据影像指南统一标记标准,由2位具有5 a以上工作经验的影像科医生使用MRIcroGL软件标记ESCC患者CT影像资料中的淋巴结。
1.3.2 图像分割 为确保分割的准确性及节省人力物力,本研究使用CNN对标记的淋巴结区域进行自动分割,进而提取ESCC淋巴结的影像组学特征。
1.3.3 影像资料提取 为减少各种CT扫描仪的影响,统一将DICOM图像转换为灰度值图像,并对每个患者自动提取感兴趣区(region of interest, ROI)的影像特征进行进一步分析,使用CNN自动提取ESCC的CT淋巴结特征,共提取到999个影像组学特征值用于后续研究。
1.4 统计学处理 采用SPSS 24.0软件及R(4.10)软件进行统计分析。使用χ2检验比较2组患者临床特征的分布情况。本研究使用CNN对标记的淋巴结区域进行自动分割,并自动提取ESCC淋巴结的影像组学特征;进而分别使用LASSO回归和随机森林筛选影像组学特征构建ESCC LNM的预测模型。根据最佳λ纳入LASSO回归筛选的影像组学特征;根据十折交叉验证拟合的模型误差及increase in mean squared error(%IncMSE)纳入随机森林筛选的影像组学特征。以曲线下面积(area under the curve, AUC)作为判断模型的效能;并使用Delong法比较,选择最佳模型进行后续研究;根据Cox回归(向后法)算法所得回归系数构建与筛选出ESCC预后相关的影像组学标签(radiomics signature),并使用Cox回归探讨影像组学标签与ESCC预后的关联;以C指数评价由多因素Cox回归分析变量所建列线图的预测能力。
2 结 果
2.1 临床特征比较 训练集和验证集在性别、年龄、肿瘤部位、肿瘤直径、分化程度和T分期分布均衡可比(P>0.05,表1)。

表1 训练集和验证集患者的临床特征比较
2.2 筛选与ESCC LNM相关影像组学特征构建ESCC LNM的预测模型
2.2.1 使用LASSO回归筛选影像组学特征构建ESCC LNM的预测模型 使用LASSO回归筛选影像特征,最佳λ为0.024(图1)。使用LASSO回归,共筛选出19个影像特征,分别为:173、417、457、473、504、521、546、583、585、611、678、695、761、812、884、905、932、974和987。

CNN:卷积神经网络。
使用上述19个影像特征构建预测模型,在全模型、训练集和验证集中,其AUC (95% CI)分别为:0.747(0.694,0.801)、0.751(0.686,0.817)和 0.766(0.672,0.860)(图2)。
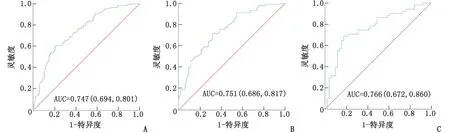
ESCC:食管鳞癌;LNM:淋巴结转移;AUC:曲线下面积。A:全模型;B:训练集;C:验证集。
2.2.2 使用随机森林筛选影像组学特征构建 ESCC LNM的预测模型 根据随机森林计算结果(图3)。以十折交叉验证曲线中模型误差的稳定性及%IncMSE作为筛选影像特征的依据,选择排序为前9位的影像特征用于构建ESCC LNM预测模型,9个影像特征分别为987、306、505、438、111、546、799、566和850。
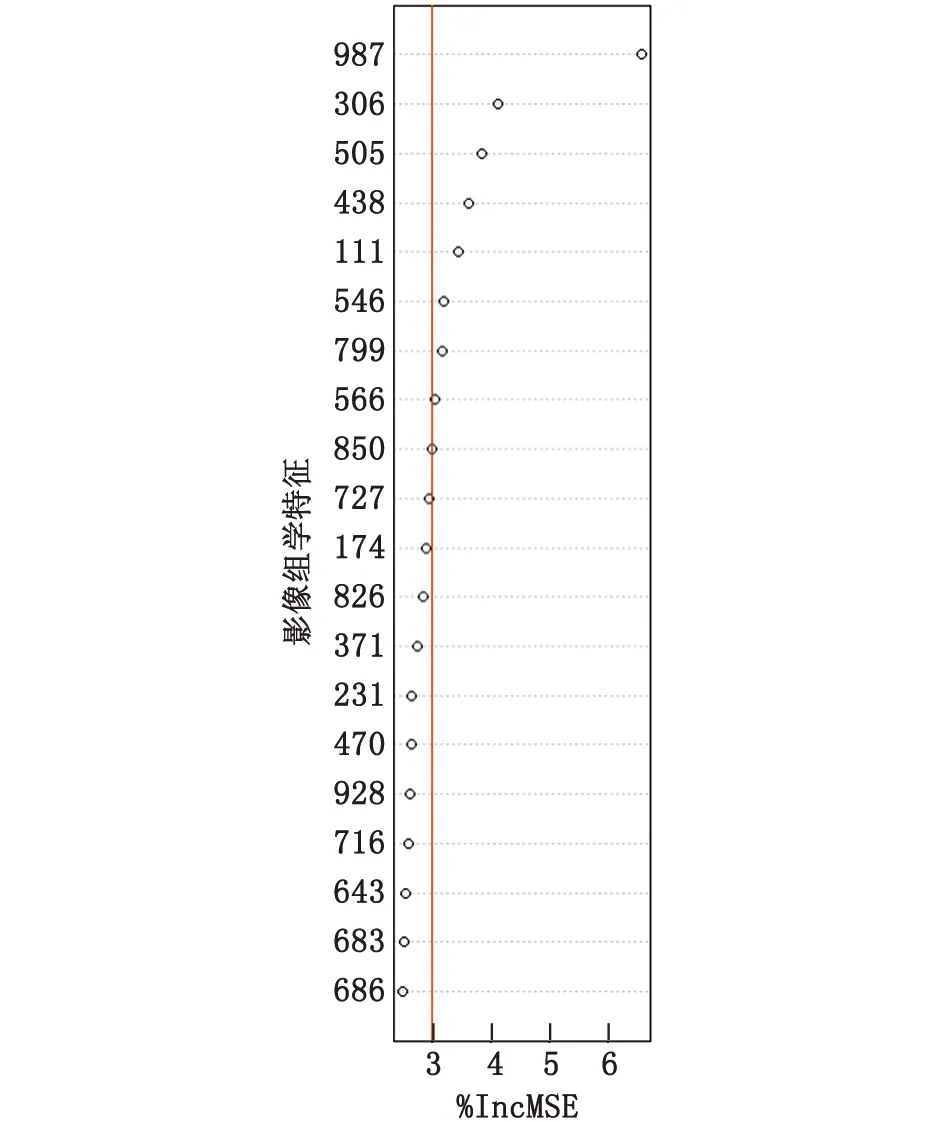
%IncMSE:增长的错误率平方均值;CNN:卷积神经网络。
使用上述9个影像特征构建预测模型,在全模型、训练集和验证集中,其AUC (95% CI)分别为:0.692(0.633,0.751)、0.683(0.610,0.755)和 0.723(0.624,0.822)(图4)。

ESCC:食管鳞癌;LNM:淋巴结转移。AUC:曲线下面积。A:全模型;B:训练集;C:验证集。
2.2.3 筛选最佳预测模型 使用Delong法比较LASSO回归与随机森林筛选的影像组学特征构建的ESCC LNM预测模型。分析发现,使用LASSO回归所构建模型的AUC优于使用随机森林所构建的模型(Z=-3.955,P<0.001)。因此,本研究选择使用LASSO回归筛选的变量进行后续的分析。
2.3 使用ESCC LNM相关的影像组学特征构建ESCC预后相关的影像组学标签 通过Cox回归(向后法)算法进一步筛选出与ESCC预后相关的影像组学特征,根据回归系数构建影像组学标签,具体计算方式如下:
影像组学标签=β1X1+β2X2+β3X3+…+βnXn
(1)
式中Xn表示影像组学特征变量,βn表示所对应的回归系数[14]。
2.4 影像组学标签与ESCC预后的关联分析 本研究对象的生存时间中位数为 17.430 个月,1、3及5 a生存率分别为77%(95% CI:0.720, 0.830)、52%(95% CI:0.450, 0.590)和42%(95% CI:0.310, 0.520)。根据影像组学标签的中位数,将其分为低风险影像组学标签组和高风险影像组学标签组。
调整性别、年龄、肿瘤直径、分化程度、T分期、术后放化疗和影像组学标签,多因素Cox回归分析发现,高风险的影像组学标签与ESCC不良的预后相关(P<0.05,表2)。

表2 多因素LASSO Cox回归分析
2.5 列线图 使用多因素Cox回归分析的变量构建列线图(图5,图6),影像组学标签能很好地预测食管癌的预后,总生存率(overall survival, OS)C指数(95% CI)在全模型、训练集和验证集中分别为0.710(0.670,0.749)、0.765(0.742,0.788)和0.652(0.568,0.736),无病进展生存率(disease free survival,DFS)C指数(95% CI)在全模型、训练集和验证集中分别为0.775(0.746,0.804)、0.754(0.726,0.781)和0.774(0.760,0.789)。

A:全模型;B:训练集;C:验证集。OS:总生存率。

A:全模型;B:训练集;C:验证集。DFS:无病进展生存率。
3 讨 论
影像组学在医疗领域应用越来越广泛,其在疾病的辅助诊断、疗效评估和预后预测方面已有初步效果[8-9]。本研究通过CNN提取影像组学特征,并使用LASSO回归筛选与ESCC LNM相关的影像组学特征,构建ESCC LNM的预测模型,发现该模型识别LNM的效果较好(AUC=0.750, 95% CI:0.690,0.800)。同时,多因素Cox回归分析发现,高风险影像组学标签与ESCC不良预后有关。此外,列线图结果显示,影像组学标签在预测ESCC预后方面有较高的价值。
近年来,多项研究[5,15-17]证实影像组学在LNM的预测价值。如ZHAO等[18]基于ESCC患者临床特征及计算机断层扫描提取到的影像组学特征,构建LNM预测模型,训练集和验证集的AUC分别为0.870和0.840。赵博[19]的研究发现,基于CT影像组学可以较好地预测ESCC LNM,且对“隐匿”LNM的预测也有一定的潜力,训练组和验证组的AUC分别为0.870和0.840。然而,上述研究大多采用手动分割对CT影像资料进行图像分割和特征提取,耗时又耗力,且手动分割存在分割不准确、对边界判断具有主观性等问题[10-12]。本研究通过CNN来分割并提取ESCC LNM的影像组学特征,其分割精准,且能节省时间,并择优选择LASSO回归对提取到的999个特征值进行筛选,共筛选出19个影像特征并构建预测LNM的预测模型,其AUC (95% CI)在全模型、训练集和验证集中分别为0.747(0.694,0.801)、0.751(0.686,0.817)和0.766(0.672,0.860)。
使用Cox回归筛选与预后相关的影像组学特征,并根据回归系数构建影像组学标签,根据中位数将其分为低风险和高风险影像组学标签,结果显示,与低风险影像组学标签比较,高风险影像组学标签与不良预后相关,说明影像组学标签与ESCC预后存在关联,可用于ESCC预后的预测,与文献 [8-9] 结论一致。此外,基于影像组学标签,构建ESCC预后预测的列线图,分析发现总OS C指数(95% CI)在全模型、训练集和验证集中分别为0.710(0.670,0.749)、0.765(0.742,0788)和0.652(0.568,0.736),DFS C指数(95% CI)分别为0.775(0.746,0.804)、0.754(0.726,0.781)和0.774(0.760,0.789),说明影像组学标签能较好地预测ESCC的预后。但本研究为小样本、单中心研究,模型的外推使用及稳定性有待进一步进行多中心、大样本的验证。
