整合宏基因组学分析生猪养殖粪污发酵过程中耐药基因的动态变化
2023-05-05赵素君李江凌张金灵王秋实于吉锋
赵素君,曹 冶,李江凌,张金灵,刘 锐,王秋实,于吉锋
(1.四川省畜牧科学研究院,成都 610066;2.动物遗传育种四川省重点实验室,成都 610066)
【研究意义】抗生素是可以抑制或杀死细菌的小分子,这些小分子通常被用于治疗细菌感染。由于抗生素使用不当,造成大量细菌产生抗生素抗性,这些抗性主要是通过耐药基因形成。耐药基因是编码耐药性状的一段核苷酸序列(DNA片段)。与其他遗传物质一样,耐药基因在细菌分裂增殖过程中得到复制。耐药基因可位于细菌的染色体上,也可位于染色体外的质粒上,质粒携带的耐药基因可通过接合、转化、转导等方式在同种细菌甚至不同种细菌的菌株之间传播。耐药基因在环境中的大量存在和抗生素残留已严重威胁到人类健康。据报道,世界上每年有多达70万人因耐药性问题死亡[1]。实际上,多个国家和全球公共卫生组织把抗菌素耐药性归为迫在眉睫的危险,并一致认为,追踪其耐药性和耐药菌流行对最大限度地减少其对人类健康的威胁至关重要。堆肥是一种常用的养殖场固体废弃物处理技术,这种技术除了在降解有机废弃物方面行之有效外,还能有效降解抗生素及消减耐药基因,充分了解堆肥过程中抗生素及耐药基因消减的规律、影响因素,不仅对养殖场固体废弃物利用具有重要意义,也为最大限度减少抗生素及耐药基因扩散风险提供重要参考。【前人研究进展】由于抗生素的使用不当,导致养殖场固体废弃物中微生物耐药基因出现和高浓度残留[2]。若未经有效处理便直接将大量耐药基因和耐药菌输入到环境中,将导致卫生事件的发生。堆肥是一种有效的处理方式,可以大幅降低沼渣[3]和城市固体垃圾[4]以及大部分畜禽粪污[5-6]等有机固废中的耐药基因。但部分耐药基因,特别是sul1在畜禽废弃物[7]和城市污泥[8]等多种有机固废堆肥中都很难清除,且堆肥腐熟期往往发生反弹[9],导致出现消减效率较低甚至在堆肥后不降反升等问题[10]。【本研究切入点】大多研究是对耐药基因在具体试验处理中的消减特征进行表述,但不同堆肥试验中耐药基因的消减效果往往差别较大,且耐药基因种类单一,其具体分子机理尚不明确。【拟解决的关键问题】本研究采用四川大量存在的生猪养殖粪污为材料,用宏基因组学分析方法来探讨堆肥对耐药基因的消减规律,以期全面了解四川生猪养殖粪污中各种耐药基因的消减规律,同时对其消减的机理进行探讨,对生猪的安全生产和畜牧业的可持续发展具有重大意义。
1 材料与方法
1.1 菌种
有效微生物(EM)菌由中国农业科学院肥料应用工程技术研究中心提供,包括芽孢杆菌、纳豆菌、放线菌、酵母菌和木霉菌等多种有益微生物及各种分泌性胞外酶类,有效活菌数>1×1010个/mL。
1.2 主要试剂和仪器
粪便DNA提取试剂盒(Stool DNA Isolation Kit)、样品均质研磨仪(MP FastPrep-24)、Illumina Hiseq 4000测序仪(Illumina)、Qubit®3.0 Fluorometer(Invitrogen Qubit 3)、安捷伦2100生物分析仪(Agilent 2100)、全自动紫外凝胶成像系统(BIO-RAD Gel Doc XR+)。
1.3 方法
1.3.1 堆肥制备 在四川省畜牧科学研究院养猪所养猪基地,将同一猪圈的新鲜猪粪进行粪尿干湿分离,分离后的干猪粪混合均匀,再随机分为添加EM菌的实验组和不添加EM菌的对照组,两组分别堆成圆锥形粪堆,粪堆高1.2 m,底部直径2 m,并保持无其他污物混入。实验组按照1 t粪便1 kg EM菌发酵的比例添加EM菌,添加时先根据粪便的干湿度,用水稀释EM菌原液(稀释比例1∶10~1∶20),洒施并均匀混合,最终粪便湿度控制在40%~50%。
1.3.2 样品采集及总DNA提取 在堆肥第1、4、8、12、16、22、29天采样。每次分别在堆肥上层(离顶部5 cm)、中层(离顶部35 cm)和底层(离底部5 cm)处按照多点采集原则采集样品,然后将样品全部混合,每次采集样本的总混合量为500 g,采集后的样品立即置于冰盒中运往实验室,-20 ℃保存,按DNA提取试剂盒说明书操作步骤提取和纯化样品总DNA。提取DNA用于宏基因组高通量测序分析。
1.3.3 文库构建和上机测序 取1 μg DNA样品,使用NEB Next®Ultra DNA Library Prep Kit for Illumina (NEB, USA)进行文库构建,用Covaris超声波破碎仪随机打断成长度约为350 bp的片段,依次将DNA片段的末端进行修复、添加Poly A尾、在片段两端添加测序接头、纯化DNA片段、PCR扩增片段等来完成整个文库制备。
文库构建完成后,须进行文库质量检测。先使用Qubit 3.0对文库进行初步定量,稀释文库至2 ng/μL,随后使用Agilent 2100对文库的insert size进行检测,insert size符合预期后,使用Q-PCR方法对文库的有效浓度进行准确定量(文库有效浓度>3 nmol/L),以保证文库质量。库检合格后,把不同文库按照有效浓度及目标下机数据量的需求pooling后用Illumina Hiseq 4000测序。
1.3.4 数据处理 使用Readfq (V8, https://github.com/cjf!elds/readfq)对Illumina HiSeq测序平台获得的原始数据(Raw data)进行预处理,获得有效数据(Clean data)。使用MEGAHIT软件(v1.0.4-beta)对Clean data进行组装分析,将组装得到的Scaffolds从N连接处打断,得到不含N的Scaftigs。使用MetaGeneMark (v2.10, http://topazatech.edu/GeneMark/)对各样品的Scaftigs (≥500 bp)进行开放阅读框(ORF)预测,并过滤掉预测结果中长度小于100 nt的信息。对ORF预测结果,采用CD-HIT软件(v4.5.8, http://www.bioinformatics.org/cd-hit/)进行去冗余,获得非冗余的初始gene catalogue。使用Bowtie2 (Bowtie2.2.4)将各样品的Clean data比对至初始gene catalogue,计算得到基因在各样品中比对上的reads数目,过滤掉各个样品中reads数目≤2的基因,获得最终用于后续分析的gene catalogue (Unigenes)。依据比对上的reads数目及基因长度,计算得到各基因在各样品中的丰度信息,基于gene catalogue中各基因在各样品中的丰度信息,进行基本信息统计等分析。
使用CARD数据库提供的Resistance Gene Identifier(RGI)软件将Unigenes与CARD数据库(https://card.mcmaster.ca/)进行比对(默认evalue <1E-30),根据比对结果,结合Unigenes的丰度信息,统计出各抗生素耐药性本体(ARO)的相对丰度。
2 结果与分析
2.1 测序数据质控及基因组装结果
样品经初检合格后,进行文库构建、检测并采用Illumina PE150进行测序。测序得到的原始数据(Raw data)进行质控及宿主过滤,得到有效数据(Clean data)。由表1可知,检测的Clean Q20[Clean data中测序错误率小于0.01(质量值大于20)的碱基数目的百分比]达96.7%以上,Clean Q30[Clean data 中测序错误率小于0.001(质量值大于30)的碱基数目的百分比]达91%以上,有效率[有效数据( Clean data)与原始数据( Raw data)的百分比]达99%以上,说明数据真实可靠。

表1 数据预处理统计Table 1 Data preprocessing statistics
采用Bowtie2软件比对至各样品组装后的Scaftigs上,过滤掉500 bp以下的片段,并进行统计分析。由表2可知,样品中Scaftigs数量达200 000以上,平均长度达1080 bp以上,N50长度达1100 bp以上,N90长度达560 bp以上。说明,组装分析准确可靠。
2.2 堆肥期间耐药基因的变化
使用CARD数据库提供的Resistance Gene Identifier (RGI)软件[11-12]将Unigenes与CARD数据库(https://card.mcmaster.ca/)进行比对,结合Unigenes的丰度信息,共发现613个耐药基因,分别对32类抗生素产生耐药(表3)。

表3 堆肥期间耐药基因丰度变化Table 3 Changes of antibiotic resistance gene during composting

续表3 Continuedtable 3
对照组在第1、4、8、12、16、22、29天的耐药基因总丰度比例分别为3.10E-03、1.13E-03、2.17E-03、2.54E-03、9.84E-04、8.79E-04、6.96E-04,耐药基因丰度在第1天最高,第4天迅速下降,在第8天迅速反弹,在第12天达到第二个高峰,然后迅速下降,丰度回归低值。实验组在第1、4、8、12、16、22、29天的耐药基因总丰度比例分别为3.93E-03、1.43E-03、3.11E-03、1.36E-03、1.14E-03、7.41E-04、6.94E-04,其变化趋势与对照组类似,不过第二个峰值提前到第8天。
对照组在第1、4、8、12、16、22、29天的丰度较高的耐药基因类型分别为:青霉烷类抗生素、头孢菌素类抗生素、四环素类抗生素,磷霉素、头孢菌素类抗生素、青霉烷类抗生素,青霉烷类抗生素、头孢菌素类抗生素、四环素类抗生素,青霉烷类抗生素、头孢菌素类抗生素、氨基糖苷类抗生素,头孢菌素类抗生素、糖肽类抗生素、青霉烷类抗生素,头孢菌素类抗生素、青霉烷类抗生素、糖肽类抗生素,磷霉素、头孢菌素类抗生素、青霉烷类抗生素。而实验组在第1、4、8、12、16、22、29天的丰度较高的耐药基因类型分别为:青霉烷类抗生素、头孢菌素类抗生素、氨基糖苷类抗生素,磷霉素、青霉烷类抗生素、头孢菌素类抗生素,青霉烷类抗生素、头孢菌素类抗生素、氨基糖苷类抗生素,青霉烷类抗生素、头孢菌素类抗生素、磷霉素,青霉烷类抗生素、头孢菌素类抗生素、氨基糖苷类抗生素,磷霉素、头孢菌素类抗生素、糖肽类抗生素,磷霉素、头孢菌素类抗生素、青霉烷类抗生素。
2.3 堆肥期间青霉烷类抗生素(Penam)耐药基因的变化
共发现133个对青霉烷类抗生素产生抗性的耐药基因。按其耐药产生原理,共有5个类型。分别为抗生素外排(Antibiotic efflux)、抗生素失活(Antibiotic inactivation)、降低抗生素渗透性(Reduced permeability to antibiotic)、改变抗生素靶位点(Antibiotic target alteration)、替换抗生素靶位点(Antibiotic target replacement)。抗生素外排类型基因共有76个,抗生素失活类型基因共有90个,降低抗生素渗透性类型基因共有7个,改变抗生素靶位点类型基因共有8个,替换抗生素靶位点类型基因共有7个。这些类型的基因丰度在第1天最高,在第4天丰度迅速下降,然后丰度再次升高,实验组的各个类型耐药基因和对照组降低抗生素渗透性类型基因、改变抗生素靶位点类型基因、替换抗生素靶位点类型基因在第8天达到第二个高峰,然后持续下降。对照组抗生素外排类型基因和抗生素失活类型基因,在第12天达到第二个高峰,然后持续下降(图1)。

图1 堆肥期间青霉烷类抗生素(Penam)耐药基因变化Fig.1 Changes of Penam resistance genes during composting
2.4 堆肥期间头孢菌素类抗生素(Cephalosporin)耐药基因的变化
共发现141个对头孢菌素类抗生素产生抗性的耐药基因。按其耐药产生原理,共有5个类型。分别为抗生素外排、抗生素失活、降低抗生素渗透性、改变抗生素靶位点、替换抗生素靶位点。抗生素外排类型基因共有35个,抗生素失活类型基因共有100个,降低抗生素渗透性类型基因共有7个,改变抗生素靶位点类型基因共有8个,替换抗生素靶位点类型基因共有7个。这些类型的基因丰度在第1天最高,在第4天丰度迅速下降,然后丰度再次升高,实验组的各个类型耐药基因和对照组降低抗生素渗透性类型基因、改变抗生素靶位点类型基因、替换抗生素靶位点类型基因在第8天达到第二个高峰,然后持续下降。对照组抗生素外排类型基因和抗生素失活类型基因,在第12天达到第二个高峰,然后持续下降(图2)。

图2 堆肥期间头孢菌素类抗生素(Cephalosporin)耐药基因变化Fig.2 Changes of Cephalosporin resistance genes during composting
2.5 堆肥期间氨基糖苷类抗生素(Aminoglycoside antibiotic)耐药基因的变化
共发现102个对氨基糖苷类抗生素产生抗性的耐药基因。按其耐药产生原理,共有3个类型。分别为抗生素外排、抗生素失活、改变抗生素靶位点。抗生素外排类型基因共有22个,抗生素失活类型基因共有74个,改变抗生素靶位点类型基因共有6个。这些类型的基因丰度在第1天最高,在第4天丰度迅速下降,然后丰度再升高,实验组的耐药基因丰度在第8天达到第二个高峰,而对照组的耐药基因丰度在第12天达到第二个高峰,然后持续下降(图3)。
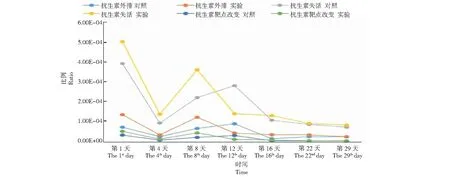
图3 堆肥期间氨基糖苷类抗生素(Aminoglycoside antibiotic)耐药基因的变化Fig.3 Changes of Aminoglycoside antibiotic resistance genes during composting
2.6 堆肥期间磷霉素类抗生素(Fosfomycin)耐药基因的变化
共发现7个对磷霉素类抗生素产生抗性的耐药基因。按其耐药产生原理,共有3个类型,分别为抗生素外排、抗生素失活、改变抗生素靶位点。抗生素外排类型基因共有1个,抗生素失活类型基因共有5个,改变抗生素靶位点类型基因共有1个。抗生素失活类型基因丰度在第4天最高,在第8天丰度迅速下降,实验组的丰度在第16天达到第二个高峰,而对照组的耐药基因丰度在第12天才达到第二个高峰,然后持续下降,维持在一个较高的水平上。其它2种类型基因丰度在第1天最高,在第4天丰度迅速下降,然后丰度再次升高,实验组的耐药基因丰度在第8天达到第二个高峰,而对照组的耐药基因丰度在第12天达到第二个高峰,然后持续下降(图4)。
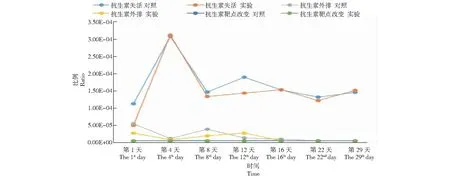
图4 堆肥期间磷霉素类抗生素(Fosfomycin)耐药基因变化Fig.4 Changes of Fosfomycin resistance genes during composting
2.7 堆肥期间糖肽类抗生素(Glycopeptide antibiotic)耐药基因的变化
共发现43个对糖肽类抗生素产生抗性的耐药基因。按其耐药产生原理,只有1个类型,为改变抗生素靶位点。该类型的基因丰度在第1天最高,在第4天丰度迅速下降,然后丰度再次升高,实验组的耐药基因丰度在第8天达到第二个高峰,对照组的耐药基因丰度在第12天达到第二个高峰,然后持续下降(图5)。
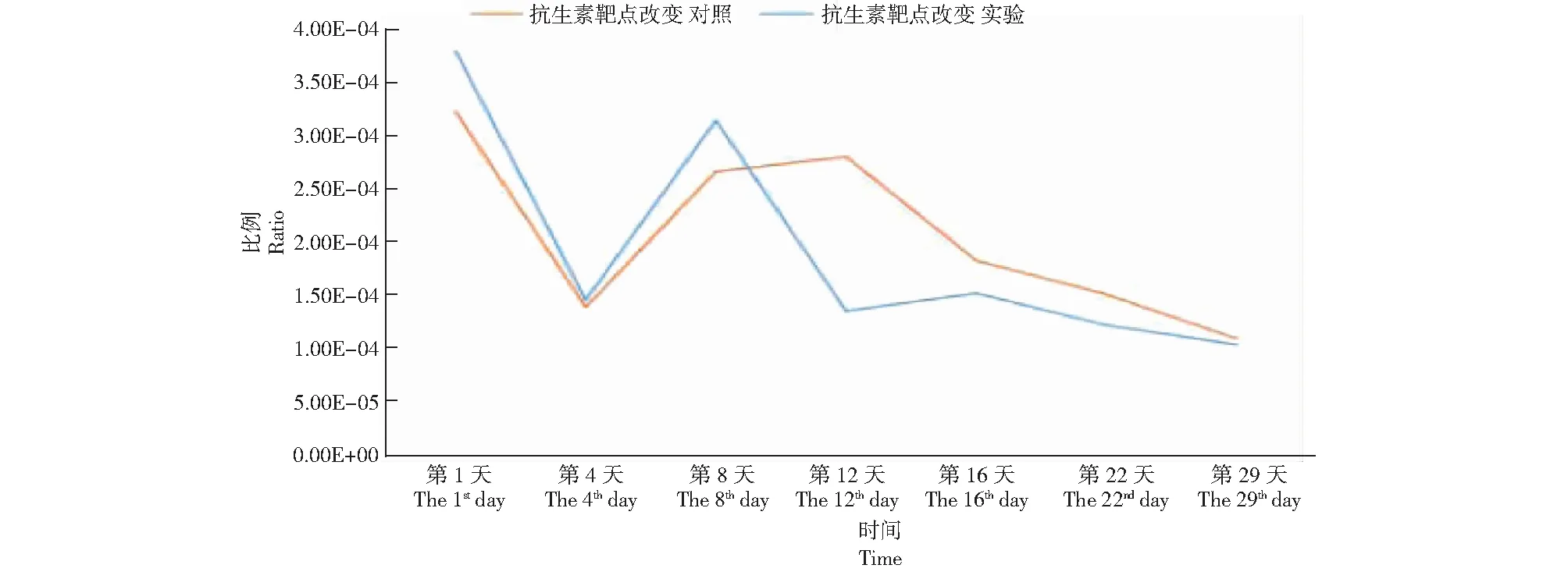
图5 堆肥期间糖肽类抗生素(Glycopeptide antibiotic)耐药基因变化Fig.5 Changes of Glycopeptide antibiotic resistance genes during composting
2.8 堆肥期间四环素类抗生素(Tetracycline antibiotic)耐药基因的变化
共发现91个对四环素类抗生素产生抗性的耐药基因。按其耐药产生原理,共有5个类型,分别为抗生素外排、抗生素失活、降低抗生素渗透性、改变抗生素靶位点、抗生素靶位点保护。抗生素外排类型基因共有77个,抗生素失活类型基因共有2个,降低抗生素渗透性类型基因共有3个,改变抗生素靶位点类型基因共有7个,抗生素靶位点保护类型基因共有12个。这些类型的基因丰度在第1天最高,在第4天丰度迅速下降,然后丰度再次升高,实验组的各个类型耐药基因和对照组改变抗生素靶位点、降低抗生素渗透性、抗生素失活类型基因,在第8天达到第二个高峰,然后持续下降。对照组抗生素外排类型和降低抗生素渗透性类型基因,在第12天达到第二个高峰,然后持续下降(图6)。
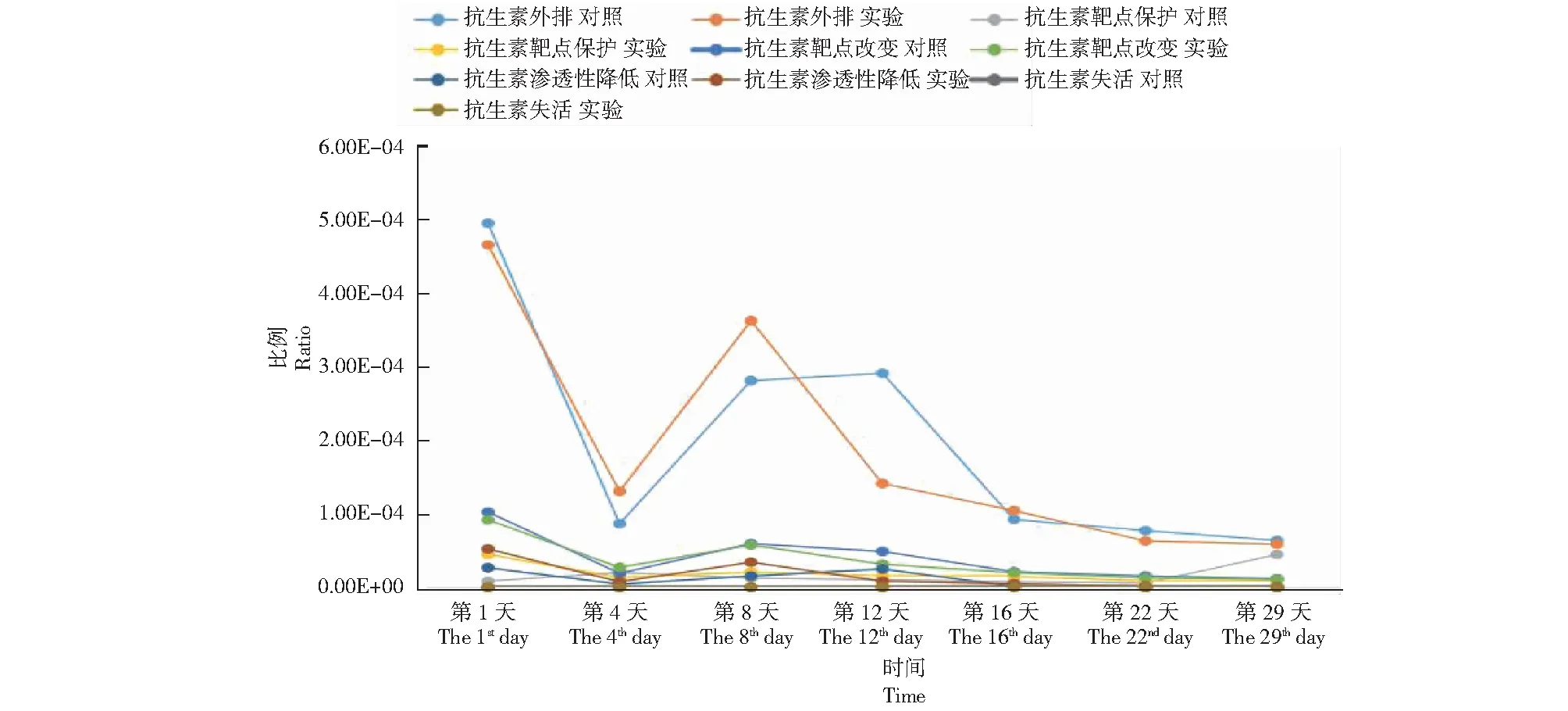
图6 堆肥期间四环素类抗生素(Tetracycline antibiotic)耐药基因变化Fig.6 Changes of Tetracycline antibiotic resistance genes during composting
3 讨 论
传统环境耐药性基因检测通过PCR反应(Polymerase chain reaction)[13]、分子杂交[14]等方法来实现。但这些方法需要依赖目标基因的保守区域设计引物或DNA探针来实现,一次性检测量较少,操作比较麻烦。而宏基因组学技术,避免了这些麻烦,且可以用来鉴定新的耐药基因,其通量高、速度快,具有传统方法所不能具备的优势,因此在本次研究中采用了该方法来进行研究。
本次研究中发现养殖废弃物经堆肥处理后,耐药基因的丰度下降到低值。说明,堆肥可有效降低粪污中的耐药基因,避免耐药性的广泛传播。这与一些研究[6,15]发现在粪污堆肥发酵过程中,大部分耐药基因的相对丰度有所降低的结果相似。
但本研究发现养殖废弃物中的耐药基因的消减出现两个峰值,第一个耐药基因丰度峰值出现在试验起始时,在第4天迅速下降。这可能是由于耐药基因大部分由样品原始菌群中的耐药菌提供,而这些原始菌群经4 d发酵,大量自然凋亡,在菌群中的比例下降,新的耐药菌还未大量产生,从而导致样品中耐药基因的丰度下降到低值。随着发酵的继续进行,样品中细菌数量进一步增加,新的耐药菌也大量出现,所以对照组第12天、实验组第8天的耐药基因丰度又达到第二个峰值,然后这一批细菌又大量自然凋亡,样品中耐药基因的丰度又下降到低值。这说明,养殖废弃物中的耐药基因的消减可能随着废弃物中的细菌凋亡规律而出现波状起伏,耐药基因的丰度一波比一波低,最终消减下去。研究发现耐药基因的消减是通过改变堆肥过程中的微生物菌群来实现[7,16-18],这与本研究的结果一致。但是可能是由于这些研究的采样周期设计较长,从而忽略了耐药基因消减的波状起伏规律。
研究发现在养殖废弃物中有大量的抗生素耐药基因存在,如四环素类抗生素(Tetracycline antibiotic)、磺胺类抗生素(Sulfonamide antibiotic)、氟喹诺酮类抗生素(Fluoroquinolone antibiotic)、内酰胺类抗生素(Monobactam)、大环内酯类抗生素(Macrolide antibiotic)等,各个样品中耐药基因的种类及丰度并不相同,这可能与当地使用的抗生素种类及剂量有关[19-25]。在四川,青霉烷类抗生素(Penam)、头孢菌素类抗生素(Cephalosporin)、氨基糖苷类抗生素(Aminoglycoside antibiotic)、糖肽类抗生素(Glycopeptide antibiotic)、四环素类抗生素(Tetracycline antibiotic)、磷霉素类抗生素(Fosfomycin)是现实中最常用的抗生素种类,使用范围广、使用量大。长期使用的结果导致细菌耐药性的广泛产生,从而使这些抗生素耐药菌在环境中占比较高。在本次研究中,发现这几种抗生素耐药基因是丰度较高的耐药基因,本研究的分析结果与上面推断一致。
磷霉素类抗生素(Fosfomycin)是一种小分子广谱抗菌药物,对多种革兰阴性、革兰阳性菌均有良好抗菌作用,于1969年被首次报道。目前有3种磷霉素制剂,即口服制剂磷霉素钙、磷霉素氨丁三醇和静脉制剂磷霉素钠,主要用于治疗肠道感染和尿路感染等。细菌暴露于中、低剂量磷霉素中极易发生诱导耐药[26]。在本研究中发现磷霉素类抗生素耐药基因丰度一直维持在较高的水平,主要通过靶位点失活来产生耐药性。这可能是由于样品中存在低剂量的磷霉素,而这些磷霉素在堆肥过程中不易降解,导致细菌持续暴露在低剂量磷霉素中,从而导致耐药。
4 结 论
养殖废弃物经堆肥处理后,耐药基因的丰度呈现波动式消减,最终下降到低值,堆肥可有效避免耐药性的广泛传播。
