SNP芯片评估柯尔克孜羊群体遗传多样性和遗传结构
2023-02-27李隐侠牙生江那斯尔赛里克都曼曹少先王伟列孟春花张建丽
李隐侠,牙生江·那斯尔,赛里克·都曼,钱 勇*,曹少先,王伟列,孟春花,张 俊,张建丽
(1.江苏省农业科学院畜牧研究所, 南京 210014; 2.新疆克孜勒苏柯尔克孜自治州克州畜牧兽医局, 阿图什 845350;3.新疆克孜勒苏柯尔克孜自治州祥泰牧业有限公司, 阿图什 845350)
柯尔克孜羊是新疆地方特色绵羊品种,以四肢高长、体型匀称、紧凑而著称,具有耐粗饲、抗病和抗逆性强、肉质好、风味独特等优良特性[1-2]。研究发现,柯尔克孜羊体格较大,周岁公羊平均体重为36.65 kg,体高为64.77 cm,体长为65.63 cm[3]。柯尔克孜羊公羊体重与体尺指标均大于母羊,且体重与各项体尺相关性显著[4]。柯尔克孜羊肉粗脂肪、粗蛋白、不饱和脂肪酸、油酸、亚油酸和微量元素(钙、铜和铁)含量优势明显,重金属含量低于国家标准,在生产有机食品上具有品种优势[5-6]。虽然柯尔克孜羊从上世纪90年代已经开始选育,但是从未对其进行过系统保种、遗传多样性和遗传结构评估等工作,导致其品种特性退化严重,现有群体遗传结构不清晰,严重阻碍了柯尔克孜羊的资源保护和利用。
单核苷酸多态性(single nucleotide polymorphism, SNP)是最早广泛应用于基因组水平的遗传标记基因分型方法,通过对群体内的个体进行全基因组范围内的SNPs位点分析,得到群体的遗传多样性、亲缘关系及家系结构[7-9]。SNP芯片具有密度大、覆盖广、价格低等优点,是现阶段重要的育种工具之一,在猪[10-11]、牛[12-13]、山羊[14-15]上均有广泛应用。利用SNP芯片评估马身猪保种群的遗传结构,发现其遗传多样性丰富,但近交程度高,家系较少,需要从原种场引入新的血统,扩大保种群数量,降低近交系数[10]。基于SNP芯片对里岔黑猪进行遗传多样性与遗传结构分析,表明里岔黑猪遗传多样性丰富,但群体近交现象明显[11];利用SNP芯片信息评估新疆近交牛基因组纯合度,为新疆近交牛遗传资源的育种规划及未来该群体的开发利用提供了科学依据[12]。基于高通量SNP芯片建立了一种可评估牛早期胚胎染色体质量和生产性能的方法,为胚胎牛育种提供了技术支撑[13],使用60K山羊SNP 芯片分析云上黑山羊的遗传结构,显示其在基因组水平具备较好的品种一致度和群体稳定性,已具备独立的品种特点[14];利用50K芯片解析巴基斯坦山羊品种的遗传多样性和群体结构,为巴基斯坦国家育种计划提供了宝贵的基因库[15]。
ROH(runs of homozygosity)指长的纯合基因型的连续片段,被定义为来自同一个祖先,是使用基因型评估遗传多样性的最新方法[16-18]。基于ROH的基因组近交系数FROH与基于系谱的近交系数相比更接近于真实的近交系数[19],长ROH片段反映最近世代发生过近交,而短ROH说明较远世代产生的近交[20-22]。
新疆克州阿图什市柯尔克孜羊良繁中心现有种公羊31只,能繁母羊1 000余只,其遗传多样性、亲缘关系和家系结构并不清晰,不利于后续保种和选育工作的开展。本试验采用高密度SNP芯片对该柯尔克孜羊群体遗传多样性和遗传结构进行分析,为柯尔克孜绵羊的保护、选育及种质创新利用等提供分子水平的参考依据。
1 材料与方法
1.1 试验动物
本研究中柯尔克孜羊来自新疆克州阿图什市柯尔克孜羊良种繁育中心,共采集61头年龄2~3岁种羊,其中公羊31头,母羊30头,所有个体的饲养标准和环境条件一致。采集61头种羊的静脉血于EDTA抗凝管中,-80 ℃保存备用。
1.2 DNA提取和鉴定
采用磁珠法提取柯尔克孜羊DNA,通过NanoDrop2000、Agilent 2100和Qubit方法测定基因组DNA的浓度和纯度,采用琼脂糖凝胶电泳检测基因组DNA的完整性。
1.3 柯尔克孜羊基因组DNA的SNP分型
DNA基因组分型在北京康普森生物技术有限公司进行,采用InfiniumTM Ovine SNP50K Genotyping Array v3芯片进行测序和基因分型。
1.4 SNP 芯片数据质控
数据分析前进行重复样本的检测,使用Plink (V1.90)软件对基因型数据进行质控,质控标准为:使用常染色体上的位点,最小等位基因频率(MAF)≥ 0.01,哈代温伯格平衡检验P值≥0.000 001,SNP检出率(call rate)≥90%,个体检出率≥90%。
1.5 柯尔克孜羊群体遗传多样性分析
采用Plink (V 1.90) 软件对质控后的数据进行分析,计算柯尔克孜羊群体的多态标记比例 (PN)、期望杂合度 (He)、观察杂合度 (Ho)、多态性信息含量 (PIC)、有效等位基因数 (Ne) 和最小等位基因频率(MAF)。
1.6 柯尔克孜羊群体ROH分析
使用Plink (V1.90) 软件计算柯尔克孜羊群体中每个样本基因组上的长纯合片段ROH,统计ROH在柯尔克孜羊群体中的分布、长度、数目等,根据每个样本中ROH片段的总长度占常染色体基因组总长的比例计算基于ROH的近交系数(FROH)[23],采用R软件(https://www.r-project.org/)绘制FROH小提琴图。
1.7 柯尔克孜羊群体IBS距离矩阵和G矩阵分析
利用Plink (V1.90) 软件计算群体个体间的状态同源性(identical by state, IBS),计算个体间的遗传距离,同时根据VanRaden[24]提出的基因组关系G 矩阵构建方法构建柯尔克孜羊群体G矩阵,分析群体个体间的亲缘关系,并使用R软件绘制IBS距离矩阵和G矩阵结果热图。
1.8 柯尔克孜羊群体家系构建分析
采用MegaX (V10.0)[25]软件对质控后的SNP 位点进行分析,采用邻接法 (neighhor-Joining, NJ) 构建群体进化树,分析柯尔克孜羊群体中种公羊的家系结构,统计不同家系种公羊的数量。
2 结 果
2.1 柯尔克孜羊DNA提取和结果检测
采用磁珠法提取柯尔克孜种公羊和种母羊的静脉血DNA, 检测发现DNA样品平均浓度在50 ng·L-1左右,OD260 nm/OD280 nm为1.8~2.0。经1%的琼脂糖凝胶电泳检测发现DNA条带清晰,无拖带现象,说明柯尔克孜羊基因组DNA质量较好,达到芯片检测要求,可以用于后续的试验研究(图1)。

M. DNA相对分子质量标准; 1~61. 柯尔克孜羊基因组DNA
2.2 柯尔克孜羊群体基因组DNA的SNP分型和质控
使用InfiniumTM Ovine SNP50K Genotyping Array v3芯片对柯尔克孜羊基因组DNA进行基因分型和质控检测,结果见表1。61只柯尔克孜羊共检测到64 734个SNPs,表明此款芯片适用于分析柯尔克孜羊基因组的SNP位点;质控后的SNPs数量为56 763,其中1号染色体上SNPs位点分布最多(6 305个),24号染色体上SNPs位点分布较少(845个)(图2)。

图2 柯尔克孜羊SNPs在每条染色体上的分布
2.3 柯尔克孜羊群体的遗传多样性分析
柯尔克孜羊群体的遗传多样性见表2。SNPs位点的平均多态信息含量为0.273±0.112, 其中多态性信息含量在0~0.15之间的SNPs位点较少(占15.6%);多态性信息含量在0.30~0.45之间的SNPs位点较多(56.9%),27.5%的SNPs位点的多态性信息含量处于0.15~0.30之间(图3A)。具体来说,多态性信息含量在0~0.25之间的SNPs位点有12 419个,占比21.9%,多态信息含量在0.25~0.5之间的SNPs位点有44 344个,占比78.1%,说明柯尔克孜羊群处于中度遗传性。61只柯尔克孜羊共检测到有效等位基因数为96.567,平均有效等位基因数为1.583。SNPs位点的多态性标记比例为0.889,此群体的平均期望杂合度和观察杂合度均为0.368,说明现在的柯尔克孜羊群体无外源血统的引入和近交现象发生。平均最小等位基因频率为0.263±0.147,分布相对均匀,0.1~0.2占比最少(17.0%),0.4~0.5占比最多(23.0%)(图3B)。

表2 柯尔克孜羊群体遗传多样性
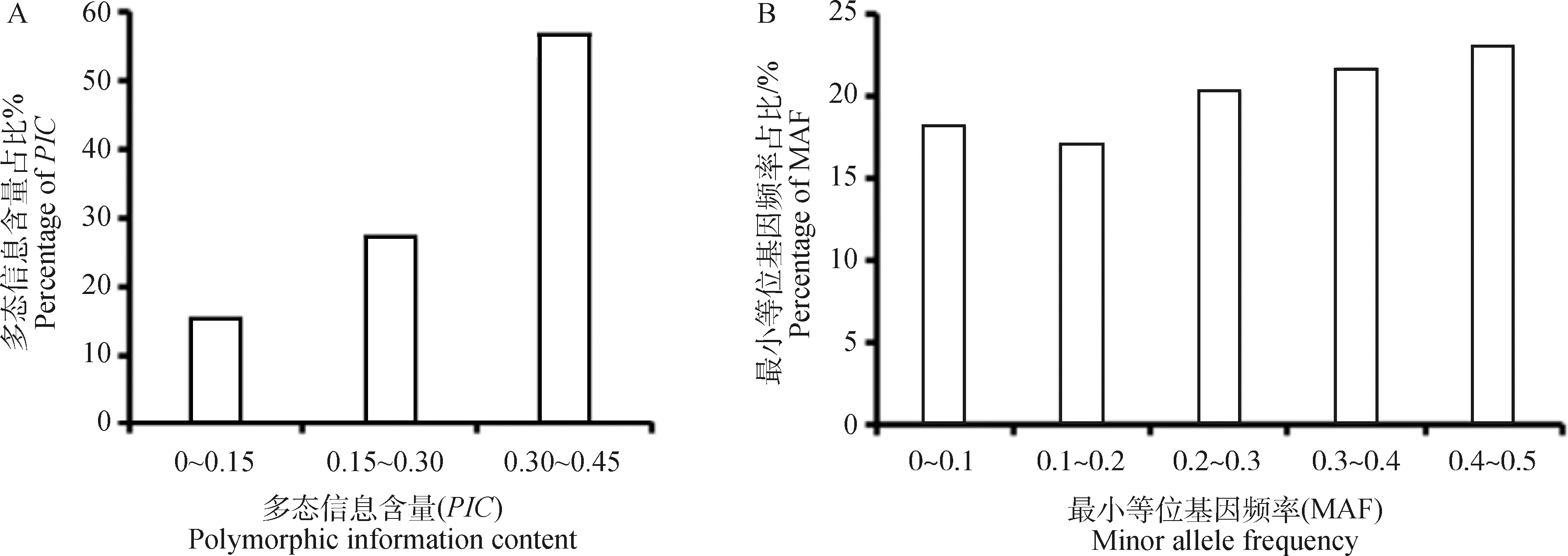
A.多态信息含量分布;B.最小等位基因频率分布
2.4 柯尔克孜羊群体的亲缘关系分析
2.4.1 ROH分析 61只柯尔克孜羊共检测到200个ROHs,ROHs在柯尔克孜羊群体的染色体上均有分布,其中在2号染色体上分布最多,24号染色体上分布最少(图4A)。在61只柯尔克孜羊中,56只羊的ROH长度在0~50 Mb之间(图4B)。在柯尔克孜羊群中,长度1~5 Mb的ROHs最多,占67.5%左右,长度为15~20 Mb的ROHs最少,占4% 左右 (图4C)。柯尔克孜羊群体基于ROH的平均近交系数FROH为0.008 19±0.018 8,其中D8392的FROH最大为0.115,31只柯尔克孜公羊的FROH为0.004 65±0.008,其中9只公羊的FROH近交系数为0(图5)。

A. ROH在染色体上分布; B. 柯尔克孜羊个体ROH长度的分布情况; C. 柯尔克孜羊不同长度ROH分布

图中心的白色点代表群体FROH的中位数,中间黑色方框的上缘和下缘分别为群体FROH的上四分位数和下四分位数。小提琴图的宽窄表示群体FROH的概率密度分布,图越宽表示处于该水平的样本数目越多,反之越少
2.4.2 基于G矩阵的柯尔克孜羊群体间的亲缘关系分析 利用全基因组SNPs位点构建的G矩阵能真实反映个体间的亲缘关系,柯尔克孜羊群体亲缘关系结果见图6,颜色越接近紫色表示亲缘关系越近,G 矩阵结果显示柯尔克孜羊群体间亲缘关系均较远。

该图展示两两个体间的基因组亲缘关系系数,颜色越接近紫色表示亲缘关系越近
2.4.3 基于IBS距离矩阵的柯尔克孜羊群体遗传距离分析 61只柯尔克孜羊之间的IBS遗传距离在0.229~0.307之间,平均遗传距离为0.294;31只种公羊间IBS遗传距离在0.229~0.306之间,平均遗传距离为0.294。柯尔克孜羊群体IBS距离矩阵的结果见图7,结果显示柯尔克孜羊个体间IBS遗传距离较远,说明此群体近交风险较低。

该图展示两两个体间的遗传距离,颜色越接近绿色表示亲缘关系越近
2.5 柯尔克孜羊群体中公羊的家系结构分析
鉴于公羊对于整个群体的重要性,采用MegaX (V10.0) 软件构建柯尔克孜羊31只种公羊群体进化树,结合基因组亲缘关系分析结果,以公羊间基因组亲缘关系系数大于等于0.1为标准进行聚类分析,将现有种公羊划分为25个家系(图8),其中家系17包含4只公羊,家系18包含2只公羊,家系20包含3只公羊,其余家系目前均只有1只公羊。

图8 基于基因组亲缘关系系数的柯尔克孜公羊群体进化树
3 讨 论
柯尔克孜羊是新疆优良的绵羊品种之一,具有独特的形态特征,但是其遗传多样性和群体结构尚未得到系统的研究,尤其是在基因组水平上。本研究采用50K高密度SNP芯片技术检测阿图什市良繁中心柯尔克孜羊,结果发现了大量的全基因组SNPs,并基于这些高质量SNPs对柯尔克孜羊的遗传多样性和种群结构进行评估。研究发现,遗传杂合度越高,群体的变异越大,杂合度高于0.5表明群体没有受到高强度选择[26]。本研究发现,柯尔克孜羊群体平均预期杂合度和观测杂合度均为0.368,说明此柯尔克孜羊群无血统流失或者外来血统的引入。赵冰茹等[27]采用9个微卫星检测了南疆地区4个绵羊品种的平均杂合度,发现柯尔克孜羊的平均杂合度为0.737,预期杂合度为0.857,与本研究的结果相差很大,这可能与微卫星位点的选择有关,也与采样地区和样本量有关。不同地方绵羊群体的杂合度不同,西藏浪卡子绵羊的观测杂合度为0.235,青海山谷型藏羊的观测杂合度为0.323[28],甘肃滩羊的观测杂合度高达0.875[29],说明不同品种绵羊的群体变异与地域环境及其选育程度有较大的关系。
多态信息含量(PIC)是衡量基因片段多态性的常用有效指标[30-32], Botstein 等[33]认为,对于微卫星位点来说,PIC>0.5时定义为高度多态位点,PIC<0.25时定义为低度多态位点,PIC位于0.5和0.25之间为中度多态位点,柯尔克孜羊SNPs位点的平均多态信息含量为0.273,但是56.9%的SNPs多态信息含量分布在0.30~0.45之间,78.1%的多态信息含量在0.25~0.5之间,说明柯尔克孜羊群处于中度遗传[33],且柯尔克孜羊群体间个体差异很大。以上结果显示,阿图什良繁中心柯尔克孜羊群体具有中度遗传变异,虽然没有经过系统选育、合理利用和保种,但是从二十世纪90年代开始对柯尔克孜羊进行不断的选育,导致其遗传变异下降。
根据ROH覆盖的基因组比例评估近亲繁殖系数被认为是检测近亲繁殖效益的一种强大而准确的方法[34],也是一种有效的替代谱系近亲繁殖的系数[35-36]。全基因组SNP芯片显示,通城猪平均ROH长度为23.7 Mb, 范围为11.26~69.02 Mb,基于ROH的平均近交系数为0.04%,表明近交水平较低,研究结果为制定通城猪的管理和保种策略提供了理论依据[37]; 用ROH检测8个印度牛品种的遗传多样性,表明在较小的本地种群中近交水平达到临界状态,研究结果为保护生物多样性提供了有用的工具[38]。本研究在61只柯尔克孜羊群中发现,91.8 %的ROH长度在0~50 Mb之间,ROH总长度较短,且基于ROH的近交程度分析发现,此柯尔克孜羊群体平均近交系数为0.008,近交系数较低。但是随着保种时间延长,加上群体规模局限性和相对封闭的运行模式,近交系数必然上升[39]。所以在日常管理中应该加强系谱档案的管理,完善配种记录,避免系谱错误导致近亲繁殖,维持柯尔克孜羊群体的遗传多样性。同时,加强柯尔克孜羊本品种选育,进一步提纯复壮。
遗传距离是指不同种群或个体间基因差异的程度,是探究物种起源、构建系统发育树和分析群体间亲缘关系的基础,对育种工作中指导亲本选育有重要作用[40]。本研究也分析了柯尔克孜羊群体间IBS遗传距离,以此评价个体间的亲缘关系[33]。柯尔克孜羊群体的IBS平均遗传距离为0.294 2,31只公羊间平均遗传距离为0.294 4,说明柯尔克孜羊种公羊和种母羊之间具有适中的遗传距离[18]。因为种公羊在整个群体中的重要性,基于31只种公羊的遗传距离构建了系统进化树,按照亲缘关系小于0.1的标准将种公羊划分为25个家系,大部分家系只有1只种公羊,后期应注意后代的选育,避免血统流失。而测序的30只母羊与所有检测的公羊血缘关系都比较远,可以任意进行交配。
4 结 论
本研究基于InfiniumTM Ovine SNP50K Genotyping Array v3芯片分析了柯尔克孜羊群体的遗传多样性和遗传结构,发现柯尔克孜羊群体遗传多样性较丰富,群体内近交程度低,虽然家系较多,但是每个家系种公羊数量太少,需要加强种公羊的后代选育,避免血统流失。
