白芨滩地区不同生物土壤结皮类型对微生物群落结构和组成的影响
2023-02-10李靖宇段晓敏刘建利刘秉儒
李靖宇,杨 瑞,段晓敏,刘建利,3,刘秉儒
(1.北方民族大学生物科学与工程学院,宁夏 银川 750021;2.宁夏特殊生境微生物资源开发与利用重点实验室,宁夏 银川 750021;3.国家民族事务委员会黄河流域农牧交错区生态保护重点实验室,宁夏 银川 750021)
宁夏灵武白芨滩国家级自然保护区通过治沙造林、控制流沙等方式筑起了阻止毛乌素沙地南移和西扩的“绿色长城”,在维护周边生态安全、改善周边生态环境等方面发挥着重要作用[1]。植物措施是荒漠化地区固定流沙和改良土壤的最有效、最经济、最持久的方法[2]。除高等维管植物外,在荒漠生态系统中广泛分布着不同类型的生物土壤结皮[2]。生物土壤结皮是由细菌、真菌、藻类、地衣和苔藓等低等生物与土壤颗粒相互作用形成的有机复合体,其生态适应性强,耐高温、辐射,可抵抗干旱、盐碱,广泛分布于全球干旱和半干旱荒漠地区,是这类地区景观的重要组成成分,在部分地区甚至可覆盖地表面积的70%以上[2-3]。生物土壤结皮不仅具有防止风蚀和水蚀、维持土壤水分、促进植被演替等功能,还能够通过光合作用及固氮过程增加土壤中有机碳和有机氮[4]。生物土壤结皮作为荒漠生态系统的工程师,在某种程度上其形成是干旱半干旱区土壤稳定的重要标志,生物土壤结皮在联结地表生物与非生物成分中起着不可替代的作用[5-6]。
土壤微生物与植物及土壤养分形成植物-土壤微生物-土壤养分模式驱动生态系统能量流动与物质循环,维持生态系统结构和功能稳定[7]。土壤微生物作为生物土壤结皮的主要组成部分,其群落结构(即不同种类的生物及其多度)在生物土壤结皮形成和发育过程中发挥着重要生态系统功能,包括协调分解和矿化过程以及调节营养的可利用性和初级生产力[8-9]。在生物土壤结皮不同发育阶段,细菌和真菌承担的作用有所差异[10]。作为初级生产者,蓝藻可以固定碳和氮,而真菌以自由生活的方式通过丝状菌丝将土壤颗粒结合在一起,或与蓝藻/藻类共生形成地衣,进而提高土壤肥力[10]。在干旱生态系统的生物土壤结皮演替过程中,细菌在碳、氮循环的调节中起着至关重要的作用,而真菌对木质素进行补充降解,从而共同刺激生物土壤结皮演替后期土壤中碳和氮代谢的增加[11]。微生物多样性对于多种生物地球化学循环和生态系统功能至关重要,而高通量测序技术的快速发展极大地促进了对微生物多样性生态作用的普遍理解[9,12]。WANG等[13]研究表明,腾格里沙漠生物土壤结皮中真菌群落多样性和多度随着发育时间的延长而显著增加,并在发育后期接近最高水平,与自然栖息地水平一致。XU等[14]采用Illumina MiSeq测序技术对古尔班通古特沙漠不同发育阶段生物土壤结皮中细菌与真菌群落结构相似性变化的研究结果表明,土壤生物结皮的发育促进了原核生物群落的趋同演替。然而,白芨滩地区植被恢复过程中生物土壤结皮中细菌和真菌群落的演替规律及其生态功能的研究还相对较少。
生物土壤结皮发育程度与枯落物覆盖、植被盖度、土地利用方式和地形地貌等关系密切[15]。大多数旱地生态系统的特点是以植被斑块和裸地区域为主的两相镶嵌结构,而多年生植被斑块对生物土壤结皮群落的多样性和覆盖率通常具有积极的促进作用[16]。以白芨滩国家级自然保护区及其周边植被恢复良好且镶嵌有大量生物土壤结皮的区域作为研究对象,采用Illumina MiSeq测序技术对不同生物土壤结皮类型中细菌和真菌群落进行表征,以期阐明不同生物土壤结皮类型对细菌和真菌群落结构和组成的影响,有助于深入认识研究区生态恢复过程中生物土壤结皮参与生物地球化学循环以及发挥生态功能的微生物多样性基础。
1 材料与方法
1.1 研究区概况与样品采集
宁夏灵武白芨滩国家级自然保护区地处毛乌素沙地西南边缘(37°49′05″~38°20′54″ N、106°20′22″~106°37′19″ E),是以柠条(Caraganakorshinskii)、猫头刺(Oxytropisaciphylla)和沙冬青(Ammopiptanthusmongolicus)等荒漠沙生植物为主要保护对象的国家级自然保护区,其南部以沙地丘陵为主,北部以山地荒漠为主,平均海拔为1 250 m。该区域年平均降水量为192.9 mm,年平均气温为10.4 ℃,积温为3 551.3 ℃,年平均日照时数为2 717 h,属中温带干旱气候区,四季分明。区域最显著特点为干燥、风大、沙多,降水量少而集中,蒸发量大、冬寒长、夏热短、温差大、日照长、光能丰富,土壤类型以灰钙土和风沙土为主[1,17]。
研究样地主要设在白芨滩国家级自然保护区及其周边区域,具体位置见表1。样品采集于2021年9月5号,共设置13个采样点,每个采样点分别取藓结皮和藻结皮样品各1个,共计26个样品。在2 m×2 m样方中按照五点取样法进行取样,将土样装入无菌袋带回实验室,过2 mm孔径筛,将处理后的样品冷冻保存,用于后续宏基因组DNA提取和Illumina MiSeq测序(细菌和真菌)。

表1 取样点位置信息
1.2 土壤宏基因组DNA提取和Illumina MiSeq测序
按照FastDNA Spin Kit for Soil (MP Biomedicals,Santa Ana,CA,USA)提取试剂盒说明书提取土壤宏基因组DNA。细菌16S rDNA V3-V4区PCR扩增反应体系:2.0 μL 2.5 mmol·L-1dNTPs,0.8 μL 5 μmol·L-1338F引物(5′-ACTCCTACGGGAGGCAGCAG-3′),0.8 μL 5 μmol·L-1806R引物(5′-GGACTACHVGGGTWTCTAAT-3′),4.0 μL 5×FastPfu缓冲液,10 ng模板以及0.4 μL TransStartFastPfu Polymerase,0.2 μL BSA,最后加ddH2O至20 μL。反应条件:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,27个循环;72 ℃ 10 min。真菌ITS1区PCR扩增反应体系:2.0 μL 2.5 mmol·L-1dNTPs,0.8 μL 5 μmol·L-1ITS1F引物(5′-CTTGGTCATTTAGAGGAAGTAA-3′),0.8 μL 5 μmol·L-1ITS2R引物(5′-GCTGCGTTCTTCATCGATGC-3′),2.0 μL 10×缓冲液,10 ng模板以及0.2 μL TaKaRarTaq DNA Polymerase,0.2 μL BSA,最后加ddH2O至20 μL。反应条件:95 ℃ 3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,35个循环;72 ℃ 10 min[18]。将PCR产物用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量,将每个样品等比例混合,采用NEXTFLEX®Rapid DNA-Seq Kit进行建库,最后利用Illumina公司的MiSeq PE300平台进行测序(上海美吉生物医药科技有限公司)[18]。
1.3 Illumina MiSeq测序数据处理与分析
采用fastp软件[19]和FLASH软件[20]对原始测序序列进行质控和拼接,随后采用Qiime2流程中的DADA2[21]插件(默认参数)对质控拼接后的优化序列进行降噪处理,尽可能去除优化数据中的PCR扩增错误或测序错误,以获得样本中真实序列信息,即扩增子序列变体(amplicon sequence variant,ASV)。基于Sliva 16S rRNA数据库(https:∥www.arb-silva.de/documentation/release-1381/)和UNITE(https:∥unite.ut.ee),采用Qiime2流程中Naive bayes分类器对细菌和真菌ASVs分别进行物种分类学分析。所有分析均在上海美吉生物医药科技有限公司云平台(www.majorbio.com) Qiime2流程中进行。
1.4 统计分析
对ASV进行物种分类学注释后,统计各样本中各ASV注释结果的丰度信息,按照最小样本序列数抽平后计算各样本中细菌和真菌群落α和β多样性指数。反映群落丰富度的Chao和ACE指数计算公式分别为
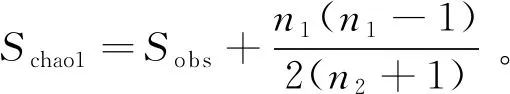
(1)
式(1)中,Schao1为估计的ASV总数;Sobs为实际观测到的ASV数;n1为只含1条序列的ASV数;n2为只含2条序列的ASV数。
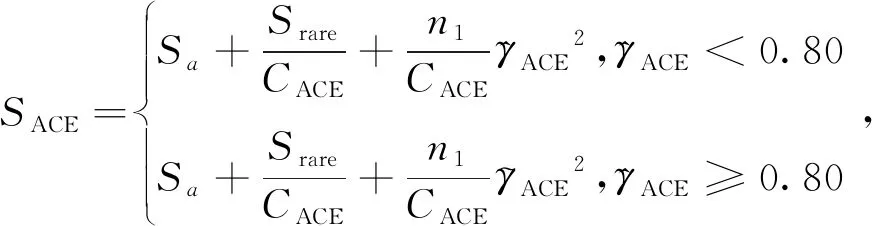
(2)

(3)

(4)

(5)

(6)
式(2)~(6)中,SACE为用来估计群落中物种(如ASV)数目的指数;CACE为样本覆盖度的估计值;γACE2为稀有物种的变异系数;ni为含有i条序列的ASV数;Nrare为序列数不超过a条的所有物种(ASV)所含的序列总数;Srare为含有不大于a条序列的ASV数;Sa为含有大于a条序列的ASV数;a为优势ASV序列的阈值,默认值为10。
反映群落多样性的指数Shannon(H)和Simpson(D)指数计算公式分别为

(7)
式(7)中,Sobs为实际观测到的ASV数;ni为第i个ASV所含序列数;N为所有序列数。

(8)
反映群落均匀度的Shannon even(E)和Simpson even(J)指数计算公式分别为
E=Dens/S。
(9)
式(9)中,Dens为Simpson有效物种(ASV)数,即与给定数据集中物种(ASV)丰度平均占比相等的均匀群落中的物种(ASV)数量;S为群落物种(ASV)丰富度指数。
J=H/Hmax。
(10)
式(10)中,H为Shannon指数;Hmax为物种(ASV)丰富度相同的情况下,Shannon指数的最大值。
采用美吉生物云平台进行土壤细菌和真菌群落主成分分析(principal component analysis,PCA),并采用Bray-Curtis距离算法通过相似性分析(analysis of similarities,ANOSIM)计算两组样品间的距离,用来检验组间差异是否显著大于组内差异,从而判断分组是否有意义。采用Wilcoxon秩和检验对两组样品中的物种进行显著差异分析,并对P值进行校正。置换多因素方差分析(perMANOVA)采用Bray-Curtis距离矩阵对总方差进行分解,分析不同生物土壤结皮类型对细菌和真菌群落结构差异的解释度,并采用置换检验(置换次数为999)对其统计学意义进行显著性分析。
2 结果与分析
2.1 不同生物土壤结皮微生物群落α多样性
通过测序共得到细菌群落30门77纲193目302科574属1 139种,包含10 181个ASV。图1显示,藓结皮ACE指数(286.62±25.98)显著高于藻结皮(265.54±25.48),藓结皮Chao指数(286.62±25.98)显著高于藻结皮(265.54±25.48);藓结皮Shannon和Simpson指数以及Shannon even和Simpson even指数与藻结皮之间没有显著差异。
通过测序共得到真菌群落11门37纲96目216科489属815种,包含4 222个ASV。图1显示,藓结皮ACE、Chao、Shannon、Simpson、Shannon even和Simpson even指数与藻结皮之间没有显著差异。
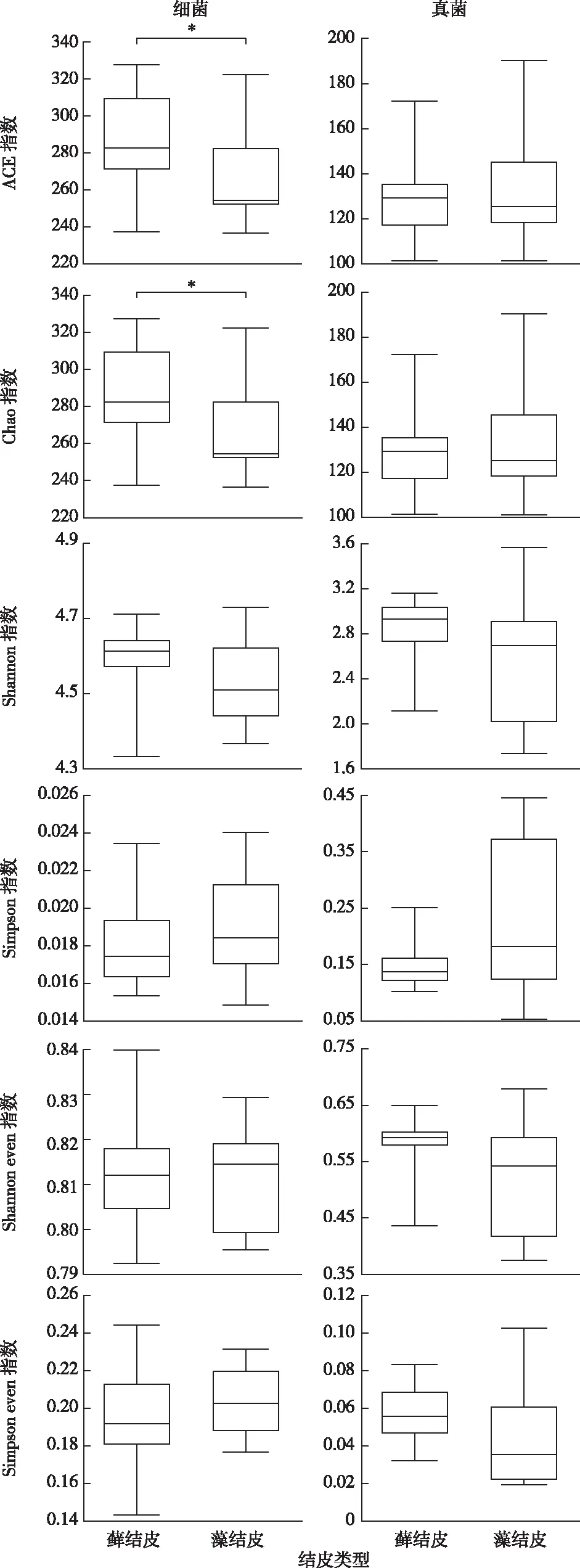
箱体上、中和下线分别表示上四分位数、中位数和下四分位数。*表示P<0.05。
2.2 不同生物土壤结皮微生物群落组成
如图2所示,不同生物土壤结皮细菌群落在门水平上以放线菌门、变形菌门、绿弯菌门、酸杆菌门、蓝细菌门和拟杆菌门为主要类群。如图3所示,不同生物土壤结皮细菌群落在属水平上以微枝形杆菌属、红色杆菌属、地嗜皮菌属、芽球菌属、鞘氨醇单胞菌属、土壤红杆菌属、微红微球菌属、类诺卡菌属和苔藓杆菌属为主要类群。如图4所示,在门水平上,藓结皮中酸杆菌门相对丰度(12.23%)显著高于藻结皮(6.94%),而芽单胞菌门(2.76%)和蓝细菌门(1.38%)相对丰度均显著低于藻结皮(3.55%和4.75%)。如图5所示,在属水平上,藓结皮中红色杆菌属相对丰度(3.00%)显著低于藻结皮(4.53%),而苔藓杆菌属相对丰度(1.45%)显著高于藻结皮(1.12%)。
如图2所示,不同生物土壤结皮真菌群落在门水平上以子囊菌门、担子菌门、壶菌门、被孢霉门和毛霉门为主要类群,其中,未分类真菌相对丰度仅次于子囊菌门。如图3所示,在属水平上,组成以赤霉菌属、Knufia、被孢霉属、石果衣真菌、链格孢属、Bahusakala、Phaeomycocentrospora、光黑壳属和Omphalina为主要已知类群的群落结构,而未知真菌在藓结皮中占比达到31.25%,在藻结皮中占比达到41.34%,是主要优势类群。如图4所示,在门水平上,藓结皮中壶菌门相对丰度(2.46%)显著低于藻结皮(4.01%)。如图5所示,在属水平上,藓结皮中Knufia(5.82%与1.57%)、Bahusakala(2.45%与0.72%)、Omphalina(2.40%与0.21%)、Lamprospora(1.32%与0%)、Vishniacozyma(1.08%与0.14%)、Phoma(0.87%与0.04%)、Entoloma(0.29%与0.25%)和Trichoderma(0.41%与0.02%)等真菌相对丰度显著高于藻结皮;而藓结皮中Powellomyces(0.69%与1.48%)、Sporormiella(0.40%与0.72%)、Thielavia(0.12%与0.25%)、Iodophanus(0.03%与0.24%)、Agaricus(0.03%与0.11%)和Acrophialophora(0.02%与0.11%)等真菌相对丰度显著低于藻结皮。
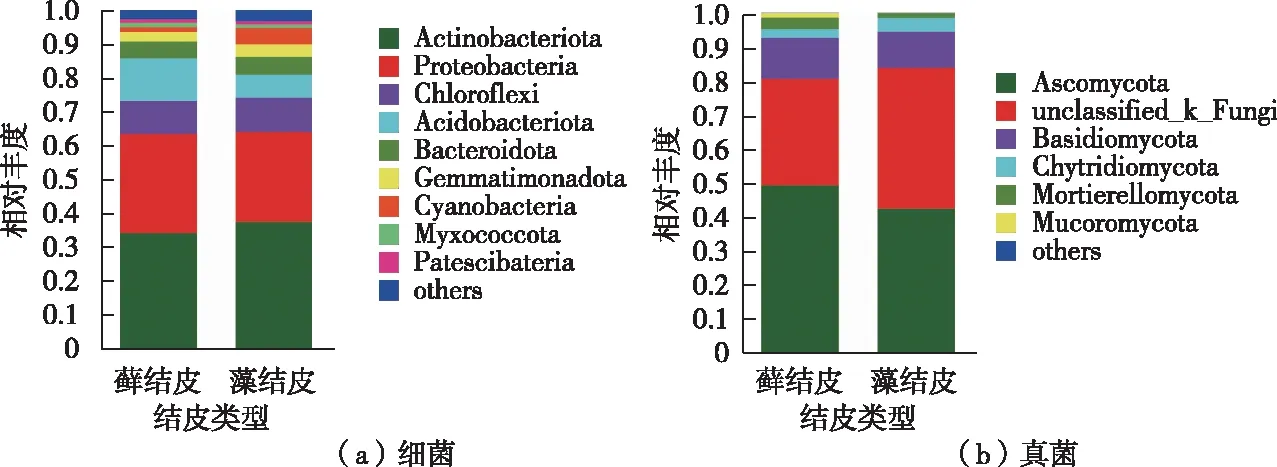
图2 不同生物土壤结皮中细菌和真菌群落在门水平上的群落组成
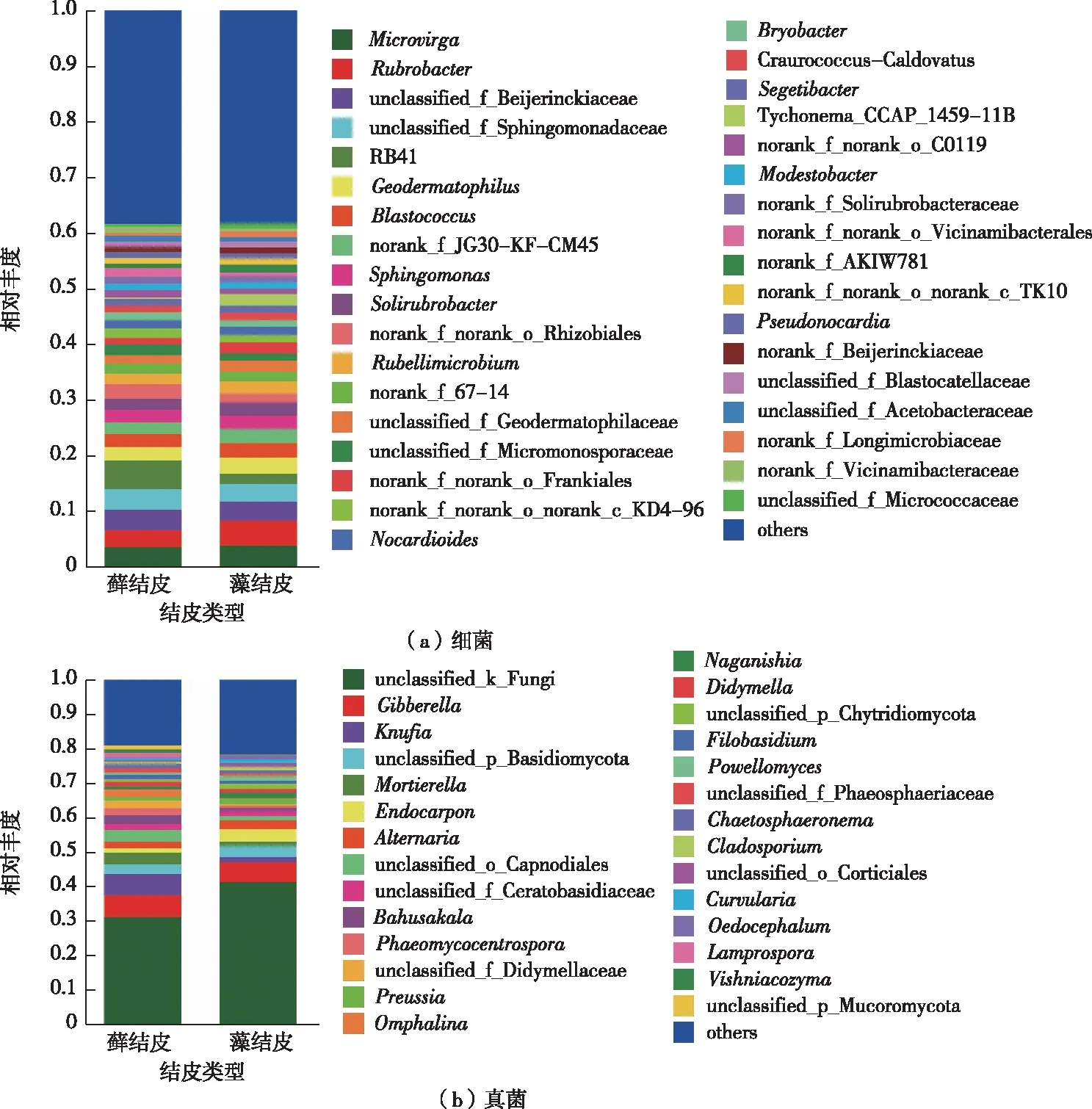
图3 不同生物土壤结皮中细菌和真菌群落在属水平上的群落组成

*表示P<0.05,**表示P<0.01,***表示P<0.001。
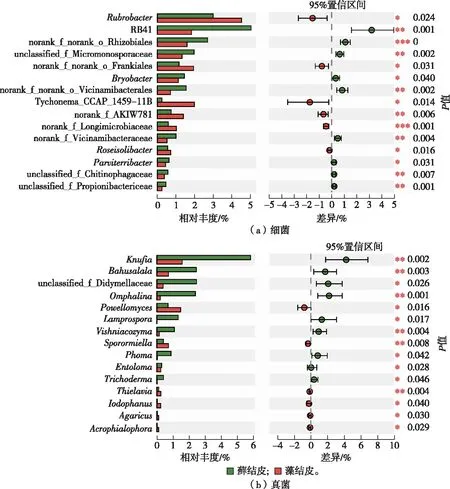
*表示P<0.05,**表示P<0.01,***表示P<0.001。
2.3 不同生物土壤结皮微生物群落β多样性
如图6所示,PCA分析结果表明,主成分1(PC1)和主成分2(PC2)分别可以解释细菌群落结构差异的11.90%和10.81%,且细菌群落结构在藓结皮和藻结皮之间存在显著差异;PC1和PC2分别可以解释真菌群落结构差异的8.83%和7.62%。如表2所示,置换多因素方差分析结果表明,生物土壤结皮类型分别可以解释细菌和真菌群落结构差异的14%和8%,且生物土壤结皮类型对细菌和真菌群落结构具有显著影响。

表2 不同生物土壤结皮类型对细菌、真菌群落属水平组成影响perMANOVA评估结果
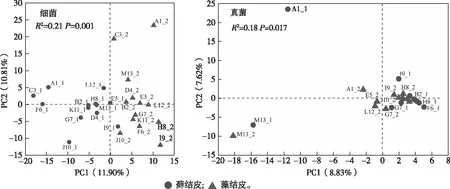
图6 不同生物土壤结皮中细菌和真菌群落的PCA分析
3 讨论
笔者研究选取白芨滩国家级自然保护区及其周边区域植被恢复良好且广泛分布着藓结皮和藻结皮镶嵌形成的地表覆盖区域作为研究对象,对藓结皮和藻结皮分别进行微生物群落结构表征,结果表明生物土壤结皮类型对细菌群落结构以及真菌群落结构产生显著影响。与腾格里沙漠东南缘生物土壤结皮类型对微生物群落结构的影响[22-23]相比,笔者研究中生物土壤结皮类型对微生物群落结构差异的解释度较低,说明研究区其他环境因子对生物土壤结皮中微生物群落结构具有重要影响。
作为生物土壤结皮的重要组成部分,真菌通过丝状菌丝聚集土壤颗粒,从而有助于提高土壤稳定性[24]。对腾格里沙漠生物土壤结皮的研究[13]发现,在演替过程中,真菌群落多样性发生变化,其丰富度可能是预测固沙植被生物土壤结皮发育程度的一个潜在指标。笔者研究中,真菌群落α多样性指数在藓结皮和藻结皮之间没有显著差异,表明研究区不同生物土壤结皮中形成了比较稳定的真菌多样性。真菌种群严格依赖于土壤性质的恢复,因此,土壤理化特性的改善可以有效促进真菌多样性及其功能[24]。这表明在植被恢复背景下,研究区真菌群落结构α多样性随着生物土壤结皮演替时间的延长接近或者达到自然种群水平[24]。
然而,α和β多样性指数不足以说明不同样本组之间的群落变化,分离这些指数的组成部分对分析和理解真菌群落中物种演替至关重要[25]。笔者研究中藓结皮和藻结皮真菌群落结构组成主要优势类群为子囊菌门真菌。在内盖夫沙漠、奇瓦瓦沙漠、科罗拉多高原以及怀俄明州和犹他州草原的研究[23]表明,无论子囊菌群起源如何,其都是生物土壤结皮的主要真菌定殖者。在属水平上,藓结皮真菌群落结构中显著富集Knufia属真菌,其属于子囊菌门,为干旱地表普遍存在的黑色小菌落真菌,其能够抵抗环境压力,包括极端温度、干燥(复水)、低营养可用性和强烈的太阳辐射[26]。Knufiapetricola常见于地中海环境的大理石上,其中,菌株A95和CBS726.95因其具有黑色小菌落真菌的所有特征,已被广泛用于研究矿物风化与Nostocpunctiforme蓝藻的共生作用[26-27]。除了分解作用外,真菌也是土壤颗粒聚集的重要参与者,在没有蓝藻和地衣的情况下,真菌还有助于土壤结构稳定和生物土壤结皮发育[28]。
反映细菌群落丰富度的指数在藓结皮中显著高于藻结皮,表明细菌α多样性是生物土壤结皮发育阶段的重要指征。作为优势类群的放线菌在藓结皮和藻结皮之间没有显著差异,这与腾格里沙漠东南缘沙坡头地区藓结皮和藻结皮之间放线菌类群的差异[23]类似。而放线菌门中在藓结皮和藻结皮之间存在显著差异的主要类群为红色杆菌属,该属也是沙坡头地区藓结皮和藻结皮之间的主要差异放线菌类群,而不同的是,沙坡头地区生物土壤结皮中的优势类群地嗜皮菌属在笔者研究中不是优势放线菌类群,且在不同生物土壤结皮之间没有显著差异[29]。笔者研究中,α-变形菌亚门根瘤菌目甲基杆菌科的微枝形杆菌属作为优势属在不同生物土壤结皮之间没有显著差异,在沙坡头地区藓结皮和藻结皮之间存在显著差异[23]。该属目前已描述的17个有效物种,主要分离自沙质干旱土壤或沙漠土壤、根瘤、粪便样本、热含水层、空气和温泉等环境中[30],而来源于生物土壤结皮的Microvirgasp. BSC39菌株具有趋化性和胞外多糖合成能力,可用于生物膜粘附[31],微枝形杆菌属中有一些菌株能够有效进行固氮[32]。红色杆菌属和微枝形杆菌属表现出对高温和干旱环境的普遍适应性,一些红色杆菌属物种具有高度的抗辐射性[33]。在古尔班通古特沙漠、柴达木盆地、腾格里沙漠、科尔沁沙地和库布齐沙漠的生物土壤结皮中已鉴定出的蓝藻中,具鞘微鞘藻(Microcoleusvaginatus)是优势种[22],腾格里沙漠东南缘不同生物土壤结皮中微鞘藻属相对丰度受季节变化和生物土壤结皮类型的影响[34]。笔者研究中藓结皮和藻结皮中微鞘藻属并不是优势属,且相对丰度较低,这可能是由于降水、土壤属性和植被盖度方面的差异造成[22]。苔藓杆菌属成员主要分离自酸性湿地,其在轻度酸性条件下生长最好[35]。笔者研究发现藓结皮中苔藓杆菌属相对丰度显著高于藻结皮,说明藓结皮微环境可能更适合该属生长,这也可能与苔藓植物本身有关。腾格里沙漠东南缘沙坡头地区藓结皮中也检测到低相对丰度的苔藓杆菌属细菌[36]。该属细菌是否与生境或者苔藓植物种类有关需要在不同区域取藓结皮样品开展比较研究来进一步说明。
4 结论
白芨滩国家级自然保护区及其周边植被恢复良好区域镶嵌分布的藓结皮和藻结皮细菌和真菌群落的α多样性指数趋于一致,仅反映细菌群落丰富度的ACE和Chao指数在藓结皮中显著高于藻结皮,而不同生物土壤结皮类型会对细菌和真菌群落结构产生显著影响。在生物土壤结皮发育的高级阶段——藓结皮样品中显著富集的细菌类群在已有数据库中尚没有明确分类,而富集的已知真菌类群主要为Knufia属。研究区藓结皮和藻结皮中未知分类的细菌和真菌尚有待开展后续分离、纯化和鉴定等基础性工作,这将有助于完善现有数据库和更加深入理解不同微生物类群在藻结皮和藓结皮以及不同生物土壤结皮类型演替过程中发挥的功能和作用。
