国境口岸截获象牙地理溯源分析初探
2022-11-07高瑞芳陈进会刘建丽徐艳春
苏 倡 高瑞芳 陈进会 刘建丽 刘 荭 徐艳春,4,5*
(1.东北林业大学野生动物与自然保护地学院,哈尔滨,150040;2.深圳海关动植物检验检疫技术中心,深圳,518045;3.黄埔海关技术中心,东莞,523070;4.国家林业和草原局野生动植物检测中心,哈尔滨,150040;5.国家林业和草原局野生动物保护与利用工程技术研究中心,哈尔滨,150040)
大象是非洲生态系统中的重要物种,象牙的非法贸易导致了非洲象种群数量的急速下降[1-3],并引发一系列生态、经济和安全问题[4]。为了保护非洲象,早在1989年,《濒危野生动植物种国际贸易公约》(CITES)就把大象及其制品的国际贸易进行严格控制。2015年7月30日,联合国大会通过了关于“打击野生动植物非法交易”的历史性决议。
识别非法象牙来源地,确定走私热点地区,描绘走私通道是打击象牙非法交易的重要前提[5],这些工作都有赖于象牙的地理溯源,即通过对象牙的分析判断其来源于哪个国家或地区。海关是查获象牙非法贸易的主要部门,如果能够对查获象牙进行地理溯源,则可以把走私行为与非洲来源地联系起来,描绘国际走私通道。目前,国际上通用的象牙溯源方法有分子生物学方法和稳定同位素分析方法。分子生物学方法是以各个地理种群的核DNA基因型[4-8]或线粒体DNA(mtDNA)单倍型[9-10]频率为依据,通过象牙样本的基因型计算属于各个地理种群的概率。稳定同位素分析方法是对象牙中的碳、氢、氧、氮、锶和硫等同位素含量分析,再与来源地的背景数据比较,确定象牙的地理来源[11-15]。mtDNA是母系遗传的,雄性非洲象成年后会离开它们出生的群体进行扩散,而雌性则始终留在出生时的群体中[16-18],因此,每个地区的群体都会有其特征性的mtDNA多样性,成为地理来源的信息[9,19-20]。由于在每个细胞中mtDNA的拷贝数远高于核DNA,所以针对较低DNA丰度或较差质量的象牙样本时,通过mtDNA进行溯源更具优势。LoxodontaLocalizer数据库(www.loxodontalocalizer. org)[10]是一个开放获取的非洲象mtDNA地理溯源数据库,其基于线粒体控制区第一高变区(HVR1)的 316 bp序列,可以区分最近分化的谱系[21-23]。该数据库的参考数据包括125个单倍型,涵盖24个国家。这些单倍型中有62%是国家特有的[9],可用于推断象牙的地理来源。LoxodontaLocalizer数据库的方法和可开放获取的政策,目前已被大量应用于非洲象牙地理来源追溯的工作中[24-26]。
本研究利用mtNDA溯源方法,分析象牙样本HVR1的序列,利用LoxodontaLocalizer数据库推断可能的地理来源,建立国境口岸截获象牙的地理信息数据库,分析输入中国的象牙遗传特征,进而推断大象栖息地、猎捕地和走私通道等信息,为打击国际象牙走私链条提供参考。
1 材料与方法
1.1 材料及其预处理
试验所用象牙样本和象牙DNA均由国家林业和草原局野生动植物检测中心和南京森林警察学院提供,合计126份。将象牙块先在0.3%次氯酸钠溶液中浸泡杀菌120 s,用无菌脱脂棉擦拭象牙块,以去除象牙块的表面污垢、残留的组织和其他可能由于切割、装样等导致的来自其他样品的粉末或人源DNA污染[27]。随后用无菌水浸泡冲洗2次,无水乙醇浸泡冲洗1次,放置于超净工作台上使乙醇和水分完全挥发。使用SPEX 6770液氮研磨仪(SPEX SamplePrep,美国)将象牙块研磨成粉,采用2 min预冷3 min研磨的方案,研磨后的象牙粉末置于10 mL顶空瓶中室温保存备用。
1.2 象牙样品的脱钙
将200 mg象牙粉末置入2 mL离心管中,向其中加入1.5 mL EDTA溶液(0.5 mol/L,pH 8.0),对于质量较好的象牙样本使用改进的2 h快速脱钙法[27-28]:在56 ℃下孵育1 h,转移到37 ℃下继续孵育1 h,每小时更换1次EDTA溶液,12 000 r/min离心3 min,弃上清1 000 μL,再补加1 000 μL EDTA溶液,最后一个孵育步骤后同样方法离心,去除上清液1 000 μL,底部剩余液体及沉淀用于后续DNA提取。对于质量较差的样本,使用3~10 d的慢速脱钙法[27]:象牙在4 ℃条件下震荡孵育过夜,每天更换1次EDTA溶液,最后1次离心后,采用同样方法保留底部剩余液体及沉淀用于后续DNA提取。
1.3 象牙总DNA的提取与PCR扩增
向原2 mL离心管中,加入320 μL TNE缓冲液(10 mmol/L Tris-HCL、10 mmol/L EDTA、100 mmol/L NaCl,pH 8.0),80 μL SDS溶液(10%),50 μL蛋白酶K(10 mg/mL),漩涡震荡混匀,56 ℃水浴中消化6 h以上直至象牙沉淀完全溶解,最后用动物基因组磁珠提取试剂盒(中科雷鸣,北京)提取象牙总DNA。
PCR扩增反应采用Brandt等[29]的引物CR-F1(5′-TGGTCTTGTAAGCCATAAATGAAA-3′),CR-R1(5′-GCTTTAATGTGCTATGTAAGACTATG-3′);CR-F2(5′-TCGTGCATCACATTATTTACCC-3′),CR-R2(5′-TGGT-CCTGAAGAAAGAACCAG-3′)。根据LoxodontaLocalizer说明书中的建议[10],DNA质量较好的样本,用CR-F1/CR-R2引物对扩增522 bp的mtDNA控制区片段;而DNA质量较差的样本则使用2个单独的扩增反应,CR-F1/CR-R1引物对扩增318 bp的mtDNA控制区片段,CR-F2/CR-R2引物对扩增306 bp的mtDNA控制区片段,二者存在102 bp的重叠片段。
PCR反应以10.0 μL的反应体系进行,包含正反向引物各0.4 μL(10 μmol/L),2×RapidTaqMaster Mix(诺唯赞,南京) 5.0 μL,ddH2O 2.2 μL,DNA 2.0 μL,混匀后瞬时离心。扩增采用PE-9700型DNA扩增仪(GeneAmp,美国),反应程序:94 ℃预变性3 min;94 ℃变性30 s,54 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃延伸5 min。扩增产物用1%琼脂糖凝胶电泳分离(100 V 稳压,20 min),将目的条带明显的扩增产物回收,采用原引物进行双向Sanger测序(委托吉林省库美生物科技有限公司完成)。
1.4 数据分析
测序结果使用DNASTAR软件包中的SeqMan软件校对测序峰图,并将正反向测序结果拼接,得到最终序列。使用MEGA 5.2软件将控制区序列剪切为316 bp的片段,利用DnaSP 6.0软件分析确定单倍型,计算单倍型数、多态性位点数、单倍型多样性、核苷酸多样性和平均核苷酸差异。
从GenBank下载653条具有非洲地理位置信息的4 258 bp非洲象线粒体DNA序列 (JQ438119~JQ438771),包含13个非洲国家的22个地点;下载1条猛犸象线粒体基因组(KX027533)作为外群,在MEGA 5.2软件中将上述序列剪切为与试验样本测序序列相同的大小,与样本序列分别使用邻接法(NJ)和最大似然法(ML)构建系统发育进化树进行分析。在MEGA 5.2软件中构建NJ树,自举检验1 000次。使用PhyloSuite[30]软件中的MAFFT进行所有序列之间的比对,使用ModelFinder中的AIC(akaike information criterion)选择最佳的替代模型,HKY+I+G模型被评估为最优模型,使用IQ-TREE构建最大似然树,自举检验由5 000次重复检验获得。采用同样方法将LoxodontaLocalizer数据库中下载的所有125个单倍型与修剪为316 bp片段的样本序列构建ML树和NJ树。使用iTOL软件进行系统发育树的编辑和美化。
1.5 Loxodonta Localizer 数据库溯源分析
把前述分析中确定的各个单倍型,导入LoxodontaLocalizer数据库中,与各个地理区域的参考序列进行查询比对。在数据库中参看样本序列的匹配情况,获得其地理来源的相关信息。
2 结果与分析
2.1 系统发育分析
使用邻接法和最大似然法对象牙样本和不同地理区域非洲象间亲缘关系进行分析,构建系统发育分析树(图1)。126条象牙样本的序列分属2个大的支系和7个mtDNA进化枝。F支系(F-clade)为所有非洲森林象(Loxodontacyclotis)和部分非洲草原象(L.africana)所有,共有31条序列,分属中西部进化枝(n=15)、中北部进化枝(n=7)、中东部进化枝(n=2)和中南部进化枝(n=7);S支系(S-clade)来自大部分草原象,共有95条序列,分属北部大草原进化枝(n=1)、大草原进化枝(n=27)和东南大草原进化枝(n=67)。由于缺乏更多的多态性位点,分属于中西部进化枝、大草原进化枝和东南大草原进化枝中的大部分样本序列并没有完全地匹配到各个国家和地区之间。

图1 基于GenBank数据库中有地理信息的序列与样本序列构建的ML树Fig.1 The maximum likelihood phylogenetic tree based on sequences with geographic information in the GenBank database and ivory sample sequences in this study 注:外群为猛犸象,NJ树与其具有相似的拓扑结构不另做展示;进化树中红点表示后验概率大于95%,自举值大于75;不同的进化枝以不同的外圈颜色表示;mtDNA的F和S支系以黑色字体标注;不同的国家地区以不同的进化枝和文字颜色展示;本研究的126份样本的树枝标为砖红色;为方便展示,地理信息完全相同的亚进化枝折叠处理 Note:The outgroup is mammoth.The Neighbor-joining tree has a similar topology and is not shown separately.The red dots indicate that the posterior probability is greater than 95% and the bootstrap value is greater than 75.Different clades are represented by different colors of the outer ring.The F and S clades of mtDNA are marked in black font.Different countries or regions are displayed in different branche and text colors.The branches of the 126 samples in this study are brick-red.For the convenience of display,the subclades with the same geographical information were folded
2.2 单倍型分析
从126条象牙样本mtDNA控制区HVR1的316 bp的序列中,共定义和匹配了22个地理单倍型(表1)。在这22个单倍型中,共包含变异位点34个,单倍型多样性为(0.742±0.039),核苷酸多样性为0.022,平均核苷酸差异为7.056。同样,使用邻接法和最大似然法对126条序列和LoxodontaLocalizer数据库中的125个单倍型合并分析,得到系统发育树(图2)。所构建进化树的系统发育分析与之前的单倍型分析结果完全一致。
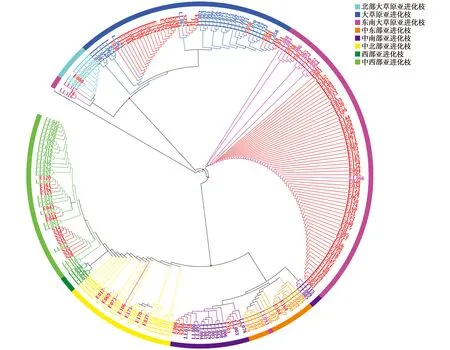
图2 基于Loxodonta Localizer 数据库中125个单倍型与样本序列构建的ML树Fig.2 The maximum likelihood phylogenetic tree based on sample sequences and 125 haplotypes in the Loxodonta Localizer database 注:外群为猛犸象,NJ树与其具有相似的拓扑结构不另作展示;进化树中红点表示后验概率大于95%,自举值大于75;不同的进化枝以不同的文字颜色和外圈颜色表示;本研究的126份样本的树枝标为砖红色 Note:The outgroup is mammoth.The Neighbor-joining tree has a similar topology and is not shown separately.The red dots indicate that the posterior probability is greater than 95% and the bootstrap value is greater than 75.Different clades are represented by different text and outer circle colors.The branches of the 126 samples in this study are brick-red
2.3 溯源定位结果
经LoxodontaLocalizer数据库溯源分析,有21个单倍型共109份样本可进行有效的地理溯源,共涉及13个国家,包含42个国家公园或保护区(表2,图3)。单倍型LL062分布极广(图4),从北非的厄立特里亚,到东非中部的乌干达、肯尼亚和坦桑尼亚,再到中南部的纳米比亚、博茨瓦纳、赞比亚、津巴布韦和莫桑比克,直至南非的最南端,因此所提供的有效地理溯源信息不足,属于这一单倍型的17份样品均无法确定确切的地理来源。这一点在LoxodontaLocalizer数据库的说明中也有提示。


续表2

图3 匹配的地理单倍型在非洲的地理分布(地图来源Loxodonta Localizer数据库)Fig.3 The geographic distribution map of African elephant mtDNA geographical haplotypes matched by the samples of this study (map source:Loxodonta Localizer database) 注:不同颜色的圆点表示不同的地理单倍型,饼图表示一个地区存在多个单倍型 Note:Different geographical haplotypes are displayed with dots of different colors,and when there are multiple haplotypes in a region,it is represented by a pie chart

图4 地理单倍型LL062在非洲的地理分布(地图来源Loxodonta Localizer 数据库)Fig.4 Geographical distribution map of the geographic haplotype LL062 in Africa (map source:Loxodonta Localizer database)
图3显示,126份象牙样本主要来源于3个热点地区,第1个地区为中非刚果盆地的Tridom (Tri-National Dja-Odzala-Minkébé) 跨境生态保护区及其周边地区,包括加蓬、刚果(布)、喀麦隆、中非共和国和刚果(金)等国,该地区也是非洲森林象的主要分布区[31];第2个地区是东非的肯尼亚、坦桑尼亚和乌干达等国;第3个地区是非洲南部的Kavango-Zambezi (KAZA)跨境保护区,包括纳米比亚、津巴布韦、博茨瓦纳和莫桑比克。此外,也有少量样品被溯源到其他国家和地区,如厄立特里亚和南非等。但样品数量较少,未能反映来源地是否有集中连片的态势。
3 讨论
3.1 提高地理溯源的精度和准确性
本研究首次对我国国境口岸截获的部分象牙进行地理溯源。因为象牙样本的质量变化较大,采用核基因微卫星遗传标记产生的数据质量不够高[4-5,32-33],故此,最终采用mtDNA控制区的序列溯源。
虽然LoxodontaLocalizer数据库的信息检索中有62%的单倍型是特定国家或地区独有的[9-10],如本研究中的LL042和LL116等,但有很多单倍型在多个国家或地区广泛分布,如LL062单倍型。分布越广泛的单倍型溯源有效性越低,追溯的地理来源信息越不可靠。mtDNA单倍型的多样性与所检测的序列长度有关,越长的序列可能包含的多态性位点越多,定义的单倍型也越多。本研究中采用了316 bp的控制区序列溯源,125个单倍型在该区域内有42个多态性位点。但是在LoxodontaLocalizer 数据库中的653条4 258 bp的序列中有多达424个多态性位点[9],如果把检测的范围进一步扩大,采用多区域联合溯源,可能会提高溯源的精准度。
另一方面,还可以通过采用多种方法联合溯源的策略来获得更多的地理来源信息,提高溯源精准度,mtDNA与核DNA标记的进化模式不同,可以从两个侧面提供谱系地理学的信息[34-36];稳定同位素提供了各地区的生态系统元素构成的信息[11-15]。因此,如果3种方法联合使用将会极大地提高地理溯源的精准性[9,15]。
但在实践中材料的消耗量也是一个重要的考量因素。象牙中的DNA都是孤独片段化的,且含量较少。需要较多的材料才能获得足够用的DNA。如果mtDNA分析采用传统的PCR产物Sanger测序,不得不把每个区段分别扩增,而每个区段能支持的扩增长度都在400 bp以下[37],故此所考察的区域越长,扩增的次数越多,DNA的耗费量也随之越大。微卫星的分析虽然采用复合扩增,但不同位点的扩增程序差异较大,无法完全复合到一个反应中,DNA的消耗量也很大[4-5,32-33]。今后如果采用二代测序技术,可以把不同区段的扩增复合到一个体系中,从而节省大量的DNA。
3.2 国境口岸截获象牙的地理来源
本研究成功获得了109份国境口岸截获象牙样本的地理来源,推测了3个热点地区:地区一是中非刚果盆地的Tridom生态保护区及其周边地区;地区二主要是东非的肯尼亚、坦桑尼亚和乌干达等国;地区三是非洲南部的KAZA跨境保护区,这与Wasser等[5,33,38]的研究结果基本一致。Wasser等[5,33,38]对2006年以来缉获的大宗非洲象牙溯源结果分析发现,该时期的非洲象牙主要来源于两大热点地区和一个新兴热点地区,其中坦桑尼亚东南部和毗邻的莫桑比克北部及其周边地区为一块热点地区;非洲森林象牙的偷盗猎主要来源于Tridom跨境生态保护区域[5,38];KAZA跨境保护区是近几年发现的又一块新兴的象牙盗猎热点地区[33,39-40]。
根据Wasser等[33]的研究,非洲象牙盗猎的3个主要国际跨国犯罪组织分别在肯尼亚的蒙巴萨、乌干达的坎帕拉和多哥的洛美。本研究推测的热点地区——Tridom跨境生态保护区,盗猎象牙主要是从多哥的洛美向外出口走私;地区二肯尼亚、坦桑尼亚和乌干达等国,偷盗猎象牙主要从肯尼亚的蒙巴萨或乌干达的恩德培向外出口走私;地区三KAZA跨境保护区是近年新出现的偷盗猎热点地区,该地区象牙大多是在刚果民主共和国的金沙萨装入集装箱开始走私运输。同时,非洲跨国犯罪组织间存在广泛地联系,走私出境港口也随着执法打击的深入不断变化,如在东非走私的集装箱运输港口近年从坦桑尼亚转移到肯尼亚再转移到乌干达,通过卡车或铁路运输至蒙巴萨。而西非的走私网络也从多哥突然转移到尼日利亚,成为西非走私出口业务的主要枢纽[33]。
跨国犯罪组织利用世界贸易的快速增长及全球化发展,在每年全球运输的10亿个集装箱中隐藏其违禁品,他们将来自非洲各地偷盗猎的大量象牙合并且集装箱化,出口国家通常不是偷猎大象所在国,而是其他的非洲国家[8,33]。因此,通过识别缉获象牙的地区,结合跨国犯罪组织象牙走私的贩运网络,可以增加走私象牙的缉获率,有利于未来在走私象牙离开非洲之前阻止其贸易。
致谢:感谢南京森林警察学院刑事科学技术学院提供DNA样本。
