特早熟玉环柚“早玉文旦”的荧光SSR标记鉴别
2022-10-12宋其岩陈友吾李元春叶碧欢沈建军胡传久杨希宏李海波
宋其岩,陈友吾,李元春,叶碧欢,沈建军,胡传久,杨希宏,李海波
(1 浙江省林业科学研究院,杭州,310023;2 玉环市自然资源和规划局,浙江玉环,317606;3玉环市农业农村和水利局,浙江玉环,317606)
中国是柚类Citrusgrandis(C.maxima)起源中心之一,栽培历史悠久,种质资源丰富,形成了包括福建、浙江、台湾的东南沿海柚产区,以广西及广东为主的华南柚产区,以四川、重庆、湖南、贵州为主的西南柚产区[1]。我国柚类的遗传资源高度多样化,大致分为沙田柚、文旦柚和杂种柚3个品种群,同时具有特早熟、早熟、中熟和晚熟配套的柚类资源结构[2]。特早熟玉环柚“早玉文旦”是从普通玉环柚“玉环文旦”芽变中选育出的优良新品种[3]。与普通玉环柚相比,“早玉文旦”可以提早一个半月上市,含糖高,化渣性好,酸甜适中,丰产性,适应性强。柚类品种的鉴别手段包括叶片(果实)的形态学和生物化学方法[4]以及高光谱成像技术[5-6],这些方法存在准确度不高、成本较高且耗时耗力、计算工作量繁琐且稳定性较低的缺陷。基于PCR的分子标记技术如RAPD和ISSR[7]、SNP基因分型[8]、SRAP[9-11]和SSR(微卫星)[12]也相继用于柚类品种的亲缘关系和遗传多样性分析,但RAPD、ISSR和SRAP引物的特异性不高,重复性较差;尽管目前的技术获得SNP位点较易,但单个SNP位点最多只有4种变异,多态信息含量也不高。因此这些分子生物学技术也并非柚类品种鉴别的理想选择。生产上,“早玉文旦”和“玉环文旦”需等到挂果后才可以鉴别。近几年,随着“早玉文旦”种苗需求的增加,市场上与“玉环文旦”混杂或直接用“玉环文旦”冒充的现象屡见不鲜,因此迫切需要开发“早玉文旦”的早期鉴别技术。
SSR(Microsatellite,微卫星)为共显性标记,具有多态性丰富、操作简单、结果可靠、重复性好等优点,在DNA指纹图谱的构建、遗传多样性分析、基因定位、分子标记辅助育种等方面得到广泛应用[13]。荧光SSR标记是基于DNA测序平台的毛细管电泳检测方法,是以不同颜色的荧光基团(如FAM、HEX、TAMRA等)来标记一对SSR引物中的一个引物末端,利用荧光检测器对产物检测,自动识别扩增产物的大小,具有快速、高效、精确、灵敏等技术优势。因此,本研究拟利用荧光SSR标记技术揭示“早玉文旦”和“玉环文旦”的基因型差异,对于“早玉文旦”品种鉴别与知识产权保护具有重要意义。
1 材料与方法
1.1 植物材料特早熟玉环柚“早玉文旦”和普通玉环柚“玉环文旦”无性系来自浙江省玉环市苔山塘文旦柚基地,采集时间为2021年4月。每个品种均采集3株,每株取2片幼嫩叶,混合后放置于密封袋,硅胶干燥保存,至完全干燥后提取基因组DNA。
1.2 主要试剂与仪器植物基因组DNA提取试剂盒(BioTeke,北京)、2×T5 Super PCR Mix(PAGE)(TSINGKE,北京)、pMD-18T载体(TAKARA,大连)等。在SSR正向引物上加注的荧光染料是FAM(蓝)和HEX(绿),SSR引物由TSINGKE公司合成。毛细管电泳检测内标试剂是Hi-DiTMFormamide(Applied Biosystems)、GeneScanTM-500 LIZ Size Standard(Applied Biosystems)。PCR仪(Life ECO,Bioer,杭州;Veriti 9902,ABI,USA)、DNA分析仪(ABI 3730 XL Genetic Analyzer,Applied Biosystems,USA)、NanoDrop 2000(Thermo Scientific,USA)等。
1.3 基因组DNA提取将各品种的幼嫩叶片混合粉碎,取混合样提取基因组DNA。DNA提取方法参照植物基因组DNA提取试剂盒的操作说明。提取后的DNA测定浓度,并经琼脂糖凝胶电泳检测,于-20 ℃保存备用。
1.4 SSR-PCR分析SSR标记Cg_U110来源于对NCBI数据库中柚类C.maxima的转录组unigenes筛选,CT02来源于Barkley等[14]的报道(见表1)。
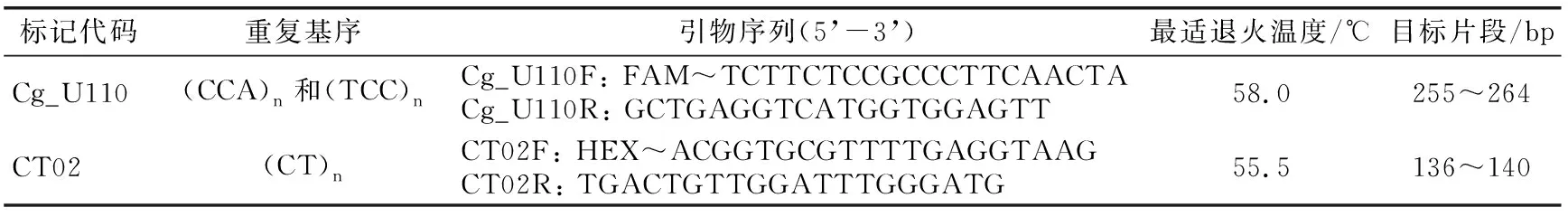
表1 用于柚类品种“早玉文旦”和“玉环文旦”鉴别的SSR标记引物
Cg_U110独立标记的20 μL SSR-PCR扩增体系为:2×T5 Super PCR Mix(PAGE)10 μL,荧光标记(FAM)的正向引物Cg_U110F和反向引物Cg_U110R(均为10 μmol·L-1)各为1.5 μL,DNA模板(20 ng·μL-1)3 μL,加ddH2O补齐到20 μL;PCR反应条件为:98 ℃预变性2 min后;98 ℃变性10 s,58 ℃退火10 s,72 ℃延伸10 s,共30个循环;最后于72 ℃补平2 min,终止温度为4 ℃。
CT02独立标记的SSR-PCR扩增体系同上,其中荧光标记(FAM)的正向引物CT02F和反向引物CT02R(均为10 μmol·L-1)各为1.5 μL,退火温度为55.5 ℃。
Cg_U110和CT02组合标记的25 μL多重SSR-PCR扩增体系为:2×T5 Super PCR Mix(PAGE)12.5 μL,荧光标记(FAM)的正向引物Cg_U110F和反向引物Cg_U110R(均为10 μmol·L-1)分别为0.65 μL和0.4 μL,荧光标记(HEX)的正向引物CT02F和反向引物CT02R(均为10 μmol·L-1)分别为0.45 μL和0.5 μL,DNA模板(30 ng·μL-1)3 μL,加ddH2O补齐到25 μL。优化后的PCR反应条件为:98 ℃预变性2 min后;98 ℃变性10 s,56.3 ℃退火15 s,72 ℃延伸15 s,共35个循环;最后于72 ℃补平2 min,终止温度为4 ℃。
1.5 毛细管电泳检测内标配制:参照油茶品种SSR鉴别报道的方法[15-16]和多花黄精SSR分子标记报道的方法[17]。具体为:取10 mL Hi-Di 和80 μL GeneScanTM-500 LIZ Size Standard混匀,离心,以每孔10 μL分装于96孔内标板,离心。将SSR-PCR产物电泳,根据电泳胶图做一定稀释(最低可检测标准为0.1 ng/μL),离心。取稀释产物0.5 μL加入到分配好的内标板中,混匀,离心,放入PCR仪,于96 ℃变性5 min。20 ℃下迅速冷冻2 min,离心。置入DNA分析仪进行毛细管电泳检测。用Data Collection 3.0软件收集数据。
1.6 Cg_U110标记的单克隆测序采用DNA凝胶回收试剂盒从1.5%琼脂糖凝胶中回收2个柚类品种Cg_U110标记的PCR扩增片段,回收产物用pMD-18T载体连接,转化于大肠杆菌(Escherichiacoli)DH5α菌株感受态细胞,经蓝白斑筛选和PCR鉴定后,每个样品均随机挑选16个阳性单克隆交付杭州有康生物技术有限公司完成测序。
1.7 数据分析用GeneMapper 4.1软件对Data Collection 3.0软件收集的原始数据进行分析。软件系统将根据目标峰的位置与同一泳道中的内标GeneScanTM-500 LIZ Size Standard进行比较,直接给出目标SSR片段大小的准确数值(bp)。纯合位点的等位变异数据记录为X/X,其中X为该等位变异的数值大小;杂合位点的等位变异数据记录为X/Y,其中X、Y为该位点2个不同等位变异的数值大小。
2 结果与分析
2.1 Cg_U110和CT02独立标记鉴别基于柚类物种C.maxima的转录组数据,识别unigenes中的SSR位点,并进一步设计SSR引物,以筛选出可用于柚类品种鉴别的SSR标记。通过对40对SSR荧光引物的PCR扩增和琼脂糖凝胶电泳检测,29对引物可以扩增出清晰明亮、稳定的SSR条带。收集SSR-PCR扩增产物,经测序验证,其中的24对引物能够扩增出目标重复序列。对24对SSR荧光引物的PCR扩增和毛细管电泳检测,结果显示,仅Cg_U110标记在“玉环文旦”和“早玉文旦”二个品种间扩增出了差异性的基因型,即“玉环文旦”为253/259,而“早玉文旦”为259/262。为进一步筛选可鉴别“玉环文旦”与“早玉文旦”的多态性SSR标记,对Barkley等[14]报道的18对SSR引物进行多态性筛选,结果显示仅CT02标记在“玉环文旦”与“早玉文旦”品种间扩增出了不同的基因型,即“玉环文旦”为136/136,而“早玉文旦”为136/140。这表明,SSR标记Cg_U110和CT02皆可作为分子特异性SSR指纹来鉴别“玉环文旦”与“早玉文旦”。
2.2 Cg_U110标记的测序验证对2个品种Cg_U110标记单克隆测序结果显示,“玉环文旦”的CCA重复基序有二种,分别为(CCA)7和(CCA)4;TCC重复基序也有二种,分别为(TCC)6和(TCC)7。“早玉文旦”的CCA重复基序有一种,为(CCA)7;TCC重复基序有二种,分别为(TCC)7和(TCC)6(见图1)。“玉环文旦”的基因型为255/261,“早玉文旦”的的基因型为261/264。这一结果与毛细管电泳检测出的结果(253/259和259/262)基本一致,仅有2个碱基的误差。

图1 Cg_U110标记在2个柚类品种的DNA序列及基因型
2.3 Cg_U110和CT02组合标记鉴别为提高“玉环文旦”和“早玉文旦”的鉴别效率,进一步建立了Cg_U110和CT02标记的多重SSR-PCR扩增体系。通过对反应体系和退火温度的优化,确定了在25 μL的多重PCR反应体系中,Cg_U110F/Cg_U110R(10 mol·L-1)的体积配比为0.65 μL/0.4 μL,CT02F/CT02R(10 mol·L-1)的体积配比为0.45 μL/0.55 μL;最适退火温度为56.3 ℃。扩增结果显示,2个柚类品种在SSR位点CT02和Cg_U110可扩增出2种不同的基因型组合,即“玉环文旦”为136/136和255/261,而“早玉文旦”为136/140和261/264(见图2)。为保证多重PCR结果的可靠性,在56.3 ℃的退火温度下,分别使用了来自不同公司的PCR反应酶(TSINGKE,北京和TAKARA,大连)和PCR扩增仪(Life ECO,Bioer,杭州和Veriti 9902,ABI,USA),均显示扩增结果稳定、可重复。可见,Cg_U110和CT02标记可作为分子特异性SSR指纹组合通过上述多重SSR-PCR扩增体系来鉴别“玉环文旦”与“早玉文旦”。
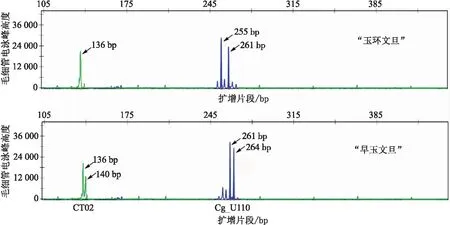
图2 Cg_U110和CT02引物组合扩增的2个柚类品种基因型
3 讨论
良种是现代农业发展的基础,优良品种的选育工作对于高产、优质、高效农业的可持续发展至关重要。柚类植物为木本果树,新品种的选育不仅需财力支持,更需要投入大量的时间和精力。有效保护好选育的新品种,才会调动育种者的积极性,有利于育种技术的持续创新和优良新品种的不断选育[18]。
“早玉文旦”是从“玉环文旦”芽变中选育出的新品种。果树芽变的实质是体细胞遗传物质发生改变,包括染色体结构数目的变异和基因突变。由于芽变品种与芽变母本的遗传背景几乎一致,常规的分子标记并不一定能覆盖到变异位点,芽变品种鉴别的难易程度与变异类型和变异程度有很大关系。利用RAPD和SSR分子标记,“沙田柚”(C.maxima)的芽变品种“真龙柚”被成功鉴定[19]。利用ISSR和SRAP分子标记,皮球桃(Prunuspersica)的黄肉微小芽变品种被成功鉴定[20]。本研究对柚类转录组数据进行大量筛选,仅获得1个可用于“早玉文旦”和“玉环文旦”鉴别的核心SSR标记Cg_U110,表明“早玉文旦”与芽变母本“玉环文旦”具有几乎一致的遗传背景,遗传差异很小,也证明了“早玉文旦”确实为“玉环文旦”的自然突变体,而非环境引起的饰变。果树芽变品种鉴别的工作量和难度均较大,随着越来越多的物种被基因组测序,以及测序技术的不断更新,除了通过转录组数据获得SSR位点外,也可通过SNP位点或基因组重测序,从全基因组范围内挖掘变异位点,用于果树芽变品种的鉴别[21]。
与普通PCR法相比,多重荧光PCR技术的应用不仅可提高工作效率,也减少了各种试剂和模板DNA的用量。在多重荧光PCR反应体系中的引物可以用不同颜色的基团(FAM、HEX、NED、PET等)进行修饰,只要扩增片段的大小不重叠,在同一毛细管电泳的泳道中可检测多达20对以上的扩增片段,从而大幅度提高检测效率[22]。本研究利用多重荧光PCR技术整合了核心SSR标记Cg_U110和CT02的应用,实现了柚类良种“早玉文旦”的高效鉴别。
SSR标记已被用于许多植物的DNA指纹数据库构建[23-30],SSR标记也是UPOV(国际植物新品种保护公约)推荐的DNA指纹数据库构建标记。植物品种DUS(Distinctness,Uniformity,Stability,简称DUS)测试指南是植物新品种特异性、一致性和稳定性测试的技术标准,是指导DUS测试开展工作的行动指南[31]。在本研究基础上,可以基于NCBI的转录组或基因组测序数据,进一步发掘更多的多态性SSR标记,应用于更多柚类品种的鉴别,并拓展到柚类品种分子标记DUS测试指南研制和柚类品种DNA指纹数据库构建,为柚类新品种申请、测试与鉴定提供补充依据。
4 结论
基于柚类物种C.maxima的转录组测序数据筛选出了多态性SSR标记Cg_U110和CT02,利用多重荧光PCR技术将Cg_U110和CT02整合应用,母本品种“玉环文旦”及其芽变品种“早玉文旦”在这2个SSR位点(Cg_U110和CT02)分别扩增出2种不同的基因型组合,即255/261//136/136和261/264//136/140,可一次性鉴别2个柚类品种。本研究为“早玉文旦”优良柚类品种的早期鉴别与知识产权保护提供了高效便捷的分子手段。
