8种植物类甜蛋白家族基因进化及密码子特征分析
2022-06-12刘潮韩利红盛巧陶雾
刘潮 韩利红 盛巧 陶雾



摘要:类甜蛋白(thaumatin-like proteins,TLP)被归为病程相关蛋白第5家族,在植物防御和生长发育过程中发挥作用。使用Mega X、CodonW及EMBOSS网站在线软件,分析8种植物(莱茵衣藻、小立碗藓、拟南芥、水稻、谷子、高粱、小麦、玉米)类甜蛋白家族进化和密码子使用偏性。结果表明,植物类甜蛋白主要归为10个聚类组,其中聚类组Ⅴ、Ⅵ、Ⅶ中的TLP数目相对较多;5种禾本科作物TLP家族基因主要以G和C作为第3位密码子,密码子具有较强的偏好性,多数类甜蛋白基因ENC值较小,GC3s值分布较集中;8个最优密码子第3位均为G或C结尾,禾本科植物类甜蛋白家族基因密码子偏好G/C结尾。本研究分析了8种植物中类甜蛋白家族基因的进化特征及密码子使用偏性,为该家族基因的分子进化以及转基因应用提供了理论依据。
关键词:类甜蛋白;系统进化;密码子偏性;最优密码子
中图分类号: S188 文献标志码: A
文章编号:1002-1302(2022)10-0044-08
类甜蛋白(thaumatin-like proteins,TLP)属于病程相关蛋白第5(pathogenesis-related protein,PR5)家族,因与产自西非的翅果竹芋(Thaumatococcus danielli)的甜蛋白(thaumatin)氨基酸序列相似度较高而得名。TLP广泛存在于多种动植物和微生物中[1-3]。研究发现,TLP具有抗真菌和渗透调节活性等,或作为其他抗真菌蛋白的激发子[4-7],在植物的生长发育和抗逆过程中发挥重要作用[8-10]。不同物种基因组中密码子的使用频率不同,这种密码子非均衡使用的普遍现象称为密码子使用偏性(codon usage bias)[11]。同义密码子的选择性使用提高了翻译的准确性和效率,生活在相似环境条件下的物种具有相似的密码子偏好,能够生活在多种生境中的生物,翻译效率降低,密码子使用偏性对基因的表达起重要的调节作用[12]。密码子使用偏性分析对于基因表达预测、异源表达最适宿主选择和密码子优化均具有重要意义[13]。不同植物物种编码TLP数量有很大差异,如胡萝卜(Daucus carota)和黄瓜(Cucumis sativus)等蔬菜中有约30个,水稻(Oryza sativa)和谷子(Setaria italica)等作物中多于50个[14]。植物基因组中TLP基因数量除了与基因组大小有关外,植物在进化过程中所处的环境也对TLP数量有重要影响,当植物面临较大的环境胁迫压力时,对抗环境胁迫的相关基因通过基因间和基因内重组,导致了基因数目的增加,从而使植物发生了快速的适应性进化[15],如RLK家族近期的扩张与植物防御/抗病基因有关[16]。密码子偏性主要受突变压力[17]和自然选择压力影响[9]。突变—选择—漂变平衡假说认为,突变的发生具有方向性,突变—选择平衡导致持续进化的种群具有稳态适应性突变[18]。通过基因工程等手段对基因密码子进行有目的的改造,能大大提高外源基因在宿主中的表达水平[19],因此密码子偏性的研究对基因工程具有重要的应用价值。
随着现代分子生物学的发展,人类已进入了后基因组时代,大量植物基因组数据的公布为研究植物基因家族进化提供了可能。植物TLP家族在植物的生长发育和抵御环境胁迫中发挥了重要作用,然而,目前对不同物种中该家族基因的进化和密码子偏性研究鲜见报道。本研究从蛋白氨基酸和基因的碱基组成出发,从不同类群的物种中选择莱茵衣藻、小立碗藓、拟南芥、玉米、水稻、小麦、高粱和谷子进行TLP家族进化、编码序列(coding sequence,CDS)密码子的组成及使用偏性分析,为TLP家族基因的开发利用和优良抗性作物品种的培育奠定基础。
1 材料与方法
1.1 基因序列获取与鉴定
2021年1月通过Superfamily网站(http://supfam.org/SUPERFAMILY)和基因组序列数据库(http://plants.ensembl.org/index.html)以thaumatin为关键词搜索数据库,获得并下载莱茵衣藻(Chlamydomonas reinhardtii)、小立碗藓(Physcomitrella patens)、拟南芥(Arabidopsis thaliana)、水稻(O. sativa)、谷子(S. italica)、高粱(Sorghum bicolor)、小麦(Triticum aestivum)、玉米(Zea mays)中TLP家族蛋白及CDS序列,使用SMART数据库对蛋白功能域进行确认,去掉不含类甜蛋白结构域的序列。
1.2 基因家族进化分析
所有蛋白通过Clustal X比对,应用Mega X构建邻接(neighbor-joining,NJ)系统发生树。NJ进化树采用泊松校验,分析步长值为1 000。
1.3 基因同义密码子偏性分析
1.3.1 密码子组成和使用特性分析
使用CodonW软件分析基因CDS密码子使用偏性,分析参数:同义密码子在第3位上相应碱基的出现频率(A3s、G3s、C3s、T3s)、密码子适应指数(codon adaptation index,CAI)、有效密码子数(effective number of codon,ENC)、密码子第3位的G+C含量(GC3s)和基因的G+C含量(GC)等。通過EMBOSS在线软件对TLP家族基因同义密码子相对使用度(relative synonymous codon usage,RSCU)进行分析。使用统计软件SPSS 23.0对密码子组成和使用偏性各参数进行相关性分析。
1.3.2 ENC-plot绘图分析
分别以GC3s和ENC为横纵坐标,绘制ENC-plot,检测碱基组成对密码子偏性的影响[20]。图中曲线表示密码子偏性仅受碱基影响时的ENC预期值,此时ENC计算公式为ENC=2+GC3s+29/[GC3s2+(1-GC3s)2],靠近标准曲线表示密码子偏性主要受碱基突变影响,远离标准曲线表示密码子偏性主要受自然选择影响。
1.3.3 最优密码子确定
选择ENC值前后各10%作为低表达和高表达基因,分别计算2组基因中TLP基因密码子的RSCU值。ΔRSCU=高表达组RSCU-低表达组RSCU,当ΔRSCU>0.3,且在高表达组中RSCU>1,在低表达组中RSCU<1,可确定该密码子为最优密码子(optimal codon)[3,21]。
2 结果与分析
2.1 TLP家族基因数量分析
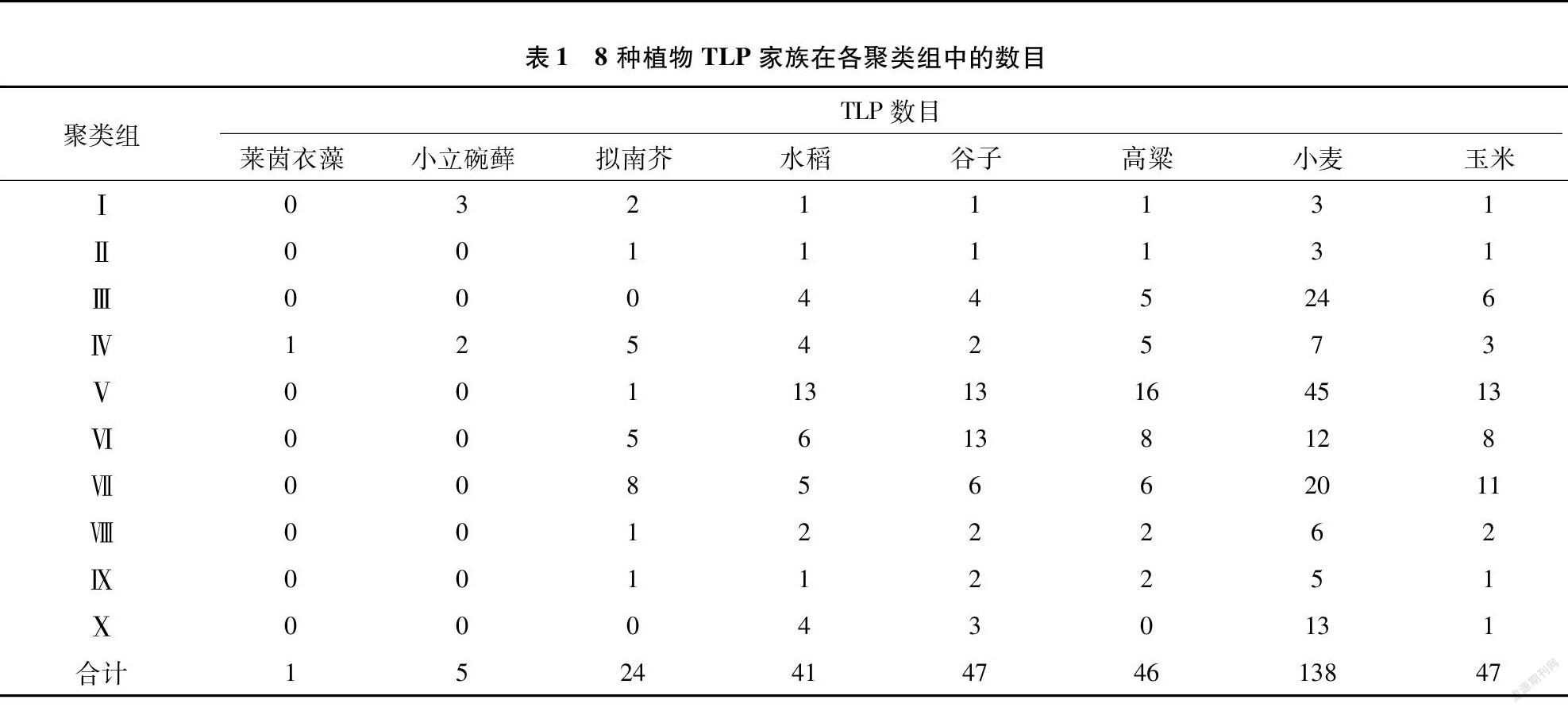
通过搜索Superfamily数据库,鉴定到植物TLP蛋白数量分别为莱茵衣藻1个、小立碗藓5个、拟南芥24个、水稻41个、谷子47个、高粱46个、小麦138个、玉米47个。植物TLP数量与基因组大小呈正相关,随基因组的增大,TLP数目增加。因小麦为六倍体,其基因组较大,染色体数目较多,单位Mb染色体上的TLP蛋白较少,只有0.010 3个。虽然拟南芥中总TLP数目只有24个,但因其基因组相对较小,且染色体只有10条,单位Mb染色体上的TLP蛋白数量最多,达到0.196 9个。水稻、高粱和谷子中的染色体数目比较接近,其TLP蛋白数量也相差不是很大。
2.2 TLP家族进化分析
使用MEGA 5.0软件通过蛋白序列对8种植物TLP家族成员进行进化分析。参考前人研究[1,15]将植物TLP家族蛋白归为10个聚类组(图1)。分析发现,水稻、谷子、小麦和玉米在全部10个聚类组中均有成员分布,高粱在除聚类组Ⅹ外的聚类组中均有成员分布,拟南芥在聚类组Ⅲ和Ⅹ中没有成员分布(表1)。研究发现,各植物TLP在10个聚类组中分布很不均衡,聚类组Ⅴ、Ⅵ、Ⅶ各组中的TLP数目相对较多,其中水稻、谷子、高粱、小麦、玉米5种作物聚类组Ⅴ中的成员均明显较多,说明这些聚类组中的基因出现了特异性的基因扩张。聚类组Ⅴ中的OsTLP基因数量最多,该组中的一些TLPs能响应病原或环境胁迫[16],说明这些基因可能与植物对抗胁迫有关,有待深入研究。
2.3 TLP家族密码子偏性分析
2.3.1 8种植物TLP家族基因密码子使用参数分析
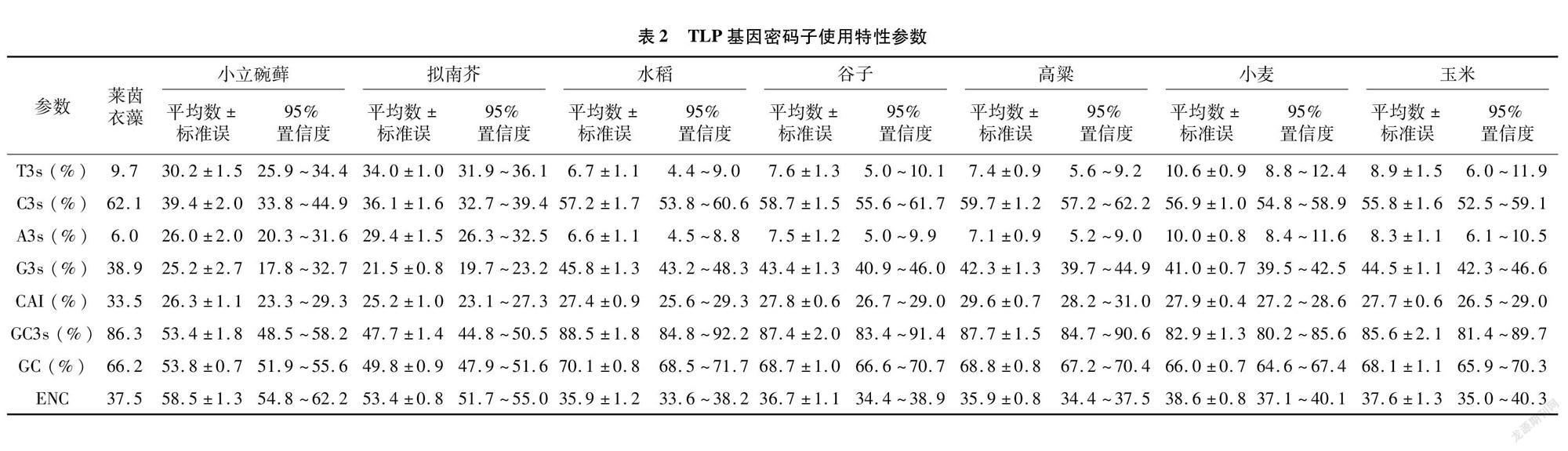
为全面了解植物TLP家族基因密码子使用性,对8种植物的同义密码子在第3位上相应碱基的出现频率、CAI、ENC、GC3s等参数进行分析(表2)。分析密码子第3位的核苷酸发现,小立碗藓和拟南芥对A、T、C、G 4种核苷酸的使用比例相当,第3位密码子偏性不是很明显,但5种禾本科作物种C3s和G3s数值明显高于T3s和A3s,主要以C和G作为第3位密码子, 其中以C作为第3位密码子的概率比G要高一些。8种植物TLP家族基因GC含量也不相同,拟南芥TLP家族基因GC含量为49.8%,小立碗藓为53.8%,而5种禾本科作物均不低于66%。各物种中GC3s与GC含量类似,5种禾本科作物中均较高,且均高于82%,水稻中甚至达到88.5%。
CAI用于衡量基因与高表达基因密码子使用的接近程度,该值越高表示基因表达水平越高,反之,表达水平越低。拟南芥和小立碗藓TLP家族基因CAI值分别为25.2%和26.3%,莱茵衣藻的TLP家族基因CAI值为33.5%,5种禾本科作物TLP家族基因CAI值均不低于27.4%,高粱TLP家族基因CAI值高达29.6%。ENC值反映基因编码对密码子选择性强弱[18],ENC值越小表示该基因表达潜力越高,基因对密码子的使用偏性越强[20]。小立碗藓和拟南芥TLP家族95%的基因ENC值均高于50,说明其TLP家族基因密码子使用偏性较弱,多数属于低表达基因,而水稻、谷子、高粱、小麦和玉米TLP家族95%的基因ENC值均介于33~40之间,平均值介于35~39之间,说明这些TLP基因密码子使用偏性较强,具有较高的表达潜力,可能在植物生长或抵御逆境过程中发挥作用。
2.3.2 有效密码子数与同义密码子第3位GC含量关联分析
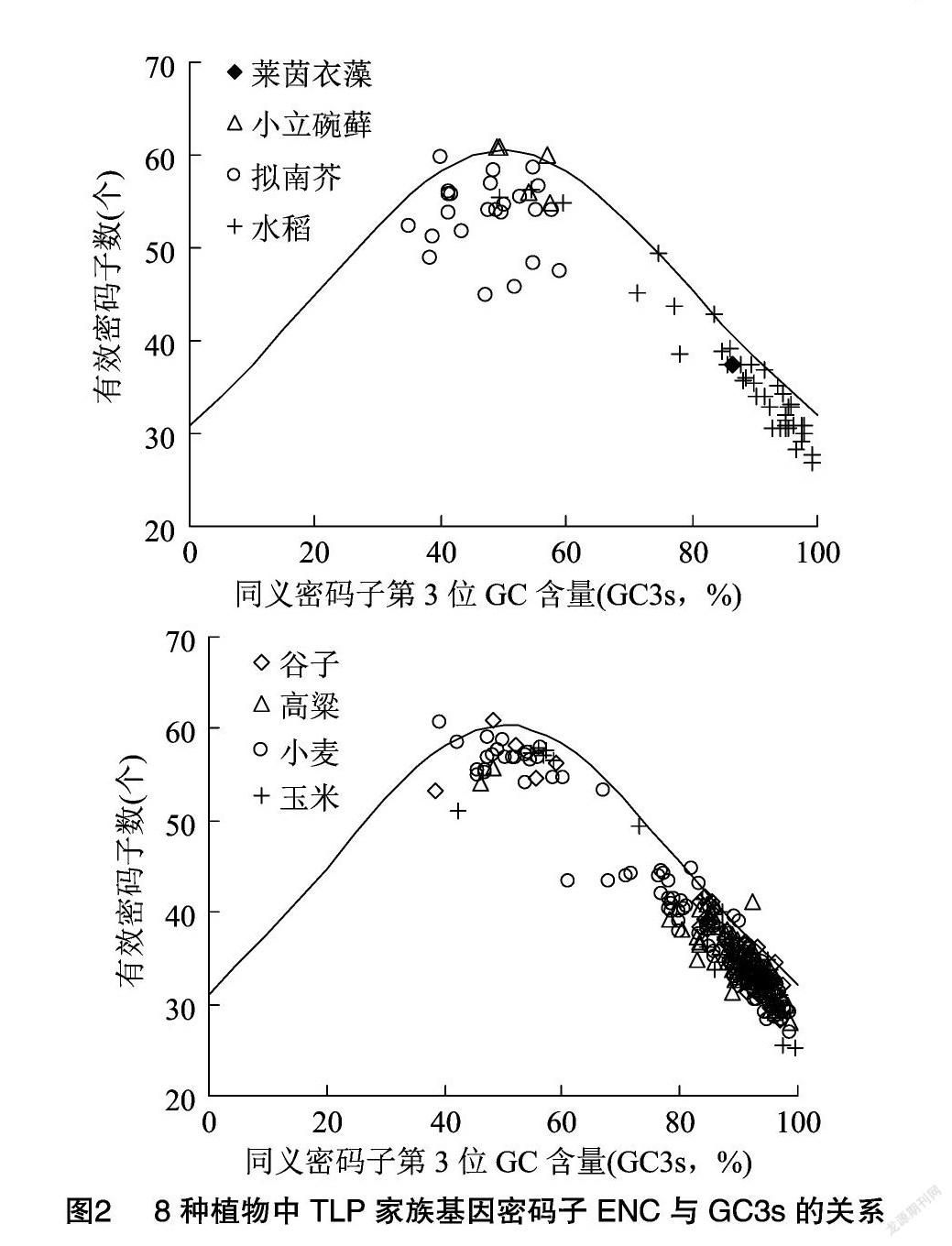
GC3s分布反映了植物所受的选择压力,GC3s分布越分散,代表密码子使用偏性主要受碱基突变压力影响,GC3s分布越集中,代表密码子使用偏性主要受自然选择压力影响[3,22]。ENC-GC3s关联分析中,基因分布越靠近标准曲线,表示密码子使用偏性受到较小的自然选择压力影响,反之表示基因受到选择压力或其他因素的影响越大[23]。莱茵衣藻只有1个TLP基因,其ENC值为37.51,GC3s值为86.3%(图2),与本研究中其他植物大部分基因集中在同一位置。小立碗藓有5个TLP基因,其中3个分布位置非常接近标准曲线(图2),说明在小立碗藓中TLP基因受自然选择压力影响较小。拟南芥基因分布于标准曲线下方,但GC3s分布较广泛(图2),说明拟南芥TLP基因受到突变选择和自然选择的双重影响。值得注意的是,水稻、小麦、高粱、谷子、玉米几种主要作物中大多数TLP基因ENC值较小,GC3s值分布较集中,且位于标准曲线下方,说明这些物种中TLP基因主要受到自然选择压力的影响。
2.3.3 类甜蛋白家族基因同义密码子相对使用度分析
若RSCU>1,密码子出现的频率比其他同义密码子高,反之,出现频率低。TLP基因RSCU值分析表明,不同植物物种氨基酸密码子RSCU值不同,5种单子叶禾本科作物RSCU值一致性较高,TLP家族基因25个RSCU>1的氨基酸密码子均以G或C结尾,拟南芥作为双子叶植物的代表与单子叶植物密码子RSCU值趋势多數截然相反(表3)。如编码丙氨酸的密码子,拟南芥偏好GCT,而其他7种植物偏好GCG或GCC;编码半胱氨酸的密码子,拟南芥偏好TGT,而其他7种植物偏好TGC。
2.3.4 最优密码子分析
通过对高表达和低表达组密码子RSCU值进行比较,发现8个最优密码子分别编码丙氨酸(Ala,GCG)、谷氨酸(Gly,GGG)、组氨酸(His,CAC)、脯胺酸(Pro,CCC和CCG)、精氨酸(Arg,CGG)、 色氨酸(Ser,TCG)、 苏氨酸(Thr,ACG)(表3)。其中6个密码子第3位为G,2个密码子为C,表明所分析的植物,尤其禾本科作物优先使用G或C结尾的密码子。
3 讨论
类甜蛋白在植物的发育和对抗胁迫过程中发挥作用[7-8,24-25],该家族基因数目的多少除了与基因组大小有关外,植物面临的环境胁迫压力往往也影响了基因的扩张[1]。本研究中莱茵衣藻和小立碗藓编码了相对更少量的TLP家族基因,其次是小麥和玉米,而小麦的基因组在进化过程中均曾多次发生染色体加倍现象[26],玉米很可能是一种异源四倍体起源的生物[27]。生物的生存策略有2种,一种是通过快速增加个体,从而提高下一代的个体数量,另一种是通过发展抵抗胁迫的防御系统,提高单个个体的存活概率。植物TLP家族基因在植物的防御反应中发挥作用,而一些禾本科作物可能通过增加TLP家族基因数目增强防御能力,提高后代存活概率,因此含有较多的TLP家族基因。
在长期的进化过程中,为了适应环境和应对胁迫,植物对编码特定蛋白氨基酸的密码子使用出现偏性,使用频率最高的密码子称为最优密码子[28]。
8种植物TLP家族基因密码子使用偏性有很大不同。拟南芥和小立碗藓TLP家族基因GC含量和GC3s值相对较低,而5种禾本科作物这2类参数均较高。5种禾本科作物主要以G或C作为第3位密码子首选。双子叶植物偏爱A/T结尾的密码子,单子叶植物偏爱G/C结尾的密码子[22,29],较高的GC3s值表示基因表达受DNA甲基化或基因改变影响,加速了基因的进化[30]。本研究结果与前人研究一致,禾本科植物TLP家族基因偏爱GC结尾的密码子,可能是因为单、双子叶植物受植物早期进化选择影响,TLP家族基因密码子使用偏性具有种属特异性。禾本科作物基因GC含量和GC3s影响了基因密码子的偏性,高表达基因偏爱使用G/C结尾的密码子,是选择性驱动了密码子的偏性[31]。本研究通过ΔRSCU法共鉴定了8个最优密码子,其中6个密码子以G结尾,2个以C结尾,这可能与8种植物TLP家族基因较高的GC含量有一定关系[32]。但在其他植物的某些基因中也有相反的结果,如在小麦[23]、水稻[33]和玉米[34]中,基因组GC含量较高,最优密码子中富含AT。基因密码子偏性参数ENC、RSCU和最优密码子分析均表明,5种禾本科作物TLP基因偏好G/C结尾的密码子。ENC与GC3s关联分析发现,小立碗藓TLP家族基因受正向选择压力较小,拟南芥受到突变和正向选择压力双重影响,而5种禾本科植物TLP家族基因密码子具有较高的GC3s值,并远离标准曲线,主要受正向选择压力影响,进一步说明正向选择作用在禾本科作物TLP家族基因进化过程中发挥了显著作用。由于密码子偏性对基因的表达有较大影响,因此开展基因异源表达研究时,须要结合密码子偏性进行设计,否则可能无法获得准确的表达量信息,甚至影响表达产物的获得。
参考文献:
[1]Shatters R G,Boykin L M,Lapointe S L,et al. Phylogenetic and structural relationships of the PR5 gene family reveal an ancient multigene family conserved in plants and select animal taxa[J]. Journal of Molecular Evolution,2006,63(1):12-29.
[2]刘 潮,王 慧,杨 静,等. 水稻类甜蛋白基因家族鉴定及表达的初步分析[J]. 分子植物育种,2017,15(10):3845-3855.
[3]刘 潮,韩利红,王海波,等. 谷子类甜蛋白基因家族的鉴定与密码子偏性分析[J]. 西北农业学报,2018,27(1):52-61.
[4]Liu Y F,Liu L J,Asiegbu F O,et al. Molecular identification and antifungal properties of four thaumatin-like proteins in spruce (Picea likiangensis)[J]. Forests,2021,12(9):1268.
[5]Sun W B,Zhou Y,Movahedi A,et al. Thaumatin-like protein(Pe-TLP) acts as a positive factor in transgenic poplars enhanced resistance to spots disease[J]. Physiological and Molecular Plant Pathology,2020,112:101512.
[6]Faillace G R,Turchetto-Zolet A C,Guzman F L,et al. Genome-wide analysis and evolution of plant thaumatin-like proteins:a focus on the origin and diversification of osmotins[J]. Molecular Genetics and Genomics,2019,294(5):1137-1157.
[7]Menu-Bouaouiche L,Vriet C,Peumans W J,et al. A molecular basis for the endo-β1,3-glucanase activity of the thaumatin-like proteins from edible fruits[J]. Biochimie,2003,85(1/2):123-131.
[8]刘 潮,韩利红,王海波,等. 植物类甜蛋白基因家族研究进展[J]. 生物技术通报,2018,34(3):9-17.
[9]de Jesús-Pires C,Ferreira-Neto J R C,Pacifico Bezerra-Neto J,et al. Plant thaumatin-like proteins:function,evolution and biotechnological applications[J]. Current Protein and Peptide Science,2020,21(1):36-51.
[10]Guo J,Zhao X,Wang H L,et al. Expression of the LePR5 gene from cherry tomato fruit induced by Cryptococcus laurentii and the analysis of LePR5 protein antifungal activity[J]. Postharvest Biology and Technology,2016,111:337-344.
[11]Kimura M.Possibility of extensive neutral evolution under stabilizing selection with special reference to nonrandom usage of synonymous codons[J]. Proceedings of the National Academy of Sciences of the United States of America,1981,78(9):5773-5777.
[12]Arella D,Dilucca M,Giansanti A. Codon usage bias and environmental adaptation in microbial organisms[J]. Molecular Genetics and Genomics,2021,296(3):751-762.
[13]吳宪明,吴松锋,任大明,等. 密码子偏性的分析方法及相关研究进展[J]. 遗传,2007,29(4):420-426.
[14]刘 潮,韩利红,王海波,等. 胡萝卜类甜蛋白家族鉴定与生物信息学分析[J]. 中国蔬菜,2017(2):38-44.
[15]Zhao J P,Su X H. Patterns of molecular evolution and predicted function in thaumatin-like proteins of Populus trichocarpa[J]. Planta,2010,232(4):949-962.
[16]Shiu S H,Karlowski W M,Pan R S,et al. Comparative analysis of the receptor-like kinase family in Arabidopsis and rice[J]. Plant Cell,2004,16(5):1220-1234.
[17]Fedorov A,Saxonov S,Gilbert W. Regularities of context-dependent codon bias in eukaryotic genes[J]. Nucleic Acids Research,2002,30(5):1192-1197.
[18]Desai M M,Fisher D S. Beneficial mutation-selection balance and the effect of linkage on positive selection[J]. Genetics,2007,176(3):1759-1798.
[19]Zelasko S,Palaria A,Das A. Optimizations to achieve high-level expression of cytochrome P450 proteins using Escherichia coli expression systems[J]. Protein Expression and Purification,2013,92(1):77-87.
[20]Wright F. The ‘effective number of codons’ used in a gene[J]. Gene,1990,87(1):23-29.
[21]Duret L,Mouchiroud D. Expression pattern and,surprisingly,gene length shape codon usage in Caenorhabditis,Drosophila,and Arabidopsis[J]. Proceedings of the National Academy of Sciences of the United States of America,1999,96(8):4482-4487.
[22]Kawabe A,Miyashita N T. Patterns of codon usage bias in three dicot and four monocot plant species[J]. Genes & Genetic Systems,2003,78(5):343-352.
[23]Zhang W J,Zhou J,Li Z F,et al. Comparative analysis of codon usage patterns among mitochondrion,chloroplast and nuclear genes in Triticum aestivum L.[J]. Journal of Integrative Plant Biology,2007,49(2):246-254.
[24]Cui Z C,Liang F,Zhang J R,et al. Transgenic expression of TaTLP1,a thaumatin-like protein gene,reduces susceptibility to common root rot and leaf rust in wheat[J]. The Crop Journal,2021,9(5):1214-1218.
[25]Nawrot R,Musidlak O,Barylski J,et al. Characterization and expression of a novel thaumatin-like protein (CcTLP1) from papaveraceous plant Corydalis cava[J]. International Journal of Biological Macromolecules,2021,189:678-689.
[26]齊莉莉,刘大钧. 小麦基因组研究进展[J]. 麦类作物学报,1999,19(1):1-5.
[27]田清震,谢传晓,李新海,等. 玉米基因组学研究进展[J]. 玉米科学,2006,14(3):1-5,9.
[28]Fuglsang A. The ‘effective number of codons’ revisited[J]. Biochemical and Biophysical Research Communications,2004,317(3):957-964.
[29]刘汉梅,赵 耀,顾 勇,等. 几种植物waxy基因的密码子用法特性分析[J]. 核农学报,2010,24(3):476-481.
[30]Tatarinova T V,Alexandrov N N,Bouck J B,et al. GC3 biology in corn,rice,Sorghum and other grasses[J]. BMC Genomics,2010,11:308.
[31]Guo X Y,Bao J D,Fan L J. Evidence of selectively driven codon usage in rice:implications for GC content evolution of Gramineae genes[J]. FEBS Letters,2007,581(5):1015-1021.
[32]Rao Y,Wu G,Wang Z,et al. Mutation bias is the driving force of codon usage in the Gallus gallus genome[J]. DNA Research,2011,18(6):499-512.
[33]Liu Q P,Feng Y,Zhao X A,et al. Synonymous codon usage bias in Oryza sativa[J]. Plant Science,2004,167(1):101-105.
[34]Liu H M,He R,Zhang H Y,et al. Analysis of synonymous codon usage in Zea mays[J]. Molecular Biology Reports,2010,37(2):677-684.
