基于TCGA数据库构建和评估口腔癌的免疫相关预后模型
2022-04-08张核子余晨笛卢晓萍操利超
巴 颖,张核子,余晨笛,卢晓萍,操利超
(深圳市核子基因科技有限公司,广东 深圳 518071)
口腔癌(oral cancer)是头颈部较常见的恶性肿瘤之一,已引起全球关注[1]。在国际分类定义中,口腔癌是包括嘴唇、舌头、牙龈、口腔底部、颊粘膜、口腔前庭或磨牙后区等部位的癌症[2]。尽管目前对口腔癌的诊断和治疗方面已取得了一定的进展,但口腔癌患者的死亡率依然很高,主要原因之一是缺乏有效的预后生物标志物[3]。因此,探索口腔癌潜在的预后标志物和治疗靶点是当前亟需解决的问题。免疫检查点抑制剂(immune checkpoint inhibitors,ICIs)是一种很有前景的免疫疗法,通过抑制负性调节受体发挥作用,如程序性细胞死亡受体1(programmed cell death protein 1,PD-1)和细胞毒性T淋巴细胞抗原4(cytotoxic T lymphocyte antigen 4,CTLA4),从而激活抗肿瘤免疫[4]。免疫治疗在口腔癌中具有更好的疗效,有关口腔癌免疫学的研究不断增加[5,6]。然而,由于肿瘤免疫微环境的异质性和复杂性,只有一小部分患者受益于免疫治疗,持续缓解的患者仍很少。许多免疫相关生物标志物被认为是口腔癌患者有用的预测指标,但目前尚缺乏一种理想的生物标志物用于临床[7-10]。为了预测和改善口腔癌患者的预后,本研究基于癌症基因组图谱-头颈部鳞状细胞癌(the cancer genome atlas-head and neck squamous cell carcinoma,TCGA-HNSC)队列构建和评估了口腔癌免疫相关预后模型,分析该预后模型与口腔癌患者临床病理特征之间的关系,探索肿瘤免疫微环境的特征,包括肿瘤浸润细胞构成、肿瘤突变负荷(tumor mutational burden,TMB)、以及PD-1/PD-L1/CLTA4的mRNA 表达水平,现报道如下。
1 材料与方法
1.1 数据获取 癌症基因组图谱(the cancer genome atlas,TCGA)中mRNA 表达数据集和对应的临床信息从UCSC Xena 平台(https://xenabrowser.net/datapages/)下载,选择队列为癌症基因组数据共享系统(genomic data commons,GDC)TCGA-HNSC,剔除非口腔癌相关部位的样本,样本信息见表1。从免疫学数据库和分析平台(the immunology database and analysis portal,ImmPort)(https://immport.niaid.nih.gov/)下载免疫相关基因列表,总计1509 个。
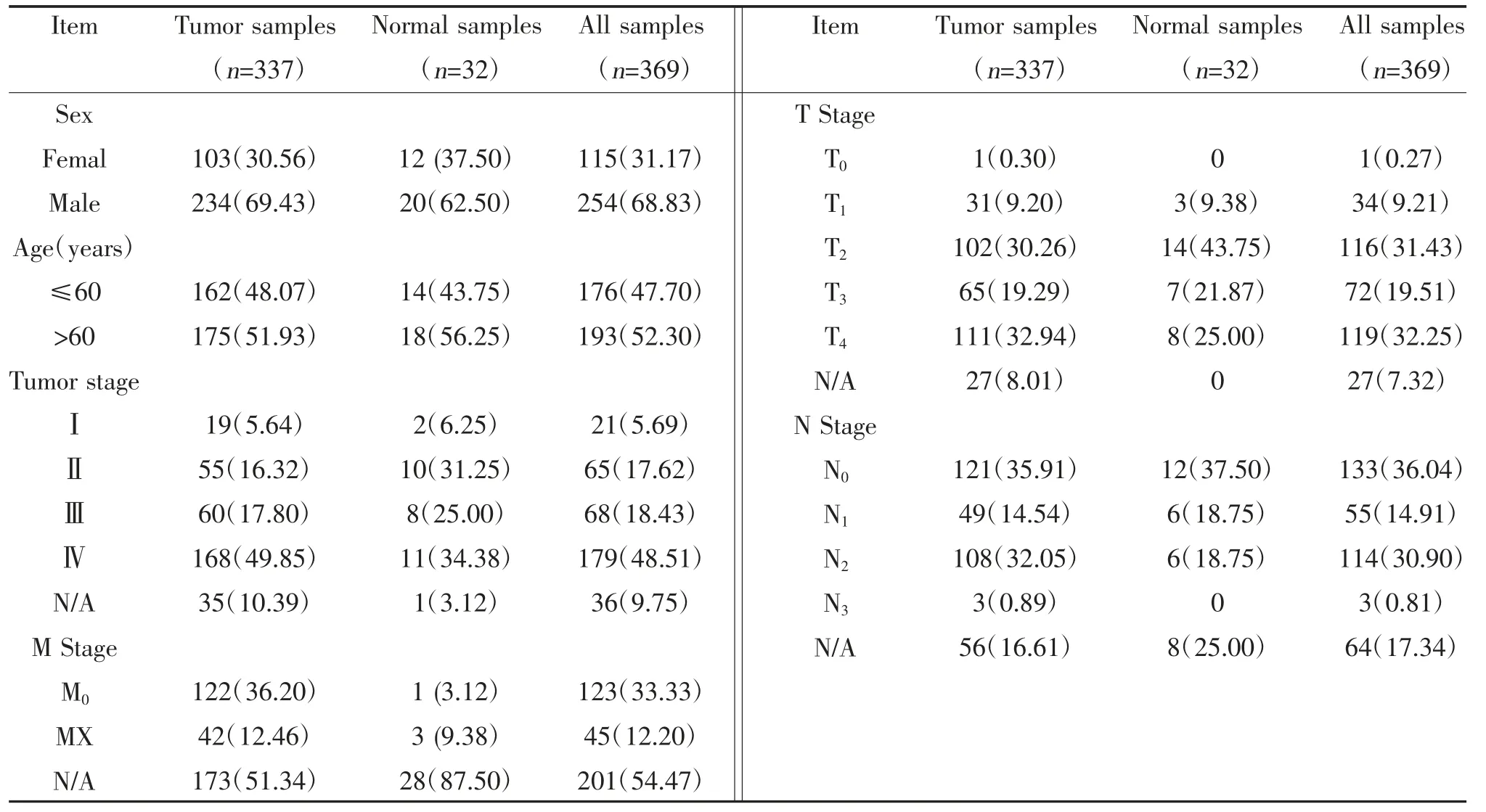
表1 TCGA数据集中样本信息[n(%)]
1.2 构建免疫相关的预后风险模型 首先,利用R包对TCGA数据集进行差异基因分析,过滤标准为adjustedP-value<0.05 和差异倍数大于1.5 倍(|Log2FC|>0.585),然后与免疫基因列表取交集,得到免疫相关的差异基因。接着,使用R 包Survival 进行单变量Cox 比例风险回归模型,选取与生存预后相关的差异基因。通过多因子回归分析确定每个预后因子的回归系数,建立预后风险评估模型,预测患者生存率。公式为:
风险分数=∑差异基因的回归系数χi×归一化处理后的基因表达量βi
1.3 绘制生存曲线和ROC 曲线 为了评估构建的风险预后模型的性能,利用R 包survivalROC 绘制受试者工作特征曲线(receiver operating characteristic curve,ROC),在ROC 曲线的转折点选择最佳风险评分临界值,转折点处真阳性和假阳性之间的差异最大。高于临界值的患者属于高风险评分组,低于临界值的患者属于低风险评分组,并使用R 包Survminer 绘制两组的生存曲线。
1.4 构建和验证列线图 为了提高预后模型的性能,绘制列线图,使用R 包rms,通过整合风险评分模型和临床信息,包括年龄、性别和肿瘤分期,可视化不同患者特征的预后价值。此外,基于单因子回归分析的森林图分析临床信息与总生存期(overall survival,OS)间的关系。其中,一致性指数(concordance index,C-index)可反映列线图的预测准确性。
1.5 肿瘤免疫侵润细胞类型的构成比较 基于构建的预后风险模型,将肿瘤样本分为高风险评分组和低风评分组,并采用CIBERSORT 算法[11],获取22 种肿瘤浸润免疫细胞的比例。随后,通过非配对t检验比较高风险评分组和低风险评分组之间的免疫图谱。
1.6 肿瘤免疫微环境的特征分析 使用R 包maftools可视化高风险评分组和低风险评分组的突变谱[12]。计算TMB 值,并通过非配对t检验统计高风险评分组和低风险评分组间TMB的差异;使用Kaplan-Meier 方法比较高风险评分组和低风险评分组间OS的差异;采用Wilcoxon 检验比较高风险评分组和低风险评分组间免疫检查点及其配体的mRNA 表达水平。
2 结果
2.1 构建口腔癌的免疫相关预后模型 共得到1533个差异表达基因,其中上调差异基因数量为726 个,下调基因数量为807 个,见图1A;与免疫基因列表取交集后,共得到73 个免疫相关基因,其中51 个基因下调,22 个基因上调。单因子回归和多因子回归分析表明,有6 个免疫相关的差异基因与OS 有关,见表2。其中,回归系数见图1B~图1D。根据逐步回归模型,Akaike 信息标准(Akaike information criterion,AIC)为1536.21,C-index 为0.63,见图1E。

图1 鉴定预后相关的差异表达基因

表2 与口腔癌预后相关的基因信息
2.2 预后模型的性能评估 基于已构建的预后风险模型,将口腔癌患者分为高风险评分组和低风险评分组,其中,cutoff 值设为0.02。可以看出,随着风险得分的增加,生存时间呈现缩短的趋势,并且高危组的死亡比例比低危组高,见图2A、图2B;基因CD40LG 和CMA1 在低风险评分组表达量高,在高风险评分组表达量低,而其余3 个基因趋势相反,见图2C;高风险评分患者的OS 比低评分患者预后较差,见图2D。
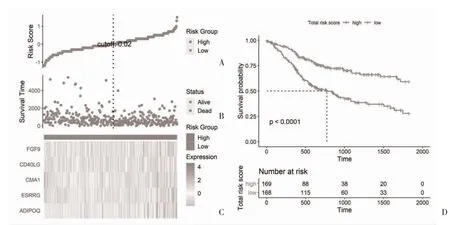
图2 预后风险评分分组和评估
2.3 预后模型的统计分析 绘制ROC 曲线和肿瘤分层分析进一步评估风险模型的性能,结果显示,ROC曲线下面积(area under the curve,AUC)在3年时为0.678,4年时为0.671,5年时为0.683,见图3A。Wilcoxon 检验表明,预后风险评分与N 分期(区域淋巴结转移)密切相关(P=0.034),但与病理分期和T 分期无相关性(P>0.05),见图3B、图3C、图3D。
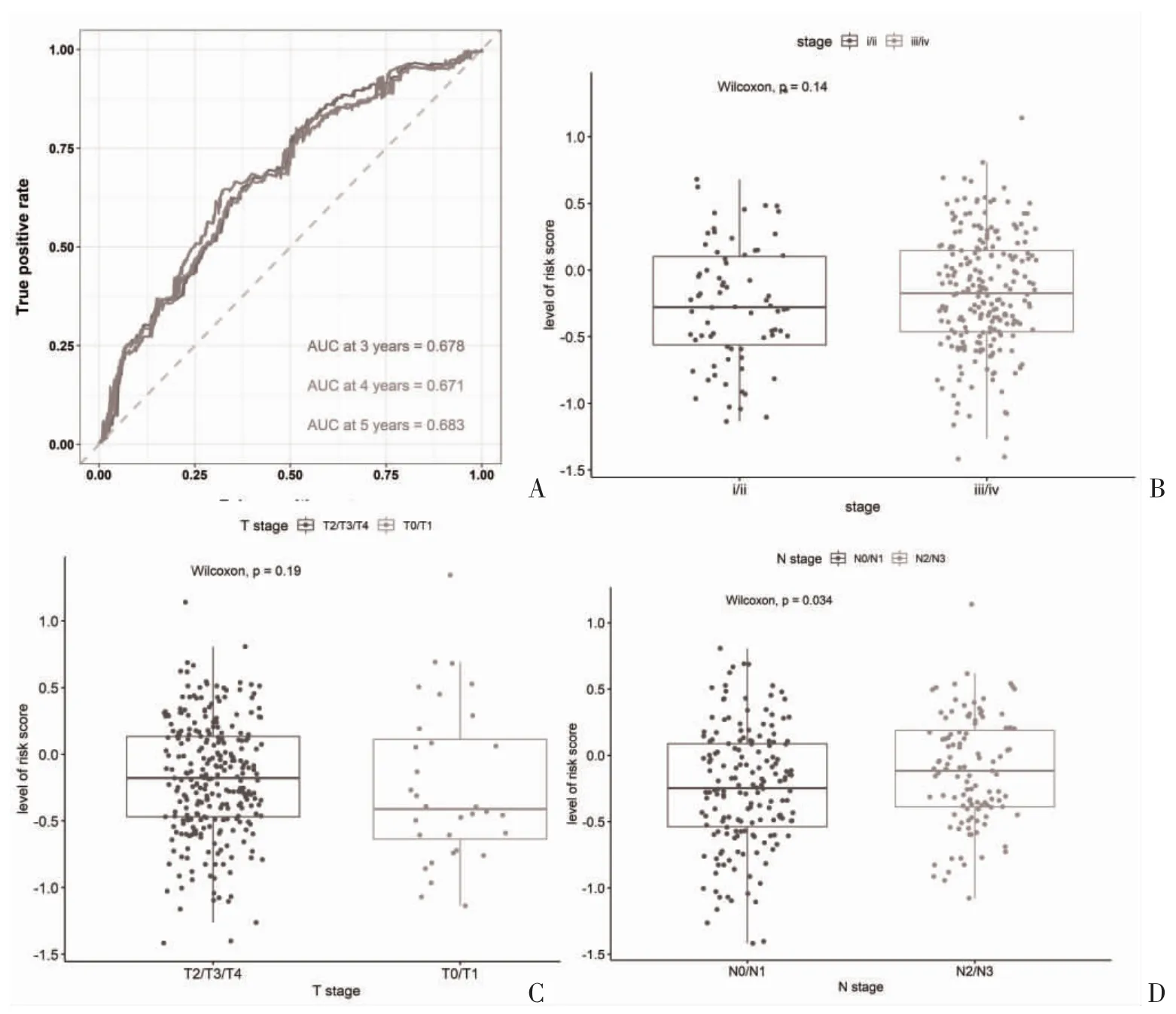
图3 预后风险评估模型性能评价
2.4 构建和评估列线图模型 在列线图中,每个变量的得分可以在分数表上找到,然后通过计算总分来估计3、4 和5年的生存概率,见图4A;森林图显示,患者年龄(>60 岁)、肿瘤Ⅳ期和风险评分与OS 相关(P<0.05),见图4B。另外,绘制校准曲线来验证列线图的性能,结果显示,构建的列线图模型性能良好,预测曲线接近理想曲线,见图4C~图4E。此外,该列线图(C-index:0.67)的预测准确性高于风险评分模型(C-index:0.63)。
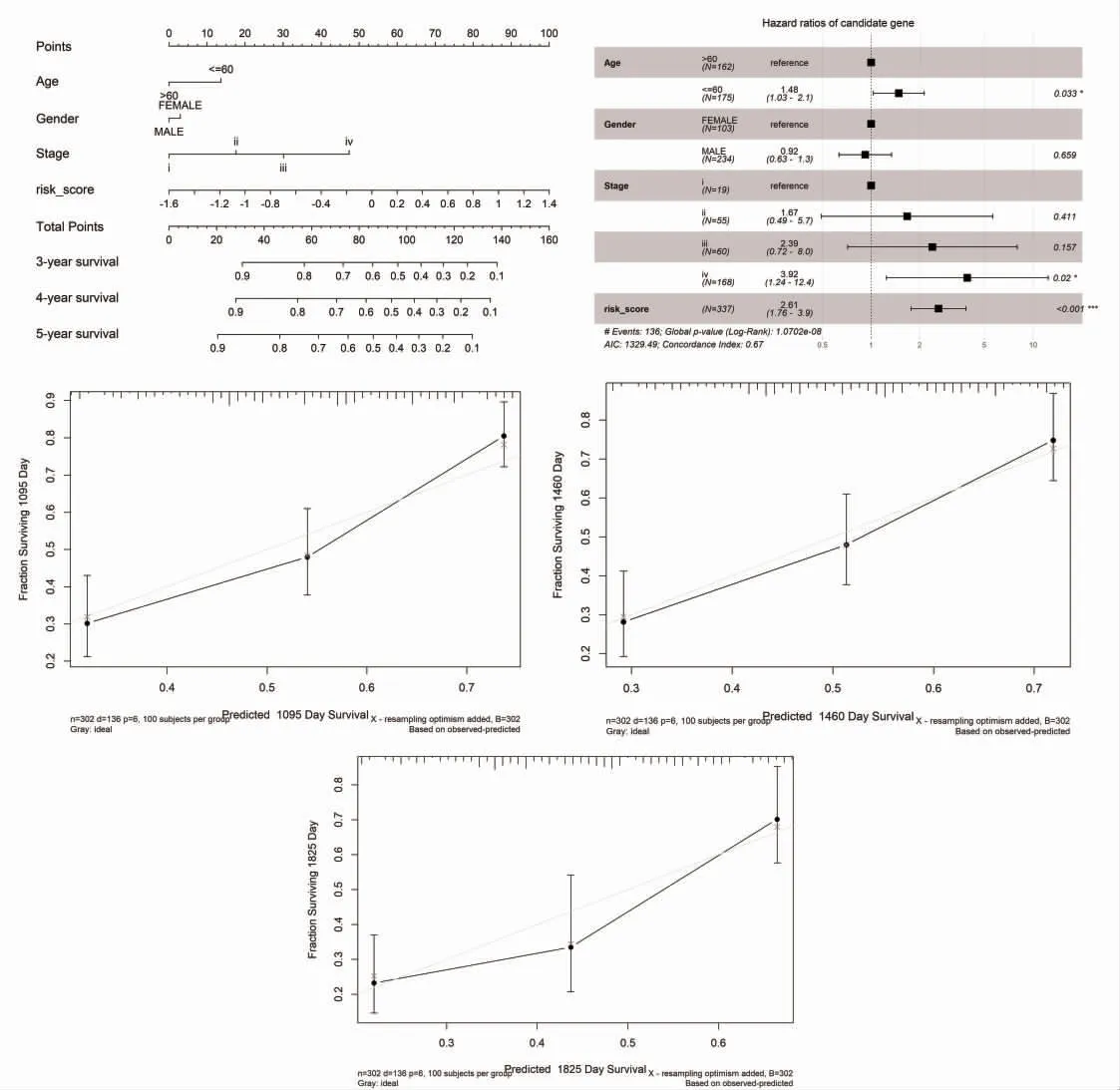
图4 列线图模型的构建与验证
2.5 体细胞突变统计及其与口腔癌生存预后的关系基于建立的免疫预后模型,将肿瘤样本分为高风险评分组和低风险评分组,分别统计体细胞突变类型、突变模式和基因突变率。结果显示,突变以错义突变为主,主要突变类型为单核苷酸变异(single nucleotide variants,SNV),突变模式主要为C>T。在高风险评分组中,突变率排名前10的基因为TP53、TTN、MUC16、FAT1、CDKN2A、KMT2D、LRP1B、NOTCH1、DNAH5 和SYNE1,在低风险评分组中,突变率排名前10的基因为TP53、TTN、FAT1、NOTCH1、CDKN2A、PIK3CA、CASP8、SYNE1、CSMD3 和LRP1B,见图5A,图5B;其中基因MUC16 和NOTCH1 在高风险评分组和低风险评分组中的突变率比较,差异有统计学意义(P<0.05),见图5C;对基因MUC16 和NOTCH1 分别进行生存曲线分析,两个基因均不能作为独立的预后因子(P>0.05),见图5D,图5E;高风险评分组和低风险评分组的TMB 比较,差异无统计学意义(P>0.05),见图5F;但生存曲线分析显示,TMB 值可作为独立的预后因子,见图5G。

图5 体细胞突变分析统计及其与OS的关系
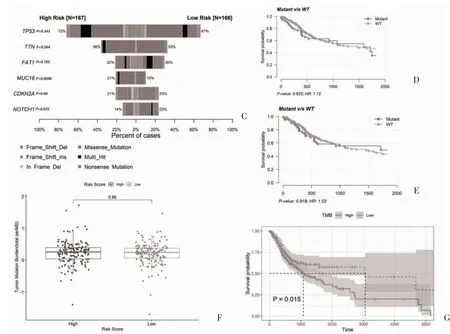
图5 体细胞突变分析统计及其与OS的关系(续)
2.6 口腔癌肿瘤免疫微环境 基于CIBERSORT 算法,估算每个口腔癌患者中22 种免疫细胞的比例,并比较高风险评分组和低风险评分组之间的免疫细胞比例,结果显示,有6 种免疫细胞类型存在差异(P<0.05),见图6A;另外,虽然免疫细胞NK.cells.activated 在高风险评分组和低风险评分组的免疫侵润程度不存在差异,但其与生存预后相关(P<0.05),见图6B。Wilcoxon 检验结果表明,高风险评分组PD-L1(P=1.8×10-7)、PD-1(P=0.0079)和CTLA-4(P=9.7×10-7)的表达水平较低,见图6C~图6E。

图6 口腔癌肿瘤免疫微环境
3 讨论
免疫细胞在肿瘤的演变中起着重要作用[4],目前ICIs 已应用于中晚期实体瘤的治疗[13]。对于口腔癌,纳武利尤单抗(nivolumab)和派姆单抗(pembrolizumab)均被批准用于治疗复发/转移性疾病[14]。然而,由于对肿瘤微环境特性的理解不足,这在一定程度上阻碍了免疫治疗的广泛应用。近年来,大量研究鉴定出了与口腔癌诊断和预后相关的免疫相关生物标志物[8,9,15,16]。但为了最大限度地发挥免疫治疗的作用,还需要探索更多可靠的生物标志物。
本研究基于5 个免疫相关差异基因构建了口腔癌预后风险评估模型,这5 个基因为FGF9、CD40LG、CMA1、ESRRG、ADIPOQ。已有研究表明[7,17,18],CD40LG、CMA1 和ESRRG 与口腔癌的预后显著相关。FGF9 是一种生长因子,属于FGFs 家族,FGF9是许多癌症(包括肺腺癌、前列腺癌和肝癌)肿瘤演化的关键因子[19]。CD40LG 是一种跨膜蛋白,属于肿瘤坏死因子超家族,存在于CD4+T 细胞、B 细胞、单核细胞和自然杀伤细胞及癌细胞中,具有增强肿瘤细胞的凋亡和抗原呈递细胞的功能[17]。CMA1 是一种乳糜蛋白酶,由肥大细胞分泌,在癌症中还没有明确的研究。有研究表明[20],通过增强ESRRG的表达和功能,可在癌细胞中发挥抑癌活性。ADIPOQ 表达的脂联素可显著抑制乳腺癌生长并诱导细胞凋亡[21],但其在口腔癌中的分子机理还未见报道。
本研究结果显示,免疫细胞NK.cells.activated的免疫侵润程度和TMB 是口腔癌潜在的独立预后标志。口腔癌高风险评分患者的免疫检查点及其配体(PD-L1、PD-1 和CTLA-4)表达水平较低,这可能表明构建的免疫相关风险评分模型能够为免疫治疗提供一定的临床指导意义。
综上所述,本研究成功构建了免疫相关的口腔癌风险评估预后模型,并探索了肿瘤免疫微环境的特征,这可能有助于口腔癌患者的预后和免疫治疗。
