大杯蕈遗传多样性的ISSR分析
2022-03-19吴碧君刘小英
吴碧君, 刘小英
(莆田市农业科学研究所,福建 莆田 351144)
大杯蕈(Panusgiganteus)又名巨大革耳、猪肚菇、大杯伞、漏斗菇、大杯香菇等,是一种中高温型食用菌品种[1]。大杯蕈菌肉肥厚、口感脆爽鲜美,子实体中富含丰富的蛋白质、氨基酸、微量元素等[2-3],在市面上深受消费者喜爱。目前,市面上大杯蕈菌种管理较为混乱,同种异名、异种同名等现象较为普遍,给大杯蕈良种收集选育、资源管理与菌株鉴定带来巨大困难。分子生物技术从DNA水平上分析菌株间的遗传多样性,不受食用菌栽培环境和传统栽培周期的影响,可以快速高效地分析菌株间的亲缘关系。
其中,简单重复序列区间(inter-simple sequence repeat,ISSR)技术具有多态性丰富、稳定性强、重复性好、操作简单等优点[4],已被应用于食用菌菌株鉴定、遗传多样性分析、辅助育种等方面[5-9]。高傲等[10]采用ISSR和SRAP分子标记技术对22个鸡腿菇菌株的遗传多样性进行综合分析,供试鸡腿菇菌株在遗传相似系数为0.8时可分为4大类,聚类分析与主坐标分析基本一致。杨和川等[11]基于分子标记技术研究了10株杏鲍菇的遗传多样性,结果表明,供试菌株在遗传相似系数为0.68时可分为3类,且RAPD分析结果与主成分分析一致。张妍等[12]采用ISSR分子标记技术结合菌丝生长速度等农艺性状研究20个金顶侧耳菌株种内遗传多样性,研究显示,在遗传相似系数为0.65时,绝大部分菌株呈现较强的地域性,按照南北方来源地聚为2类;表型性状与基因型结果基本吻合,亲缘关系远的菌株农艺性状差异大。表型农艺性状是传统菌种鉴别方法之一,但极易受到环境的影响,只能鉴别到属内种间水平[13],结合ISSR等分子标记技术,可以提供更准确的遗传多样性结果。目前,有关大杯蕈的研究主要集中在栽培技术、蛋白质营养、多糖等活性物质的研究[14-16],针对大杯蕈遗传多样性的研究鲜少报道。基于此,本研究采用ISSR分子标记技术,通过计算遗传相似系数、聚类图,结合大杯蕈子实体形态,分析10个大杯蕈菌株的遗传多样性,以期为大杯蕈菌株鉴定和良种选育等提供参考依据。
1 材料与方法
1.1 材料
1.1.1 供试菌株
10个供试大杯蕈菌株编号、来源如表1所示。
1.1.2 试剂及引物
本试验所用DNA marker、TaqPCR Mix、4s Gel Red染料、Ezup柱式真菌基因组DNA抽提试剂盒、琼脂糖B、5×TBE、TE缓冲液及ISSR引物均购自上海生工生物工程股份有限公司。
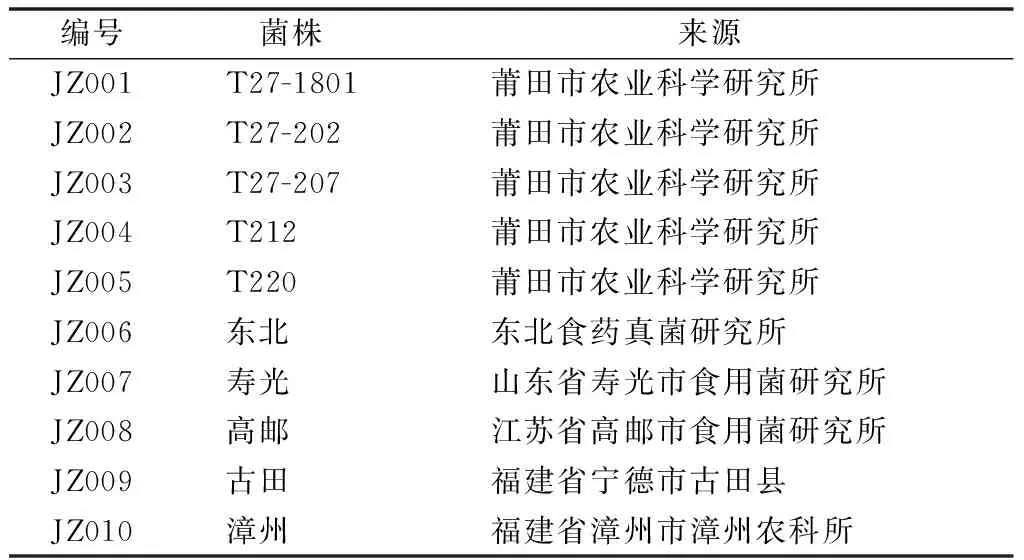
表1 供试的10个大杯蕈菌株
1.1.3 培养基
PDA培养基:马铃薯200 g、葡萄糖20 g、琼脂20 g,定容至1 000 mL,pH值自然。
原种及栽培袋培养基:63%木屑+20%棉籽壳+16%麸皮+1%石灰,原种培养基含水量为50%~55%,栽培种为60%,pH自然。
1.2 方法
1.2.1 菌丝生物学特征测定方法
在18 mm×180 mm的试管内接种供试母种,以活化菌丝体。每个菌株设3个重复,将接种完的供试菌株试管置于25 ℃恒温培养箱中培养。观察菌丝生长情况,记录菌丝颜色。
1.2.2 菌丝体培养及DNA提取
将灭菌玻璃纸铺入PDA平板,并从活化后的大杯蕈菌株斜面上挑取菌丝块接种于PDA平板上,于25 ℃恒温培养7~15 d。培养结束后,刮取平板上活化后的菌丝作为试验材料,采用真菌基因组DNA抽提试剂盒提取DNA(上海生工生物工程股份有限公司)。提取物经1.0%琼脂糖凝胶电泳检测质量后,于-20 ℃保存备用。
1.2.3 PCR反应体系和条件
ISSR-PCR反应的总体系为50 μL,反应体系为:25 μLTaqPCR Mix,4 μL引物(10 μmol·L-1),2 μL模板DNA,19 μL ddH2O。
PCR扩增反应条件:94 ℃预变性5 min;94 ℃变性1 min,45~55 ℃(退火温度依不同引物而定)退火45 s,72 ℃延伸1.5 min,40个循环;72 ℃ 延伸10 min;4 ℃保存。
1.2.4 电泳和数据分析
PCR扩增反应结束后,取扩增产物进行琼脂糖凝胶电泳分离检测,在凝胶成像系统上拍照。电泳图谱中在同一水平位置上扩增出的强带或可分辨性好的弱带,赋值“1”,未扩增出条带,赋值“0”,形成0/1数据矩阵。用NTSYSpc 2.10软件进行聚类分析。
2 结果与分析
2.1 大杯蕈菌丝及子实体形态特征
对10个大杯蕈菌株进行出菇试验,并观察菌丝及子实体形态特征(表2)。10个大杯蕈菌株菌丝颜色均为白色。菌盖颜色分为浅褐色、褐色;菌柄颜色分为浅褐色、褐色、米白色3种。按菌株的菌盖和菌柄颜色划分,除漳州菌株的菌盖和菌柄颜色不一致(菌盖为褐色,菌柄为米白色),其余9个大杯蕈菌株的菌盖和菌柄皆为浅褐色或褐色。根据菌柄类型可分为圆锥形和圆柱形,其中仅T27-1801菌株为圆锥形菌柄。
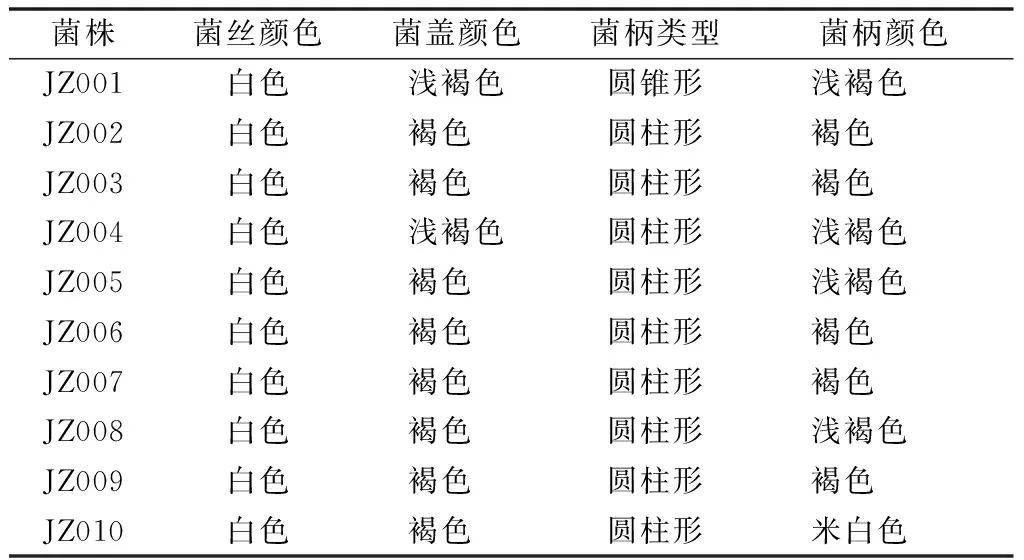
表2 大杯蕈菌丝及子实体形态特征
2.2 大杯蕈菌株ISSR-PCR分析扩增结果
从30条引物中筛选出8条(表3)可以扩增出多态性好、清晰度高条带的引物,8条引物分别为UBC 809、UBC 816、UBC 817、UBC 818、UBC 856、UBC 857、UBC 878、UBC 888。表3所示,不同引物对供试菌株的扩增条带多态性不一样。引物UBC 857扩增出26条条带,多态率为96.15%。引物UBC 818扩增的多态率最高,为100%。8条引物对10个菌株共扩增出的DNA片段大小为200~2 000 bp,共扩增出111条条带,其中多样性条带91条,占总带数的81.98%,说明10个大杯蕈菌株具有丰富的遗传多样性。每条引物对供试菌株的扩增条带为9~26条,平均每个引物扩增出11条条带。引物UBC 817和UBC 857对10个大杯蕈菌株的扩增图谱见图1。
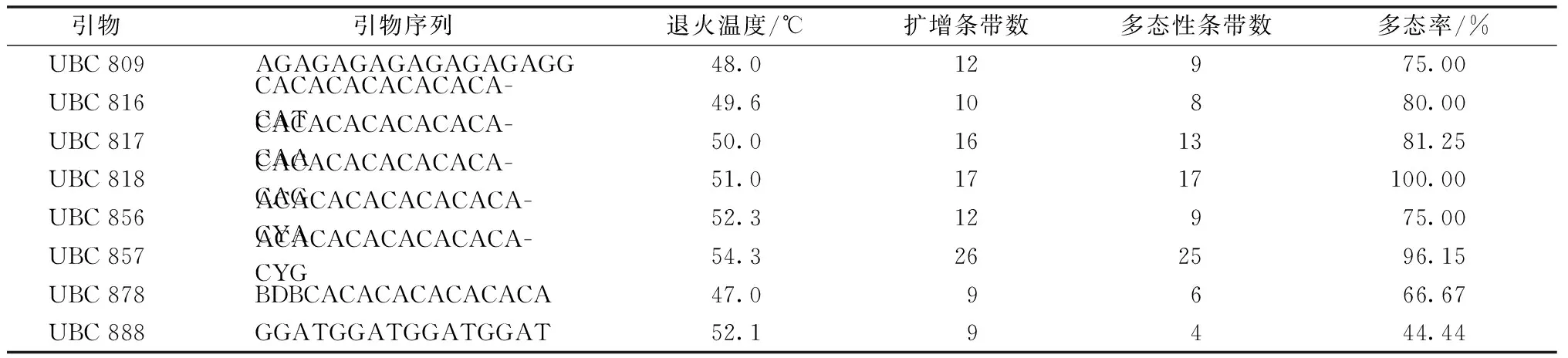
表3 不同ISSR引物对供试大杯蕈菌株的扩增结果
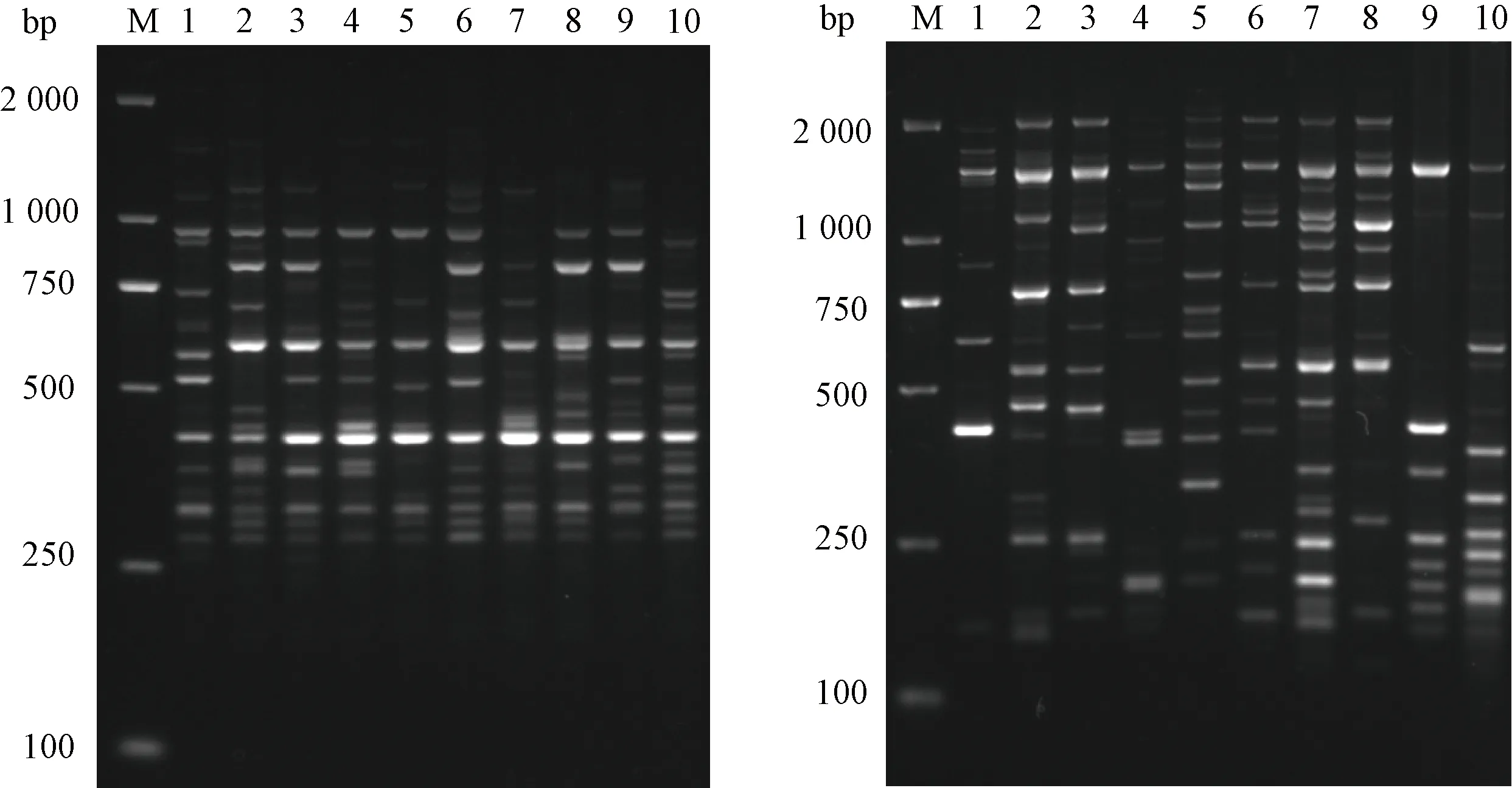
左图为引物UBC 817,右图为引物UBC 857;M为D2000 Marker,1~10为菌株编号。图1 部分引物扩增结果
2.3 供试菌株遗传相似性分析
基于ISSR-PCR的扩增结果,采用NTSYSpc 2.10软件计算供试菌株间遗传相似系数(表4)。结果显示,10个大杯蕈菌株遗传相似系数在0.477~0.856,平均遗传相似系数为0.730,表明这10个菌株遗传关系较近。菌株JZ007与JZ001相似系数最小,说明T27-1801和寿光这2个菌株的亲缘关系较远;菌株JZ003与JZ006相似度最大为0.856,说明T27-207与东北这2个菌株的亲缘关系较近。
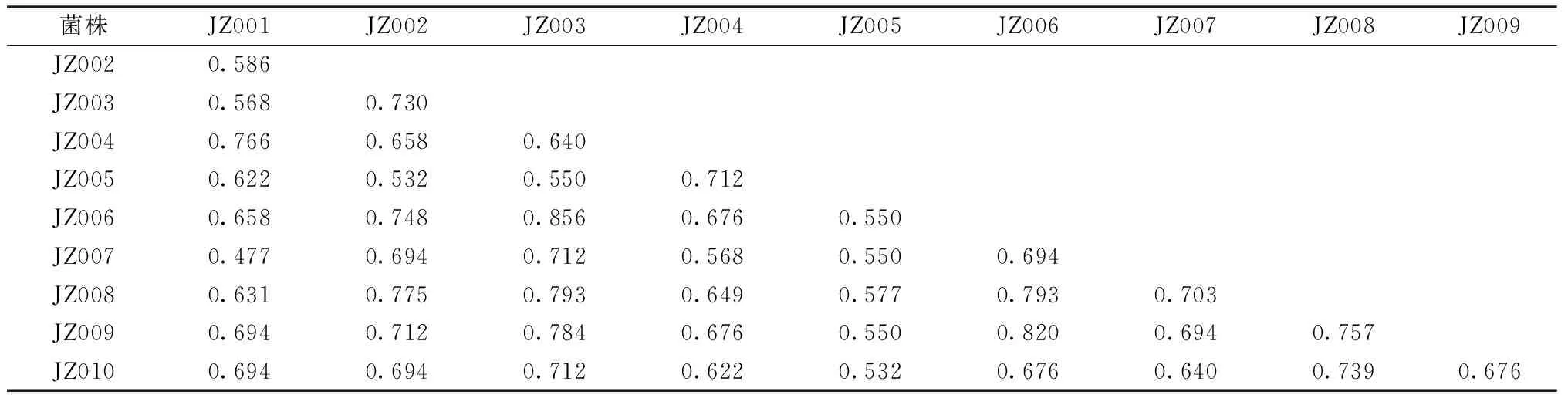
表4 10个大杯蕈菌株遗传相似系数
2.4 10个大杯蕈菌株的聚类分析
图2表明,10个大杯蕈菌株在遗传多样性上存在一定的差异。在遗传相似系数为0.60时,10个大杯蕈菌株聚为2类;在遗传相似系数为0.68水平上分为4类。其中,T27-1801、T212菌株聚为一类;T220单独为一类;T27-207、T27-202、东北、寿光、古田、高邮为第3类;漳州菌株单独形成第4类。T212为大杯蕈品种莆蕈1号自交后代,T27-1801为莆蕈1号与T212菌株的杂交后代,二者亲缘关系较近聚在一起。结合大杯蕈子实体形态,漳州菌株的菌柄和菌盖颜色不一致,菌柄米白色区别于其他9个菌株,这与聚类结果一致,单独聚为一类。T27-207、东北菌株在遗传相似系数为0.86处聚为同一类群,这有可能是由于市面上大杯蕈在引种过程中出现的同种异名所致。
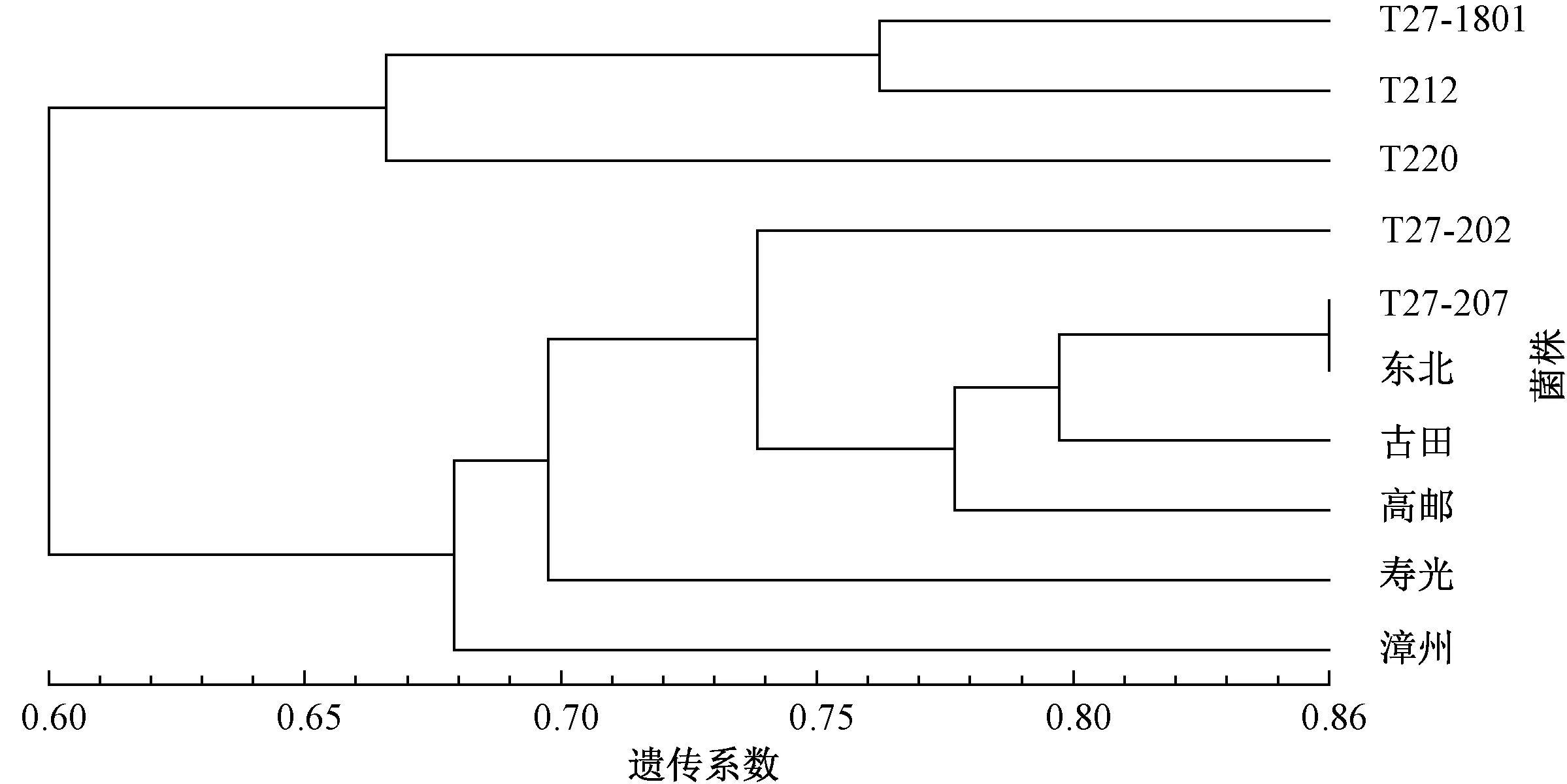
图2 10个供试菌株的UPGMA聚类分析结果
3 小结与讨论
本研究基于ISSR分子标记技术,筛选了8条多态性好、清晰度高的引物对10个大杯蕈菌株进行遗传多样性分析。8条引物共扩增出111条DNA片段,多样性条带91条,多态率为81.98%,说明供试菌株具有丰富的遗传多样性。供试菌株的平均遗传相似系数为0.730,遗传关系较近。ISSR分子标记可有效分析大杯蕈菌株遗传多样性。
菌株、品种间遗传多样性分析在食用菌良种选育和种质资源保藏中具有重要意义。研究表明,食用菌子实体颜色等农艺性状可能对遗传多样性有一定影响[17]。观察食用菌子实体颜色、形态等农艺性状结合ISSR等分子标记方法从DNA分子水平上检测菌株基因组间的遗传多样性已在多种食用菌上有所报道。目前,大杯蕈还没有系统的遗传多样性分析报道。江玉姬等[18]研究9个巨大革耳菌株的亲缘关系,经ISSR聚类分析表明9个菌株分为2类,C.m0002菌株单独聚为一类。结合子实体形态特征发现,C.m0002菌株的子实体为多脂鳞伞,为同名异种。宫志远[19]搜集35个山东主栽平菇菌株,在遗传相似系数为0.665时供试菌株的遗传关系可按照子实体颜色分为黑色、灰黑色、白色3个类群。但是,谭艳[20]利用ISSR分子标记方法分析29个金针菇菌株种间的遗传差异,ISSR分子标记技术未能按照供试菌株的子实体颜色区分开。杨和川[11]基于RAPD技术研究10个杏鲍菇菌株的遗传多样性,在遗传相似系数为0.68时将供试菌株分为3类,也未能利用RAPD技术将供试菌株按照子实体菌盖颜色进行分类。谭艳等[20]研究结果与本研究一致,未能利用分子标记技术按照子实体的颜色区分供试菌株。真菌的生长速度、子实体形态等农艺性状受多基因控制,遗传基础复杂,易受到温度、湿度等栽培环境的影响[21],采用多种分子标记综合分析的结果能够更准确地体现供试菌株间的亲缘关系。汪昊等[22]认为,综合不同的分子标记方法进行聚类分析,由于原理不同聚类结果略有差异,但可以更加准确地反映菌株间的亲缘关系,研究中综合采用了ISSR、SRAP、TRAP 3种分子标记结果,在遗传相似系数为0.75时将19个香菇菌株分为6类。武海月等[13]通过ISSR和SRAP技术综合分析19个榆黄菇品种的遗传多样性,结果表明,在遗传相似系数为0.70时,可将榆黄菇分为4类。高傲等[10]基于ISSR和SRAP的聚类分析结果将22个供试菌株分为4类,结果与主坐标分析吻合。
因此,在今后的研究工作中应从地域、亲缘关系等方面考虑选取材料,扩大研究群体,增加引物数量,结合农艺性状观察和拮抗试验;另一方面,丰富分子标记方法,融合不同分子标记技术优势。双管齐下,优化聚类结果,提高遗传分析结果的准确性。
