基于SSR标记的澳洲坚果种质资源遗传多样性分析
2022-03-07井敏敏黄炳钰戴小红李栋梁陈晶晶
井敏敏 黄炳钰 戴小红 李栋梁 陈晶晶



摘 要:澳洲堅果原产于澳大利亚亚热带雨林,是我国新兴的重要经济作物,其种质资源丰富,但遗传背景复杂,亲缘关系混乱。开展澳洲坚果种质资源遗传多样性分析,可拓展澳洲坚果种质资源创新利用途径,有效加快澳洲坚果育种进程。形态表型分析是最早用于种质鉴定和管理的标记之一,但易受环境因素影响。DNA分子标记技术在种质资源鉴定、遗传多样性分析中克服了传统表型分析易受环境影响的缺点。本研究采用9对高多态性SSR引物对91份国外引进以及自主选育品种(品系)澳洲坚果种质进行遗传多样性分析。结果表明,共扩增出73个等位基因位点,平均等位基因8个,平均基因多样性为0.689,平均杂合度为0.578,多态性信息含量PIC为0.453~0.792,平均值为0.659,表明91份澳洲坚果具有较高的多态性。UPGMA聚类分析表明,在遗传距离为0.33处,将91份澳洲坚果分为2大类,且其亲缘关系与地理来源无显著相关性,与主成分分析、Structure分析结果一致。本研究供试澳洲坚果材料群体内遗传变异显著高于群体间变异,且遗传成分来源单一,遗传结构较为简单。本研究为澳洲坚果种质的有效利用以及核心种质构建提供理论基础,为进一步加速澳洲坚果种质遗传改良以及分子辅助育种奠定基础。
关键词:澳洲坚果;种质资源;SSR标记;遗传多样性
中图分类号:S664.9 文献标识码:A
Genetic Diversity Analysis of Germplasm Resources by SSR Markers
JING Minmin HUANG Bingyu DAI Xiaohong LI Dongliang CHEN Jingjing
South Subtropical Crops Research Institute, Chinese Academy of Tropical Agricultural Sciences / National Field Genebank for Tropical Fruits / Key Laboratory of Tropical Fruit Tree Biology, Ministry of Agriculture and Rural Affairs, Zhanjiang, Guangdong 524091, China
, derived from the Australian subtropical rainforest, is a recently domesticated nut crop in China. is rich in germplasm resources. However, the genetic background of is complex and the genetic relationship is unclear. The genetic diversity analysis of germplasm resources can expand the innovative utilization of and effectively improve the breeding process. Morphological traits are among the earliest markers used in germplasm characterization and management. However, phenotypic analysis is easily affected by environmental factors, resulting in deviation in the results. DNA molecular marker technology overcomes the shortcomings of traditional phenotypic analysis vulnerable to environmental impact in germplasm resource identification and genetic diversity analysis. The genetic diversity of 91 germplasm introduced from abroad and self-breeding varieties (lines) was analyzed by 9 simple sequence repeat (SSR) loci in this study. The results showed that a total of 73 alleles were amplified by 9 pairs of SSR primers, with average of 8 per marker. The average genetic diversity was 0.689. The average heterozygosity was 0.578. The polymorphic information content (PIC) of each locus varied from 0.453 to 0.792 with an average of 0.659, indicating that germplasm had high genetic diversity. UPGMA cluster analysis showed that the tested resources could be divided into two groups at the genetic distance of 0.33. The first category includes all tetraphylla species, Nelmak1-1, Nelmak1-2 and HAMC14, with a total of 19 germplasms. The second category contains 72 germplasms, mainly the cultivated varieties in Australia and Hawaii and self-breeding varieties (lines) in China. There was no significant correlation between genetic relationship and geographical origin. The results were consistent with population genetic structure analysis and principal component analysis. For population genetic structure analysis, Bayesian clustering modelling was executed in the Structure software using data generated by SSR marker-based profiling and diversity analysis of 91 accessions. The highest log-likelihood was performed which provided the maximum peak at =2, indicating that all genotypes represent two distinct clusters. In addition, the results showed that the genetic components of germplasm were single and the genetic structure was relatively simple. AMOVA analysis showed that the genetic variation within populations was 90% and that among populations was 10%, indicating that the genetic variation of used in this research within population was significantly higher than that among population. This study provides a theoretical basis for the effective utilization of germplasm and the construction of core germplasm. In addition, we therefore aimed to use the SSR markers to accelerate genetic improvement in breeding program. These results lay a foundation for molecular marker-assisted breeding in .
; germplasm resources; SSR marker; genetic diversity
10.3969/j.issn.1000-2561.2022.02.005
澳洲坚果( F. Muell),别名夏威夷果、昆士兰栗、澳洲核桃,属山龙眼科澳洲坚果属,常绿乔木,原产于澳大利亚昆士兰州东南部和新南威尔州北部、南纬25°~31°的沿海亚热带雨林。澳洲坚果果仁含油率达60%~80%,含蛋白质、糖类约9%,并包含人体所必需的氨基酸以及丰富的钙、磷、铁和维生素,被誉为“坚果之王”,成为热带地区新兴的经济作物。澳洲坚果属包含4个种,其中 Maiden & Betche、 L.A.S. Johnson及其杂交种广泛用于商业种植。我国澳洲坚果种植起步较晚,近年来,我国云南、广西、贵州等省(区)进行了大规模引种种植,2019年种植面积达24.5万hm,全年总产量(未去壳果)0.494万t,全年总产值152 278.6万元(农业农村部农垦局统计)。澳洲坚果市场容量大,在我国具有很好的发展前景。
澳洲坚果种质资源丰富,但由于交叉引种、杂交、自然变异等因素,遗传背景复杂,亲缘关系混乱。因此,开展澳洲坚果种质资源的遗传多样性分析,可有效提升澳洲坚果育种进程。种质遗传多样性主要通过表型性状和分子标记等方式进行评价。20世纪90年代起,国外研究学者借助表型性状和同工酶开展了澳洲坚果遗传多样性分析。国内李文华对15个澳洲坚果品种幼龄期植物学形态进行观察,陈业渊编制了澳洲坚果种质资源描述规范,王维对21份澳洲坚果种质资源的主要生物學特性进行了观测研究,曾辉等主编出版了《澳洲坚果品种图谱》,涵盖‘H2’‘O.C’‘344’‘695’等主要栽培品种。
但是,表型性状受外界环境影响较大,且要求种植者具有较高的技术经验,其分析结果缺乏稳定性。近年来,分子标记技术发展迅速,AFLP、RAPD、RAF、ISSR、SSR、SNP等分子标记逐渐应用于澳洲坚果种质资源鉴定。
SSR又称微卫星序列,1~6个碱基单元重复序列,为共显性标记,具有多态性高、分布广泛、遗传稳定、操作简便以及引物通用等优点,广泛应用于作物遗传多样性分析、种质资源鉴定以及育种工作中。然而国内对澳洲坚果SSR分子标记的报道很少,仅报道了澳洲坚果SSR-PCR反应体系优化,以及杂交F1代SSR鉴定,未对种质资源进行系统性分析以及鉴定。本研究对国外引进以及自主选育品种(品系)的91份澳洲坚果材料,通过SSR分子标记技术进行遗传多样性分析,以期为澳洲坚果种质的有效利用以及核心种质构建提供理论基础,进一步为加速澳洲坚果种质遗传改良以及分子辅助育种奠定基础。
材料与方法
材料
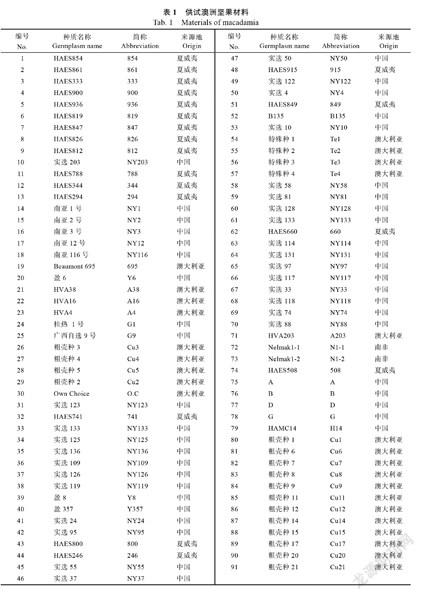
参试澳洲坚果材料共91份(表1),新鲜幼嫩叶片材料均采集于中国热带农业科学院南亚热带作物研究所,采用75%酒精擦拭干净,保存于–80℃备用。
提取与扩增
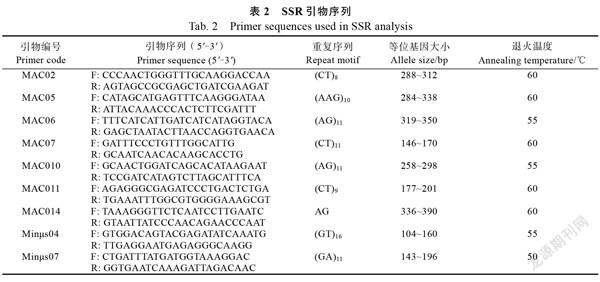
样品DNA采用QIAGEN DNeasy Plant Kit(QIAGEN, Valencia, California, USA)提取,以0.8%琼脂糖凝胶检测DNA质量,并采用Nanodrop 1000分光光度计(Thermo Fisher Scientific, Wilmington, DE, USA)测定其浓度与纯度,稀释至20 ng/μL置于–20℃保存备用。本研究所用SSR引物参考NOCK等以及SCHMIDT等的文献,筛选出9对稳定性好、多态性高的引物(表2),均由广州艾基生物技术有限公司合成。PCR扩增体系为20 μL,含20 ng DNA模板、0.5 U DNA聚合酶、10 μL PCR buffer、0.1 mmol/L dNTPs、2 mmol/L MgCl、0.2 μmol/L引物。扩增程序为:94℃预变性4 min,94℃变性30 s,退火30 s,72℃延伸40 s,28个循环;72℃延伸5 min;4℃保存。
PCR产物稀释20倍,按照HT DNA Extended Range LABCHIP(PerkinElmer, USA)以及HT DNA 1k Reagent kit, Dual protocol(PerkinElmer, USA)使用说明,使用LabChip GX touch HT 24生物大分子仪(PerkinElmer, USA)对PCR产物进行毛细管电泳检测,并在电脑上读取条带大小数据。
数据处理
采用PowerMarker v3.25软件计算每对SSR引物等位基因数(allele number)、最大等位基因频率(major allele frequency)、基因多样性(gene diversity)、杂合度(heterozygosity)、多态性信息量(polymorphism information content, PIC),并计算样本间Nei’s遗传距离,采用非加权分组算术平均法(UPGMA)进行聚类分析,使用MEGA5.2软件进行聚类树绘制。
使用GenALEx 6.4.1计算群体间分化系数(between-population differentiation, )、群体内近交系数(inbreeding coefficients of individuals relative to the sub-population, )和群体间近交系数(inbreeding coefficients of individuals relative to the total population, ),以及主成分分析、AMOVA分析。
采用Structure v2.3.4软件对91份澳洲坚果种质进行遗传结构分析,采用混合祖先模型(admixture model)和等位变异发生频率相关模型(allele frequency correlated model),设置Length of Burn-in Period和Number of MCMC rers after buin in参数值分别为10 000和100 000,值设置为1~10,重复5次,将Structure运行结果打包上传至Structure Harvester获得最佳值,并采用CLUMPP软件进行重复抽样分析,最后采用Distruct1.1绘制遗传结构图。
结果与分析
引物多态性分析
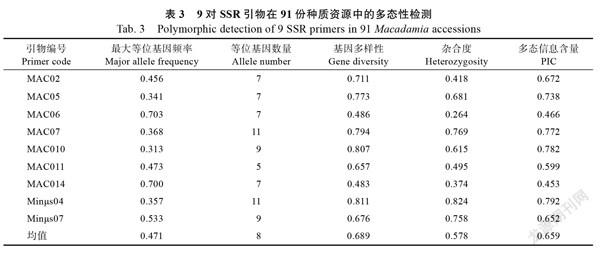
通过9对SSR引物对91份澳洲坚果种质进行PCR扩增,共检测到73个等位基因位点(表3),各引物等位基因数量为5~11个,平均等位基因8个,最大基因频率在0.313~0.703之间,平均基因多样性为0.689,平均杂合度为0.578,多态信息
含量为0.453~0.792,平均值为0.659。其中Minμs04引物等位基因为11,基因多样性为0.811,杂合度为0.824,多态信息含量PIC为0.792,各参数值相对较大,说明Minμs04具有较高的多态性,且杂合度较高,而多态性最小的MAC014基因多样性为0.483,杂合度为0.374,PIC为0.453。表明9对引物具有较高的多态性,可用于进一步研究澳洲堅果遗传多样性。
澳洲坚果种质聚类分析
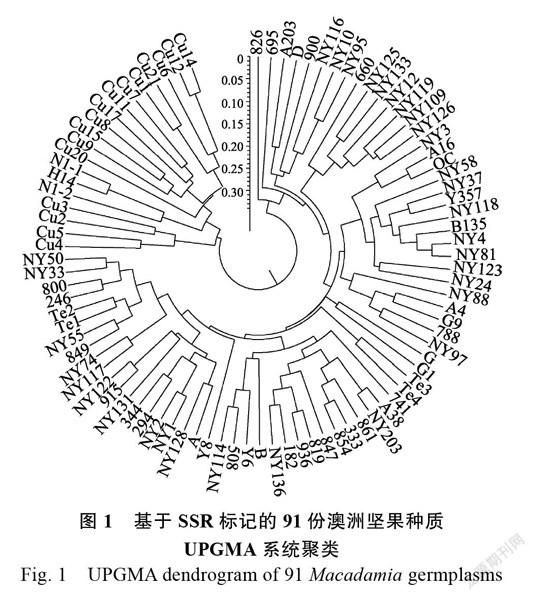
将91份澳洲坚果种质由9对引物扩增的多态性条带用PowerMarker 3.25软件以UPGMA法进行聚类分析(图1)。在遗传距离为0.33处,可将91份澳洲坚果种质分为2大类,第一大类包含所有粗壳种以及Nelmak1-1、Nelmak1-2、HAMC14,共19份种质。其中粗壳种12与粗壳种14亲缘关系很近,Nelmak1为南非选育的杂交品种,由于在实验过程中发现种植园内有2种不同基因型的种质均命名为Nelmak1,所以本研究以Nelmak1- 1、Nelmak1-2标记来区分,且HAMC14、Nelmak1-1、Nelmak1-2聚为一类,说明三者亲缘关系较近。第二大类包含72份种质,主要为澳大利亚、夏威夷主要栽培品种以及国内选育品种(品系),其中由中国热带农业科学院南亚热带作物研究所选育的南亚系列和实选系列可明确其亲缘关系较近的栽培种,可由此推断其可能父母本。从聚类图中可发现澳洲坚果种质并不是严格按照地理来源进行分类,这说明其亲缘关系与地理来源可能无显著相关性。
澳洲坚果种质主成分分析
澳洲坚果种质亲缘关系可通过主成分分析直
观表现,种质间位置远近代表其遗传相似性,位置越近,亲缘性越近,位置越远,遗传差异越大。采用GenALEx软件对91份澳洲坚果种质进行主成分分析,从图2可以看出,来源于夏威夷、澳大利亚、中国的澳洲坚果种质之间存在交叉,说明部分种质亲缘性较近。
基于贝叶斯聚类法的遗传结构分析
基于9对SSR引物获得的多态性数据,使用Structure软件中的贝叶斯聚类方法分析澳洲坚果群体遗传结构。根据Delta 折线图(图3)所示,值为2时,Delta 最大,为278,即=2为最佳值,表明91份澳洲坚果种质的群体遗传结构分析结果可分为2大类群(图4中红色代表类群1,绿色代表类群2)。类群1主要包含夏威夷的19份种质,中国44份种质,澳大利亚10份资源,以及南非1份资源。澳大利亚16份种质以及1份南非种质为类群2,并且此类群主要为粗壳种种质。根据各类群中主要颜色比率(值),将>0.6视为材料血缘比较单一,而<0.6表示材料具有混合来源,说明供试澳洲坚果种质遗传成分来源单一,遗传结构较为简单。
群体遗传分化
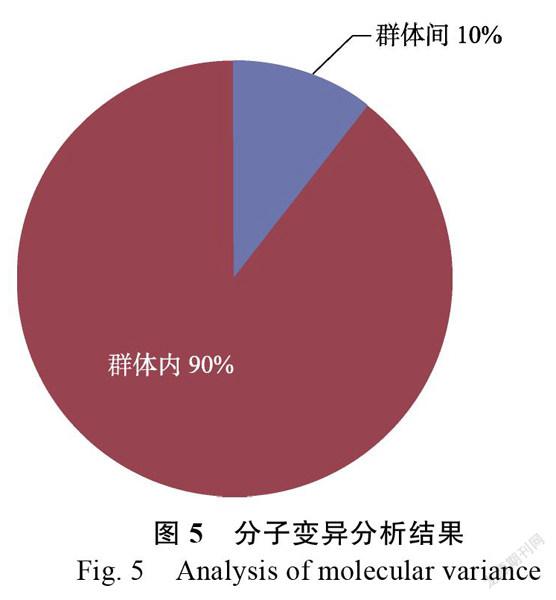
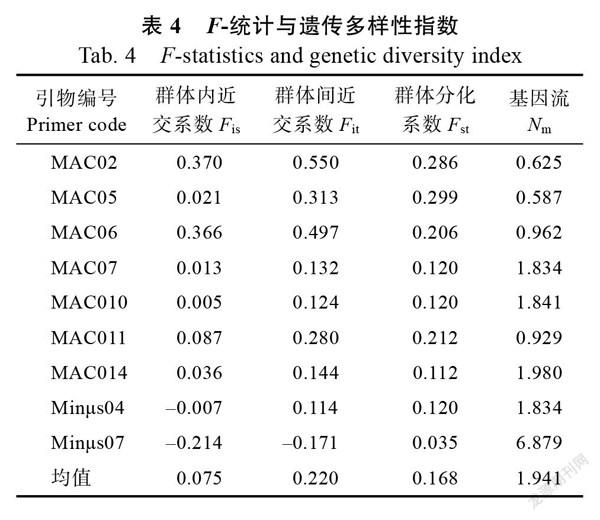
由表4 -统计结果显示,澳洲坚果种质遗传分化系数()变幅范围为0.035~0.299,平均值为0.168,群体内近交系数()变幅范围为–0.214~0.370,均值为0.075,群体间近交系数()为–0.171~0.550,均值为0.220,基因流()均值为1.941,说明91份澳洲坚果种质具有较高的遗传分化,且存在一定的基因交流。AMOVA分析结果表明,供试澳洲坚果群体内遗传变异为90%,群体间遗传变异为10%,表明澳洲坚果群体内遗传变异显著高于群体间变异。
讨论
遗传多样性的研究对于种质资源的遗传改良、杂交育种以及创新利用具有重要意义,SSR标记在澳洲坚果上的应用,有助于了解澳洲坚果种质的遗传多样性以及亲缘关系,对澳洲坚果种质资源进一步挖掘与育种工作具有参考价值。NOCK等基于二代测序技术开发了12对澳洲坚果SSR分子标记引物,在22个品种中,12对引物均具有多态性,平均观察杂合度和期望杂合度分别为0.571和0.626,共检测到71个等位基因,并且能很好地区分4种澳洲坚果种质、、以及。LANGDON等采用11对SSR引物对‘741’以及其他8个品种开放授粉后代进行父母本鉴定,指出不同品种澳洲坚果自交能力存在显著差异,并且澳大利亚种植最广泛的2个品种‘741’与‘344’之间存在一定亲缘关系。本研究采用9对SSR引物对91份澳洲坚果种质进行扩增,共检测出73个等位基因位点,基因多样性均值为0.689,PIC变化范围为0.453~0.792,平均0.659,说明9对引物可较好地鉴别澳洲坚果种质的遗传差异性。
本研究中91份澳洲坚果材料的UPGMA聚类、PCA主成分分析、以及Structure遗传结构分析的结果基本一致,且相互补充,采用的9对SSR引物将91份澳洲坚果种质分为2个类群,聚类结果表明,不同来源地种质聚集交错,与地理分布没有显著相关性,与谭秋锦等基于SNP分子标记的澳洲坚果种质遗传多样性分析中具有相同结论。同时Structure结果表明,供试澳洲坚果种质遗传成分来源单一,遗传结构较为简单,在其他研究中具有相同报道。HARDNER等报道全球澳洲坚果生产主要基于100年前夏威夷育种项目中优良种质的嫁接繁育,并且可能仅有一到三代来源于野生资源。NOCK等采用叶绿体基因组测序开发了SNP,对37份野生资源以及26份主栽品种进行亲缘关系分析,发现野生资源具有高水平的变异,但大部分夏威夷品种来源于位于昆士兰东南部Moolo和Mt Bauple的2个野生地点,来源非常狭窄。
指数反映群体等位基因杂合性水平,用来衡量种群分化程度。本研究91份澳洲坚果种质变化范围为0.035~0.299,平均值为0.168,表明澳洲坚果具有较高的遗传分化。O’CONNOR等采用SNP对29个母本及295个子代进行群体结构、遗传多样性分析,在子代中检测到高水平差异(=0.518)以及较大遗传分化(= 0.401)。AMOVA分析结果表明澳洲坚果群体间与群体内均存在遗传变异,但群体内遗传变异显著高于群体间变异。
我国澳洲坚果从20世纪80年代后期开始商业性种植,经过30多年发展,种植面积已居世界第一。目前我国已收集保存了较丰富的澳洲坚果种质资源,前期研究报道我国澳洲坚果种质资源遗传基础较窄,大致分为夏威夷类群和澳大利亚类群,本研究中供试的我国澳洲坚果种质与夏威夷、澳大利亚主要栽培品种聚为一大类,除此之外,我国澳洲坚果品种多由开放授粉的实生后代选育,杂交育种进展缓慢,对各种优质、高抗等优良性状种质资源挖掘与利用、新种质创制研究较弱。这说明今后在澳洲坚果选育中,应该加大引进优异野生资源力度,增加种质的遗传多样性,拓宽遗传基础。
本研究采用9对SSR引物对91份澳洲坚果种质进行了遗传多样性分析,初步明确了供试澳洲坚果种质的群体结构,为澳洲坚果种质资源鉴定和创新利用提供数据支撑,并且对澳洲坚果杂交育种以及优良种质创制具有重要的指导意义。
参考文献
- 万继锋, 杨为海, 曾 辉, 邹明宏, 张汉周, 陆超忠. 澳洲坚果种质资源叶片表型多样性分析及其数量分类研究[J]. 热带作物学报, 2017, 38(6): 990-997.WAN J F, YANG W H, ZENG H, ZOU M H, ZHANG H Z, LU C Z. Leaf character diversity analysis and numerical classification of spp. germplasm resources[J]. Chinese Journal of Tropical Crops, 2017, 38(6): 990-997. (in Chinese)
- HARDNER C M, PEACE C, LOWE A J, NEAL J M, PISANU P C, POWELL M, SCHMIDT A L, SPAIN C S,WILLIAMS K J. Genetic resources and domestication of [J]. Horticultural Reviews, 2009, 35: 1-126.
- 李志强, 贺熙勇, 吴 超, 耿建建. 分子标记在澳洲坚果研究中的应用[J]. 热带农业科技, 2018, 41(2): 46-49.LI Z Q, HE X Y, WU C, GENG J J. Application of molecular marker techniques in research[J]. Tropical Agricultural Science and Technology, 2018, 41(2): 46-49. (in Chinese)
- VITHANAGE V, WINKS C W. Isozymes as genetic markers for macadamia[J]. Scientia Horticulturae, 1992, 49(1/2): 103-115.
- ARADHYA M K, YEE L K, FRANCIS T, RICHARD M M. Genetic variability in [J]. Genetic Resources and Crop Evolution, 1998, 45(1): 19-32.
- 李文华. 十五个澳洲坚果品种幼龄期植物学形态观察[J]. 云南热作科技, 2001, 24(2): 39-41.LI W H. Observation on botanical morphology of fifteen varieties at young age[J]. Journal of Yunnan Crops Science and Technology, 2001, 24(2): 39-41. (in Chinese)
- WANG T, LI H T, ZHU H, QI S Y, ZHANG Y M, ZAHNG Z J, ZOU Q D. Comparative analyses of genetic variation in a tomato ( L.) germplasm collection with single nucleotide polymorphism and insertion-deletion markers[J]. Russian Journal of Genetics, 2019, 55(2): 204- 211.
- BARBOSSA M N N, TAKASHI M A. Variability in by rapd markers[J]. Crop Breeding and Applied Biotechnology, 2010, 10(3): 266-270.
- 郭凌飞, 邹明宏, 杜丽清, 曾 辉, 陆超忠, 刘晓静. 利用ISSR 分析澳洲坚果的亲缘关系[J]. 园艺学报, 2001, 38(9): 1741-1746.GUO L F, ZOU M H, DU L Q, ZENG H, LU C Z, LIU X J. Analysis on genetic relations of by ISSR markers[J]. Acta Horticulturae Sinica, 2001, 38(9): 1741-1746. (in Chinese)
- 譚秋锦, 王文林, 陈海生, 韦媛荣, 郑树芳, 黄锡云, 贺 鹏, 夏志强. 基于SNP分子标记的澳洲坚果种质遗传多样性分析[J]. 分子植物育种, 2020, 18(21): 7246-7253.TAN Q J, WANG W L, CHEN H S,WEI Y R, ZHENG S F, HUANG X Y, HE P, XIA Z Q. Genetic diversity of varieties based on single-nucleotide polymorphism[J]. Molecular Plant Breeding, 2020, 18(21): 7246-7253. (in Chinese)
- SMITH J S C, CHIN E C L, SHU H, SMITH O S, WALL S J, SENIOR M L, MITCHELL S E, KRESOVICH S, ZIEGLE J. An evaluation of the utility of SSR loci as molecular markers in maize (L.): Comparisons with data from RFLPS and pedigree[J]. Theoretical and Applied Genetics, 1997, 95: 163-173.
- 唐莹莹, 杨祥燕, 蔡元保, 李 穆, 曾黎明, 郑文武, 邱文武, 李季东, 叶维雁. 澳洲坚果SSR-PCR反应体系优化及其应用[J]. 福建农业学报, 2018, 33(2): 154-158.TANG Y Y, YANG X Y, CAI Y B, LI M, ZENG L M, ZHENG W W, QIU W W, LI J D, YE W Y. Optimization and application of SSR-PCR reaction system for [J]. Fujian Journal of Agricultural Sciences, 2018, 33(2): 154-158. (in Chinese)
- 李志强, 吴 超, 陶 丽, 陶 亮, 宫丽丹, 耿建建, 贺熙勇. 澳洲坚果杂交F1代的SSR鉴定[J]. 分子植物育种, 2019, 17(14): 4704-4707.WANG Z Q, WU C, TAO L, TAO L, GONG L D, GENG J J, HE X Y. SSR identification of hybrids F1 generation[J]. Molecular Plant Breeding, 2019, 17(14): 4704-4707. (in Chinese)
- NOCK C J, ELPHINSTONE M S, ABLETT G, KAWAMATA A, HANCOCK W, HARDNER C M, KING G J. Whole genome shotgun sequences for microsatellite discovery and application in cultivated and wild macadamia ()[J]. Applications in Plant Sciences, 2014, 2(4): 1300089.
- SCHMIDT A L, SCOTT L, LOWE A J. Isolation and characterization of microsatellite loci from [J]. Molecular Ecology Notes, 2006, 6(4): 1060-1063.
- HANCOCK J, MAGEMBE E, ZAWEDDE B M, GHISAIN M, AMARO G B, GRUMET R. Characterization of the genetic diversity of Uganda’s sweet potato () germplasm using microsatellites marker[J]. Genetic resources and Crop Evolution, 2015, 62(4): 501-513.
- 李 博. 鹅掌楸群体空间遗传结构研究[D]. 南京: 南京林业大学, 2013.LI B. The spatial genetioc structure of Sarg as revealed by SSR markers[D]. Nanjing: Nanjing Forestry University, 2013. (in Chinese)
-
收稿日期 2021-05-10;修回日期 2021-07-06
基金项目 中国热带农业科学院基本科研业务费专项(No. 1630062020001);农业农村部物种品种资源保护费项目(No. 12516
3006000160001);国家热带果树种质资源圃平台运行服务项目(No. 125163006000150007)。
作者简介 井敏敏(1990—),女,硕士,研究实习员,研究方向:热带果树种质资源。*通信作者(Corresponding author):
陈晶晶(CHEN Jingjing),E-mail:chenjingjing0704@163.com。
