侵染大豆的花生斑驳病毒公主岭分离物基因组测序及分析
2022-02-10张春雨李小宇王晨战笑蕾刘建青王永志
张春雨 李小宇 王晨 战笑蕾 刘建青 王永志





摘要
本研究利用小RNA深度测序法在大豆叶片上检测到1株花生斑驳病毒Peanut mottle virus (PeMoV),根据小RNA深度测序结果和GenBank公布的PeMoV基因组序列设计引物克隆了PeMoV公主岭分离物(PeMoVGongzhuling)的基因组序列。测序结果经拼接后获得了PeMoVGongzhuling基因组,大小为9 709个核苷酸(GenBank登录号:MT790744)。该基因组在第123位—第9 422位存在1个大的开放阅读框(ORF),编码1个多聚蛋白(分子量351.28 kD)。PeMoVGongzhuling与GenBank中已登录的其他PeMoV分离物的基因组的核苷酸序列一致性为95.88%~99.53%,氨基酸序列一致性为97.39%~99.84%,其中与韩国分离物GYBU92 (MT603819)在核苷酸和氨基酸水平上的一致性均为最高。
关键词
花生斑驳病毒;基因组;大豆
中图分类号:
S435.29
文献标识码:A
DOI:10.16688/j.zwbh.2020544
Sequencing and analysis of the genome of Peanut mottle virus (PeMoV) Gongzhuling isolate infecting soybean
ZHANG Chunyu1,LI Xiaoyu1,WANG Chen1,2,ZHAN Xiaolei1,3,LIU Jianqing1,3,WANG Yongzhi1*
(1. Jilin Academy of Agricultural Sciences, Gongzhuling136100, China; 2. Jilin Agricultural University,
Changchun130118, China; 3. Changchun Normal University, Changchun130032, China)
Abstract
In this study, the Peanut mottle virus (PeMoV) from soybean leaves was detected by small RNA deep sequencing. Primers were designed according to results of sRNA deep sequencing and the published genome sequences of PeMoV on GenBank, and the segments of genome of PeMoV Gongzhuling isolate (PeMoVGongzhuling) was amplified by RTPCR. The assembled genome of PeMoVGongzhuling was 9 709 nucleotides (GenBank accession no. MT790744). A large open reading frame (ORF) was found located between the 123th and the 9422th nucleotides, which encoded a polypeptide of 351.28 kD. By comparing it with other isolates of PeMoV, the nucleotide identity was 95.88% 99.53%, and the amino acid identity was 97.39% 99.84%, and it shared the highest identity with the South Korea isolate GYBU92 (MT603819).
Key words
Peanut mottle virus(PeMoV);genome;soybean
花生斑駁病毒Peanut mottle virus (PeMoV)是马铃薯Y病毒属Potyvirus成员,主要靠汁液接触、种子或由蚜虫以非持久方式传播。PeMoV除严重危害花生外,其寄主范围仅局限于豆科植物,例如菜豆、大豆、豌豆、豇豆等[1]。 1972年Kuhn等[2]首次在美国佐治亚州发现PeMoV侵染大豆;之后陆续在美国的弗吉尼亚州、南卡罗莱纳州,澳大利亚,东非等地发现PeMoV侵染大豆[3 4];1984年薛宝娣等[5]首次报道了PeMoV 在我国大豆上的侵染情况,并进行了PeMoV对不同品种大豆的侵染试验;2014年Lim等[6]在韩国首次发现PeMoV侵染大豆并获得了全基因组序列。PeMoV侵染大豆后叶片表现为明脉、花叶、黄脉、坏死等症状,一般可导致大豆减产12%~28%[4],一些感病品种可达到41%,还会导致大豆含油量降低,品质下降[3]。
本研究利用小RNA深度测序方法在感病大豆叶片上检测到1株花生斑驳病毒,分段克隆测序获得了全基因组序列并进行了同源性分析,为大豆花生斑驳病毒病的防控提供依据。
1材料与方法
1.1材料
感病大豆叶片采自吉林省长春市公主岭市(124°4′E,43°31′N), 80℃保存。
1.2感病大豆叶片总RNA提取
参照TRIzol(Invitrogen)说明书提取感病大豆叶片总RNA,NanoDrop微量核酸蛋白浓度测定仪(Thermo Scientific)测定浓度。
1.3小RNA文库建立、测序及分析
樣品总RNA(2个重复)质量由北京诺禾致源科技股份有限公司检测,质量合格后使用Small RNA Sample Pre Kit构建文库,文库构建完成后先使用Qubit 2.0初步定量,再使用Agilent 2100对文库中插入片段的大小(insert size)进行检测,插入片段大小符合预期后使用qPCR对文库有效浓度进行准确定量,以保证文库质量。文库检测合格后进行HiSeq/MiSeq测序,采用无参考基因组寄主病毒sRNA分析流程进行候选病毒评估。
1.4PCR扩增、测序和序列分析
根据小RNA测序获得的拼接片段和GenBank中已报道的PeMoV分离物基因组序列,分段设计4组病毒引物(表1),以大豆叶片总RNA反转录合成的cDNA为模板进行RTPCR扩增。 PCR反应体系(50 μL):2×SanTaq PCR Mix 25 μL,10 μmol/L 引物各1 μL, 模板cDNA 1 μL,ddH2O 22 μL。扩增条件:94℃预变性5 min;94℃变性30 s,55℃退火30 s, 72℃延伸1~4 min(根据目的片段大小调整时间),35个循环;72℃延伸10 min,4℃保存。扩增产物经1%琼脂糖凝胶电泳检测后, 回收目的片段由吉林省库美生物科技有限公司测序。根据测序结果设计用于扩增5′、3′非编码区的引物PeMoVR、PeMoVF,采用SMARTer RACE 5′/3′试剂盒(TaKaRa)扩增5′、3′非编码区。利用DNAMAN软件拼接序列,使用MEGA 7.0软件进行进化分析,采用neighborjoining法 构建系统发育树。
2结果与分析
2.1田间感病大豆植株症状
对公主岭大豆田进行病毒病调查时,发现1株染病大豆植株相对矮小,叶片褪绿、斑驳,叶肉呈泡状突起(图1)。
2.2大豆叶片sRNA深度测序分析
提取大豆叶片总RNA,经sRNA测序共获得18 822 100个reads,去除含有接头的、低质量的reads,获得clean reads 17 549 725个。筛选18~40 nt的序列与GenBank Virus RefSeq核酸数据库
进行比对,初步鉴定样品感染病 毒种类(表2)。其中比对到花生斑驳病毒(PeMoV)的reads数占比对到数据库的总reads数的11.8%。
2.3PeMoVGongzhuling基因组序列分析
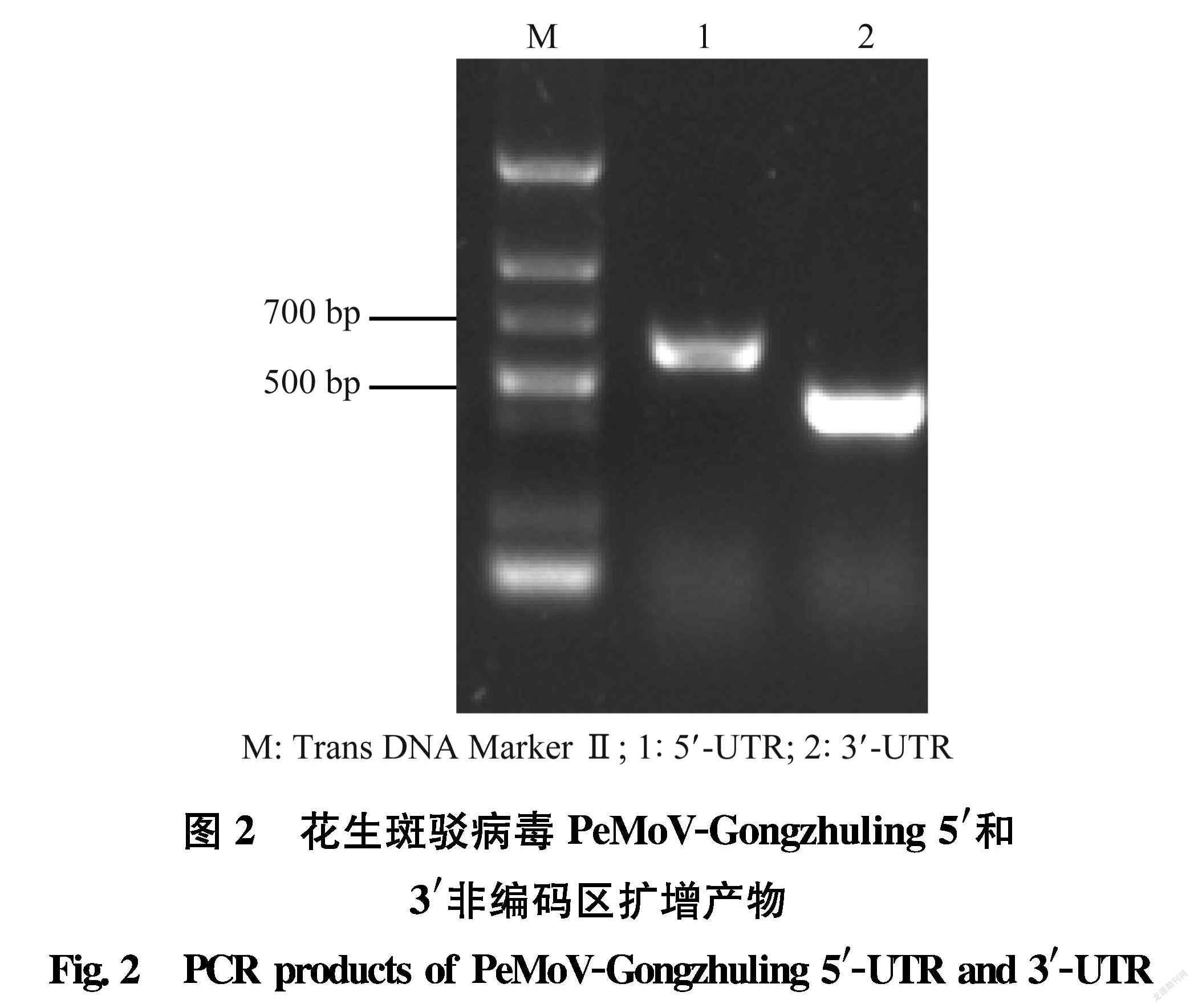
根据小RNA测序获得的拼接片段和GenBank中已报道的PeMoV分离物基因组序列,设计引物进行分段扩增,获得与预期大小一致的片段,对回收产物进行测序。根据测序结果设计5′、3′非编码区引物,采用SMARTer RACE 5′/3′试剂盒扩增目的片段(图2),回收产物测序。
对分段测序结果进行拼接、校正后,除poly(A)尾外,获得的PeMoVGongzhuling的基因组序列长9 709 bp,提交到GenBank取得的登录号为MT790744。序列中A、T、C、G 4种碱基的含量分别为32.66%、25.18%、18.17%、23.99%,5′非编码区(5′UTR)包含122个碱基,3′非编码区(3′UTR)包含287个碱基。 PeMoVGongzhuling在第123位 第9422位存在1个大的开放阅读框(ORF),编码1个由3 099个氨基酸组成的多聚蛋白(分子量351.28 kD)。该多聚蛋白自身分解为10个成熟的蛋白质,从N端到C端依次为P1(36.82 kD)、HCPro( 51.29 kD)、P3(40.31 kD)、6K1(5.75 kD)、CI(70.91 kD)、6K2(6.08 kD)、NIaVpg(21.35 kD)、NIaPro(27.75 kD)、NIb(59.58 kD) 和CP(31.60 kD)。通过与其他PeMoV分离物的氨基酸序列比对,确定了多聚蛋白的9个切割位点,分别为KIHQY/S、HYVVG/G、PVVYQ/A、TVRYQ/S、TVQYQ/S、PVKYQ/G、VVATE/G、SVEYQ/S、EVRYQ/S(图3)。
2.4PeMoVGongzhuling与PeMoV其他分离物的同源性比较
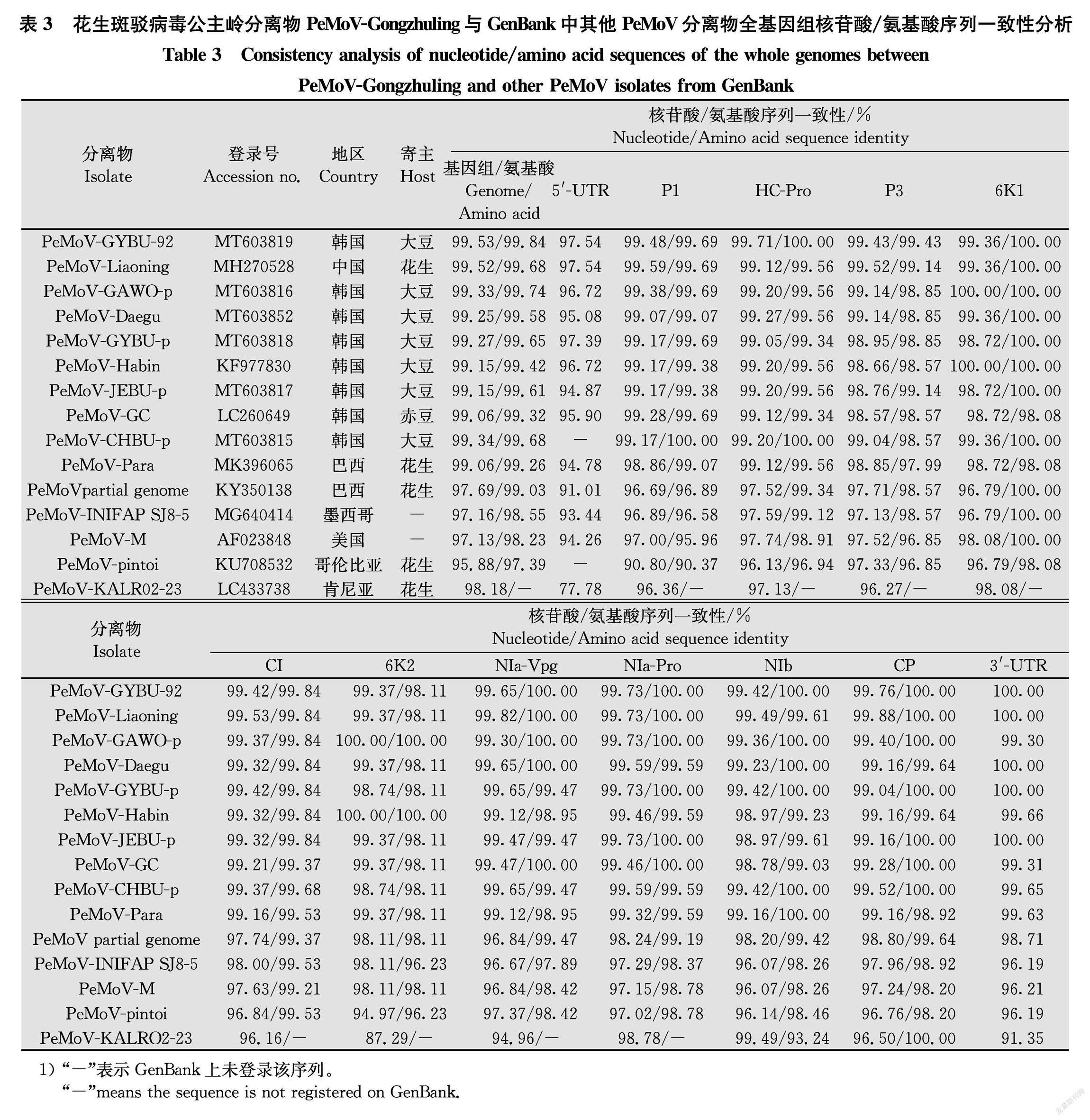
利用DNAMAN软件将PeMoVGongzhuling全基因组序列与GenBank中已公布的其他PeMoV分离物基因组进行序列一致性分析,发现PeMoVGongzhuling与它们在核苷酸水平上的一致性为95.88%~99.53%,在氨基酸水平上的一致性为97.39%~99.84%,其中与韩国分离物GYBU92 (MT603819)的同源性在该两种水平上均为最高。在单个基因区段上,HCPro、NIaPro、CP基因的序列一致性多数高于全基因组序列一致性的平均值,变异程度较低(表3)。
以马铃薯Y病毒Potato virus Y(PVY)为外组生物构建系统发育树,所有分离物聚成3个簇群,其中哥伦比亚分离物PeMoVpintoi(KU708532)单独成为1簇,美国分离物PeMoVM(AF023848)、墨西哥分离物PeMoVINIFAP SJ85(MG640414)、 巴西分离物PeMoV(KY350138)聚成1簇,其余分离物聚成1簇。PeMoVGongzhuling与PeMoVGYBU92 (MT603819)的亲缘关系最近(99.53%)(图4)。
3讨论
PeMoV侵染大豆的情况已陆续在世界各地报道,在我国各地也普遍发生,目前GenBank上已报道的PeMoV大豆分离物全基因组序列均来自韩国。本研究首次在吉林公主岭大豆上克隆得到了PeMoVGongzhuling的全基因组序列,对其进行一致性分析发现,PeMoVGongzhuling与侵染大豆的韩国分离物GYBU92 (MT603819)一致性最高(99.53%),其次是侵染花生的辽宁分离物(MH270528)(99.52%)。 对PeMoVGongzhuling的基因产物比对发现,P1、P3、CI、NIaVpg、NIaPro、NIb和CP在核苷酸水平均与辽宁分离物(MH270528)一致性最高,HCPro与韩国分离物GYBU92 (MT603819) 一致性最高,而6K1和6K2与侵染大豆的韩国分离物PeMoVGAWOp (MT603816)和PeMoVHabin (KF977830)一致性最高。
吉林省作为国家粮食主产區之一,大豆、花生、杂粮杂豆等种植面积广泛,近年来随着花生种植效益优势日益凸显,吉林省花生种植面积由2000年的不足6万hm2到2017年统计的27万hm2左右,增长了近5倍[7],2018年达到33万hm2左右,超过同期大豆的种植面积。PeMoV是侵染花生及豆科植物的重要病害,尤其在花生和大豆毗邻种植区,更易造成病毒病大面积暴发,严重影响花生、大豆的产量及种质资源。目前吉林省PeMoV侵染大豆的情况还未见报道,田间也未见大面积发病,更应予以重视,及时防控。
本研究为后续研究PeMoV基因组结构和功能,基因进化机制以及病毒与寄主的互作等研究提供了理论基础,同时对大豆的PeMoV检测、防控以及抗病育种等工作具有重要意义。
参考文献
[1]蒲绪志.花生斑驳病毒病的发生与防治[J].农业灾害研究, 2018,8(4):78-79.
[2]KUHN C W, DEMSKI J W, HARRIS H B. Peanut mottle virus in soybeans [J]. Plant Disease Reporter, 1972,56:146-147.
[3]BOERMA H R, KUHN C W. Inheritance of resistance to Peanut mottle virus in soybeans [J]. Crop Science, 1976,16(4): 533 534.
[4]DEMSKI J W, KUHN C W. A soybean disease caused by Peanut mottle virus [J]. Research Bulletin Georgia Agricultural Experiment Stations, 1977,196:36.
[5]薛宝娣,陈永萱,施志新.大豆上发生的花生斑驳病毒[J].中国油料作物学报,1987, 9(2):38-40.
[6]LIM S, LEE Y H, IGORI D, et al. First report of Peanut mottle virus infecting soybean in South Korea [J]. Plant Disease, 2014, 98(9): 1285.
[7]牛海龙,李玉发,何中国, 等.吉林省花生产业发展需求报告[J].东北农业科学,2019, 44(3):11-13.
收稿日期:2020-10-16修订日期:2020- 11- 24
基金项目:
吉林省科技厅重点研发计划(20180201013NY);吉林省农业科技创新工程(CXGC2021ZY022)
* 通信作者
E-mail:yzwang@126.com
