荔枝蝽 Tessaratoma papillosa卵黄原蛋白及受体的序列及时空表达分析
2021-11-12程林韩顺财蒋敬涛李海超彭凌飞
程林 韩顺财 蒋敬涛 李海超 彭凌飞


摘要:【目的】為荔枝蝽Tessaratorria papillosa的产卵繁殖行为提供分子层面的理论基础,并为荔枝蝽防治的靶点筛选提供有益思路。【方法】采用转录组测序的方法,对荔枝蝽小同发育时期及组织进行转录组测序。通过筛选荔枝蝽的转录组数据和分子克隆的方法获得荔枝蝽卵黄原蛋白及其受体基因,并利用实时荧光定量PCR( qRT-PCR)分析其在小同发育阶段和组织部位的时空表达情况。【结果】获得荔枝蝽3个卵黄原蛋白基因(Tpapi Vgl,Tpopi Vg2,Tpapi Vg3)和1个卵黄原受体蛋白基因(Tpapi_VgRs)。对4个基因进行分析,发现其均具有典型的保守结构域,是典型的昆虫Vg和VgRs基因。从进化关系看,荔枝蝽的Vg和VgRs基因的分子进化与物种之问的进化关系较为匹配。qRT-PCR结果显示Vgl基因在雌虫各个组织中表达量都比较高,雄成虫中仪在淋巴液中表达量较高,而Vg2和Vg3基因仪在雌虫脂肪体中较高表达。VgRs的表达与Vg的表达部位基本一致,在雌虫卵巢中表达量最高,其次是雌虫、雄虫淋巴液和若虫的脂肪体当中,在若虫触角、雄虫触角和雄虫精巢中也有少量表达。【结论】获得了荔枝蝽3个卯黄原蛋白基因和1个卵黄原蛋白受体基因,对其结构和进化关系进行探讨,并对其时空表达情况进行分析,为后续对荔枝蝽新防治靶标的筛选奠定基础,
关键词:卵黄原蛋白;卵黄原受体蛋白;转录组;进化分析;表达量分析
中图分类号:S 435
文献标志码:A
文章编号:1008-0384( 2021) 07-0793-13
Sequences and Spatiotemporal Expressions of Vitellogenin and Vitellogenin Receptor
Genes of Tessaratoma papillosa
CHENG Lin l, HAN Shuncai I. JIANG Jingtao l, LIHaichao 2, PENG Lingfei l*
(1.Biological Control Research Institute,Fujian Agriculture and Fores trv University/China Fruit Flv Research and C ontror
Center of FAO/IAEA/Key Laboratory of Biopesticide and Chemical Biology,Ministy of Education/State Key Laboratory ofEcological Pest Control for Fujian and Taiwan Crops,Fuzhou,Fujian 350002,China:2.Key Laboratory of Insect Developmentarand Evolutionary Biology/Centerfor Excellence m Molecular Plant Sciences/lnstitute of Plant Physiology and Ecology,Chinese Acaderriy of Sciences,Shanghai 200032,China)
Abstract: 【Objective】 Molecular information associated with the oviposition and reproduction of Tessaratoma papillosawas studied for control of the pest on Iychee trees. 【Method】Transcriptome sequencing of T papillosa in various tissues anddevelopmental stages was conducted. The vitellogenin and vitellogenin receptor genes were obtained by screening thetranscriptome database and applying molecular cloning methods. Temporal and spatial expressions of the genes were analyzedusing qRT-PCR【Result】Three vitellogenin genes (i.e.,,papi_ Vg1,Vg2,and Vg3) and one receptor gene,T papi VgRs,were obtained after screening Homologous analysis showed the genes contained conserved domains typical in the Vg and VgRsof insects. The molecular evolution of Vg and VgRs of T. papillosa paralleled that of the species. The temporal and spatialexpressions of Vgl shown by qRT-PCR were relatively high in all female tissues, but only the lymphatic fluid in adult males.whereas those of Vg2 and Vg3 highly expressed only in female adipose tissue. The expression of VgRs, as well as Vg, washighest in female ovary, followed by female, male lymph fluid, nymph adipose tissue. and minute in nymph antenna, maleantenna. and male testis.【Conclusion】 The structures and evolution of the 3 vitellogenin genes and one vitellogenin receptorgene were analyzed. The information on the spatiotemporal expressions of the genes would aid target selection in study for thecontrol of T. papillosa.
Key words: Vitellogenin gene: vitellogenin receptor gene; transcriptome; evolutionary analysis; gene expression
0 引言
【研究意义】荔枝蝽Tessaratoma papillosa隶属于半翅目Hemiptera荔蝽科Tessaratomidae,是我国及东南亚等地荔枝、龙眼产区的重要害虫。荔枝蝽以成虫和若虫刺吸危害果树幼枝、嫩芽、花和幼果,导致果树嫩枝萎蔫、落花落果。成、若虫臭腺分泌物还可灼伤植株幼嫩部位,造成枯死,同时还可传播龙眼鬼帚病[1-2],荔枝蝽常年危害严重,造成巨大的经济损失。在发生严重年份,荔枝、龙眼可减产20%~30%,甚至部分地区减产达到70%~80%,甚至绝收[3]。【前人研究进展】卵黄原蛋白(vg)是一类大分子糖脂复合蛋白,广泛存在于非哺乳类性成熟卵生雌性动物的血淋巴、脂肪体和卵中,是几乎所有卵生动物卵黄蛋白的前体,能为正在发育的胚胎提供氨基酸、脂肪、碳水化合物、維生素和微量元素等营养和功能性物质[4-8]。目前已知的昆虫卵黄原蛋白序列约30条,主要有蜚蠊目的美洲大蠊Periplanetaamericana[9],德国小蠊Blattella gerrrianica[10];鳞翅目的柞蚕Anthereapernyi[11],野桑蚕Bombyxmandarina[12],眉纹天蚕蛾Samia cynthia,绿尾大蚕蛾Actiasningpoana,半目大蚕蛾Antheraea yamamai,舞毒蛾Lymantria dispar;膜翅目的意大利蜜蜂Apis mellifera[13],蚜小蜂Aphytissp.;双翅目的埃及伊蚊Aedes aegypti[14],疟蚊Anopheles sp.;半翅目的褐飞虱Nilaparvata lugens[15]等。昆虫卵黄原蛋白一般在雌性成虫的脂肪体合成,随后进入血淋巴,经血淋巴运输到卵巢,与膜上卵黄原蛋白受体结合,经胞吞作用,使卵黄原蛋白进入卵巢细胞,并行使功能[7-8.16-17]。昆虫卵黄原蛋白的mRNA全长大约为6 000 bp,在整个加T过程中,经蛋白水解,在完全变态昆虫和不完全变态昆虫中切割成两段或多个小的片段[7,l7-18]。根据卵黄原蛋白前体被切割的形式,可以将其分为三类:①没有被酶切的蛋白前体,仅含有一个大分子量亚基[7];②酶切为一个大分子量的亚基( >180 ku)和一个小分子量的亚基( <50 ku),绝大多数完全变态昆虫中的vg蛋白前体属于这一类型[7.19];③酶解为几个分子量约为80~110 ku的多肽,大部分不完全变态昆虫中的vg蛋白前体属于这一类型[17]。昆虫的卵黄原蛋白受体( VgRs)属于低密度脂蛋白受体家族成员,昆虫的VgRs具有LDLR(low density lipoproteinreceptor)家族典型的保守结构域[17.20-21]。一般来说,VgRs在成虫羽化前的卵巢中合成,随后经过血淋巴运输到卵巢与卵黄原蛋白结合,昆虫的VgRs具有卵巢特异性,是卵黄原蛋白Vg的专一性胞吞作用受体,可介导vg进入昆虫卵母细胞,而后沉淀积累形成昆虫生殖必需的卵黄蛋白[8.22]。VgRs与昆虫卵巢激活,卵黄发生,卵子形成密切相关,同时在昆虫信息交流、社会分化、行为构建以及免疫调控等行为过程中起到至关重要的作用。【本研究切入点】荔枝蝽新成虫大规模发生以后,由于抗药性强[23],在采取化学防治时不能产生较好的防治效果。而通过人T释放平腹小蜂Anastatus fulloi进行生物防治较为可行,但平腹小蜂抗寒能力较弱,容易被农药杀伤,野外种群需要较多营养使种群复壮,需要在防治时错峰喷洒农药才能产生较好的防治效果[24]。在愈加重视食品安全和绿色植保的今天,筛选出新的荔枝蝽防治靶标迫在眉睫。本研究通过分子生物学技术,从基因层面分析荔枝蝽的产卵过程。【拟解决的关键问题】通过转录组获得荔枝蝽卵黄原蛋白及其受体基因序列,并分析其进化关系、结构和时空表达情况。为了解荔枝蝽产卵的生理过程提供分子层面的信息,同时亦可为从卵期防控荔枝蝽的发生奠定理论基础。
1材料与方法
1.1供试昆虫
野外虫源采白福建省福清市龙眼果园内,将荔枝蝽雌、雄成虫若虫分别保存等待解剖处理。
1.2荔枝蝽的解剖与处理
荔枝蝽的解剖采用蜡盘解剖法,解剖时先用剪刀剪去翅、足、中胸小盾片和腹侧板,随后自腹部两侧剪开,再横向在消化道上方剪断前胸背板,并用镊子掀开背板,用昆虫针将剩余的组织固定在蜡盘中,注入清水至淹没虫体,在昆虫双目解剖镜下解剖,取得荔枝蝽雌雄成虫和若虫的脂肪体、中肠,雌雄成虫的卵巢和精巢、若虫和雄成虫触角[25]。解剖后得到的组织置于1.5 mL离心管中,在80℃冰箱保存,样品来源及名称如表1所示。
1.3 RNA抽提及全长cDNA的扩增
总RNA的提取使用TRIz01.RJ Reagent( Invitrogen)进行,并严格按照实验手册要求进行。在1 mL TRIz01中加入用研磨柱(使用前用DEPC浸泡并高温灭菌)充分研磨的液氮速冻过的样品,充分混匀,并室温静置5 min。在混合溶液中加入200 uL氯仿,充分震荡混匀,并室温静置5 min(氯仿要在阴凉避光处保存,避免暴露空气中被阳光照射产生毒气)。将混合溶液用4℃低温离心机离心15 min,12 000 r·mm-1。离心后溶液分为3层,下层为有机相、中层为白色界面、上层为水相,RNA全部在水相中溶解。将上层溶液转移至新的离心管中,加入500 uL预冷的异丙醇(无水乙醇),充分震荡混匀,置于80℃超低温冰箱静置30 min,使RNA沉淀。将样品取出后,4℃,12 000 r·mm-1,高速离心10 min,倒掉上清,注意不要将沉淀倒出。用500 uL预冷的无RNase 75%乙醇洗涤沉淀,涡旋仪震荡10 s,充分溶解沉淀中的盐离子。4℃,12 000 r·mm-1,高速离心5 min,小心吸取上清后在室温下静置5 min,使沉淀干燥,加入适量无RNase ddH20溶解沉淀,即可得到总RNA样品。在分光光度计下检测吸光度,1%琼脂凝胶电泳检测总RNA质量, 80℃保存待用。
使用试剂盒ReverTra Ace?qPCR RT Master Mixwith gDNA Remover( TOYOBO),并按照说明书进行实验操作。初次使用时,将试剂盒中的4×DNMaster Mix与gDNA Remover按照50:1的比例混合。将2 ug RNA样品在65℃水浴锅中热变性5 min,立即置于冰上冷却。在冰上配制反应液( RNA template,2ug;4×DN Master Mix( gDNA Remover),4uL;Nuclease-free Water补足16 UL;在加入5×RT MasterMix II,4uL),将反应液轻轻混匀后,在37℃条件下孵育5 min。
将配置好的反应液轻轻混匀后,按照以下程序进行逆转录反应(37℃,15 min;50℃,5 min;98℃,5 min)。反应结束后,将样品稀释10倍放置在20℃备用。
1.4 PCR扩增
引物设计及全长序列扩增:用Geneious R11软件设计引物,本试验所用引物见表2。以荔枝蝽cDNA为模板进行PCR扩增,反应体系:cDNAtemplate 2uL;2×PCR MIX 25 uL; Primer F luL;Primer R luL; Nuclease-free Water 21 uL。用高保真Taq酶(Takara)擴增目标片段全长序列,反应程序为:95℃1 min. 95℃30 s,58℃30 s,72℃1 mm.72℃7 min,4℃保存,反应结束后,将样品进行测序反应,测序委托上海铂尚生物技术有限公司进行。
1.5基因序列的生物信息分析
利用ORF finder预测ORF全长(https://www.ncbi.nlm.nih.gov/orffinder/),利用SignalP5.0进行信号肽预测(http://www.cbs.dtu.dk/services/SignalP/),EXPASY{十算pl/Mw( https://web.expasy.org/compute pi/),利用SMART软件分析基因的蛋白结构域(http://smart.embl-heidelberg.de/),用Clustal X v2.1软件对荔枝蝽3个卵黄原蛋白的氨基酸序列进行同源比对分析,利用MEGAX软件,建立NJ树(Bootstrap值1000)进行系统进化分析。
1.6 实时定量PCR(Real-time quantitative PCR,qRT-PCR)
qRT-PCR反应使用试剂盒为SYBR?Premix ExTaq rM II( Takara),所有操作过程使用的试剂均放置在冰上进行。反应所用的PCR 96孑L板或八连管为荧光定量试验专用,引物见表3。
按如下组分配置反应液:cDNA template 2uL;SYBR? R.Premix Ex Taq rM 11 12.5 uL; PCR ForwardPrimer( 10 umol-L-l)1uL; PCR Reverse Primer( 10umol.L-1)1uL; Nuclease-free Water 8.5 uL。整个过程保持在冰上进行(通常模板量在100 ng以下,引物终浓度为0.4 umol·L-1时结果通常较好),轻轻混匀,避免产生气泡。采用两步法PCR程序进行实时定量PCR反应,反应体系如下:95℃30 s,95℃5s,60℃30 s,40个循环。
为了消除个体差异,每个试验组的样品为3个同一组织RNA反转录的cDNA等量混合,作为一个样品池进行检测。每个样品3次重复,其表达水平分析选用18S rRNA的表达量进行均一化处理。反应结束后根据Real Time PCR的扩增曲线和溶解曲线判断引物质量。数据分析参照2 -△ACT法进行[26]。数据间的差异显著性分析通过T.Test进行,通过计算均值与数据间的标准偏差(SD)得到相应结果。
2结果与分析
2.1 Vg基因全长序列CDS的获得及分析
通过筛选荔枝蝽的转录组数据(数据未发表)获得3个vg序列和1个VgRs序列,其mRNA分别为Vgl 5 764 bp,Vg2 6 216 bp,Vg3 6 278 bp和VgRs3 239 bp,将其分别命名为T papi Vgl,T papi_Vg2,T papi Vg3,T papi VgRs,将获得的序列进行PCR扩增,随后测序验证,最终将获得的序列进行ORF预测,蛋白二级结构分析,信号肽,等电点等分析,结果如表4所示。
随后,用SMART软件分析了4个基因的蛋白结构域,结果显示,荔枝蝽3个vg蛋白包含了卵黄原蛋白的3个典型结构域,一个完整的Vitellogenin_NSupperfamliy domain,一个DVF1943结构域和一个VWD结构域,所不同的是,Vg2在DUF1943结构域前面,包含一个low complexity( 774-785)结构域,而Vg3在VWD结构域之后,包含一个low complexity( 1752-1756)结构域,卵黄原受体蛋白包含了8个LDLRA,3个EGF,7个LY和1个GRAM结构域(图1)。
2.2序列一致性及进化分析
用Clustal X v2.1软件对荔枝蝽3个卵黄原蛋白的氨基酸序列进行同源比对分析(图2),结果显示3个卵黄原蛋白之间序列一致性为59.73%,其中 T papi_Vgl与T papi_ Vg2的序列一致性为50%,T papi_Vgl与T papi_Vg3的序列一致性为42%,T papi_Vg2与T papi_Vg3的序列一致性为45%。
随后,利用34个其他物种昆虫vg蛋白和28个其他物种昆虫的VgRs蛋白分别进行进化树分析(表5、6),利用MEGAX软件,建立NJ树(Bootstrap值1 000)(图3),结果显示,在所获得的具有全长的37个vg蛋白中序列之中,卵黄原蛋白分成3个大群,其中荔枝蝽的3个卵黄原蛋白与蝽科的豆类点蜂缘蝽Riptortus pedestris和大眼长蝽Geocorispallidipennis聚集在了一支上,其次是负蝽科的大田负蝽Lethocerus deYrolli盲蝽科的绿盲蝽Lygus lucorum和黑肩绿盲蝽Cyrtorhinus lividipennis聚集在一起。还发现,鳞翅目的荔枝蒂蛀虫Conopomorphasinensis与绿盲蝽Lygus lucorum的Aluco Vg2蛋白很好地聚集在一起,随后与其他半翅目昆虫灰飞虱Laodelphax striatellus、褐飞虱Nilaparvata lugens和油蝉Graptopsaltria nigrofuscata聚集形成了一个大群。说明宿主可能会改变昆虫的一些基因序列,从而产生进化中的相似性。其次,鳞翅目昆虫除了荔枝蒂蛀虫Conopomorphasinensis之外的其他昆虫聚集形成一个大群,膜翅目昆虫和半翅目的烟粉虱Bemisiatabaci以及脉翅目的大草蛉Chrysopa septempunctata聚集形成一个大群。
同样,对T papi_ VgRs的进化树分析(图4)结果显示,29个VgRs分别聚集,主要分为两大类,鳞翅目与双翅目昆虫聚集为一个大类,而荔枝蝽与半翅目其他昆虫烟粉虱B.tabaci、灰飞虱T.striatellus、褐飞虱Ⅳ.lugens和白背飞虱Sogatellafurcifera聚集在一起,随后与鞘翅目、蜚蠊目和啮虫目昆虫聚集形成一个大群,同目昆虫的VgRs进化关系更为接近,E值绝大多数都在50以上,说明VgRs与物种进化一致。
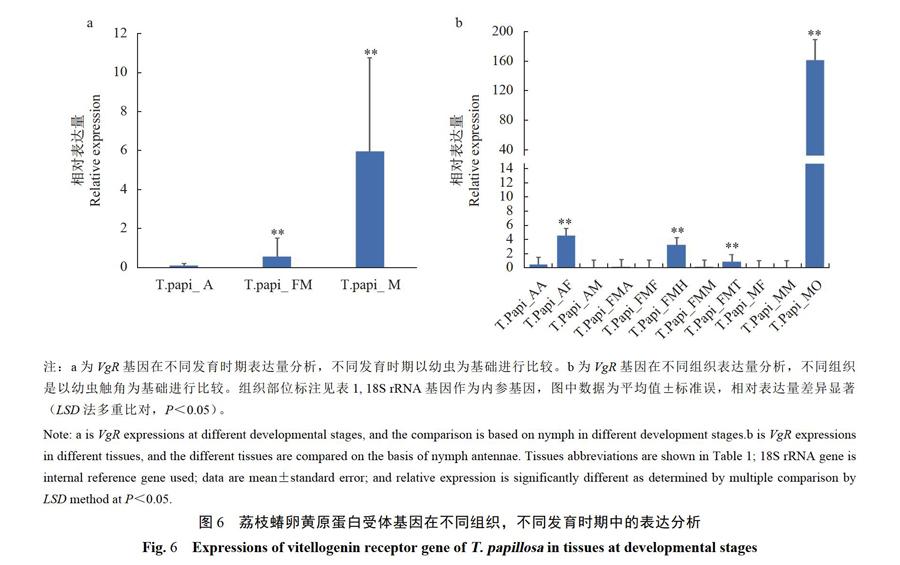
2.3荔枝蝽vg及VgRs基因的时空表达分析
用实时荧光定量PCR( qRT-PCR)检测荔枝蝽14个不同发育阶段的不同组织中的vg基因和VgRs基因的表达情况(图5、6),结果表明,Vgl基因在各个组织中表达量相对比较高,尤其是在雄成虫淋巴液、雌虫、雌虫脂肪体、雌虫中肠和卵巢中,而Vg2和Vg3基因仅在雌虫脂肪体中较高表达。VgRs的表达量,与vg的表达部位基本一致,在雌虫卵巢中,表达量最高,其次是雌虫,雄虫淋巴液和若虫的脂肪体当中,在若虫触角,雄虫触角和雄虫精巢中,也有少量表达。
3讨论与结论
卵黄原蛋白基因是编码昆虫和许多其他卵生生物卵内的主要蛋白一卵黄蛋白前体。在昆虫中,vg基因以性别、组织和阶段特异性的方式在脂肪体内和卵巢外表达。在生殖阶段,vg蛋白mRNA大量表达,然后被翻译,分泌到血淋巴中,并最终通过受体介导的内吞作用被发育中的卵母细胞吸收。Vgs以卵黄蛋白(Vn)的形式储存,卵黄蛋白(Vn)是发育中的胚胎的主要营养储备[27]。
Vgs和VgRs已在许多脊椎动物和无脊椎动物中被广泛鉴定,并且昆虫的Vgs和VgRs得到了广泛的研究,在蜚蠊目、直翅目、鞘翅目、双翅目、鳞翅目、半翅目和膜翅目等昆虫中均有相关基因被报道。通常,昆虫vg的氨基酸结构和组成高度保守[27]。它通常具有20个残基的信号肽,N端区域的脂质结合结构域( LPD N),C端区域的von Willebrand因子D型结构域( VWD)[27],以及未知的功能域( DUF1943)。
本研究通过筛选荔枝蝽T papillosa的转录组数据(数据另文发表),利用分子克隆的方法获得了荔枝蝽3个vg基因和1个VgRs基因,在序列结构方面荔枝蝽与其他报道的昆虫Vgs相似,3个荔枝蝽vg基因均含有20 bp左有的信号肽,并且均具有3个典型的vg蛋白的功能结构域,高度保守的卵黄蛋白原结构域LPD N和VWFD结构域[28]。以及目前仅在少数昆虫中(例如果斑螟Cadra cautella和白背飞虱S furcifera等)被报道发现的未知功能的DUF1943结构域[29-32]。同时,3个荔枝蝽vg蛋白序列也具有不同的特征,例如:Vg2在DUF1943结构域N端区域包含一个low complexity( 774-785)结构域,而Vg3在VWD结构域之后,C端区域的包含一个lowcomplexity(1752-1756)结构域。
对其他昆虫物种的研究结果显示,vg蛋白的基因数量因昆虫而异[33]。例如,意大利蜜蜂A.mellifera[34]、佛罗里达弓背蚁Campotusfloridanus[35],家蚕B.mori[36]和德国小蠊B.germanica[37]僅有1个vg基因,而美洲大蠊P.americana[38]带有2个vg基因。最近的研究表明,褐飞虱N.lugens具有3个vg基因[39]。目前,埃及伊蚊A.aegypti[40]和阿根廷蚁Linepithema humile[35.41]中基因数最高的是5个。这些基因数差异被认为是基因复制事件的结果。本研究在荔枝蝽中发现的3个vg基因,序列一致性为59.73%,T.papi Vgl与其他物种的结构相似性最高,具有比价保守的3个功能结构域,与在NCBI数据库中获得的几种蝽科昆虫的序列一致性相对较高,而T.papi Vg2与T.papi Vg3中均在不同的位置中插入了一个low complexity结构域,LCRs功能域是常见的通过减数分裂形成的重组事件,这些区域的动态多样化和无中间的变异和多态性水平较高,被认为是物种间表型变异的重要来源[42],这一结构特征,可能说明T papi Vg2与T.papi Vg3是在长期的进化过程中,由T papi_ Vgl基因复制及变异而来的,基因复制是基因组进化的重要机制,并且是基因表达模式和功能进化新颖的重要手段[43]。因此,这3个基因在具有类似的生理功能的同时,发生了什么样的改变,值得进一步探讨。
对荔枝蝽VgRs序列的分析表明,它由多个保守的模块化元件组成,与其他昆虫VgRs相似,并且是LDLR超家族的典型成员[17]。鳞翅目中的VgRs的特征是在两个LBD结构域中存在11个富含半胱氨酸的LDLRA重复,其中在第一和第二LBD结构域中分别为4个和7个重复。但是,LDLRA重复序列的数量和排列方式与其他昆虫顺序有很大不同。蜚蠊目和双翅目中有5和8个LDLRA重复序列,膜翅目中有2个、4个和8个重复序列,鞘翅目有8个重复序列[44-45]。与鳞翅目昆虫VgRs相比,荔枝蒂蛀虫C.sinensis在2个EGF前体结构域中的EGF/钙结合样重复序列的数量和排列都不同。通常,在第1个EGF前体结构域中出现了2个或3个类似EGF的重复序列,但在荔枝蒂蛀虫VgRs中多了1个额外的重复序列[46]。本研究在荔枝蝽中发现,T.papi VgRs包含了8个LDLRA、3个EGF、7个LY和1个GRAM结构域,这与半翅目昆虫的结构域较为类似。
本研究获得的4个基因,均具有典型的保守结构域,是典型的昆虫vg和VgRs基因,通过构建两个系统进化树,推断了3个荔枝蝽vg基因和1个荔枝蝽VgRs基因与其他目昆虫Vgs和VgRs的进化关系。从进化关系来看,vg和VgRs基因的分子进化与物种之间的进化关系较为匹配,不论是荔枝蝽vg基因还是荔枝蝽VgRs基因均与半翅目昆虫较好地聚集在一起,从荔枝蝽vg基因进化树来看,先后与蝽科、负蝽科、盲蝽科昆虫聚集在一个大支上,荔枝蝽VgRs基因进化树也很好地支持这一结果,这一结论也很好地说明荔枝蝽的vg和VgRs基因具有较为保守的功能。
同时,发现荔枝蒂蛀虫C.sinensis的Vg2基因在分子进化水平上没有和鳞翅目其他昆虫显示出更为接近的关系,反而是聚集在与荔枝蝽更为接近的半翅目分支上,其与绿盲蝽的Vg2和荔枝蝽的Tpapi Vg2在进化关系上更为接近,但在VgRs基因支序图中并没有和半翅目在相近的分支,根据vg基因的功能特征,推测其原因可能是在长期的进化过程中,由于昆虫长期取食专一性宿主,在遗传变异的过程中,对生殖相关的卵黄原蛋白造成了相应的结构变异造成的。VgRs基因由于具有极强的卵巢特异性,所以可能在昆虫生殖系统的进化中由于不同目昆虫生殖方式及转运物质的需求不同,从而产生了不同的基因结构变异。因此,这些基因可能的功能,还需要进一步的深入研究。
卵黄原蛋白的结构、合成、摄取过程与激素的调控激励是昆虫生理学研究的热点之一,昆虫卵黄原蛋白基因存在着明显的分子多态性,同一种昆虫也存在几个编码卵黄原蛋白的基因,因此,常常被作为分子进化研究的对象,同时,卵黄原蛋白作为昆虫体内的贮藏蛋白,为胚胎发育提供营养,这为害虫防治提供了新的思路,4种杀虫剂对荔枝蒂蛀虫C.sinensis产卵的亚致死作用的研究结果表明,在测试的各种杀虫剂中,对荔枝蒂蛀虫的毒性各不相同,毒死蜱的LCso为0.23 mg·kg-l、p一氯氰菊酯的为20.00 mg·kg-l、苯甲依氨菌素( EB)对产卵率,存活率和卵巢有明显的负影响,LC30剂量下荔枝蒂蛀虫C.sinensis的发育和交配率有显著的影响[46]。进一步的研究表明,在EB暴露后24、48和72 h,CsVg和CsVgR的转录水平受到不同程度的损害,这一结果与亚致死浓度EB-下荔枝蒂蛀虫的减少是一致的[46]。一些杀虫剂可以通过破坏昆虫的脂肪体,从而抑制vg的合成,如印楝素处理可以导致溪岸蚰嫂Labidurariparis脂肪体细胞解体[47],硫丹处理也会破坏剑角蝗Poekilocerus pictus的脂肪体[48],同样,SfVg和SfVgR沉默抑制了白背飞虱S.furcifera的卵巢发育、产卵数和孵化率。噻虫嗪LCio显著抑制了SfVg-like和SfVgR的表达。相反,三唑磷LC 25显著促进了白背飞虱Sfurcifera中SfVg、SfVg-1ike和SfVgR的表達,并增加了卵黄蛋白原的含量。因此,杀虫剂可以通过改变SfVg和SfVgR的表达来调节白背飞虱的繁殖,从而影响白背飞虱Sfurcifera的种群密度。这些发现有助于加深对杀虫剂对害虫繁殖和复发的影响的分子机制的理解奠定基础,可以说,昆虫的vg基因,已成为潜在的害虫控制和鉴定的新靶点[49]。
vg基因的调节在转录水平上直接受激素控制。参与vg基因转录的激素是保幼激素(JH)、蜕皮类固醇和一些神经肽。总体上根据vg基因转录的激素调节系统,可以将昆虫分为3类:(i)仅使用JH进行vg基因转录的昆虫,如鞘翅目和不完全变态昆虫;(ii)同时需要JH和蜕皮类固醇来调节vg的昆虫,如双翅目昆虫;( iii)需要JH、蜕皮类固醇和其他激素来调节其生殖生物学的昆虫,如鳞翅目昆虫。然而为什么昆虫物种在使用不同的激素来控制其生殖生理方面会发生分歧尚不清楚[25]。本研究获得了vg和VgRs基因在荔枝蝽不同组织中的时空表达情况,均显示其在卵巢和雌虫脂肪体中的高效高表达的现象,这与其他的昆虫的表达模式类似,所不同的是,T papi Vg2和T.papi Vg3在卵巢中并没有出现显著的高表达趋势,在雌虫脂肪体中,呈现特异的高表达特性。这从另一个角度说明,这两个基因在结构域产生变异之后,功能亦随之发生了变化,这些现象的发现,为更好地理解荔枝蝽Tpapillosa的产卵繁殖行为提供了基因层面的理论基础,同时亦可为荔枝蝽防治的靶点筛选提供一些有益的思路。
参考文献:
[1] 陈景耀,柯冲,陈菁瑛荔枝鬼帚病的初步调查及传病试验[J]植物病理学报,1992 (1):26
CHEN J Y,KE C, CHEN J Y A preliininary study on the incidenceand transinission of litchi witches broom [J] Acto PhyopathologicaSinica. 1992 (1) 26 (in Chinese)
[2] 许长藩,陈景耀,夏雨华,等荔枝蝽传播龙眼鬼帚病的研究[J]植物病理学报,1994,2(2):60-64
xu C F,CHEN J Y,XIA Y H,et al On transmission of Longyanwitches broom byTessaratoma papillosa(Drury)[J].ActaPhytopathologico Sinica,1994,2 (2):6064. (in Chinese)
[3] 黎荣欣,赵冬香,王玉沽,等荔枝蝽防治研究进展[J]热带作物学报,2013,34 (1):195200
LI R X,ZHAO D X.WANG Y J et al Research progress ofcontrolling Tessaratoma popillosa(Drury) [J]. Chinese Journal ofTropical Crops. 2013. 34(1):195200.( in Chinese)
[4] AMDAM G v,PAGE JR R E,FONDRK M K,et al Hormoneresponse to bidirectional selection on social behavior[J]Evolution&Development. 2010. 12(5):428436
[5]戈林泉,吴进才.昆虫卵黄蛋白及其激素调控的研究进展[J].昆虫知识,2010,47 (2):236-246
GE L Q,WU J C Research progress in insect vitellin and its hormoneregulation EJl. Chinese Bulletin of Entomology,2010,47(2):236246. (in Chinese)
[6] MATOZZO v,GAGNEF MARIN M G,et al_Vitellogenin as abiomarker of exposure to estrogenic compounds in aquaticinve11ebrates:AreⅥew[J].Environment International,2008. 34 (4):531-545
[7] TUFAIL M TAKEDA M Molecular characteristics of insectvitellogenins[J]Journal of Insect Physiology,2008. 54 (12):14471458
[8] TUFAIL M TAKEDA M Insect vitellogenin/lipophorin receptors:Molecular structures. role in oogenesis, and regulatoryinechanisms[J]Journol of Insect Physiology,2009,55 (2):87103
[9] TUFAIL M. TAKEDA M Molecular cloning. characterization andregulation of the cockroach vitellogenin receptor during oogenesis EJl .Insect Molecular Biology. 2005. 14 (4) : 389401 .
[IO] CIUDAD L. PIULACHS M D. BELLES X Systemic RNAi of thecockroach vitellogenin receptor results in a phenotype slmllar to thatof the Drosopbila yolkless mutaIlt [J]. The FEBS Journal. 2006.273 (2) : 325335.
[11] LIU Q N, ZHU B J. LIU C L. et al. Characterization of vitellogeninreceptor (VgR) from the Chinese oak silkworm. Antheraeo pernyi [J].Bulletin of lnsectology, 2011. 64 (2) : 167-174
[12] QIAN C. FU W W. WEI G Q. et al. Identification and expressionanalysis of vitellogenin receptor from the wild silkworm. Bombyxmondarina [J]. Archives of Insect Biocbemistry and Physiology. 2015.89 (4) : 181-192
[13] WEINSTOCK G M. ROBINSON G E. GIBBS R A. et al Insights intosocial insects from the genome of the honeybee Apis mellifera [J].Nature. 2006. 443 (7114): 931-949
[14] CHO K H. RAIKHEL A S Organizanon and developmentalexpression of the mosquito vitellogenin receptor gene [J]. InsectMolecular Biology. 2001. 10 (5): 465474
[15] LU K. SHU Y. ZHOU J. et al. Molecular characterization and RNAinterference analysis of vitellogenin receptor from Niloparvata lugens(Stal) [J]. Journol of lnsect Physiology, 2015. 73 : 20-29
[16] BROWN M S. GOLDSTEIN J L. A receptor - mediated pathway forcholesterol hoineostasis (Nobel lecture) [J]. Angeivandte ChemieInternotional Edition in English. 1986. 25 (7): 583602
[17] SAPPINGTON T W. RAIKHEL A S Molecular characteristics ofinsect vitellogenins and vitellogenin receptors [J]. Insect Biocbemistryand Molecular Biology. 1998. 28 (5-6) : 277300
[18] TUFAIL M. TAKEDA M Vitellogenin of the cockroach, Leucophaeamaderae: Nucleotide sequence. structure and analysis of processing inthe fat bodv and oocytes [J]. Insect Biochemistry and MolecularBiology, 2002. 32 (ll) : 1469-1476.
[19] HIRAI M. WATANABE D. KIYOTA A. et al Nucleotide sequence ofvitellogenin mRNA in the bean bug, Riptortus clavatus: Analysis ofprocessing in the fat bodv and ovary [J]. Insect Biochemistry andMolecular Biology. 1998. 28 (8): 537-547
[20] RAIKHEL A S. DHADIALLA T S Accumulation of yolk proteins inmsect oocytes EJl. Annual Review of Entomology. 1992. 37: 217-251.
[21] SNIGIREVSKAYA E S. RAIKHEL A S. Receptor-mediatedendocytosis of yolk proteins iii insect oocytes[J] Progress invitellogenesis[J]. Reproductive biology of invertebrates. 2005. 12(PartB): 199-228.
[22] ROEHRKASTEN A. FERENZ H J. Role of the lysine and arginineresidues of vitellogenin in high affmity binding to vitellogeninreceptors in locust oocyte membranes [J]. Biochimica et BiopbysicoActa. 1992. 1133 (2): 160-166的研究[J].昆蟲学报,1973. 16 (l): 15-24
ZHANG W Q, LIU X Q. Studies on the seasonal changes of thephysical properties and the reducing power of the haemolymph of thelvchee stinkbug,Tessorotoma papillsoa drury (Heiniptera:Pentatomidae) [J]. Acto entomologico sinica. 1973.16 (I) : 1524. (in Chinese)
[24]佘春仁.潘蓉英 .占德祥 .等 .利用平腹小蜂防治荔若枝技术问题探时 [J].福建农业大学学报. 1997. 26 (4): 441-445.
SHE C R. PAN R Y. GU D X. et al. Several technological problems ofapplying Anastatus sp. to control Tessaratoma papillosa [J]. Journalof Fujion Agricultural University. 1997, 26 (4): 441-445 (inChinese )
[25]佘春仁,潘蓉英荔枝蝽的解剖及其在测报的应用[J].福建学院学报,1993(1):59-63
SHE C R. PAN R Y. Svstematic dissection for forecasting the pestTessaratoma popillosa Drury [J].Journal of Fujian Agriculturalcollege. 1993 (l) : 59-63 (in Chinese)
[26] LIVAK K J. SCHMITTGEN T D Analysis of relative gene expressiondata using real-time quantitative PCR and the 2- AACI method [J].Methods. 2001. 25 (4) : 402-408.
[27] TUFAIL M. NAGABA Y. ELGENDY A M. et al. Regulation ofvitellogenin genes in insects [J]. Entomological Science, 2014.17(3) : 269-282.
[28] UPADHYAY S K. sINGH H. DIXIT S. et al. Molecularcharacterization of vitellogenin and vitellogenin receptor of Bemisiatabaci [J]. PLoS One. 2016. ll (5) : e0155306.
[29] HU K. TIAN P. TANG Y. et al. Molecular characterization ofvitellogenin and its receptor in Sogotella furcifera, and their functionm oocvte maturation [J]. Frontiers in Physiology, 2019.10: 1532
[30] ROBERTSON J L. PREISLER H K. Pesticide bioassays witharthropods[M]. CRC Press. 1992: 127
[31] THOMPSON J R. BANASZAK L J. Lipid-protem mteractions mlipovitellin [J]. Biochemistry. 2002, 41 (30) : 93989409.
[32] HUSAIN M. RASOOL K G. TUFAIL M. et al RNAi-mediatedsilencing of vitellogenin gene curtails oogenesis in the almond mothCadra cautella [J]. PIoS one. 2021. 16(2): e0245928.
[33] MORANDIN C. HAVUKAINEN H. KULMUNI J, et al. Not only foregg yolk -fimctional and evolutionary insights from expression.selection. and stmctural analyses of Formica ant vitellogenins [J].Molecular Biology and Evohition. 2014. 3l (S): 2181-2193.
[34] PIULACHS M D. GUIDUGLI K R. BARCHUK A R. et al Thevitellogenin of the honey bee. Apis mellifera: Structural analysis of thecDNA and expression studies [J]. Insect Biochemistry and MolecularBiology. 2003.33(4): 459-465
[35] CORONA M. LIBBRECHT R. WURM Y. et al. Vitellogeninunderwent subfunctionalization to acquire caste and behavioralspecific expression in the harvester ant Pogonomyrmex borbatus [J].PLoS Genetics. 2013. 9(8):el003730.
[36] YANO K. SAKURAI M T. WATABE S. et al. Structure andexpression of mRNA for vitellogenin in Bombyx mori[J]. Biochimicaet Biophysica Acta. 1994. 1218(l):1-10
[37] MARTIN D. PIULACHS M D. COMAS D. et al. Isolation andsequence of a partial vitellogenin cDNA from the cockroach. Blottellagermanica (L) (DicO-optera, Blattellidae). and characterization of thevitellogenin gene expression [J]. Archives of Insect Biochemistry andPhysiology. 1998. 38 (3):137-146
[38] TUFAIL M. HATAKEYAMA M. TAKEDA M. Molecular evidencefor two vitellogenin genes and processing of vitellogenins in theAmerican cockroach, Periplaneta americano [J]. Arcbives of InsectBiochemistry and Physiology, 2001. 48(2): 7280
[39] SHEN Y. CHEN Y Z. LOU Y H. et al Vitellogenin and vitellogenin-like genes in the brown planthopper [J]. Frontiers in Physiology.2019. 10: 1181.
[40] ROMANS P. TU Z. KE Z. et al. Analysis of a vitellogenin gene of themosquito, Aedes aeglpti and comparisons to vitellogenins from otherorgaiusms[J]. Insect Biochemistry and Molecular Biology. 1995.25(8) : 939-958
[41] SMITH C D. ZIMIN A. HOLT c, et al. Draft genome of the globallywidespread and invasive Argentine ant (Linepithema bumile)[J]Proceedings of the National Academy of Sciences. 2011. lO8(14):5673-5678.
[42] COLETTA A. PINNEY J W. SOLIS D Y W. et al. Low-complexityregious within protein sequences have position-dependent roles EJl .BMC Sy'stems Biology. 2010, 4 (l): 1-13
[43] LYNCH M. CONERY J S The evolutionarv fate and consequences ofduplicate genes [J]. Science. 2000. 290(5494) : 1151-1155
[44] CONG L. YANG W J. JIANG X Z. et al. The essential role ofvitellogenin receptor in ovary development and vitellogenin uptake inBoctrocera dorsalis (hendel)[J]. International Journal of MolecularSciences. 2015. 16 (8) : 18368-18383.
[45] ZHANG W. MA L. XIAO H. et al Molecular characterization andfuiiction analvsis of the vitellogenin receptor from the cottonbollworm. Helicoverpa armigera (Hubner) (Lepidoptera. Noctuidae)[J]. PIoS ONE. 2016. 11 (5): e0155785.
[46] YAO Q. XU S. DONG Y Z. et al. Characterization of vitellogenin andvitellogenin receptor of Conopomorpba sinensis Bradlev and theirresponses to sublethal concentrations of insecticide [J]. Frontiers inPhysiology. 2018. 9:1250.
[47] SAYAH F. FAYET C. IDAOMAR M. et al. Effect of azadirachtin onvitellogenesis ofLabidura riparia (Insect Dermaptera) [J]. Tissue andCell. 1996. 28 (6):741-749
[48] AMIR M Histopathological effect of some toxicants on the femalereproductive system of Sarcophago ruficornis Fabricius (Diptera:Sarcophagidae) [J]. Cibtech Journal of Zoology ISSN, 2014. 3(2):231938831
[49] ZHOU C. YANG X B. YANG H. et al Effects of sublethalconcentrations of insecticides on tlie fecundity of Sogatello furcifero(Hemiptera: Delphacidae) via the regulation of vitellogenin and itsreceptor [J]. Journal oflnsect Science. 2020. 20(5): 14.
收稿日期:2021-0401初稿;2021-0505修改稿
作者简介:程林( 1996-),男,硕士研究生,研究方向:昆虫生态与害虫综合治理( E-mail: 76782253l@qq.com)
通信作者:彭凌飛( 1982-),男,博士,讲师,研究方向:昆虫分类与系统学、害虫防治( E-mail: lingfeipeng@fafu edu.cn)
基金项目:福建省昆虫生态重点实验室开放课题(FIE201703)
