基于Pacbio第三代测序技术的厚朴基因组测序分析
2021-09-13尹彦棚丁乔娇罗加伟林新娜张敏彭成高继海
尹彦棚 丁乔娇 罗加伟 林新娜 张敏 彭成 高继海

摘 要:厚樸为著名的传统药用植物,归于木兰科、木兰属,于我国广泛种植,其树皮、根皮、枝皮、叶片、花、果实均能入药或食用。为获取厚朴全基因组序列信息,该文以厚朴叶片DNA为材料,采用Pacbio Sequel第三代测序技术构建厚朴全基因组数据库,并利用生物信息学方法对获得的核苷酸序列进行组装、功能注释以及进化分析研究。结果表明:(1)原始测序数据过滤后获得140.91 Gb三代数据,Read N50约为13 784 bp,经过组装得到厚朴基因组大小为1.68 Gb,Contig N50约为222 069 bp,单拷贝基因完整性为81.0%。(2)组装后的序列通过与NR、KOG、KEGG等功能数据库比对,共有98.40%的基因得到了功能注释,其中KOG功能注释结果发现厚朴的蛋白功能主要集中在一般功能预测、翻译后修饰、蛋白质转换、伴侣以及信号转导机制;GO功能分类表明厚朴的基因集中在细胞组分及生物学过程;KEGG分析发现厚朴参与代谢通路的基因占主要地位。(3)通过与葡萄、拟南芥、水稻、杨树、银杏、无油樟、茶树及牛樟基因组的比对分析,发现厚朴23 424个基因中有20 801个基因可以分类到12 129个家族,其中有515个基因家族为厚朴所特有,而厚朴与牛樟(樟科)亲缘关系较近,两者的分化时间约在122.5百万年前(mya)。该研究首次利用第三代测序技术对厚朴全基因组解析,有利于对其进一步进行深入的开发与利用,也为研究其他药用植物全基因组奠定了基础。
关键词:厚朴,基因组,第三代测序技术,基因注释,药用植物
中图分类号:Q943.2
文献标识码:A
文章编号:1000-3142(2021)08-1251-12
Abstract: Magnolia officinalis is a famous traditional medicinal plant, belonging to the Magnoliaceae family and Magnolia L. genus and being widely cultivated in China. Its barks, root barks, branch barks, leaves, flowers and fruits could be used as medicine or food. However, the whole genome information is little known for this plant species. In order to obtain the whole genome sequence information of M. officinalis, the leaf DNA was used as the material, and the third-generation sequencing technology of Pacbio Sequel was used to establish its nucleotide sequence database. Then genome assembly, function annotation and evolution analysis were carried out by bioinformatic methods. The results were as follows: (1) 140.91 Gb the third-generation data were obtained after the original sequencing data, with the Read N50 about 13 784 bp. The assembled M. officinals genome size was 1.68 Gb, contig N50 being about 222 069 bp, and the integrity of single-copy gene being 81.0%. (2) 98.40% of the genes from the assembled sequence got gene annotation after being compared with functional databases such as NR, KOG and KEGG. The result of KOG gene annotation was that the protein function of M. officinalis concentrated in the general functional prediction only, posttranslational modification, protein turnover, chaperones signal transduction mechanisms. GO functional classification indicated that the genes of M. officinalis concentrated on cell components and biological processes. KEGG analysis found that the M. officinalis genes mostly involved in metabolic pathways. (3) By comparative genomics analysis, the genomes of Vitis vinifera, Arabidopsis thaliana, Oryza sativa, Poplar trichocarpa, Ginkgo biloba, Amborella trichopoda, Camellia sinensis and Cinnamomum kanehirae were aligned. It was found that 20 801 of 23 424 genes in M. officinalis could be classified into 12 129 families, 515 gene families being unique to M. officinalis. The genetic evolution tree constructed from the genomes of the selected reference species pointed that the M. officinalis (Magnoliaceae) was closely related to Cinnamomum kanehirae (Lauraceae), and the divergence time between the two species was about 122.5 mya. It is the first time to use the third-generation sequencing technology to analyze the whole genome of M. officinalis in the study, which is conducive to its further development and utilization, and also provides the information for the study of the whole genome of other medicinal plants.
Key words: Magnolia officinalis,genome,the third-generation sequencing technology,gene annotation, medicinal plant
隨着人类基因组计划的完成,基因组测序技术的不断发展和逐渐成熟,尤其以第三代测序技术发展为单分子实时测序,这加速了植物全基因组研究的进程。基因组大小是指某个物种单倍体基因组的全部DNA碱基对数,是研究物种基因组学的基础。木兰科在植物进化及分类学中的地位属于比较原始的科,近年来关于木兰属物种叶绿体基因组测序在国内外研究较多,如李西文等(2012)、Li et al.(2013)通过454 FLX第二代高通量测序平台建立一种厚朴叶绿体基因组的标准测序流程用于区分厚朴及近缘物种,并利用测序平台获得了木兰属物种荷花玉兰(Magnolia grandiflora)的叶绿体全基因组序列,为其优良品种的选育、叶绿体基因工程、分子标记的开发和系统发育分析提供了有价值的信息;Cui et al.(2019)对厚朴同属物种天女木兰(M. sieboldii)进行叶绿体全基因组测序,获得了111个独特的基因,包括78个蛋白编码基因、29个tRNA 基因和4个rRNA 基因。
厚朴 (Magnolia officinalis) 为木兰科(Magnoliaceae)木兰属(Magnolia L.)植物,主产于四川东部、湖北西部等地,野生厚朴是我国二级保护植物(薛珍珍等,2019)。厚朴树皮、枝皮、根皮、芽等均可入药,在临床中广泛使用。此外,厚朴花大美丽,被列入保健食品名录,其种子可榨油,有明目益气之功效。同时厚朴作为道地药材,其主要活性成分是以厚朴酚及和厚朴酚代表的酚类,研究表明这两种活性成分具有良好抗菌、抗炎、抗肿瘤和抗病毒等药理作用(王立青等,2005)。查良平等(2015)通过转录组学研究了厚朴萜类化合物的生物合成途径,揭示其中的甲羟戊酸 (MVA)途径相关基因调控萜类次级代谢产物的合成机制;时小东等(2018)在此基础上深入研究厚朴次级代谢产物中苯丙素途径和萜类合成途径,获得了代谢途径中相关酶和基因的信息。
厚朴在自然环境下生长周期长,产量低,但市场需求量较大,所以人工繁殖培育的厚朴较多,种质资源丰富(张龙辉等,2013)。然而,当前关于厚朴的研究对其遗传信息、进化历程及性状形成等相关分子生物学基础缺乏认识和了解,导致厚朴的厚朴酚、和厚朴酚等核心次级代谢产物的合成调控机理尚有诸多不清楚,对其在分子辅助育种,发掘相关生长发育、抗病抗逆等优良性状基因等方面的问题也得不到有效解决,造成厚朴资源利用度低,开发不够深入。因此,本研究基于厚朴遗传基因组信息匮乏,初步对厚朴进行全基因组测序研究,获得的基因组信息将会进一步丰富厚朴遗传进化研究资料,为探索药用植物优良品种选育、有效成分的生物合成途径与调控机制及综合开发利用等奠定基础。
1 材料与方法
1.1 厚朴样品及DNA提取
厚朴植株选取于成都中医药大学药用植物园,采摘新鲜幼嫩无病害的叶片,蒸馏水清洗表面后,再使用75%乙醇清洗3次,擦干,-80 ℃冻存备用。
采用CTAB法(沙丽萍,2018)提取厚朴叶片DNA,步骤如下:(1)样品使用液氮研磨后分装至离心管;(2)向离心管中加入十六烷基三甲基溴化铵溶液(CTAB),65 ℃水浴1 h,10 800 r·min-1离心10 min,取上清;(3)离心后加入等体积氯仿∶异戊醇 (24∶1),充分混匀,4 ℃、10 800 r·min-1离心10 min,取上清,重复两次;(4)向上清中加入异丙醇和乙酸钠溶液,离心,弃上清;(5)加入75%乙醇,离心,弃上清;(6)晾干,加入TE缓冲液溶解,4 ℃保存备用。
1.2 文库构建及测序
使用g-TUBE剪切管打断厚朴DNA样品,对打断的DNA样品(5 mg)使用建库试剂盒(SMRTbell Template Prep Kit)进行损伤修复、末端修复及连接接头;对连接接头产物使用BluePippin Size-Selection System进行目的片段筛选,并通过AMpure PB磁珠进行纯化回收;回收产物使用损伤修复试剂盒(SMRTbell Damage Repair Kit)进行二次损伤修复,并对修复产物进行AMpure PB磁珠纯化回收;最终文库即二次损伤修复产物进行浓度(Qubit)及大小(Agilent 2100)的文库质量检测,即得到测序文库。采用第三代测序平台Pacbio Sequel进行单分子测序,原始数据进行评估、过滤后得到高质量的数据用于基因组组装与质量评估。
1.3 基因组组装及评估
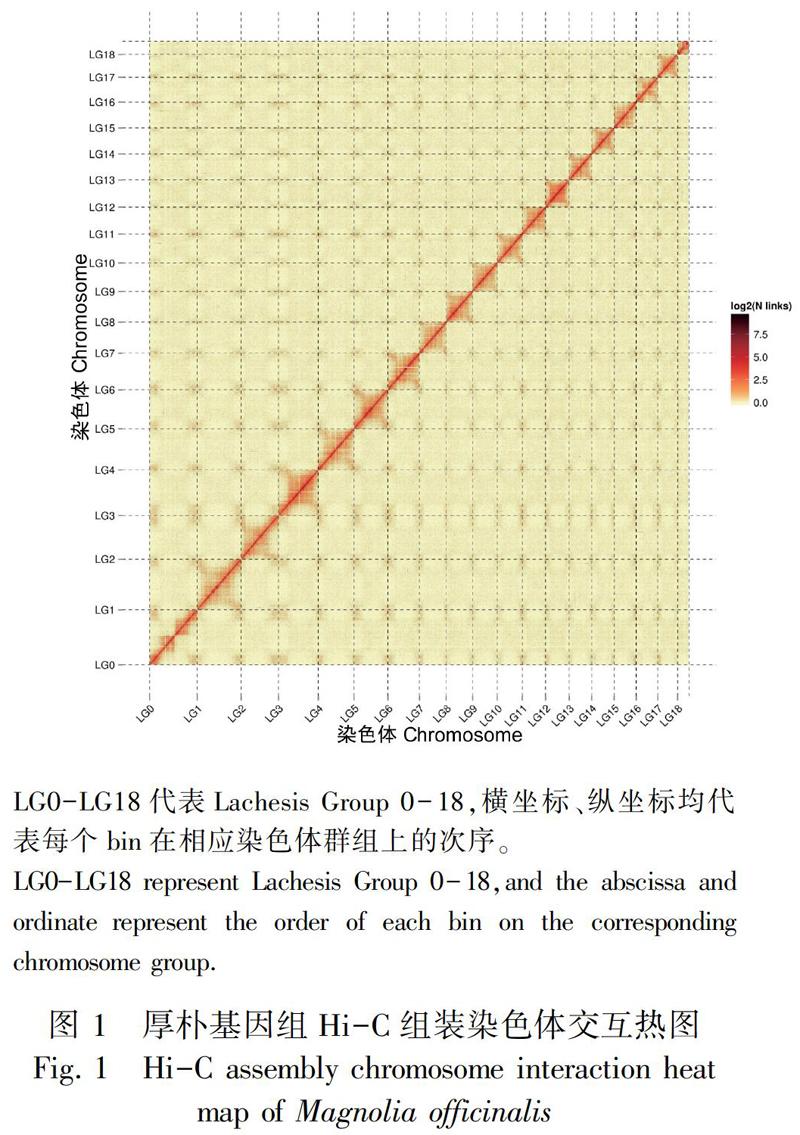
对PacBio测序平台产生的原始数据进行过滤低质量和短片段后,首先,利用Canu(Koren et al.,2017)软件对过滤后的数据进行初步组装;然后,采用LACHESIS(Belton et al.,2012)软件对初步组装后的序列进行群组的划分、排序和定向。将每个Scaffold按照等长50 kb打断,利用Hi-C(high-throughput chromosome conformation capture)技术(Marbout & Koszul,2015)重新组装,将无法还原为最初组装序列的位置列为候选错误区域,然后鉴定此区域中低Hi-C覆盖深度的位置即为错误点,从而完成对初步组装基因组的纠错,以提高基因组组装质量。对组装结果利用BUSCO v5.0(Simao et al.,2015)软件来评估组装基因组的完整性,与Embryophyta_odb10数据库中含有的植物1 614个保守的核心基因比对,并绘制互作热图来评估Hi-C组装结果。[LACHESIS软件使用具体参数为(1) CLUSTER MIN RE SITES=52;(2)CLUSTER MAX LINK DENSITY=2;(3)CLUSTER NONINFORMATIVE RATIO = 2;(4) ORDER MIN N RES IN TRUN=46;(5) ORDER MIN N RES IN SHREDS=42。]
1.4 序列预测
使用LTR FINDER v1.05(Zhao & Wang,2007)、RepeatScout v1.0.5(Price et al.,2005)、PILER-DF v2.4(Edgar & Myers,2005)软件,首先,基于结构预测和从头预测(Ab initio)的原理构建重复序列数据库,对构建好的重复序列库通过PASTEClassifier(Wicker et al.,2007)进行分类;然后,基于重复序列数据库Repbase(https://www.girinst.org/repbase/)合并作为最终的厚朴基因组的重复序列数据库;最后,通过RepeatMasker v4.0.6(Tarailo & Chen,2009)软件基于构建好的数据库对厚朴进行重复序列的预测。
基于从头预测(Ab initio)和同源物种预测(Homolog)两种原理对厚朴基因组进行基因预测,并对预测结果进行评估。首先,使用Genscan(Burge & Karlin,1997)、Augustus v2.4(Stanke & Waack,2003)、GlimmerHMM v3.0.4(Majoros et al.,2004)、GeneID v1.4(Blanco et al.,2007)、SNAP(version 2006-07-28) (Blanco et al.,2007)进行从头预测;然后,使用GeMoMa v1.3.1(Jens et al.,2016)进行基于同源物种的预测;最后,利用EVM v1.1.1整合上述方法得到的预测结果。同时针对非编码RNA预测,包括了microRNA、rRNA及tRNA等已知功能的RNA,分别基于Rfam(Griffithsjones et al.,2005)数据库和miRBase(Griffithsjones et al.,2006)数据库并利用Infenal 1.1(Nawrocki & Eddy,2013)进行rRNA和microRNA预测;利用tRNAscan-SE v1.3.1 (http://lowelab.ucsc.edu/tRNAscan-SE/)(Lowe & Eddy,1997)识别tRNA。
1.5 功能基因注释
对预测得到的基因序列与NR(Non-Redundant Protein Database)(Aron et al.,2011)、KOG(EuKaryotic Orthologous Groups)(Tatusov et al.,2001)、KEGG(Kyoto Encyclopedia of Genes and Genomes)(Minoru & Susumu,2000)、TrEMBL(Boeckmann et al.,2003)等功能数据库做BLAST v2.2.31(Altschul et al.,1990)比对(设置比对筛选阈值e-value<1e-5),得到基因功能注释。基于NR数据库比对结果,应用软件Blast2GO(Conesa et al.,2005)进行GO(Dimmer et al.,2012)数据库的功能注释。
1.6 比较基因组学分析
拟南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、杨树(Populus trichocarpa)、银杏(Ginkgo biloba)、无油樟(Amborella trichopoda)、茶树(Camellia sinensis)、牛樟(Cinnamomum kanehirae)]的蛋白序列比对(NCBI数据库https://www.ncbi.nlm.nih.gov/),基于序列比对结果,对已知基因的序列和结构进行比较,分析物种间的进化以及物种特有基因的分类。
使用OrthoMCL(Li et al.,2003)软件(参数:Pep_length: 10, Stop_coden: 20, PercentMatchCutoff: 50, EvalueExponentCutoff: -5, Mcl: 1.5 #1.2-4.0)对上述9个物种的蛋白序列进行家族分类,寻找厚朴基因组特有的基因家族。先利用OrthoMCL聚类的结果提取单拷贝蛋白序列,再将单拷贝蛋白序列使用Muscle(http://www.ebi.ac.uk/Tools/msa/muscle/)软件进行序列比对,使用PHYML(Stéphane et al.,2010)软件(参数:-gapRatio 0.5, -badRatio 0.25, -model HKY 85, -bootstrap 1 000)通過ML(最大似然法)构建进化树,研究物种间的进化关系。利用Timetree(http://www.timetree.org/)查询已有物种之间的化石时间,并通过mcmctree(http://abacus.gene.ucl.ac.uk/software/paml.html)估算出物种间的分化时间。采用MCScanX(Wang et al.,2012)软件分别对自身(参数:-s 10 ,-b 1, 其他参数默认)及与近缘物种牛樟(Cinnamomum kanehirae)(参数:-s 10 ,-b 2, 其他参数默认)基因组做共线性分析,统计相应的共线性基因数目和共线性区块(Block)数目。
2 结果与分析
2.1 基因组测序
通过三代测序平台对厚朴叶片进行全基因组测序,对原始数据的reads质量值进行初步过滤,去掉低质量和短片段的reads,统计得到140.91 Gb三代原始数据,Read N50为13 784 bp,最长reads的长度为128 492 bp,平均长度为8 654 bp,测序质量符合后续组装要求。
2.2 基因组组装及评估
借助Canu软件对厚朴的初步组装结果见表1,初步组装的序列经过Hi-C纠错组装后基因组大小约为1.68 Gb,Contig N50为222 069 bp,最长的Contig为2 700 203 bp,GC含量为40.65%。Hi-C组装后其中共有1.67 Gb的序列长度的基因组序列被定位到19条染色体上,占比99.66%,而对应的序列数目为11 470条,占比99.20%。在定位到染色体上的序列中,能够确定顺序和方向序列长度为1.53 Gb,占定位染色体序列总长度的91.21%,对应的序列数目为8 689条,占定位染色体序列总数目的75.75%。
组装后的基因组采用BUSCO软件评估,在组装的基因中共找到1 391个完整的BUSCO基因,其中完整单拷贝的1 307个,Fragmented BUSCO 75个基因,有148个基因在Embryophyta_odb10数据库中没有找到,BUSCO评估基因组完整度为86.2%,说明组装结果较完整。通过Hi-C辅助组装热图分析(图1),厚朴19个染色体分组可以明显区分,且每一分组对角线的交互强度信号要高于非对角线位置,说明Hi-C组装的染色体结果中邻近的序列间(对角线位置)交互强度高,而非邻近的序列之间(非对角线位置)的交互信号强度弱,证明基因组组装效果较好。
2.3 基因预测结果
利用RepeatMasker v4.0.6软件进行重复序列预测得到包含1.37 Gb重复序列的厚朴基因组,占比81.60%。其中长散在重复序列(LINE)得到重复序列数目为450 863条,占比 8.47%;短散在重复序列(SINE)数目为18 530条,占比0.2%;长末端重复序列(LTR)数目为997 318 条,占比44.04%;末端反向重复序列(TIR)数目为145 539条,占比 4.5%;简单重复序列(SSR)数目为10 506条,占比0.47%。
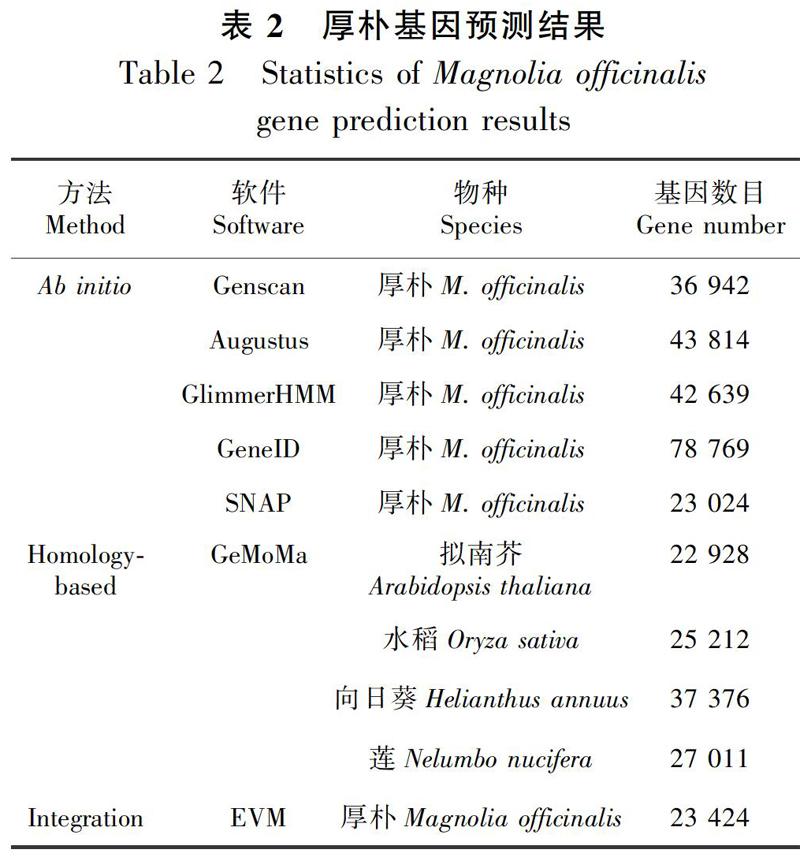
对厚朴的基因预测结果(表2)显示,获得了23 424个蛋白编码基因及1 096个非蛋白编码基因,包括72个microRNA基因,575个tRNA基因和449个rRNA基因。
2.4 基因功能注释与分析
通过KOG功能注释(图2),厚朴基因组的13 845个基因获得注释,占预测到的总基因数的59.11%。从图中可以看出,厚朴的蛋白功能主要集中在“翻译后修饰、蛋白质转换、伴侣”(posttranslational modification, protein turnover, chaperones)(O),占比10%;“信号转导机制”(signal transduction mechanisms)(T)占比9%,其次为“碳水化合物转运和代谢”(carbohydrate transport and metabolism)(G)与“转录”(transcription)(K)等功能,各占比5%。“一般功能预测”(general function prediction only)(R)占比22%。这些基因差异性表达可以对今后深入探究厚朴在进化过程中对环境响应的机制提供数据支持。
通过厚朴基因组GO注释(图3),共有13 438个基因具有GO注释功能,占预测到的总基因数的57.37%。功能分布在“细胞”(cell)、“结合”(binding)、“催化活性”(catalytic activity)、“细胞过程”(cellular process)、“代谢过程”(metabolic process)等功能的基因占据优势地位,而在整个分类中细胞组分占33%,分子功能占21%,生物学过程占45%。由此可见,初探到厚朴的基因主要富集在生物学过程中的代谢过程。
通过KEGG通路注释(图4),对厚朴的8 253个基因进行了通路注释,占预测到的总基因数的35.23%。其注释结果分别为5.40%的“细胞过程”(cellular process)、4.50%的“环境信息处理”(environmental information processing)、29.85%的“遗传信息处理”(genetic information processing)、55.09%的“代谢” (metabolism)、5.16%的“机体系统”(organismal systems)。通过KEGG的通路注释进一步了解厚朴基因在生物学过程上的功能,其中参与代谢通路上的基因占主要地位,淀粉和蔗糖代谢(ko00500)、氨基酸的生物合成(ko01230)及碳代谢(ko01200)为主要的代谢通路。
2.5 比较基因组学分析
通过对厚朴与葡萄、拟南芥、水稻、杨树、银杏、无油樟、茶树及牛樟的蛋白序列比对,发现在预测得到的23 424个基因中有20 801个基因可以分类到12 129个家族,其中有515个基因家族是厚朴所特有的,蛋白预测分类结果见图5和表3。
为进一步确定厚朴的种属关系,以单拷贝蛋白序列进行比较分析,选择上述8个已知基因组信息的物种,构建出遗传进化树(图6),结果表明厚朴与牛樟聚为一支,两者间物种亲缘关系较近。根據物种分化时间分析(图7),两者分化时间约在122.5百万年前(mya)。通过绘制出的共线性图(图8),比较厚朴与牛樟基因组的同源性。比对结果的共线性图中的每一条线代表同源基因之间的连线,没有线条的空白区域代表没有比对上的序列,代表物种之间存在差别的基因区域。从图中看出两者基因组片段能比对上的片段较少,发现两者基因组存在较大差异。
3 讨论与结论
基因组测序技术和生物信息学技术的进一步发展、测序成本的降低和分析方法的完善极大地推动了厚朴这种非模式药用植物的基因组测序研究。目前常用的测定基因组大小的方法有流式细胞术(林瀚等,2019)、第二代高通量测序技术(李西文等,2012)及已发展至第三代的单分子测序技术(柳延虎等,2015),本研究通过第三代测序技术测得的厚朴全基因组大小约为1.68 Gb,与叶林江等(2015)通过流式细胞术检测的木兰属物种凹叶厚朴 (M. officinalis subsp.biloba) (2n=2x=38)的基因组大小为1.59 Gb相符合。物种的基因组大小与其倍性水平和染色体数目存在一定的正相关性(叶林江等,2015),王跃华等(2005)利用厚朴新生的愈伤组织制作染色体切片,显微观察结果显示其染色体数为2n=38,上述提到的凹叶厚朴也属于二倍体,染色体数为38条,表明本次测得的厚朴基因组大小符合其倍性水平和染色体数目。
基因组功能注释是对物种功能基因解析的一重要方面。本研究通对厚朴的基因组功能注释分析发现,在GO功能注释中厚朴的基因集中在生物学过程中 “代谢过程”,这与KEGG通路注释的结果显示在“代谢通路”上的基因占主要地位相符,其中淀粉和蔗糖代谢、氨基酸的生物合成及碳代谢为主要的代谢通路。杨旭等(2019)利用Illumina高通量测序技术对厚朴根、茎、叶不同组织进行转录组分析,功能注释的结果显示厚朴的主要生物代谢途径为碳水化合物代谢、氨基酸代谢和能量代谢,与本研究得到的厚朴代谢通路注释结果相对应。厚朴基因组和转录组分析相结合,有利于厚朴的功能基因的进一步发掘和分析。
目前,关于厚朴等药用植物研究较多还是其叶绿体基因组。由于核基因组包含丰富的遗传信息,所以基因组很大,组成结构复杂,且多倍性与高度的重复序列片段也给测序带來很大困难(陈勇等,2014)。本研究获得的厚朴全基因组序列是较高质量的序列,这也是木兰属物种中首个核DNA全基因组序列,为后续分析研究木兰属甚至木兰科物种起源和进化关系提供了参考基因组序列。厚朴全基因组测序的完成,是进行药物植物的分子辅助育种的重要一步,基于基因组学、蛋白组学和种质信息等相关数据,利用生物信息学方法分析,最终筛选出最佳基因型和育种方案(马小军和莫长明,2017),这对在临床上需求较大的药用植物是一个新颖的培育方法。全基因组序列也为后续研究厚朴的功能基因组学(王勇波等,2009)提供了数据支撑。通过转录组学和代谢组学对药用植物的次生代谢产物合成的关键酶鉴定和代谢途径解析,并筛选出关于生长发育、抗病抗逆等优良性状基因位点,是解决对厚朴资源开发利用不够深入的一个有效策略。本研究通过对厚朴的全基因组测序可以从分子层面加深对物种的认识,为其他药用植物的全基因组测序提供参考,也为今后进一步开发利用中药资源提供相关分子生物学基础,且对促进中药材的现代化具有重要作用。
参考文献:
ALTSCHUL SF, GISH W, MILLER W,et al., 1990. Lipman DJ: Basic local alignment search tool [J]. J Mol Biol, 215: 403-410.
ARON MB, SHENNAN L,ANDERSON JB, et al., 2010. CDD: A conserved domain database for the functional annotation of proteins [J]. Nucl Acid Res, 39(Suppl.1): D225-D229.
BELTON JM, MCCORD RP, GIBCUS JH, et al., 2012. Hi–C: A comprehensive technique to capture the conformation of genomes [J]. Methods, 58(3): 268-276.
BLANCO E, GENIS P, RODERIC G, 2007. Using geneid to identify genes [J]. Current Protocols, 18(1): 4-3.
BOECKMANN B, BAIROCH A, APWEILER R, et al., 2003. The SWISS-PROT protein knowledgebase and its supplement TrEMBL in 2003 [J]. Nucl Acid Res, 31(1): 365-370.
BURGE C, KARLIN S, 1997. Prediction of complete gene structure in human genomic DNA [J]. J Mol Biol, 268(1): 78-94.
CONESA A, GOTZ S, GARCIA-GOMEZ JM, et al., 2005. Blast2GO: A universal tool for annotation, visualization and analysis in functional genomics research [J]. Bioinformatics, 21(18): 3674-3676.
CUI YF, LI C, ZHANG Y, et al., 2019. The complete chloroplast genome of Siebolds magnolia: Magnolia sieboldii (Magnoliaceae), a highly ornamental species with attractive aromatic flowers [J]. Conserv Genet Resour, 11(3): 299-301.
CHEN Y, LIU YS, ZENG JG, 2014. Progress in plant genome sequencing [J]. Life Sci Res, 18(1): 66-74. [陈勇, 柳亦松, 曾建国, 2014. 植物基因组测序的研究进展 [J].生命科学研究, 18(1): 66-74.]
DIMMER, EMILY C, 2012. Eberhardt R: The UniProt-GO annotation database in 2011 [J]. Nucl Acid Res, 40(D1): 565-570.
EDGAR RC, MYERS EW, 2005. PILER: Identification and classification of genomic repeats [J]. Bioinformatics, 21(Suppl. 1): i152-i158.
GRIFFITHSJONES S, GROCOCK RJ, DONGEN SV, et al., 2006. miRBase: MicroRNA sequences, targets and gene nomenclature [J]. Nucl Acid Res, 34(Suppl. 1): 140-144.
GRIFFITHSJONES S, MOXON S, MARSHALL M, et al., 2005. Rfam: Annotating Non-Coding RNAs in complete genomes [J]. Nucl Acid Res, 33(Database issue): D121-124.
JENS K, MICHAEL W, ERICKSON JL, et al., 2016. Using intron position conservation for homology-based gene prediction [J]. Nucl Acid Res, 44(9): e89-e89.
KOREN S, WALENZ BP,BERLIN K, et al., 2017. Canu: scalable and accurate long-read assembly via adaptive k-mer weighting and repeat separation [J]. Genome Res, 27(5): 722-736.
LI XW, GAO HH, WANG YT, et al., 2012. High throughput sequencing and structural analysis of the whole chloroplast genome of Evergreen magnolia [J]. Chin Sci: Life Sci, 42 (12): 947-956. [李西文, 高歡欢, 王一涛, 等, 2012. 荷花玉兰叶绿体全基因组高通量测序及结构解析 [J]. 中国科学: 生命科学, 42(12): 947-956.]
LI XW, GAO H, WANG Y, et al., 2013. Complete chloroplast genome sequence of Magnolia grandiflora and comparative analysis with related species [J]. Sci Chin Life Sci, 56(2): 189-198.
LI XW, HU ZG, LIN XH, et al., 2012. Whole chloroplast genome sequencing of Magnolia officinalis based on 454 FLX high throughput technology and its application [J]. Acta Pharm Sin, 47(1): 124-130. [李西文, 胡志刚, 林小涵, 等, 2012. 基于454FLX高通量技术的厚朴叶绿体全基因组测序及应用研究 [J]. 药学学报, 47(1): 124-130.]
LI L, STOECKERT CJ, ROOS DS, 2003. OrthoMCL: Identification of ortholog groups for eukaryotic genomes [J]. Genome Res, 13(9): 2178-2189.
LIN H, HAN XW, LAN SR, et al., 2019. Determination of genome size of two orchids based on flow cytometry [J]. J For Environ, 39(6): 616-620. [林瀚, 韩晓文, 兰思仁, 等, 2019. 基于流式细胞技术两种兰属植物基因组大小的测定 [J]. 森林与环境学报, 39(6): 616-620.]
LIU YH, WANG L, YU L, 2015. Principle and application of single molecule real-time sequencing [J]. Genetics, 37(3): 259-268. [柳延虎, 王璐, 于黎, 2015. 单分子实时测序技术的原理与应用 [J]. 遗传, 37(3): 259-268.]
LOWE TM, EDDY SR, 1997. tRNAscan-SE: A program for improved detection of transfer RNA genes in genomic sequence [J]. Nucl Acid Res, 25(5): 955-964.
MAJOROS WH, PERTEA M, SALZBERG SL, 2004. TigrScan and GlimmerHMM: Two open source ab initio eukaryotic gene-finders [J]. Bioinformatics, 20(16): 2878-2879.
MARBOUTY M, KOSZUL R, 2015. Metagenome analysis exploiting high-throughput chromosome conformation capture (3C) data [J]. Trends Genet, 31(12): 673-682.
MINORU K, SUSUMU G, 2000. KEGG: Kyoto Encyclopedia of Genes and Genomes [J]. Nucl Acid Res, 28(1): 27-30.
MA XJ, MO CM, 2017. Prospects for molecular breeding of medicinal plants [J]. Chin J Trad Chin Med, 42(11): 2021-2031. [马小军, 莫长明, 2017. 药用植物分子育种展望 [J]. 中国中药杂志, 42(11): 2021-2031.]
NAWROCKI EP, EDDY SR, 2013. Infernal 1.1: 100-fold faster RNA homology searches [J]. Bioinformatics, 29(22): 2933-2935.
PRICE AL, JONES NC, PEVZNER PA, 2005. De novo identification of repeat families in large genomes [J]. Bioinformatics, 21(Suppl. 1): i351-i358.
SHA LP, 2018. Examples of CTAB method, SDS method and salting-out method for crude extraction of plant DNA [J]. Teachnol Midd School Biol, (21): 65-67. [沙丽萍, 2018. 例谈植物DNA粗提取的CTAB法、SDS法与盐析法 [J]. 中学生物教学, (21): 65-67.]
SHI XD, GU YX, DAI J, et al., 2018. Gene mining and analysis of Magnolia officinalis secondary metabolite pathway based on transcriptome [J]. Lishizhen Med Mat Med Res, 29(1): 247-250. [时小东, 顾雨熹, 代娇, 等, 2018. 基于转录组的厚朴次级代谢产物途径基因挖掘及分析 [J]. 时珍国医国药, 29(1): 247-250.]
SIMAO FA, WATERHOUSE RM, IOANNIDIS P, et al., 2015. BUSCO: Assessing genome assembly and annotation completeness with single-copy orthologs [J]. Bioinformatics, 31(19): 3210-3212.
STANKE M, WAACK S, 2003. Gene prediction with a hidden Markov model and a new intron submodel [J]. Bioinformatics, 19(Suppl. 2): ii215-ii225.
STEPHANE G, DUFAYARD JF, LEFORT V, et al., 2010. New algorithms and methods to estimate maximum-likelihood phylogenies: Assessing the performance of PhyML 3.0 [J]. Syst Biol, 59(3): 307-321.
TATUSOV RL, NATALE DA, GARKAVTSEV IV, et al., 2001. The COG database: New developments in phylogenetic classification of proteins from complete genomes [J]. Nucl Acid Res, 29(1): 22-28.
TARAILO GM, CHEN N, 2009. Using RepeatMasker to identify repetitive elements in genomic sequences [J]. Current Protocols, 25(1): 4.10. 1-4.10. 14.
WICKER T, SABOT F, HUA V, et al., 2007. A unified classification system for eukaryotic transposable elements [J]. Nat Rev Genet, 8(12): 973-982.
WANG LQ, JIANG RG, CHEN HF, 2005. Research progress on pharmacological effects of magnolol and honokiol [J]. Chin Trad Herb Drugs, (10): 155-158. [王立青, 江榮高, 陈蕙芳, 2005. 厚朴酚与和厚朴酚药理作用的研究进展 [J]. 中草药, (10): 155-158.]
WANG YB, LIU Z, ZHAO AH, et al., 2009. Application of functional genomics in the study of secondary metabolites of medicinal plants [J]. Chin J Trad Chin Med, 34(1): 6-10. [王勇波, 刘忠, 赵爱华, 等, 2009. 功能基因组学方法在药用植物次生代谢物研究中的
应用 [J]. 中国中药杂志, 34(1): 6-10.]
WANG Y, TANG H , DEBARRY JD, et al., 2012. MCScanX: A toolkit for detection and evolutionary analysis of gene synteny and collinearity [J]. Nucl Acid Res, 40(7): e49-e49.
WANG YH, XU WJ, MA DW, et al., 2005. Chromosome production and karyotype analysis of Magnolia officinalis [J]. J Sichuan Norm Univ (Nat Sci Ed), 28(2): 242-244. [王跃华, 徐文俊, 马丹炜, 等, 2005. 川厚朴染色体制作和核型分析研究 [J]. 四川师范大学学报(自然科学版), 28(2): 242-244.]
XUE ZZ, ZHANG RX, YANG B, 2019. Research progress of Magnolia officinalis authenticity [J]. Chin J Chin Mat Med, 44(17): 3601-3607. [薛珍珍, 张瑞贤, 杨滨, 2019. 厚朴道地性研究进展 [J]. 中国中药杂志, 44(17): 3601-3607.]
YANG X, YANG ZL, TAN M, et al., 2019. Analysis of transcriptome characteristics of Magnolia officinalis and development of EST-SSR markers [J]. J Nucl Agric, 33 (7): 1318-1329. [杨旭, 杨志玲, 谭美, 等, 2019. 厚朴转录组特征分析及EST-SSR标记的开发 [J]. 核农学报, 33(7): 1318-1329.]
YE LJ, ZHANG ZR, SUN ZX, et al., 2015. Determination of nuclear DNA content (2C value) in the main genera of Magnoliaceae [J]. J Plant Classif Resour, 37(5): 605-610. [葉林江, 张志荣, 孙志霞, 2015. 木兰科主要属种核DNA含量(2C-值)的检测 [J]. 植物分类与资源学报, 37(5): 605-610.]
ZHA LP, YUAN Y, HUANG LQ, et al., 2015. Identification and bioinformatics analysis of Magnolia officinalis MVA related genes [J]. Chin J Chin Mat Med, 40(11): 2077-2083. [查良平, 袁媛, 黄璐琦, 等, 2015. 厚朴MVA途径相关基因鉴定及生物信息学分析 [J]. 中国中药杂志, 40(11): 2077-2083.]
ZHANG LF, HUANG SJ, JIANG JL, et al., 2013. Study on the current situation and resource development of Magnolia officinalis forest [J]. Fujian For, (2): 28-30. [张龙辉, 黄树军, 蒋建立, 等, 2013. 厚朴营林现状及资源开发的研究 [J]. 福建林业, (2): 28-30.]
ZHAO X, WANG H, 2007. LTR_FINDER: An efficient tool for the prediction of full-length LTR retrotransposons [J]. Nucl Acid Res, 35(Suppl. 2): W265-W268.
(责任编辑 李 莉)
