团头鲂高密度遗传连锁图谱的建立及性别QTL定位
2021-04-21曹景龙王卫民
曹景龙,王卫民
华中农业大学水产学院/农业动物遗传育种与繁育教育部重点实验室/农业农村部淡水生物繁育重点实验室,武汉 430070
团头鲂(Megalobramaamblycephala),俗称武昌鱼,隶属鲤形目(Cypriniformes)鲤科(Cyprinidae)鲂属(Megalobrama),是我国特有的草食性养殖经济鱼类。目前已有2个团头鲂新品种在全国推广应用,年总产量近80 t。由于团头鲂生长快,肉质细嫩,营养价值高,深受广大消费者的喜爱。然而团头鲂自然分布非常狭窄,主要分布在长江中下游及其附属水体,其遗传多样性低,近亲繁殖极易引起经济性状衰退[1]。各地团头鲂养殖群体先后出现了生长速度减慢、性成熟提早、抗逆性能降低、体型变薄和肉质品位下降等种质衰退现象,故开展团头鲂种质资源和遗传改良相关研究势在必行。
鱼类的生长和生殖关系密切[2],许多重要的经济性状都在雌雄间表现出明显的差异,了解此类鱼的性别决定机制对于研究其性别控制育种至关重要。2015年梅洁[3]总结出14种养殖鱼类雌性个体明显大于雄性,例如:半滑舌鳎(Cynoglossussemilaevis)[4]、鲤(Cyprinuscarpio)、褐牙鲆(Paralichthysolivaceus)[5]和泥鳅(Misgurnusanguillicaudatus)[6]、大黄鱼(Larimichthyscrocea)[7]等。有11种养殖鱼类雄性个体显著大于雌性,如黄颡鱼(Pelteobagrusfulvidraco)和尼罗罗非鱼(Oreochromisniloticus)[8]等。在传统的养殖模式中,部分鱼类雌雄个体生长速度不同,雌性较雄性个体性成熟早,进行繁育活动消耗大量的能量,导致其生长速度减缓以及影响群体均匀度[9]。通过调控此类鱼类性别比例,实现单性化养殖,可避免鱼类生长过程中因生殖活动而消耗能量,使吸收的营养物质主要在肌肉中沉积,提高鱼体生长速度、肉质和经济价值[10]。人工养殖条件下团头鲂1~2 a就可以达到性成熟,但是雌性团头鲂因为怀卵导致个体生长速度下降,抗应激能力下降,活鱼运输容易死亡。因此,了解团头鲂性别决定机制以及性别相关QTL的定位对团头鲂的性别控制育种具有重要意义。
性别控制育种需要准确判别亲本的遗传性别,但对于团头鲂群体,其两性的外部形态差异不明显[11],也没有异形性染色体[12-13],依靠传统方法无法根据外部形态或组织学观察鉴定其遗传性别,甚至无法准确地判别其表型(生理)性别,所以需要开发团头鲂的性别相关的分子标记,用以辅助性别控制育种。水产养殖动物的多数重要经济性状都是数量性状,不仅由多个基因控制,且易受环境等因素的影响,例如性别、生长速度、肉质、抗病抗逆以及饲料的利用率等[14]。挖掘数量性状位点(quantitative trait locus,QTL)的最终目的是利用与目标性状紧密连锁的分子标记进行分子标记辅助选择育种(marker-assisted selection,MAS),从而促进对性状的遗传改良[15]。而要快速获得与性状相关的QTL位点,首先要构建高密度、高质量,且能覆盖整个基因组的遗传连锁图谱。
自1997年美国农业部启动5种水产养殖动物基因组计划以来,水产养殖动物的基因组研究取得快速进展,构建遗传连锁图谱的水产动物种类和图谱密度也在不断增加[16]。在2014年时就已经有40多个水产养殖物种构建了遗传连锁图谱,并绘制出超过20个水产养殖动物的重要经济性状的QTL位置[17]。近几年,更多的水产养殖动物以及更加精确的遗传连锁图谱也陆续被公布[18-21]。而团头鲂遗传连锁图谱的研究起步较晚,发展缓慢。2000年,林红[22]采取互为测交的远缘杂交作图策略及RAPD标记第一次构建了团头鲂的遗传图谱,该图谱包含14个遗传标记和4个连锁群。2002年葛芹玉[23]在林红研究的基础上,采用远缘杂交和单配子分型技术结合RAPD构建了团头鲂的遗传图谱,共获得6个连锁群,由21个标记组成。2017年,Wan等[24]构建出基于SNPs分子标记的团头鲂的高密度遗传连锁图谱,14 648个高质量的SNP标记被成功分配至24个连锁群。
本研究采用拟测交策略构建作图群体,通过简化基因组测序并参考笔者所在实验室自行组装的团头鲂第3代全基因组开发SNP标记,构建出团头鲂新一代高质量、高密度遗传连锁图谱,对性别性状进行了QTL定位,并在QTL的置信区间中筛选到一个性别相关的候选基因,以期为团头鲂性别分子标记辅助育种以及单性化养殖奠定基础,从而进一步推动团头鲂的遗传改良工作。
1 材料与方法
1.1 作图群体
试验选择团头鲂“华海1号”[1]正常生长性成熟雌雄个体,采用拟测交策略构建F1代为作图群体,本研究所有动物和实验均按照国家科学技术委员会的《实验动物管理条例》进行。于2016年在华中农业大学水产学院教学基地进行团头鲂的人工繁育,剪取父母本鳍条样本,分装于冻存管中用液氮速冻,24 h后转至-80 ℃超低温冰箱长期保存备用。繁殖后子代在此基地培育。子代1龄时,随机抽取197尾子代个体,解剖检测并记录其生理性别,剪取个体鳍条,用95%乙醇固定后,于-20 ℃保存用于DNA提取。
1.2 DNA提取
采用醋酸铵法提取DNA样本,方法步骤如下:
(1)剪取适当鳍条样本,剪碎并放置于600 μL的组织裂解液中;(2)加入6 μL蛋白酶K(20 mg/mL),混匀;(3)放于65 ℃水浴锅中水浴2~4 h,每隔15 min摇晃1次,或56 ℃过夜;(4)组织裂解后冷却至室温,加入200 μL醋酸铵(7.5 mol/L),混匀并放置冰上5 min;(5)4 ℃,12 000 r/min,离心10 min。取上清液500 μL至干净的1.5 mL离心管;(6)4 ℃,12 000 r/min,离心10 min,转移上清液于新离心管;(7)加入等体积约500 μL的异丙醇,轻轻摇动可见白色丝状沉淀;(8)4 ℃,12 000 r/min,离心10 min,弃上清;(9)加入1 mL 70%乙醇,轻摇混匀,4 ℃,12 000 r/min,离心10 min,弃上清;(10)重复步骤(9),弃上清,干燥15 min;(11)加入50 μL或100 μL ddH2O,溶解DNA;(12)使用Nandrop2000 (Thermoscientific,美国)检测DNA的浓度和质量,并用1%琼脂糖凝胶电泳检测完整性。提取合格的样本DNA送至华大公司测序建库。
1.3 文库建立及群体SNP的筛选
本研究采用RAD测序方法,使用Hiseq2500(华大基因,中国)对199个样本的DNA文库进行测序。建库成功样本包含2个亲本及197 F1子代个体。以笔者所在实验室完成的第3代团头鲂基因组为参考挖掘SNP标记。首先对原始数据进行过滤处理,去除接头污染和低质量reads,得到高质量的Clean data数据用于后续的比对分析。过滤采用华大自主研发的Soapnuke (华大基因,中国)软件进行,具体过滤条件如下:(1)去除含有adapter的reads;(2)去除低质量reads(碱基质量值小于等于15的碱基占10%);(3)去除含N碱基的比例大于10%的reads。然后将过滤到的Clean data按照reads前段4~8 bp的barcode序列信息拆分为每个文库数据,并去除barcode碱基,最后统计每个样本的数据量。使用短序列比对软件BWA(Version:0.7.12-r1039)的mem算法将Clean reads比对到参考基因组上,最后将每个样本数据经过过滤和校正后,采用GATK 3.3.0和SAMtools 2.6.2两个软件对群体的SNP进行变异检测,挖掘高质量SNP标记。
1.4 作图SNP的筛选及注释
接下来对适合构图的分子标记进行筛选。SNP标记的基因型编码根据遗传学通用的二等位编码规则,可分为8种分离模式(aa×bb、ab×cc、ab×cd、cc×ab、ef×eg、hk×hk、lm×ll、nn×np)。其中aa×bb仅适用于近交群体(如RIL、DH、F2等),其余标记适用于杂交群体(如CP)[25]。本研究作图群体属于CP群体,根据群体类型,对开发的标记进行如下筛选:
1)亲本筛选:首先过滤掉亲本中有缺失的位点,接着过滤掉亲本都为纯合的位点。
2)子代筛选:对于子代中的位点其GQ(genotype guality)<20的,标记为缺失;若子代为纯合位点,其深度<4的标记为缺失;若子代为杂合位点,其深度<5的以及次等位碱基深度<2的都标记为缺失。
3)缺失率筛选:将材料与方法“1.4 2)”中得到的结果,进行缺失率筛选,将缺失率>10%的位点过滤,并且在1 kb的物理距离上只允许有1个marker存在。
4)卡方筛选:根据CP群体各类型标记的分离情况,我们用卡方检验过滤偏分离标记,在α=0.01水平进行卡方检验,最终获得的标记用于遗传图谱的构建。
1.5 遗传图谱的构建
将上述过滤后的SNP标记,导入LepMap2.0软件进行遗传图谱的构建。首先采用该软件Filtering功能对标记进行进一步过滤,获得可用的作图标记。再采用该软件的Separate Chromosomes功能进行连锁群的划分,将LOD值设置为10时,可将剩余标记分配至不同连锁群,最后使用Order Markers功能计算各个连锁群遗传距离,最终获得团头鲂的遗传图谱。
1.6 QTL定位及候选基因的筛选
根据第F1子代雌雄性状的统计,结合得到的团头鲂高密度遗传图谱,采用MapQTL6.0分析软件的多QTL区间作图法(muliti-QTL mapping,MQM)对性别性状进行QTL定位。具体方法为:
(1)按照CP群体类型分别整理性状数据、连锁群数据、样品基因型数据成MapQTL6.0要求的格式,导入软件。(2)对性别表型进行置换检测(permutation test)检验,确定各表型在每个连锁群上的LOD阈值。(3)选择MQM mapping分析方法。 (4)根据显著性阈值,对结果进行过滤,筛选出跟性状显著关联的位点。(5)以LOD值3.0作为初始阈值,确定QTL数目及其在连锁群上的位置。
QTL命名方法按照QTL+连锁群+编号命名,其中QTL以大写Q表示。统计QTL置信区间内包含的SNP标记,参考团头鲂第3代全基因组,对标记上下游500 kb进行基因筛选,并在NR数据库内完成基因功能注释。
2 结果与分析
2.1 原始数据的产出及SNP的筛选
研究共获得1 863.65 Mb Reads数据,测序平均Q20为94.26%,平均Q30为89.69%,平均GC含量为37.51%,样本GC分布正常。碱基识别结果显示,各样本Q30(即碱基测序准确率为99.9%以上)比例均在90%左右;利用碱基类型分布分析调查了AT、GC分离情况,结果显示父本、母本和子代的GC含量分别为37.61%、37.62%和37.51%(表1)。
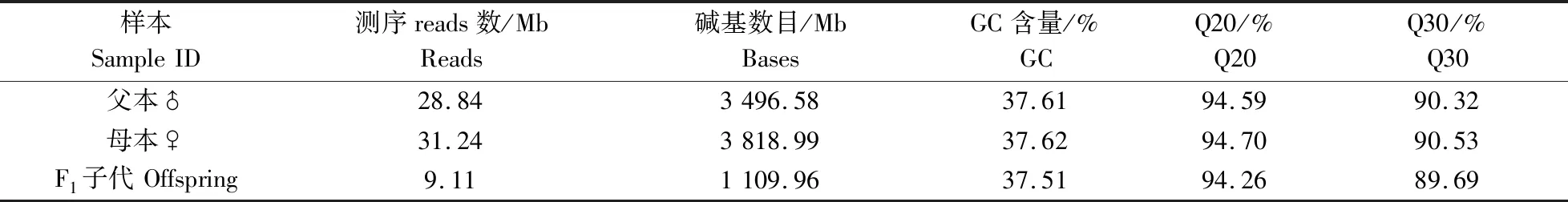
表1 各样本测序数据统计Table 1 Summary of sequencing information
将得到的Clean date与团头鲂参考基因组序列进行比对,每个样本数据经过过滤和校正后,采用GATK3.3.0和SAMtools 2.6.2两个软件对群体的SNP进行变异检测,共获得高质量SNP标记324 733个。SNP在基因组各染色体上的密度分布见图1。
2.2 作图SNP的筛选
群体变异检测共得到SNP位点324 733个,根据通用的二等位基因编码规则,对所得标记进行分型,本研究家系属于CP作图群体,选择3种亲本的多态性位点,使其对应3类基因型。雄性杂合与雌性纯合编码基因型为lm×ll,雄性纯合和雌性杂合编码基因型nn×np,雄性、雌性均杂合编码基因型为hk×hk。通过亲本筛选、子代筛选、缺失率筛选以及卡方筛选,最终获得构图标记10 809个,这些SNP中,转换标记有6 328个,占58.54%;颠换标记有4 481个,占41.46%,转换颠换之比为1.41(表2)。
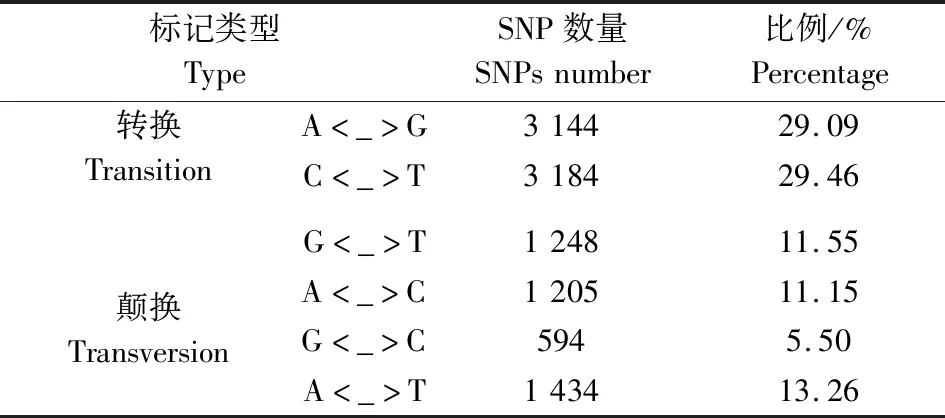
表2 SNP标记类型与分类Table 2 SNP marker types and classification
2.3 遗传图谱的构建
以筛选到的10 809个SNP标记为基础,采用LepMap2.0软件的Filtering功能对所得SNP标记进行进一步过滤,最终共获得10 795个作图标记,被分配至24个连锁群,与团头鲂单倍体染色体数目一致。该遗传连锁图谱全长2 578.45 cM,包含10 795个SNP标记,包括3 703个雌性特异标记、3 947个雄性特异标记和3 145个共有标记,可整合为4 034个簇(Bin Marker),平均遗传距离为0.24 cM(表3)。
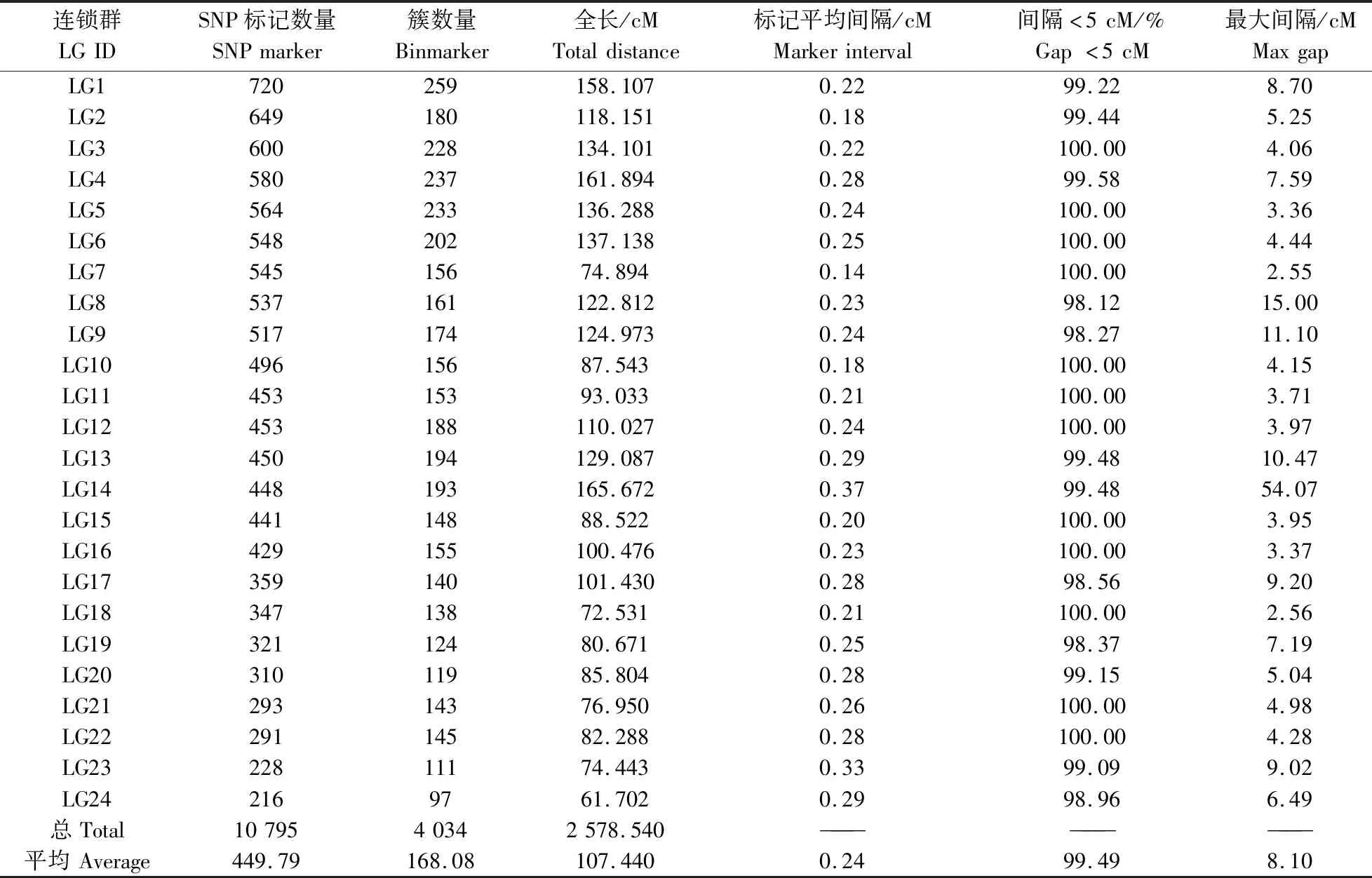
表3 遗传连锁图谱信息统计表Table 3 Information of genetic linkage map
24个连锁群长度介于61.702 cM(LG24)至165.672 cM(LG14)之间,平均长度为107.439 cM,标记间平均遗传间隔介于0.14 cM(LG7)至0.37 cM(LG14)之间。其中LG1上分布了最多的SNP标记(720个),该连锁群长度为158.107 cM,相邻标记间平均遗传距离仅为0.22 cM;而LG24上分布了最少的SNP标记(216个),其长度为61.702 cM,相邻标记间平均遗传距离为0.29 cM。24条连锁群的Max Gap长度介于2.546 cM(LG7)至54.066 cM(LG14)之间,而每个连锁群中Gap <5 cM的平均比例为99.49%(介于98.12%至100.00%之间)。使用JoinMap 4.0 软件绘制遗传连锁图谱(图2)。

图2 团头鲂高密度遗传连锁图谱Fig.2 The high-density genetic linkage map of M. amblycephala
2.4 性别相关QTL定位结果
利用软件MapQTL6.0对团头鲂性别相关QTL进行定位。取LOD显著阈值3.0进行分析,取97%的置信区间,即LOD峰值降低1时对应的区域。这样共检测到4个QTL。表4统计了团头鲂性别相关QTL的连锁群位置、置信区间大小、区间内SNP标记名称及数目、各标记的遗传位置、LOD值和表型解释率等信息。4个性别相关QTL分布于LG8、LG13、LG15、LG18连锁群上,置信区间内共包含14个SNP标记,单个SNP位点的LOD值介于3.18~4.17,解释表型变异介于5.1%~10%。根据QTL的定位信息,使用Map Chart2.2软件绘制了团头鲂性别相关QTL在连锁群上相对位置图(图3)。
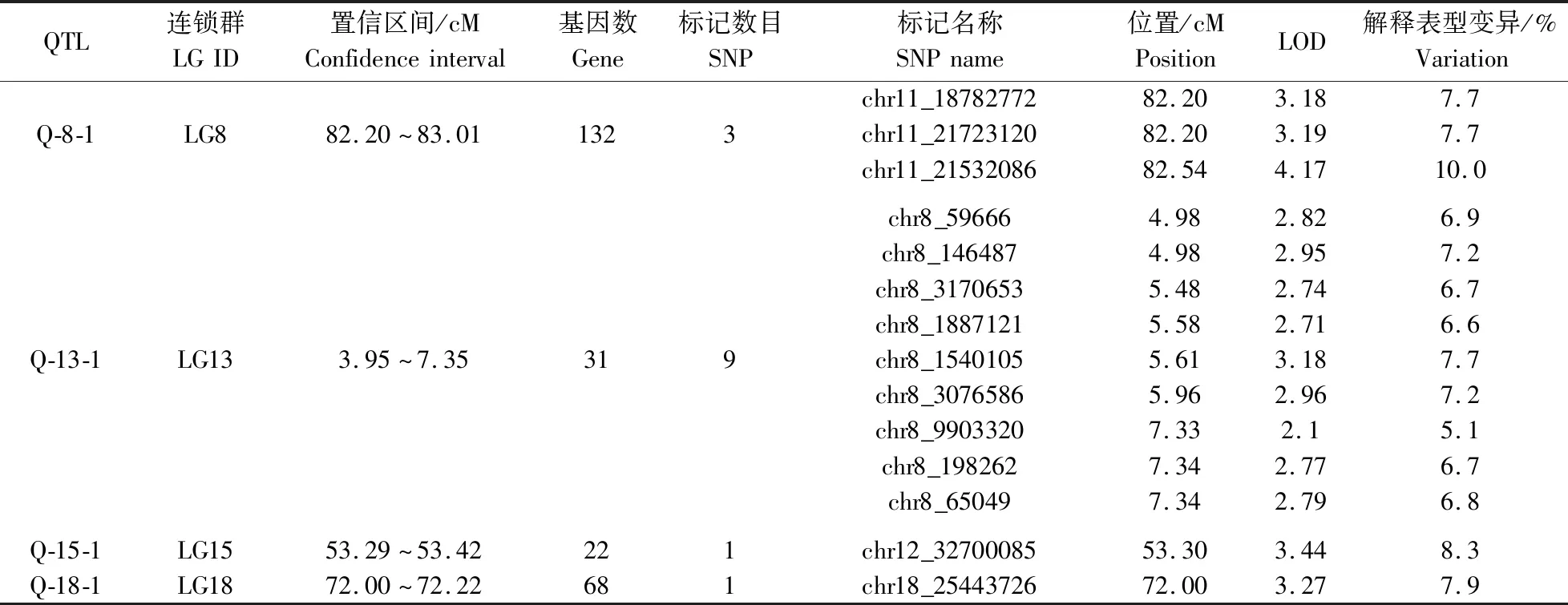
表4 团头鲂性别QTL定位结果Table 4 Sex related QTL mapping results in M. amblycephala
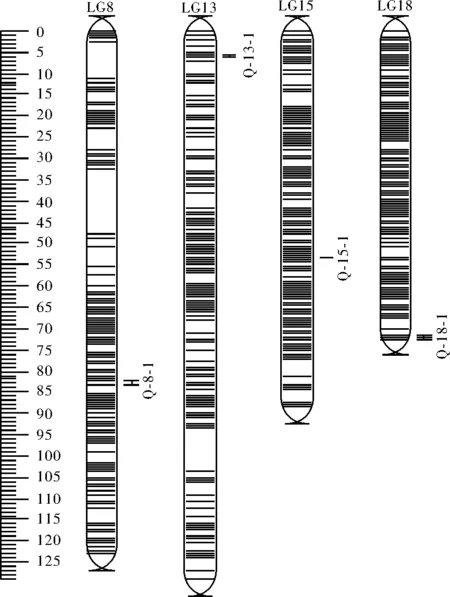
图3 性别QTL在遗传连锁图谱上的位置Fig.3 The position of gender QTL on the genetic linkage map
2.5 性别相关候选基因的筛选
参考团头鲂的第3代全基因组信息,对QTL置信区间进行基因查找,共得到253个基因(Q-8-1区间包含132个基因、Q-13-1区间包含31个基因、Q-15-1区间包含22个基因、Q-18-1区间包含68个基因),在NR数据库内完成基因功能注释。通过基因功能分析,发现1个参与生殖过程的基因,该基因位于Q-13-1号QTL区间内,chr8-3076586号SNP位点附近,编号为TTF29452。将此基因序列在NCBI数据库进行比对,注释到了基因DCTN2。DCTN2全称为动力蛋白激活蛋白2(p50)(dynactin 2 (p50)),参与微管形成,有丝分裂和细胞增殖以及细胞极性的建立和维持。此基因在斑马鱼、鲫和金线鲃(Sinocyclocheilus)等水产动物基因组中均有发现。此基因很可能是与团头鲂性别决定密切相关的关键候选基因。TTF29452基因序列信息见图4。

图4 TTF29452序列信息Fig.4 DNA sequence of TTF29452
3 讨 论
遗传图谱被广泛地应用于图位克隆、目标基因定位、比较基因组和分子标记辅助育种等研究中,高密度、高质量的遗传图谱的构建是在基因组水平进行遗传学研究的重要基础。团头鲂遗传图谱的研究较少,已发表的图谱属于中低密度的遗传图谱,标记数目少,分辨率低,无法进行QTL的精准定位、分子标记的筛选和目标基因的定位。
遗传图谱的构建受到亲本的多态性、作图群体和作图标记的影响。团头鲂达到性成熟需要1~2 a时间,世代周期长,建立近交群体难度较大。所以本研究在大规模的团头鲂养殖群体中,挑选性成熟度较好的雌雄个体作为亲本,在保证了亲本的基因信息多态性的同时,通过拟测交法构建F1子代群体为作图群体,作图群体构建简单快速,满足水产动物遗传图谱构建的要求。
简化基因组测序能大幅降低基因组的复杂度,能够低成本地开发大量的SNP位点,常用于遗传变异检测、高密度遗传图谱构建、重要性状候选基因定位和群体遗传进化分析。单核苷酸多态性(SNP)标记作为目前最具应用潜力的第3代分子标记,具有遗传稳定性高、位点丰富且分布广泛以及检测快速、易实现自动化分析等特点,被广泛地应用到遗传图谱构建、QTL定位和标记辅助选育等众多方面。本实验通过RAD-seq测序构建基因文库,并开发SNP标记。最后得到1 863.65 Mb Reads数据,样本Q30比例均在90%,测序深度分别为52.344×、56.002×和17.301×,表明此次测序质量较好。
通过筛选得到的324 733个高质量SNP标记,最终得到10 795个SNP标记,用以团头鲂遗传连锁图谱的构建。该图谱拥有24个连锁群,连锁群数目与团头鲂单倍体染色体数目一致,全长2 578.45 cM,平均长度为107.439 cM,标记的平均遗传距离为0.24 cM,表明本研究构建的遗传图谱密度高,质量好且标记分布均匀。相对于团头鲂已经发表的遗传连锁图谱,本研究构建的遗传连锁图谱在连锁群的完整度、标记数量、图谱长度以及标记平均距离等方面均有所提升,是团头鲂现阶段密度及质量最好的遗传连锁图谱。
以团头鲂高密度遗传图谱为基础,在LOD阈值>3.0下,共检测到4个性别相关QTL。其中位于8号连锁群上的QTL,Q-8-1区间峰值SNP标记chr11_21532086,LOD值最大为4.17。其表型解释率为10%。虽然Wan等[24]对团头鲂性别性状进行了QTL定位,但是其LOD值仅为2.18,表型解释率为4%,相对其结果,本研究定位的QTL更加准确。
通过基因功能分析,在Q-13-1号QTL区间内,chr8-3076586号SNP位点附近,注释到基因DCTN2(TTF29452)。该基因功能为细胞极性的建立和维持。DCTN2基因属于动力蛋白激活蛋白(dynactin,DCTN)家族的亚基之一,DCTN是拥有的6个亚基的多亚基蛋白质,包括DCTN1、DCTN2、DCTN3、DCTN4、DCTN5和DCTN6。DCTN家族基因的研究主要集中于哺乳动物肿瘤的发生、癌细胞的病变等,在水产动物研究报道较少。根据已有的报道,DCTN家族已有2个亚基被发现可能参与性别形成过程:郑波等[26]以小鼠精子为研究对象,向小鼠的生精小管内注射DCTN1的小干扰RNA(siRNA)进而干扰DCTN1的表达。3周后取附睾尾部精子进行形态学分析,发现注射DCTN1 siRNA 组的精子尾部畸形率明显上升,说明DCTN1在精子变形中发生重要作用,影响了精子尾部的形成。魏敏[27]对半滑舌鳎的DCTN5基因进行了相关的功能研究。通过RACE-PCR获得了DCTN5的2个转录本(DCTN5-tv1、DCTN5-tv2)。通过荧光定量PCR测定了2个转录本在性腺、肝、脑、鳃、皮肤、肌肉、肠、脾、肾、心脏、胃、鳍条12种组织中的表达水平。最后发现DCTN5-tv2主要在性腺中表达,在其他组织中表达量非常低,推测DCTN5-tv2可能参与性腺的发育和分化过程。本研究所筛选到的性别关键候选基因DCTN2是否参与团头鲂性别分化的具体过程,还需要进一步的验证。
综上,本研究所构建的高密度遗传连锁图谱,筛选到的性别相关QTL位点、连锁标记以及候选基因,为我们后续开展团头鲂的分子标记辅助育种、性别相关功能基因精细定位等研究提供了丰富的基础资料和重要的理论依据。
