利用纳米孔测序鉴定蓖麻蚕病害
2021-03-30陈大嵩李健雄戴建青
陈大嵩,黄 鸿,吴 华,李健雄,戴建青
(广东省科学院动物研究所,广东省动物保护与资源利用重点实验室,广东省野生动物保护与利用公共实验室,广州 510260)
蓖麻蚕Samiaricini,又叫惠利蚕、木薯蚕,大蚕蛾科Saturniidae樗蚕属的驯养绢丝与食用的非滞育经济型昆虫。原产于印度东北部的阿萨姆邦,由宽带樗蚕Samiacanningi驯化而来(Peigler & Calhoun, 2013)。我国自1951年,由中国科学院实验生物研究所朱洗教授引进饲养蓖麻蚕。蓖麻蚕茧壳奶白色,可用于生产绢纺原料,为全世界仅次于家蚕与柞蚕的第三大绢丝昆虫,其蚕丝质量仅次于家蚕BombyxmoriLinnaeus并优于柞蚕AnthereapernyiGuerin-Meneville (van Beeketal., 2000),蚕丝可设计成优良的生物材料与细胞支撑介质(Paletal., 2013),其熟蚕与蛹亦是优质昆虫食品(Longvahetal., 2011),蚕蛹油脂可提炼优质健康的食用油(王卫飞等, 2018),卵可繁殖多种寄生蜂(莫现会等, 2016)。蓖麻蚕即可室内饲养亦可放养,除家蚕外,为仅有的几种可以在室内大量圈养的绢丝昆虫。蓖麻蚕的寄主植物相较家蚕与柞蚕更广,一般以大戟科的蓖麻与木薯做饲料大量饲养,替代寄主包括大戟科的乌桕Sapiumsebiferum(L.)Roxb.、夹竹桃科的鸡蛋花Plumeriarubra、苦木科的臭椿Ajugalupulina、马桑科马桑CoriarianepalensisWall.、芸香科吴茱萸Euodiaruticarpa、菊科的蒲公英TaraxacummongolicumHand.-Mazz.和莴苣LactucasativaL. var. angustanaIrish.等。以蓖麻-蓖麻蚕与木薯-蓖麻蚕的形式进行栽培-养殖的联合增加了蓖麻与木薯的产值,是带动蓖麻与木薯种植业的重要因素之一。
蓖麻蚕生产上可能遭受侵害的毁灭性病害包括蓖麻蚕微粒子病(刘士贤等, 1964;谢伟东, 1989)、脓病(叶育昌, 1982;李敏棠等, 1985)、软化病等(吴汝章, 1987),甚至同时感染多个疾病。此外,被污染的蚕室、蚕具、蚕卵很易残留病原,且随着累代对病原物的不停积累,蚕病会呈现愈发严重的趋势并且病原物亦愈发难以清除。目前常用的分子生物学检测蚕病病原手段包括PCR、实时荧光qPCR、环介导等温扩增LAMP、巢式PCR等方法。随着高通量测序方法的发展,测序成本的不断降低,高通量检测方法被广泛应用在各种病原的检测中。在各高通量测序平台中,牛津纳米孔公司Oxford Nanopore的掌上纳米孔测序仪MinION是一款便携式测序仪,能够直接进行DNA和RNA测序,拥有超长的读长、实时测序、随时终止等特性,被广泛应用在检测多种病原体中(Warwick Dugdaleetal., 2019;Wongsurawatetal., 2019)。本文通过利用MinION测序仪对蓖麻蚕病蚕组织样品的DNA进行测序,鉴定病蚕感染的病原物,以实现利用高通量测序技术对蓖麻蚕疾病进行监控。
1 材料与方法
1.1 DNA提取
蚕病取自江苏省南京市蚕农的蚕室,接种于实验室内饲养的蓖麻蚕1龄幼虫,接种后至2~3龄幼虫集体发病,幼虫脱离寄主、拒食、拉稀,死后呈现发黑、软化等症状,死后蚕体整体用于DNA提取以获得足够量的起始DNA浓度。待测蚕体首先在80℃孵育5 min以杀灭病原以防交叉污染,每1 g蚕体组织放于10 mL CTAB裂解液(2% CTAB,100 mM Tris (pH 8.0),20 mM EDTA,1.4 M NaCl,3%疏基乙醇)中研磨,65℃水浴2 h。待降温至室温,加入等体积的酚氯仿异戊醇(25 ∶24 ∶1)萃取,震荡10 s后离心10 min,取上清液重复上一步再次加入等体积的酚氯仿异戊醇萃取。上清液加入等体积的异丙醇沉淀DNA,颠倒混匀后,离心10 min。弃上清留取DNA沉淀,加入适量70%乙醇清洗DNA,离心5 min后弃上清并用移液器吸干液体,晾干DNA后加入100 μL无菌水溶解DNA。
1.2 纳米孔测序文库构建
DNA溶液经过微量分光光度计测量DNA含量,取2 μg DNA溶液加入Covaris g-TUBE打断管上部,8 500 r/min离心1 min将DNA溶液离心到下部,颠倒后8 500 r/min离心1 min将DNA溶液重新离心到上部,DNA溶液取出1 μL加入微量分光光度计测量DNA含量并保证1 μg以上的DNA用于下一步实验。利用末端修复试剂盒(NEBNext End repair / dA-tailing Module,E7546)对打断的DNA进行末端修复,利用磁珠(AMPure XP beads)对DNA文库进行回收,取1 μL文库加入微量分光光度计测量DNA含量并保证700 ng以上的DNA用于测序。利用末端连接试剂盒(NEB Blunt/TA Ligase Master Mix,M0367)把连接测序试剂盒(1D Genomic DNA by ligation,SQK-LSK108)中的接头连接到DNA上,并用AMPure XP磁珠回收DNA文库,取1 μL文库加入微量分光光度计测量DNA含量并保证430 ng以上的DNA用于测序。DNA测序芯片(Flow Cells R.9.4.1)放入MinION测序仪并连接电脑,对机器与芯片进行质检并保证芯片有800以上的可用纳米孔,从芯片priming port孔取出20~30 μL缓冲液以去除气泡,从priming port加入800 μL测序缓冲液并静置5 min。打开sample port孔,再次从priming port加入200 μL测序缓冲液,DNA文库与连接测序试剂盒中的测序珠(LLB)与缓冲液(RBF)混合,一滴一滴加入到sample port孔,关闭sample port与priming port。
1.3 纳米孔测序与分析
MinION测序仪运行48 h,合并获得fastq文件,测序原始数据储存在国家基因库生命大数据可信计算平台(项目编号:CNP0001421,测序编号:CNR0339720)。从公共核酸序列数据库NCBI下载各微孢子、各核多角体病毒、白僵菌、柞蚕肠球菌基因组,作为病原体序列数据库,利用minimap2(v2.17)把测序序列比对到病原体数据库中,以检测测序结果。
2 结果与分析
通过MinION测序仪获得2 116 314条DNA序列约1.87 G个碱基,最长53 282 bp,中位数563,均值884,序列长度大多超过300 bp且集中在 300~1 500 bp长度(图1)。其中,4 476条序列(占总序列2.1‰)被minimap2比对到病原数据库(图2),且与比对到蓖麻蚕基因组类似,序列长度与比对碱基数量的散点图集中在一片V字区域,并没有线性关系。比对长度超过800 pb的序列中157条序列比对到家蚕微粒子Nosemabombycis基因组,139条序列比对到柞蚕肠球菌Enterococcuspernyi基因组(部分比对结果见表1),比对长度超过300 bp的序列分别有206和313条序列比对到家蚕微粒子和柞蚕肠球菌基因组(图3),这说明样品组织已经感染两种病原体。
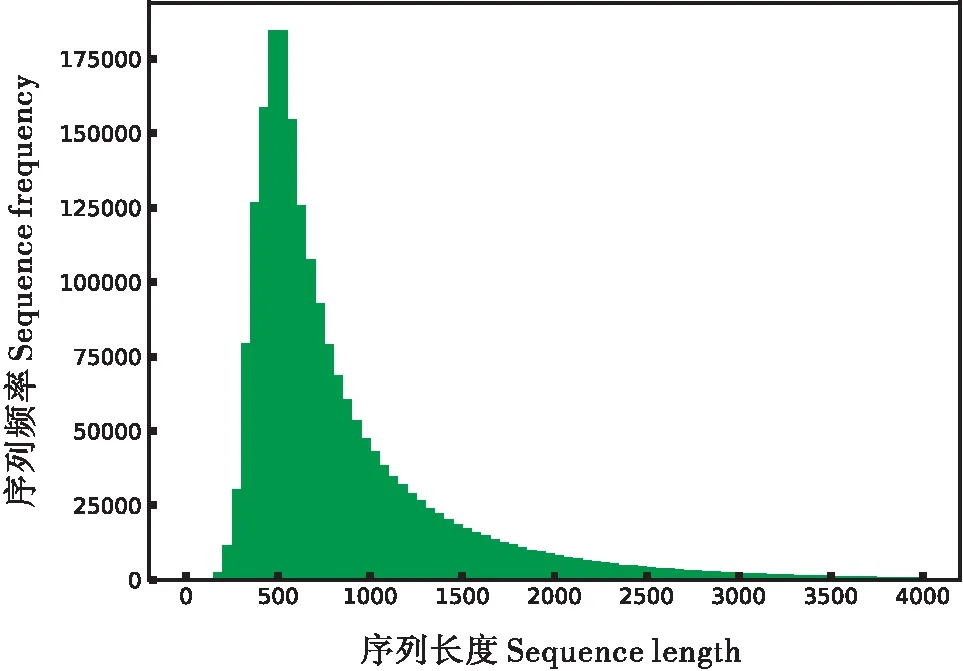
图1 MinION测序序列长度分布图Fig.1 Distribution of the sequence lengths obtained by MinION

图2 序列比对结果散点图Fig.2 Scatter plot of mapping result注:A图,序列与蓖麻蚕基因组比对结果;B图,序列与病原数据库比对结果。Note:A, mapping results to genome; B, mapping results to pathogen database.

表1 部分序列比对结果

图3 比对到病原基因组且比对长度超过300 bp的序列长度分布小提琴图Fig.3 Violin plot of the sequence length mapped to pathology genomes with more than 300 pb mapping length 注:小提琴图宽度代表序列的数量。Note:Widths of violin plot represent the sequence amount.
3 结论与讨论
昆虫组织具有高蛋白质、高脂肪的特性,并且在被病原体感染后,坏死组织的DNA被降解,然而高通量测序对DNA提取的质量有较高的要求并且起始建库的DNA须达到足够数量,因此昆虫DNA的提取质量决定了最终测序结果的优劣。本研究采用提高裂解液与增加萃取次数的方式提高DNA的提取纯度与数量,为昆虫构建高通量测序文库提供参考。
单分子纳米孔测序技术具有高通量、超长读长、可以直接检测碱基甲基化修饰、无需PCR扩增和体积较小便于携带等优势,可应用于动植物和微生物基因组测序、宏基因组测序、RNA直接测序、微生物检测、DNA与RNA甲基化检测、mRNA的polyA长度定量等研究(曹影等, 2020。然而,单分子纳米孔测序的结果具有较高的错误率,是阻碍其应用的关键。目前MinION测序仪的单碱基准确率约85%,修正后的一致性序列的准确率可达97%~99%,因此对单分子纳米孔测序的分析具有一定的挑战。在DNA文库构建时,本研究通过DNA低速离心打断管以获取更长的DNA片段以构建测序文库,有利于增加序列比对的长度以增加可信度。值得注意的是,minimap2的比对长度并未与序列长度成线性关系,可能与minimap2的算法有关,因此本研究选取的置信区间为最低300 bp的比对长度,即比对长度超过300 bp为可信比对结果。同时,比对数据库中病原物基因组序列的丰富度与病原物种类的选择,会对鉴定结果起关键作用并影响最终鉴定结果。因此,在选择病原物种核酸序列时,应尽可能涵盖所有蚕病病原的基因组序列,降低比对错误与假阳性。本实验选择真菌性病害病原、细菌性病害、微粒子基因组以及所有杆状病毒基因组序列,尽可能构建全面的病原序列数据库以降低鉴定的误差,并且通过过滤序列本身长度短且比对长度较短的序列,以增加比对结果可靠性。
本研究通过MinION测序仪对蓖麻蚕病蚕基因组进行测序,与病原基因组序列库进行比对,无需PCR扩增步骤,降低了假阳性。利用MinION等高通量测序手段对病蚕、蚕具或蚕室进行检测不仅可以一次性准确的鉴定蚕病,也摒弃了PCR方法可能具备的假阳性现象,而且其不需要模板设计引物,甚至可以鉴定新病原等的特性,可以与PCR鉴定方法形成互补,对蚕病的全面监测提供保障。
