基于边缘关注模型的语义分割方法
2021-03-07佘玉龙张晓龙程若勤邓春华
佘玉龙,张晓龙*,程若勤,邓春华
(1.武汉科技大学计算机科学与技术学院,武汉 430065;2.武汉科技大学大数据科学与工程研究院,武汉 430065;3.智能信息处理与实时工业系统湖北省重点实验室(武汉科技大学),武汉 430065;4.武汉科技大学附属天佑医院,武汉 430064)
(*通信作者电子邮箱xiaolong.zhang@wust.edu.cn)
0 引言
肝癌是世界上最常见的癌症疾病之一,每年导致大量死亡[1-2]。在核磁共振成像(Magnetic Resonance Imaging,MRI)或电子计算机断层扫描(Computed Tomography,CT)影像中可以准确地观测出一些结果,包括肝脏的形状、位置以及肝脏边缘细节,这些可以帮助医生准确地做出肝功能以及肝癌的评估和治疗计划[3]。在传统临床诊断中,这些由专家进行人工分辨,但是,分辨结果会受到主观经验、认知能力、疲劳等因素的影响,因此,计算机医学图像识别在临床实践中有助于提高识别的准确率和稳定性。
最近几年,深度学习在处理多种计算机视觉任务上取得了巨大的进展,其中大放光彩的是处理图像分类和目标检测问题的卷积神经网络(Convolution Neural Network,CNN)[4]。越来越多的研究者用CNN 以及其衍生出来的各种深度神经网络来处理各种图像问题[5],其中就包括了图像语义分割(Semantic Segmentation)。
图像语义分割是一种从预定义的一组类中对图像中的每个像素进行分类的方法,而预定义的一组类就是具有特定语义类别属性的一片区域[6]。对于医学领域的语义分割,大部分网络都是在全卷积神经网络(Fully Convolutional Network,FCN)[7]的基础上根据医学影像的特点进行改进。例如:郭树旭等[8]采用了改进的全卷积神经网络对肝脏进行分割。而最经典的网络模型则是U-Net[9]和V-Net[10]。这两个网络的特点都是类似U型结构,且都是通过skip-connection 结构将前后对应的stage 进行特征的融合。二者的区别是:U-Net 处理二维数据,V-Net处理三维数据。后来有很多学者在此基础上做了改进,例如UNet++[11]就是在U-Net 的基础上,在中间skipconnection 结构中添加了短链接,将不同层的特征进行了融合,并取得到了更好的分割效果。例如徐宝泉等[12]先后使用了两次Vnet-S网络对CT影像中的器官进行粗分割和细分割,并提出了基于级联Vnet-S 网络的单一器官自动分割算法;再例如AHCNet[13]利用了U-Net 中不同层之间的特征信息进行混合连接,并引入了注意机制[14],以此来有效地获取肝脏和肿瘤分割的混合特征,从而实现肝脏和肿瘤CT 图像的分割;另外,CE-Net[15]提出了一个上下文编码器网络,能获取更多的高层次信息并保留用于2D医学图像分割的空间信息。
上述的一些肝脏影像分割方法取得了一定的进展,但是对于肝脏影像一些具体边缘特征信息方面有待进一步完善。肝脏边缘分割的四个边缘特征难点如下:
特征1 肝脏中间部位的深色圆状物是下腔静脉,而附近的深色或白色裂纹或斑点是肝脏的软组织或者血管;前者不属于肝脏,而后者则属于肝脏。
特征2 肝脏上右区域与脾脏的界线模糊,不易分辨。
特征3 肝脏的下左方和中左部位会出现一些肿瘤或者坏死的深色区域,虽然是肿瘤或者坏死,但是这些也都是属于肝脏成分。
特征4 肝脏的上面出现裂开的现象,医学上称为肝裂,但是这些则不属于肝脏部位。
对于上面提到的边缘信息,在普通的卷积网络中,难以提取到边缘的一些细节特征,所以就需要对边缘数据进行上下文关系的解析。基于以上特征信息,以及上述已有研究的基础上,本文提出了一种边缘关注模型(Edge Attention Model,EAM)来充分利用边缘信息。
本文研究的问题是肝脏影像的语义分割,主要的工作如下:1)针对特征的空间和通道,结合注意力机制构建了EAM,并运用Encoder-Decoder(编码-解码)深度学习框架[16]结合残差网络ResNet34[17]与反卷积操作构建了边缘关注网络(Edge Attention Net,EANet),以此提高关于边缘特征信息的肝脏影像语义分割的效果;2)利用几种图像平滑操作对分割后噪声比较大的肝脏分割图像进行降噪处理;3)本实验中现有已处理好的肝脏语义分割数据集包含20 个序列,共1 150 张图片,此数据集中的肝脏标注图像均经过专家严格指导与修改。
1 EANet架构
1.1 Encoder-Decoder结构
Encoder-Decoder 是深度学习中非常常见的一个模型框架。如无监督算法的auto-encoding 就是用这种编码-解码结构设计并训练的;近期的image caption 应用就是CNN-RNN 的编码-解码框架;文献[18]中也用了此模型来做文本摘要总结;文献[19]中则用此结构来处理MRI 图像。Encoder-Decoder 可作为一类框架。Encoder 和Decoder 部分可以处理任意的文字、语音、图像、视频数据,模型可以采用CNN、循环神经网络(Recurrent Neural Network,RNN)、双向循环神经网络(Bi-directional Recurrent Neural Network,BiRNN)、长短期记忆网络(Long Short-Term Memory,LSTM)、门控循环单元(Gated Recurrent Unit,GRU)等。所以基于Encoder-Decoder,可以设计出各种各样的应用算法。Encoder-Decoder框架是一个End-to-End 学习的算法,这也是该框架最显著的特征。本文以图像-图像的方法来实现肝脏部位的语义分割。对于本文所搭建的Encoder-Decoder结构,Encoder可以理解为一个卷积操作,用来获取图像的特征信息;而Decoder 则可以理解为是一个反卷积操作,用来解析得到的特征信息,并将其展示出来,如图1所示。

图1 Encoder-Decoder基本结构Fig.1 Basic structure of Encoder-Decoder
1.2 EANet构建
前面提到本文的网络结构分为Encoder和Decoder两个模块,即图1 中的卷积操作和反卷积操作。由于医学图像数据集量比较少,所以选择的模型不宜过大,因为参数过多容易造成过拟合现象。本文将在ImageNet 上预训练的ResNet34 作为卷积操作基础模型来进行特征提取,它有助于减小模型和提高训练的速度。
在Encoder阶段中,为了有效地融合肝脏边缘多层次的特征和增强特征的表达能力,本文在ResNet34 的每层卷积阶段的输出上加上了本文提出的EAM。在本阶段的EAM 中,将第二次下采样后的第一张特征图作为EAM 中的低层特征,将同卷积模块中的最后一张特征图作为EAM 中的高层特征。图2中虚线的跳跃结构之后的两个卷积模块也进行同样的操作。在特征图大小相同的情况下,以此获取更多的边缘细节特征。
在Decoder阶段,考虑到会遗失掉编码器中的一些低层特征和经过多次卷积以及反卷积的高层特征,将低层特征和高层特征再次进行关注模块的处理,这样能结合更丰富的特征信息。在此阶段,本文将Encoder中同大小的最后一张图作为EAM 中的低层特征,将Decoder 中上采样后的特征图作为EAM 中的高层特征,这有助于提取更多边缘细节特征。此外,边缘关注模型的参数比较少,有助于节省内存。
图2 描述了EANet 的整体结构,其中用跳跃结构将EAM融入整个网络模型当中,充分利用了影像的空间特征,具有更高的效率。从原始肝脏图像的输入到肝脏区域分割结果的输出,实现了端对端的训练。
1.3 EAM结构
在传统的Encoder-Decoder 模型中,例如图像语义分割中的FCN,没有考虑全局的上下文信息和像素与像素之间的关系,忽略了在通常的基于像素分类的分割方法中使用的空间规整步骤,缺乏空间一致性;并且分割也不是在实例级别之上,效率不够高,不能适用于非结构化数据;而且在解码器的上采样操作中,会导致边缘模糊以及空间的细节丢失。文献[9]中运用跳跃结构将底层特征和高层特征直接串联起来,这有助于补充空间的细节;但是这个方法太过粗糙,没有将低层特征和高层特征进行上下文信息的获取,仍然会遗失掉一些肝脏边缘特征。为了解决这个问题,本文提出的边缘关注模型会捕获高级语义信息、着重关注分割目标的边缘细节信息。另外,包含全局上下文信息的底层特征对不同通道之间的语义关系进行编码,这有助于过滤干扰信息。通过使用包含丰富特征的信息,边缘关注模型可以加强目标区域并提高特征的表现力。
为了充分获取肝脏边缘特征信息,设计了针对空间和通道的注意力模块,并将并行处理融合后的结果与操作前的特征图进行特征融合,以提取进一步的语义依赖。对于普通的分割网络模块,空间注意模块与通道注意模块能有更好的特征图表达能力。图3是EAM的结构。

图2 EANet结构Fig.2 Structure of EANet

图3 EAM的结构Fig.3 Structure of EAM
1.3.1 EAM的算法描述
本模型有两个输入A、B,分别执行两个相同的并行操作。
1)并行阶段。
操作1:对输入的特征图A进行转置,得特征图A1,再进行1×1卷积操作,得特征图A2。
操作2:对输入的特征图A进行全局池化操作,得特征图A3;再进行1× 1 卷积操作,得特征图A4。最后将得到的A2与A4进行矩阵相乘操作得到特征图Aα。
对B进行相同操作流程可得到特征图Bα。
其中操作1将注意力放在了特征图的空间上,操作2将注意力放在了特征图的通道上。
2)融合阶段。
将Aα和Bα进行矩阵相加操作,得到特征图Cα;并用Softmax 函数进行激活得到特征图Cβ;然后用原始特征图A与Cβ进行矩阵相乘,得到特征图Cγ;最后再将特征图Cγ与B进行矩阵相加,并得到最终的返回结果C。
该算法伪代码如下:

1.3.2 空间注意模块
如图3 中模块a 的区域,低高层特征经过带有批规范化(Batch Normalization,BN)层和ReLU 层的卷积操作后,为了得到肝脏部位不同空间的特征信息,将其进行转置操作,如式(1);然后再进行一个1×1 的卷积操作,如式(2),以此来实现不同通道的交互和信息整合。此操作通过编码更广范围的语义信息到局部感受野中以增强特征图表示能力。
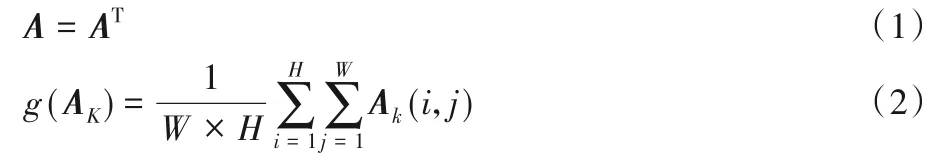
其中:A为待转置矩阵图;k代表常量1,2,…,c;W代表操作矩阵的宽,H代表操作矩阵的高。
1.3.3 通道注意模块
如图3中模块b的区域,高层特征的每一个通道映射可以看成一个类别明确的响应,并且不同的语义响应之间互相联系。通过获取不同通道映射之间的相互依赖性,可以有效增强特征图对于特定语义的表征能力。此通道操作则是进行全局平均池化(Global Average Pooling,GAP)操作提取全局上下文和语义信息[20]。将全局信息压缩成通道注意向量(Channel Attention Vector,CAV)(此注意向量包含丰富的不同通道语义特征),对语义依赖关系进行编码,这有助于加强关键特征和过滤背景信息。注意向量产生如式(3)、(4)所示:

其中:x代表高层信息特征图;y代表低层信息特征图;g()代表全局平均池化操作;δ1代表ReLU 函数;δ2代表Softmax 函数;Wx代表1× 1 卷积操作的参数;bx代表偏执量;AC代表注意力矩阵。
然后对向量进行1×1 的卷积与批量归一化,并将并行操作的结果进行矩阵相乘操作,以进一步捕获语义依赖关系。
1.3.4 特征融合
如图3中模块c的区域,为了更好地利用两个注意操作得到的全局语义信息,本文将操作输出的结果进行一个逐元素的加和实现特征的融合。然后采用了Softmax 函数[21]作为激活函数对向量进行归一化处理,如式(5)。

其中:X为待Softmax的操作数。
为了获取更多处理前遗失掉的基本特征,用操作前的底层特征图乘以得到的注意力向量,并生成注意力特征图。最后,通过添加高层次的特征图来校准注意力特征图,此操作如式(6)。

其中:sji为利用式(5)得出的结果,Di为相乘的低层信息矩阵,Aj为相加的高层信息矩阵。
与用来连接矩阵的concatenation 方法相比,特征矩阵的加法可以减少卷积的参数,有助于减少计算成本;此外,因为它只使用了全局平均池化和1×1 卷积,这个模块也没有添加太多的参数。全局平均池化将全局信息压缩成一个向量,这也降低了计算成本。
1.4 损失函数
在训练的反向传播过程,通过网络预测的输出与真实标签比较计算损失,不断迭代更新网络参数,使得输出结果接近真实值。本文采用的是交叉熵损失方法[22],由于本文实验的标签只有一个,模型最后需要预测的结果只有两种情况,对于每个类别的预测得到的概率为p和1-p,所以交叉熵公式如式(7):

其中:yi表示实际标签表示预测标签。
1.5 降噪处理
为了获取尽量多的特征,通常会增加训练迭代次数,但这样也可能导致某些肝脏边界复杂的图像产生一些噪声,如图4(a)所示,所以有必要对实验数据结果进行降噪处理。在降噪处理中,分别进行了以下五种平滑和模糊操作。对于不同分割结果,最佳降噪方法不唯一,所以本文对每个数据集分别使用五种降噪方法,并从中选取最佳的结果。
方法1:利用2D 卷积平滑[23]对图像进行平均过滤。设置一个平均滤波器内核,将此内核保持在像素上方,添加该内核下方的所有像素,取平均值并用新的平均值替换中心像素。
方法2:平均模糊由一个归一化卷积框完成,它只是用卷积框覆盖区域所有像素的平均值来代替中心元素。
方法3:高斯模糊[24]与平均模糊的不同就是把卷积核换成了高斯核,也就是卷积核里面的值由相等变成了符合高斯分布,即中心值最大,向两边递减。
方法4:中值滤波[25]是取相邻像素的点,对相邻像素的点进行排序,取中点的灰度值作为该像素点的灰度值。该滤波器经常用来去除椒盐噪声,同时又能保留边缘细节。
方法5:双边滤波[26]是一种非线性的滤波方法,是结合图像的空间邻近度和像素值相似度的一种折中处理,通过同时考虑空域信息和灰度相似性,达到保边去噪的目的。
从图4(b)中可以看到,处理后的肝脏分割图像的噪声明显减少。

图4 肝脏分割图像处理前后对比Fig.4 Comparison of liver segmentation image before and after treatment
2 实验结果与分析
2.1 数据集
本文实验的数据集包括两个公共数据集3Dircadb(https://www.ircad.fr/research/3dircadb/)和MICCAI-Sliver07(https://sliver07.grand-challenge.org/Download/)[27],以及某医院临床MRI 肝脏影像数据集。其中:3Dircadb 数据集总共有20 个序列,每一个序列大约有141 张肝脏图片,分组随机选4组为测试集,16 组为训练集;Sliver07 有20 个序列训练集,10个序列测试集,每个序列大约150 张肝脏图片;某医院数据集共有20 个序列,每个序列大约60 张肝脏图片,分组随机选4组为测试集,16为组训练集。
实验环境为高性能服务器,125 GB 内存,8 个Tesla V100 SXM2 16 GB GPU 卡,个人用户目录3 TB 内存;以及实验室计算机,操作系统为Windows 10(64 位),CPU 为i5-7500,内存8 GB。
2.2 3Dircadb数据集上的实验结果与对比
本文采用的第一个公共数据集是3Dircadb,该数据集中有20个CT 影像序列(共2 823张),其中10个为女性检查者的CT 影像序列,另外10 个是男性检查者的CT 影像序列。在这20 位检查者中,75%患有肝癌。本文实验随机选择了16 个CT 影像序列作为训练数据集,4 个序列作为测试数据集进行训练和测试。
为了验证本文提出的分割模型的可行性和有效性,用下列评价指标对分割结果进行预测统计。在图5中,A代表黄金分割图,也就是GT(Ground Truth)结果;B代表模型预测分割的结果;FN表示本该预测分割却没有分割到的区域;TP是分割正确的区域;FP是本不该分割却分割了的错误区域;TN是黄金分割和预测分割都不用分割到的区域。

图5 分割占比示意图Fig.5 Schematic diagram of division proportion
评价指标如下:
1)体积重叠误差(Volumetric Overlap Error,VOE),结果越小越好。

2)相对体积误差(Relative Volume Difference,RVD),为了方便判断,这里取绝对值,结果越小越好。

3)对称位置的平均对称表面距离(Average Symmetric Surface Distance,ASD),结果越小越好。
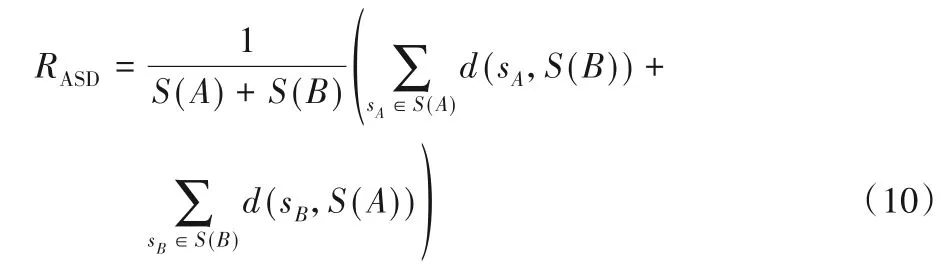
其中:S(A)代表A集合中的表面体素,d(v,S(A))表示任意体素v到S(A)的最短距离。
4)最大表面距离(Maximum Surface Distance,MSD),结果越小越好。

5)重合率(DICE),指两个物体相交的面积占总面积的比值,值域为[0,1],完美分割时该值为1,结果越大越好。

与U-Net、FCN、UNet++、AHCNet 等分割类网络结果进行对比,各种网络在3Dircadb 数据上的分割结果如图6 所示,可以看到,本文方法基本能将肝脏轮廓分割出来,且达到了比较好的分割效果。
利用上述5 个指标对分割结果进行统计,并将结果与U-Net、FCN、UNet++、AHCNet 等方法比较,结果如表1 所示,可以看到,在大部分预测标准中,本文方法都能取得较好的结果。

图6 3Dircadb数据集上的分割图像对比Fig.6 Comparison of segmentation images on 3Dircadb dataset

表1 3Dircadb数据集上分割结果的评价指标对比Tab.1 Comparison of evaluation indicators of segmentation results on 3Dircadb dataset
2.3 Sliver07数据集上的实验结果与对比
Sliver07数据集来自MICCAI2007,包含20个CT序列训练集,层间距范围为0.7~5.0 mm。采用以下指标评价实验分割结果:体积重叠误差(VOE)、相对体积误差(RVD)、平均对称表面距离(ASD)、最大对称表面距离(MSD)以及在文献[13]中提出的均方差对称表面距离(Root Mean Square Surface Distance,RMSD)。RMSD计算如式(13),结果越小越好。

这5 个评价指标的均值与标准差如表2 所示,可以看出,本文方法的效果整体较好。

表2 Sliver 07数据集上的分割结果的评价指标对比Tab.2 Comparison of evaluation indicators of segmentation results on Slicer 07 dataset
2.4 某医院临床MRI肝脏影像数据集上的实验结果与对比
此数据集来源于某医院肝脏科核磁共振(MRI)原始序列数据,选出了20 个描述肝脏较正常的dicom 序列,用python 的pydicom包将其转换成可见的png格式图片,共1 150张。
本文用labelme 标注工具进行手工标注,并在进行标注之前经过临床专家医生的指导。肝脏影像中,会在肝脏中下部位出现深色圆状物以及深色或白色裂纹或斑点等现象,并且这种现象在肝脏MRI中比较常见。图7 展示了四个肝脏影像在不同网络模型下的分割结果。
图7(a)为原始数据转换后的正常图片;图7(b)的GT 图则是经过专家指导后人工标注的GT。图7(c)为U-Net分割的结果图,可以看到在肝脏边缘的部分效果并不好。在测试UNet 语义分割实验中,尝试了多种卷积网络作为backbone,包括VGG16、ResNet34 和ResNet50 等,其中ResNet34 能得到较好的分割效果。图7(d)为UNet++分割的结果图,可以看到把第三张肝脏影像深色病变的肝脏也分割了,其实这也属于肝脏部分。图7(e)为AHCNet 的分割结果,效果要好一些,但边缘细节仍然比较粗糙。图7(f)为本文方法EANet 的分割图,可以看到和原图和GT图像相比,几个肝脏边缘的分割难点的分割效果有很大提升,边缘轮廓基本恢复,不过在某些分割边缘的细节上有待提高。图7(g)则是本文方法EANet实验结果在原图上的分割图,从上往下数第三个图中,可以看到中部肝脏病变区域也分割为肝脏部分,而将右下已坏死区域则分割为非肝脏部位,这正符合对肝脏病情中的需求。为了使精度更有说服性,这里采用五折交叉测试,将数据划分为5 组,依次让每一组都做一次测试集,另外4 组则作为训练集,最后取5组的测试精度平均数作为预测结果,结果如表3所示。

图7 某医院肝脏数据集上的分割图像对比Fig.7 Comparison of segmentation images on liver dataset of a hospital

表3 某医院肝脏数据集上分割结果的评价指标对比Tab.3 Comparison of evaluation indicators of segmentation results on liver dataset of a hospital
3 结语
本文提出了一种基于边缘关注的神经网络模块,并设计了边缘关注网络EANet 来对肝脏影像进行分割。在Encoder-Decoder 结构上:Encoder 部分运用了在ImageNet 上预训练的残差网络ResNet34,并在某些层加入边缘关注模型;Decoder部分则使用了反卷积操作和边缘关注模型(EAM)进行搭配,充分提取特征信息,最后得到肝脏影像语义分割图。在边缘关注模块中,本文利用全局平均池化和向量转换操作对空间和通道进行注意力机制处理,并搭配向量的归一化及其他特征融合操作,以充分提取到肝脏边缘的特征信息。与基于传统的深度学习的医学影像分割方法相比,本文方法充分利用了影像的空间特征,具有更高的效率;同时也可直接得到肝脏区域分割结果,实现了端对端的训练。最后还对分割后的图像进行了降噪处理。在多个数据集上的分割结果表明了本文方法的有效性。对于肝脏病灶分割中的肝脏的坏死、肿瘤、肝萎缩等具体问题,会涉及到更多的医学知识,需要后期和专家讨论,在这一方面做进一步的工作。
