基于生物信息学分析尤文肉瘤的差异基因表达及功能预测
2021-02-22韩广弢李皓桓
韩广弢,李皓桓
·论著·
基于生物信息学分析尤文肉瘤的差异基因表达及功能预测
韩广弢,李皓桓
430060 武汉大学人民医院骨科
基于生物信息学分析尤文肉瘤的基因差异表达及功能预测,为尤文肉瘤的治疗提供帮助。通过GEO 数据库寻找RNA 数据集,通过limma 包寻找差异基因并寻求可视化,对其进行信号通路分析和GO 富集分析,最后进行基因互作网络的分析。在尤文肉瘤组织中共筛选出312 个基因具有差异,其中192 个上调基因,120 个下调基因。通过细胞组成的研究,细胞外区域组分、细胞外区域、细胞外小泡、细胞外细胞器、细胞外泌体对尤文肉瘤影响较大。通过对生物进程的分析,细胞分化、系统发育、解剖结构、细胞发育、细胞发育过程影响较大。通过对分子功能的研究,蛋白结合、细胞黏附结合、蛋白质二聚体结合、胰岛素II 受体结合、细胞外基质结合影响较大。通过基因互作网络发现,ANXA2 和S100A10,ANXA2和S100A4,GRP 和CCK,LAMA5 和ITGA3,SCNN1A 和SG1C1 互作关系较为密切。ANXA2 和S100A10,ANXA2 和S100A4,GRP 和CCK,LAMA5 和ITGA3,SCNN1A 和SG1C1 互作关系较为密切,这些相互作用的基因编码蛋白可能调控尤文肉瘤的产生。
生物信息学; 尤文肉瘤; GEO
尤文肉瘤是儿童和年轻人中第二常见的恶性骨肿瘤,占所有原发性骨肿瘤的10% ~15%。发病年龄通常发生在10 ~ 20 岁之间[1]。尤文肉瘤最常见于骨骼,但不到10% 的肿瘤起源于软组织,并且有15% ~ 25% 的患者出现明显的转移[2]。没有临床转移灶的患者可能会发生微转移,因为在没有全身化疗的情况下,大多数患者会在手术切除后因远处转移性疾病复发[3]。这种传播的倾向导致尤文肉瘤患者的预后较差,因此对尤文肉瘤的研究迫在眉睫。本文将基于生物信息学分析尤文肉瘤的基因差异表达及功能预测,对尤文肉瘤的治疗提供帮助。
1 材料与方法
1.1 数据来源
GSE93075 数据集是从Gene Expression Omnibus(GEO)数据库获取,通过pubmed 进入,由NCBI 提供。此数据集由Nirmalya S 发布,我们通过搜索关键词“Ewing sarcoma”得到此数据集。此数据集由6 个尤文肉瘤患者的组织以及6 个非尤文肉瘤患者的组织组成。
1.2 方法
1.2.1 筛选差异基因 我们对芯片数据进行背景校正、标准化、汇总和探针质量控制。通过GEO 网站下载注释包,进行ID 转换,提取表达矩阵。对limma 包进行安装并加载,以< 0.05,|logFC| ≥ 1.5 为标准筛选差异基因,绘制热图和火山图。
1.2.2 利用DAVID 进行功能富集分析 DAVID 是生物信息数据库,其整合了生物学数据和分析工具,为大规模的基因或蛋白列表,并主要用于基因的功能富集,富集内容包括生物过程、细胞组成和分子功能三个部分。
1.2.3 蛋白质互作网络的构建 蛋白质互作网络通过PPI 应用10.5 版STRING 数据库分析。PPI 网络图中综合得分> 0.4 的蛋白具有统计学意义。用 Cytoscape 插件 MCODE 筛选出有意义的 PPI 模块,筛选标准为:MCODE 得分> 3,网络节点> 4。对筛选出的模块中的差异基因进行通路富集分析,< 0.05 具有统计学意义。
2 结果
2.1 尤文肉瘤组织和非尤文肉瘤组织的基因差异表达
通过limma 包对尤文肉瘤组织和非尤文肉瘤组织的基因进行筛选,共筛选出312 个差异基因,其中192 个上调基因,120 个下调基因,具体如图1 所示。
2.2 尤文肉瘤组织和非尤文肉瘤组织的功能富集
通过DAVID 数据库,我们进行功能富集。通过细胞组成的研究,我们根据差异大小进行排列,前5 个分别为:细胞外区域组分、细胞外区域、细胞外小泡、细胞外细胞器、细胞外泌体。通过对生物进程的分析,我们根据差异大小进行排列,前5 个分别为:细胞分化、系统发育、解剖结构、细胞发育、细胞发育过程。通过对分子功能的研究,根据差异大小进行排列,前 5 个分别为:蛋白结合、细胞黏附分子结合、蛋白质二聚体结合、胰岛素II 受体结合、细胞外基质结合。具体如图2 所示。
2.3 尤文肉瘤组织和非尤文肉瘤组织的基因互作网络分析
通过string 数据库进行PPI 网络图的绘制,我们得出此网络有243 个节点,152 条边。其中ANXA2 和S100A10,ANXA2 和S100A4,GRP 和CCK,LAMA5 和ITGA3,SCNN1A 和SG1C1 互作关系较为密切,这些相互作用的基因编码蛋白可能调控尤文肉瘤的产生。具体如图3 所示。
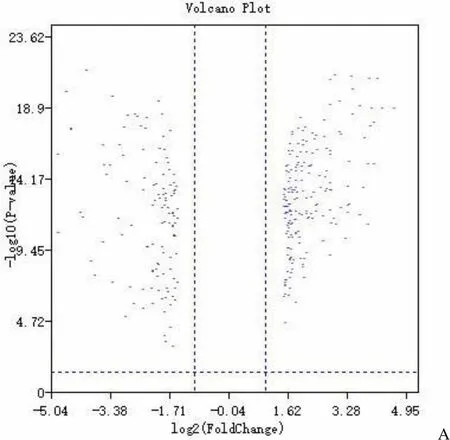
A
Figure 1 Volcano (A) and heatmap (B) of differential miRNA in Ewing sarcoma and normal tissues (Green: down-regulated miRNA; Red: up-regulated miRNA)
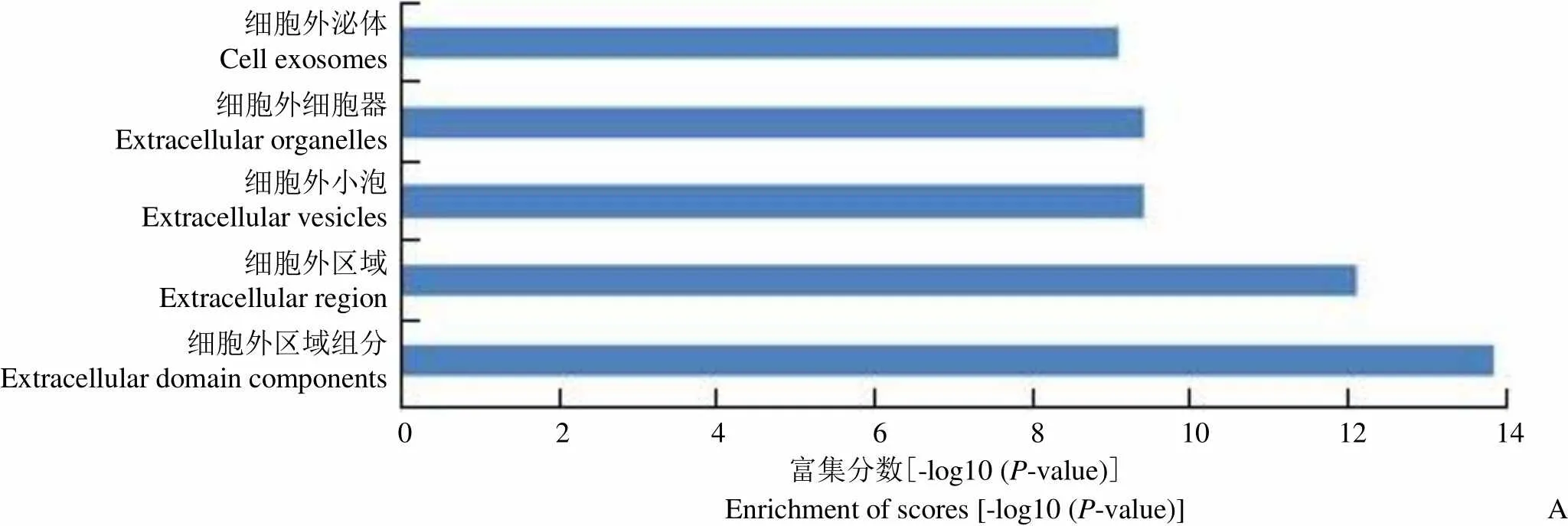
细胞外泌体Cell exosomes细胞外细胞器Extracellular organelles细胞外小泡Extracellular vesicles细胞外区域Extracellular region细胞外区域组分Extracellular domain components 0 2 4 6 8 10 12 14 富集分数[-log10 (P-value)]Enrichment of scores [-log10 (P-value)]A
Figure 2 Cell composition (A), biological process (B) and molecular function (C) analyzed by GO and KEGG
3 讨论
生物信息学的产生对于解释一些疾病的产生、发展与转归具有重要作用[4]。尤文肉瘤是仅次于骨肉瘤的第二常见的骨恶性肿瘤,在儿童和年轻人中发病,发病年龄在 15 岁时达到峰值[5]。尽管大多数尤文肉瘤都发生在骨骼中,尤其是骨盆,长骨的骨干区和胸壁的骨骼,但 15% 的原发性尤文肉瘤可能发生在各种骨外部位,包括深部软椎旁、胸廓和近端肢体组织、肾脏、膀胱、肺、前列腺和脑膜[6]。目前,尤文肉瘤可通过外科手术,放射疗法和化学疗法相结合的方法进行治疗,但是存活率仍然很差[7-10]。
细胞分化在尤文肉瘤的产生中起着重要作用。研究指出,尤文肉瘤具有神经分化潜能[11-12]。其与ETS 基因融合,分化成永生化的NIH3T3 成纤维细胞,从而增加其侵袭性,对预后产生不良影响[13]。细胞外区域对尤文肉瘤的产生起促进作用。研究指出,尤文肉瘤的结构中有许多FasL,此物质具有一个氨基末端胞质和一个羧基末端胞外区,可以促进尤文肉瘤的迁移[14]。蛋白结合在尤文肉瘤的产生和发展中具有很大的作用。尤文肉瘤中含有DNA 和RNA 结合蛋白,促进尤文肉瘤的突变,从而对尤文肉瘤的产生造成影响[15]。
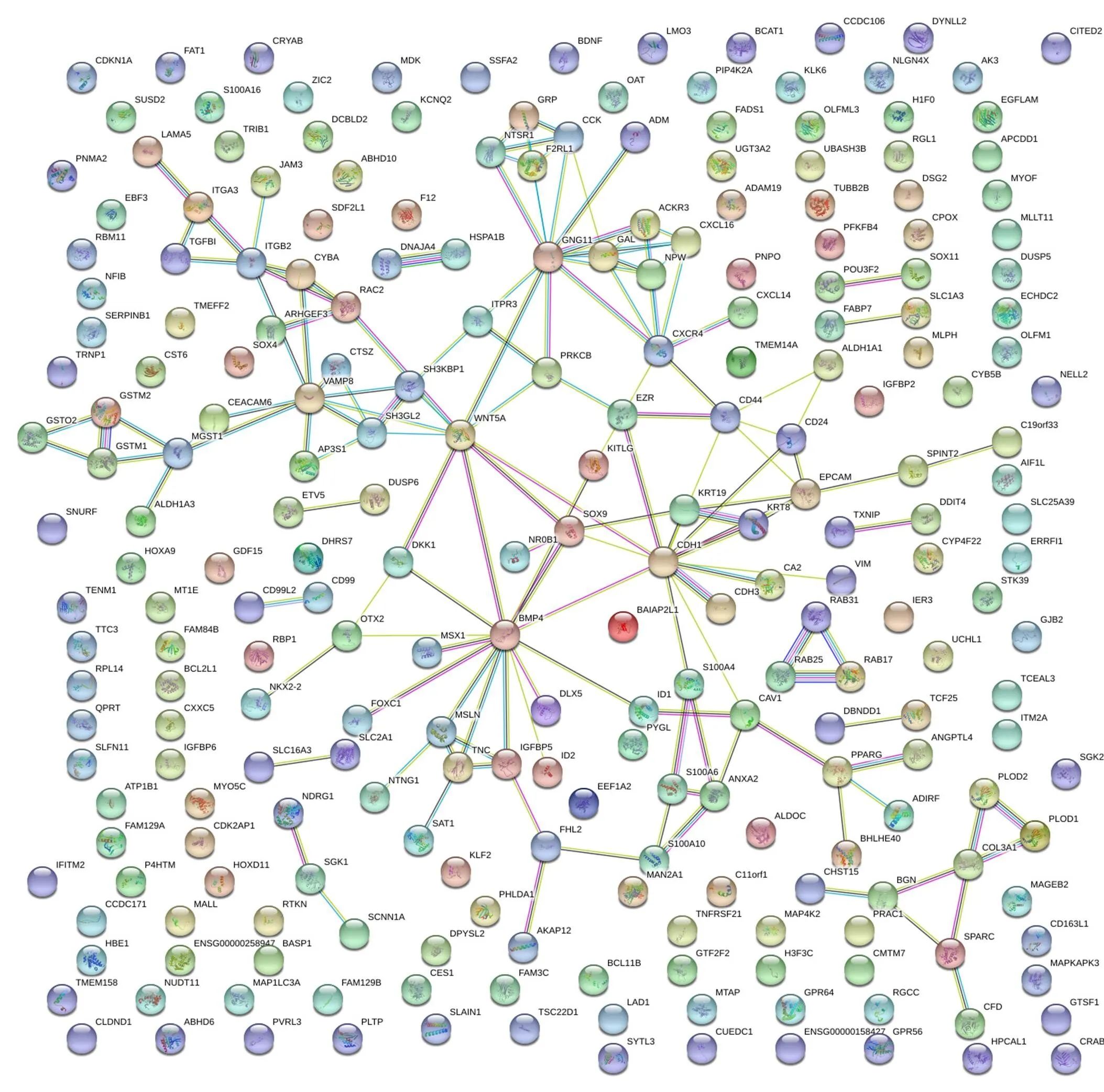
图 3 基因互作网络
Figure 3 PPI of the RNA
本研究发现ANXA2 和S100A10,ANXA2 和S100A4,GRP 和CCK,LAMA5 和ITGA3,SCNN1A 和SG1C1 互作关系较为密切,这些相互作用的基因编码蛋白可能调控尤文肉瘤的产生。随着RNA 研究技术的发展,对尤文肉瘤中的信号通路、表观遗传调控以及发病机制的进一步深入研究,部分差异表达的血清基因可以作为尤文肉瘤早期诊断潜在的生物标志物已经得到证实,恢复差异基因的生理水平可能为尤文肉瘤治疗的新策略。虽然缺乏大量临床标本证实这一观点,但仍为研究尤文肉瘤的治疗提供了新的方向。
[1] Gaspar N, Hawkins DS, Dirksen U, et al. Ewing sarcoma: current management and future approaches through collaboration. J Clin Oncol, 2016, 33(27):3036-3046.
[2] Riff AJ, Gross CE, Foucher KC, et al. Acetabular osteoarticular allograft after ewing sarcoma resection: a 15-year follow-up: a case report. JBJS Case Connect, 2016, 6(4):e89.
[3] Murthy SS, Gundimeda SD, Challa S, et al. FISH for EWSR1 in Ewing's sarcoma family of tumors: Experience from a tertiary care cancer center. Indian J Pathol Microbiol, 2021, 64(1):96-101.
[4] Huang DW, Sherman BT, Lempicki RA. Bioinformatics enrichment tools: paths toward the comprehensive functional analysis of large gene lists. Nucleic Acids Res, 2009, 37(1):1-13.
[5] Theisen ER, Pishas KI, Saund RS, et al. Therapeutic opportunities in Ewing sarcoma: EWS-FLI inhibition via LSD1 targeting. Oncotarget, 2016, 7(14):17616-17630.
[6] Pishas KI, Lessnick SL. Recent advances in targeted therapy for Ewing sarcoma. F1000Res, 2016, 5:F1000 Faculty Rev-2077.
[7] Reynolds A, Leake D, Boese Q, et al. Rational siRNA design for RNA interference. Nat Biotechnol, 2004, 22(3):326-330.
[8] Whitehead KA, Langer R, Anderson DG. Knocking down barriers: advances in siRNA delivery. Nat Rev Drug Discov, 2009, 8(2):129- 138.
[9] Flisiak R, Jaroszewicz J, Łucejko M. siRNA drug development against hepatitis B virus infection. Expert Opin Biol Ther, 2018, 18(6):609- 617.
[10] Fang T, Huang H, Li X, et al. Effects of siRNA silencing of TUG1 and LCAL6 long non-coding RNAs on patient-derived xenograft of non-small cell lung cancer. Anticancer Res, 2018, 38(1):179-186.
[11] Terashima T, Ogawa N, Nakae Y, et al. Gene therapy for neuropathic pain through siRNA-IRF5 gene delivery with homing peptides to microglia. Mol Ther Nucleic Acids, 2018, 11(C):203-215.
[12] Li S, Yang Q, Wang H, et al. Prognostic significance of serum lactate dehydrogenase levels in Ewing's sarcoma: a meta analysis. Mol Clin Oncol, 2016, 5(6):832-838.
[13] Gardiner JD, Abegglen LM, Huang X, et al. C/EBPβ-1 promotes transformation and chemoresistance in Ewing sarcoma cells. Oncotarget, 2017, 8(16):26013-26026.
[14] Von Heyking K, Roth L, Ertl M, et al. The posterior HOXD locus: Its contribution to phenotype and malignancy of Ewing sarcoma. Oncotarget, 2016, 7(27):41767-41780.
[15] Spaulding SL, Xing MH, Seo GT, et al. Extraskeletal Ewing sarcoma of the buccal space. Clin Imaging, 2020, 73:108-110.
Analysis of differential expression and functional prediction of genes in Ewing's sarcoma based on bioinformatics
HAN Guang-tao, LI Hao-huan
Department of Orthopedics, People's Hospital of Wuhan University, Hubei 430060, China
To analyze the differential expression and functional prediction of genes in Ewing's sarcoma based on bioinformatics, providing help for the treatment of Ewing sarcoma.The RNA data set was searched through GEO database, the differential genes were searched by limma package and visualized, the signal pathway analysis and GO enrichment analysis were performed, and the gene interaction network was finally analyzed.A total of 312 mRNA samples were selected from Ewing's sarcoma, among which 192 were up-regulated and 120 were down-regulated. Through the study of cell composition, the components of extracellular regions, extracellular regions, extracellular vesicles, extracellular organelles and exosomes had great influence on Ewing’s sarcoma. Through the analysis of biological processes, cell differentiation, phylogeny, anatomical structure, cell development, cell development process had a greater impact. Through the study of molecular functions, protein binding, cell adhesion binding, protein dimer binding, insulin II receptor binding and extracellular matrix binding had great influence. Through the gene interaction network, we found that ANXA2 and S100A10, ANXA2 and S100A4, GRP and CCK, LAMA5 and ITGA3, SCNN1A and SG1C1 closely interacted.ANXA2 and S100A10, ANXA2 and S100A4, GRP and CCK, LAMA5 and ITGA3, SCNN1A and SG1C1 closely interact, and these interacting gene-coding proteins may be responsible for Ewing's sarcoma.
bioinformatics; Ewing sarcoma; GEO
LI Hao-huan, Email: 1055891745@qq.com
国家自然科学基金(81171760)
李皓桓,Email:1055891745@qq.com
2020-08-25
10.3969/j.issn.1673-713X.2021.01.006
