陆地棉CNGC全基因组鉴定及表达分析
2021-01-07刘文豪田琴余渝王旭文马麒司爱君孔宪辉
刘文豪 田琴 余渝 王旭文 马麒 司爱君 孔宪辉
摘要:环状核苷酸门控通道(CNGC)基因家族是非选择性阳离子通道基因家族之一,在与植物发育和环境胁迫等有关的生理生化过程中起着至关重要的作用,但是目前尚无陆地棉CNGC基因家族的全基因组鉴定和分析。基于已知的拟南芥CNGC基因家族成员序列信息,以生物信息学方法分析陆地棉基因组中CNGC家族成员的理化性质、系统发育、染色体定位和差异表达情况。结果表明,共鉴定出33个GhCNGC基因,它们不均匀地分布在A、D染色体亚组上,其中15个基因分布在A染色体亚组上,18个基因分布在D染色体亚组上。系统发育分析结果表明,GhCNGC基因家族被分为4个主要组,由于在进化过程中不均等地扩增,Ⅳ组又分为Ⅳa和Ⅳb组。同组陆地棉、拟南芥的CNGC基因显示出相似的保守基序和基因结构,尤其是同源性越近,相似度越高。GhCNGC基因的表达谱以组织特异性模式表达,多数基因在根、叶中的表达量较高。研究结果使人们增加了对陆地棉和其他植物中CNGC基因家族的了解。
关键词:CNGC;基因家族;全基因组;陆地棉;染色体定位;差异表达
中图分类号:S562.01 文献标志码: A
文章编号:1002-1302(2021)24-0049-07
收稿日期:2021-05-10
基金项目:新疆生产建设兵团科技攻关项目(编号:2016AC027);新疆生产建设兵团重点领域创新团队建设计划(编号:2017CB011)。
作者简介:刘文豪(1992—),男,安徽界首人,硕士,助理研究员,主要从事棉花分子育种研究。E-mail:whylwh2016@163.com。
通信作者:孔宪辉,硕士,研究员,主要从事棉花育种研究。E-mail:xjkxh920@163.com。
植物中的Ca2+通过钙离子传导通道进行信号转导是一种重要的信号转导机制。信号转导有助于植物生长发育、病原体防御及植物对激素、光和盐胁迫的反应[1]。环状核苷酸门控通道(CNGC)是可渗透Ca2+的阳离子转运通道,调节植物生长并应对生物和非生物胁迫。CNGC位于细胞质膜或核膜,由Ca2+/钙调蛋白(CaM)和环状单磷酸核苷酸(cNMPs)等二级信使从细胞内部控制[2]。在植物中,CNGC由6个跨膜(TM)域和介于第5、第6个TM域之间的1个孔区域组成。环状核苷酸结合结构域(CNBD)是一个高度保守的区域,并有1个磷酸盐结合盒(PBC)和1个铰链区。CNGC通过直接结合环状核苷酸而被激活,当钙调蛋白(CaM)结合到CaM结合域时会被抑制[3]。目前,生物信息学工具已在水稻[4]、玉米[5]、拟南芥[6]、甘蓝[7]、白菜[8]、番茄[9]中用于识别CNGC基因家族成员。
之前的研究发现,CNGC是植物发育的关键组成部分。目前,大多数CNGC已通过遗传学方法得到表征,并且它们已显示出与植物生理、分子功能有关的重要作用,例如信号通路、植物发育和对环境胁迫的响应中涉及的多个生理过程。拟南芥环核苷酸门控通道2(AtCNGC2)参与拟南芥表皮细胞中茉莉酸(JA)诱导的质外体Ca2+流入[10],AtCNGC4可渗透K+、Na+,并被环磷酸鸟苷(cGMP)、环磷酸腺苷(cAMP)激活[11],AtCNGC7、AtCNGC8基因对雄性生殖力的作用至关重要[12],AtCNGC16、AtCNGC18基因可参与花粉发育[13],AtCNGC6、AtCNGC19、AtCNGC20基因参与了非生物胁迫反应[14]。
近年来,人们对植物中的CNGC基因家族进行了研究,然而,关于陆地棉(Gossypium hirsutum Linn.)CNGC(GhCNGC)基因家族的系統鉴定、起源和功能的研究却很少。本研究利用陆地棉全基因组序列信息、拟南芥CNGC家族的研究信息及综合生物信息学分析技术对陆地棉中CNGC进行全基因组鉴定来完成每个CNGC基因家族成员的深入分析,包括对编码蛋白的生理、生化特性分析。此外,本研究还分析了CNGC家族成员的表达方式,以阐明其对生物、非生物胁迫响应的机制,并鉴定出可能对育种有用的新基因。
1 材料与方法
1.1 陆地棉CNGC基因家族成员的筛选鉴定
陆地棉TM-1的基因组数据来自南京农业大学Cotton Research Institute网站 (http://mascotton.njau.edu.cn/),试验时间为2020年8月,试验地点为新疆维吾尔自治区石河子市新疆农垦科学院棉花研究所。以拟南芥CNGC基因家族成员的身份标志(ID)为探针在陆地棉TM-1基因组注释文件中查找陆地棉CNGC家族ID,用虚拟机Bio-Linux 80运行HMM-Search搜索结构域,并提取目标基因序列。
1.2 陆地棉CNGC基因家族的理化性质分析
通过ExPASY-ProtParam在线网站(https://web.expasy.org/protparam)对陆地棉CNGC蛋白家族氨基酸序列的分子式、总原子数、亲水性平均值、分子质量、蛋白不稳定系数等进行预测分析。通过SOPMA在线网站(https://npsa-prabi.ibcp.fr)分析陆地棉CNGC蛋白家族的二级结构。
1.3 陆地棉CNGC基因家族进化树的构建
将陆地棉、拟南芥的CNGC蛋白序列以fasta格式保存在1个文件中,用默认参数运行MEGA 7.0软件的ClustalW命令,进行序列对齐,采用邻接法(neighbor-joining,NJ)进行系统发育重建,以自举法(bootstrap method)进行系统发育计算,引导复制次数为1 000次,输出格式为Newick,通过Evolview在线网站(https://www.evolgenius.info/evolview/)进行进化树的编辑。
1.4 陆地棉CNGC基因家族染色体的定位及结构分析
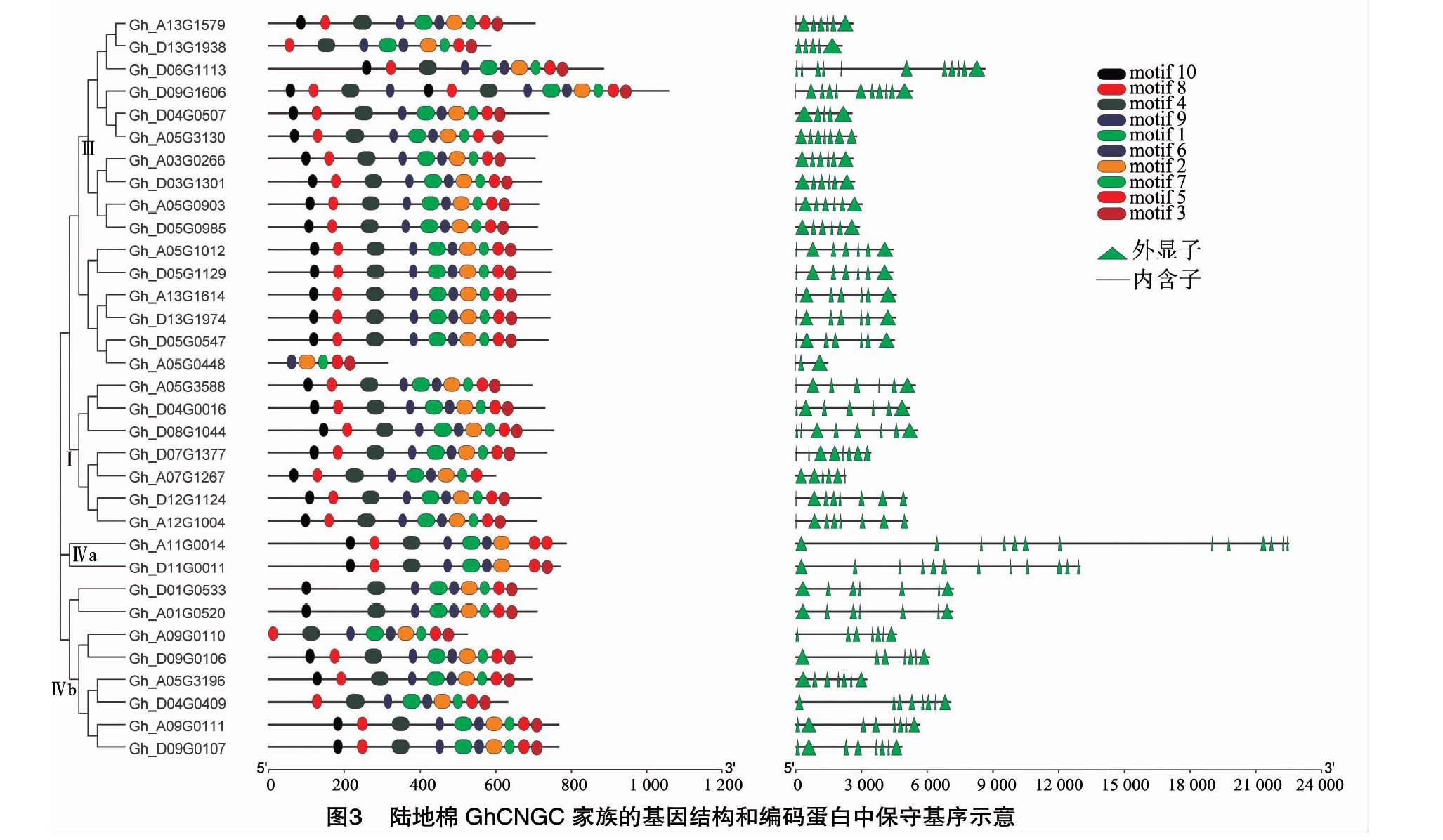
根据陆地棉基因组数据库中CNGC基因家族成员在染色体上的区域分布和染色体长度,通过Map Gene 2 Chrom在线网站(http://mg2c.iask.in/mg2c_v2.1/)绘制染色体分布图。通过MEME在线网站(http://meme-suite.org/)进行motif分析,搜索得到motif的总数为10个。用本地软件TBtools将CNGC基因家族进化树、MEME分析图和基因结构图进行合并分析。
1.5 陆地棉CNGC基因家族的差异表达分析
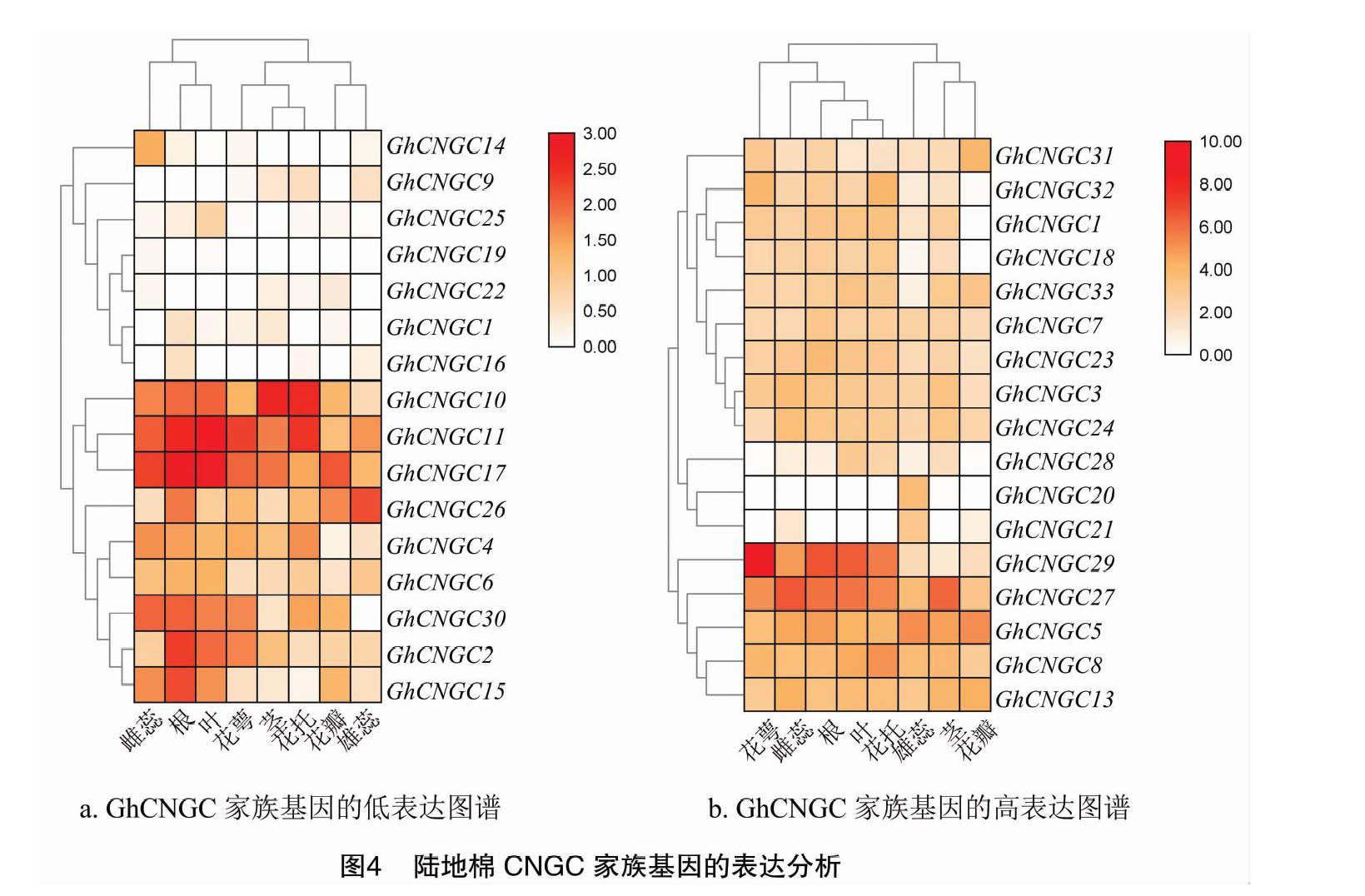
从美国国家生物信息中心(NCBI)数据库中下载TM-1标准系陆地棉转录组数据包PRJNA248163,以FPKM值作为参数,将FPKM值低于8的分为一组,高于8的分为另一组。用TB-tools软件进行基因表达图谱的绘制。
2 结果与分析
2.1 陆地棉CNGC基因家族的鉴定分析
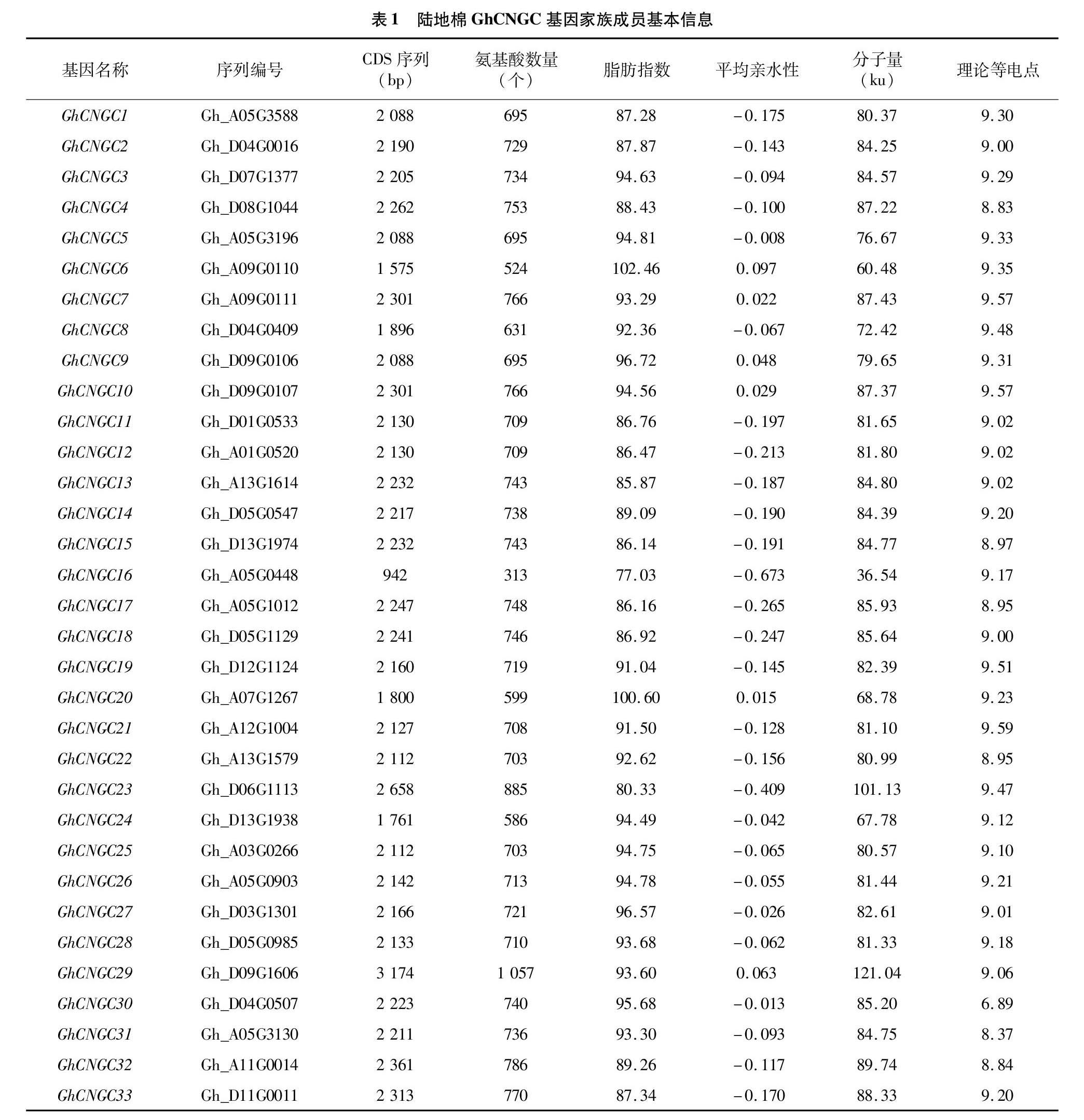
以已知的20个拟南芥CNGC基因家族成员的蛋白序列作为参考序列,在陆地棉标准系TM-1全基因组数据库中进行比对筛选,最终获得33个环状核苷酸门控通道(CNGC)基因家族成员。由表1可以看出,陆地棉环状核苷酸门控通道基因家族成员基因编码区(CDS)序列全长为942~3 174 bp,编码氨基酸313~1 057个,脂肪指数为77.03~102.46,平均亲水性为-0.673~0.097,都属于亲水性蛋白;氨基酸残基分子量为36.54~121.04 ku,理论等电点为689~9.59。
2.2 陆地棉CNGC基因家族的二级结构预测及蛋白的稳定性分析
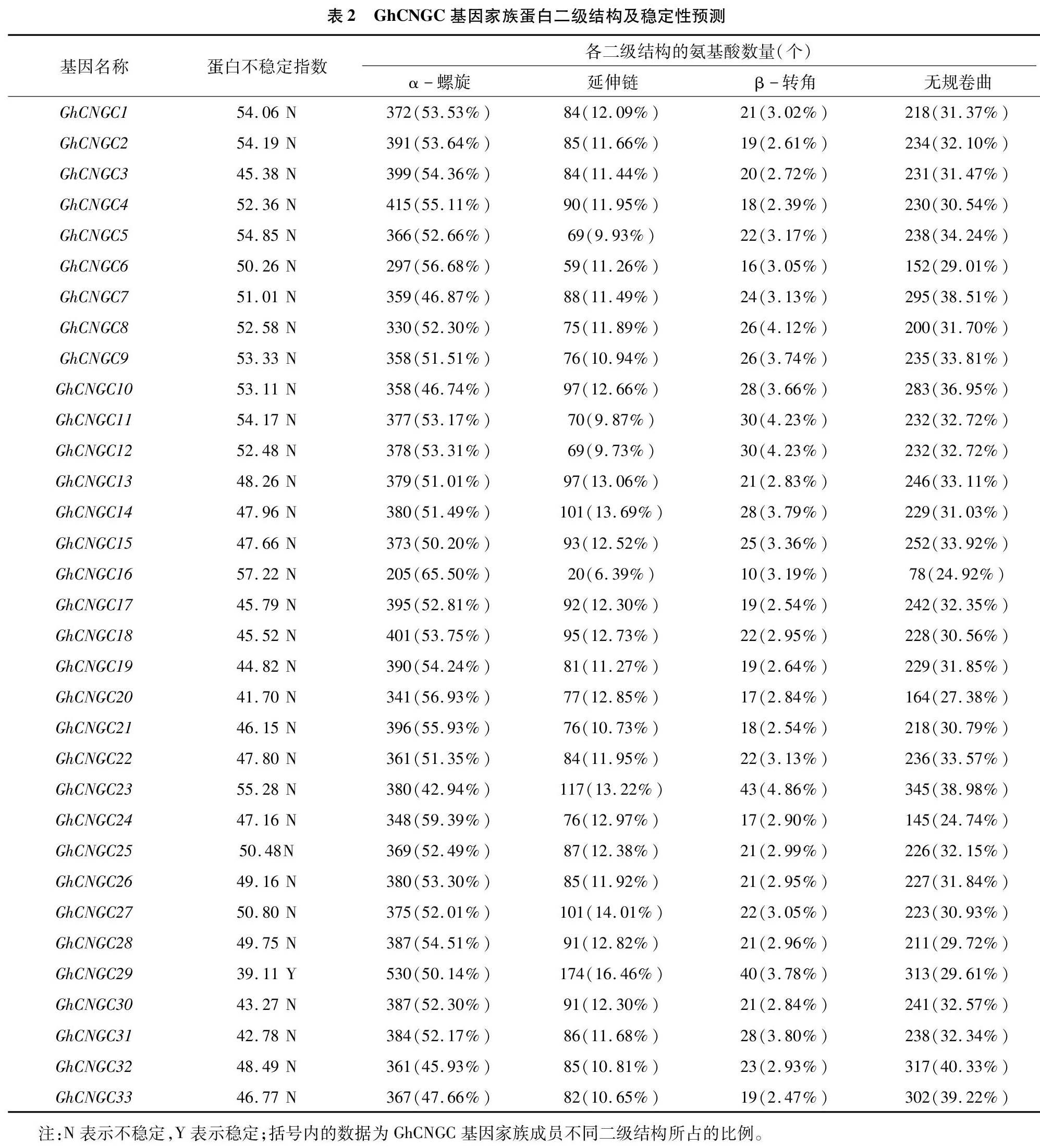
由表2可以看出,陆地棉CNGC基因家族成员的二级结构均由α-螺旋、延伸链、β-转角、无规卷曲4种结构组成,并且α-螺旋和无规卷曲是主要组成部分,所占比例最大。延伸链、β-转角包含的氨基酸残基数少,在二级结构中所占比例较小。根据蛋白不稳定指数统计结果发现,该家族成员的蛋白不稳定指数为39.11~57.22,仅GhCNGC29为稳定蛋白(稳定指数<40),其余的32个家族成员均为不稳定蛋白。
2.3 拟南芥与陆地棉CNGC基因家族的系统发育分析
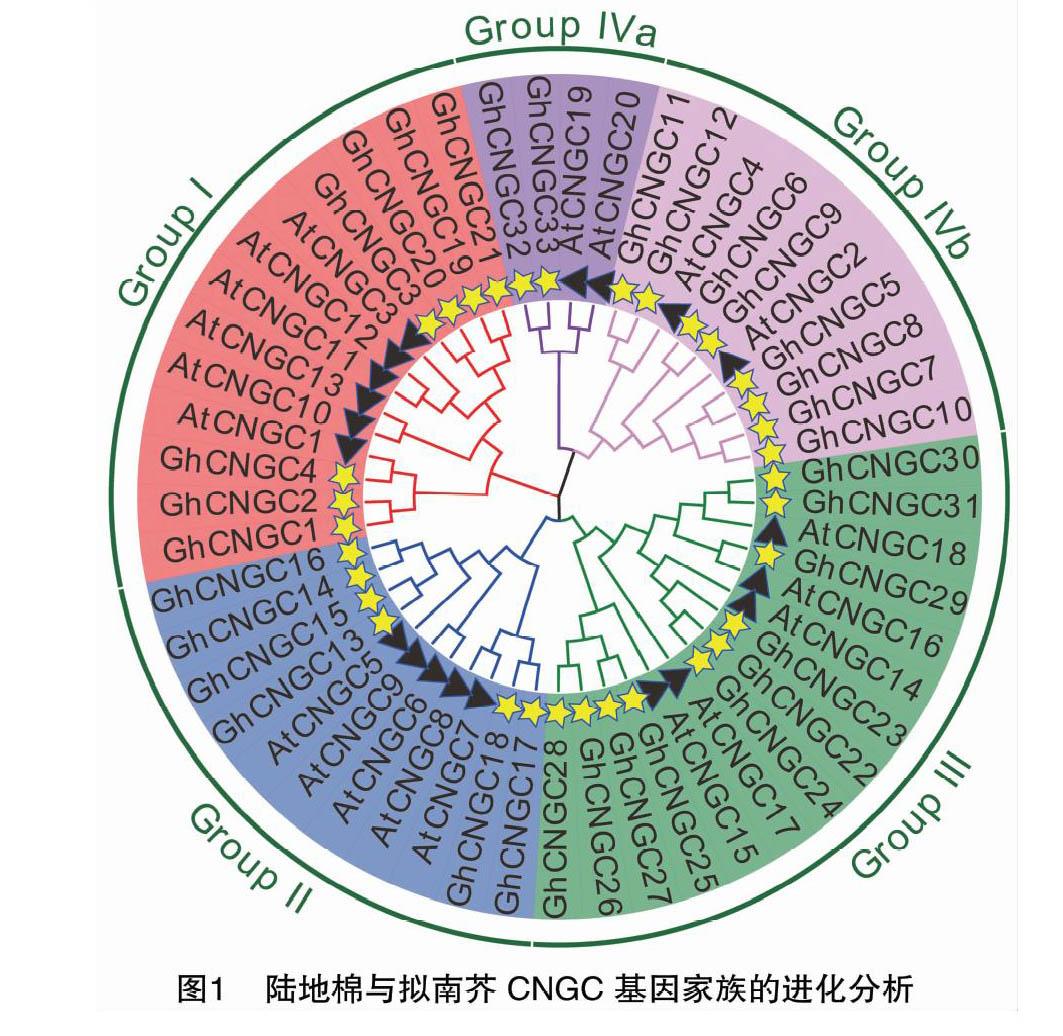
本研究从陆地棉标准系TM-1基因组数据库中筛选得到33个GhCNGC基因,根据这些基因在染色体上的分布情况,将这些基因命名为GhCNGC1~GhCNGC33。依据拟南芥AtCNGC基因家族的系统发育关系,将GhCNGC基因家族的33个成员与AtCNGC基因家族的20个成员的蛋白序列构建成系统进化树。由图1可以看出,GhCNGC基因家族成员的聚类情况与拟南芥相似,分为4个组群,分别为Group I、Group Ⅱ、Group Ⅲ、Group Ⅳ,其中Group Ⅳ又分为Group Ⅳ a和Group Ⅳ b。Group I含有13个成员,包括7个GhCNGC(GhCNGC1、GhCNGC2、GhCNGC3、GhCNGC4、GhCNGC19、GhCNGC20、GhCNGC21)和6个AtCNGC(AtCNGC1、AtCNGC3、AtCNGC10、AtCNGC11、AtCNGC12、AtCNGC13);Group Ⅱ含有6个GhCNGC(GhCNGC13、GhCNGC14、GhCNGC15、GhCNGC16、GhCNGC17、GhCNGC18)和5个AtCNGC(AtCNGC5、AtCNGC6、AtCNGC7、AtCNGC8、AtCNGC9);Group Ⅲ含有10个GhCNGC (GhCNGC22、GhCNGC23、GhCNGC24、GhCNGC25、GhCNGC26、GhCNGC27、GhCNGC28、 GhCNGC29、 GhCNGC30、GhCNGC31)和5个AtCNGC(AtCNGC14、AtCNGC15、AtCNGC16、AtCNGC17、AtCNGC18);Group Ⅳa含有2个GhCNGC(GhCNGC32、GhCNGC33)和2個AtCNGC(AtCNGC19、AtCNGC20);Group Ⅳb含有8个GhCNGC(GhCNGC5、GhCNGC6、GhCNGC7、GhCNGC8、GhCNGC9、GhCNGC10、GhCNGC11、GhCNGC12)和2个AtCNGC(AtCNGC2、AtCNGC4)。
2.4 陆地棉CNGC家族成员的染色体定位
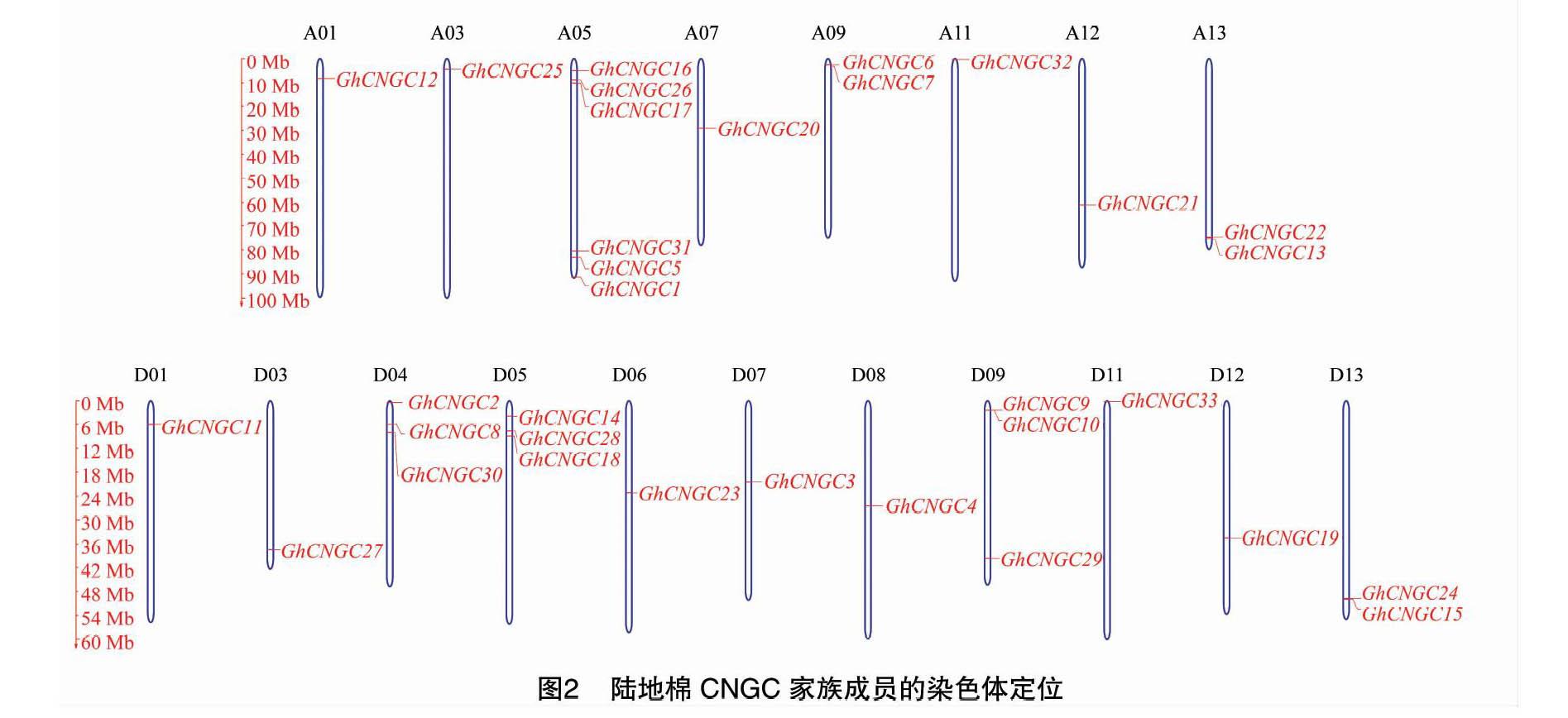
根据陆地棉标准系TM-1的基因组数据库资源,通过网站MapGene2Chrom进行在线GhCNGC家族成员的染色体定位分析[15]。将GhCNGC家族的33个成员定位在19条染色体上,并绘制该家族的基因图谱。结果(图2)显示,分布在D基因组亚组的基因最多,有18个,而这18个基因主要分布在染色体的两端,分布在上端的基因比分布在下端的基因多,分布在中间部位的基因仅有3个。分布在A基因组亚组的基因相对较少,有15个,这15个基因依然主要分布在染色体的两端。分布在D04、D05、D09和A05染色体上的基因最多,分别为3、3、3、6个,而其他染色体上仅分布1~2个该家族基因成员。
2.5 陆地棉CNGC的系统进化和基因结构分析
通过MEME在线网站[16]及TB-tools软件[17]分析GhCNGC家族成员的保守基序、内含子和外显子的数量及分布。由图3可以看出,同一亚家族成员分布在同一进化分支上,与图1中的进化分析结果相同。分析motif的数量及位置发现,同一组成员的motif数量基本相同,且分布的位置较接近,在不同组之间存在差异。该家族33个成员中的24个成员均含有10个motif,8个成员的motif数量为9个,1个成员的motif数量为5个。而motif数量为9个的成员大多分布在Ⅳ组,可能由于Ⅳ组与其他3组的亲缘关系较远。有趣的是,Ⅱ组中的GhCNGC16仅有5个motif基序。从在内含子与外显子的分布情况看出,多数同一组成员的内含子及外显子数量较为接近,I组7个成员外显子数为6~8个;Ⅱ组6个成员的外显子数量为3~7个,除GhCNGC之外,均为7个外显子;Ⅲ组的外显子数量为5~11个;Ⅳ组的外显子数量为7~13个, 但是Ⅳ a的2个成员的外显子数量分别为12、13个,Ⅳ b的成员中,5个外显子数量为7个,3个外显子数量为8个。而且同一组成员的外显子、内含子分布情况类似,差异显著性小,表明了进化的保守性。不同组之间的差异较为显著,尤其是Ⅳ组的成员之间及其与其他组相比较差异较大。
2.6 陆地棉CNGC家族成员表达分析
从NCBI网站下载陆地棉TM-1的PRJNA248163数据资源,分析GhCNGC家族基因的表达模式,选取根、茎、叶、雄蕊、雌蕊、花托、花萼和花瓣等8个组织进行预测。图4-a中16个基因的表达量较低,图4-b中17个基因的表达量较高。不同GhCNGC家族基因在不同组织中的表达量存在差异,GhCNGC10在茎、花托中的表达量高,GhCNGC11在根、叶、花托中的表达量高,GhCNGC17在茎中的表达量高,GhCNGC26在雄蕊中的表达量高,GhCNGC29在花萼中的表达量高,GhCNGC27在雌蕊中的表达量高。GhCNGC2、GhCNGC11、GhCNGC15、GhCNGC17、GhCNGC26和GhCNGC30在根中的表达量相对较高。由此可见,GhCNGC10、GhCNGC11、GhCNGC27、GhCNGC29可能参与陆地棉的生殖过程,GhCNGC17、GhCNGC2、GhCNGC15可能参与到陆地棉的营养生长过程,而GhCNGC10、GhCNGC11、GhCNGC17、GhCNGC29等基因在多个组织中的表达量均较高,可能这些基因具有一因多效的特点。
3 讨论
环核苷酸门控通道是配体门控、钙离子渗透的二价阳离子选择性通道,在非生物胁迫信号传导相关的信号传导过程中具有重要的生物学功能。本研究基于电生理和异源表达分析,用正向遗传方法明确了拟南芥中的AtCNGC2、AtCNGC4、AtCNGC11和AtCNGC12参与了植物免疫過程[18]。据报道,其生物学作用在防御反应、发育和离子体内平衡方面具有重要作用。目前,枣树[19]、水稻[20]、梨[2]、烟草[21]、小麦[22]等植物的CNGC蛋白均有相应研究。虽然CNGC在植物生存及对环境的胁迫响应中起着关键作用,但人们对陆地棉中CNGC家族所扮演的角色和功能知之甚少。陆地棉是全球重要的经济作物,本研究在陆地棉TM-1全基因组数据库[23]中筛选鉴定获得33个GhCNGC蛋白。通过分析确定33个GhCNGC蛋白的生理生化特征,陆地棉环状核苷酸门控通道基因家族成员CDS序列全长为942~3 174 bp,编码的氨基酸数量为313~1 057个。脂肪指数为77.03~102.46,平均亲水性为-0.673~0.097,都属于亲水性蛋白。理论等电点为6.89~9.59,且等电点、蛋白质电荷对于溶解度,亚细胞定位和相互作用非常重要,这取决于直系同源物之间的插入和缺失及有机体的生态[24]。根据蛋白不稳定指数统计结果可知,该家族成员的蛋白不稳定指数为39.11~57.22,仅GhCNGC29为稳定蛋白(稳定指数<40),其余32个家族成员均为不稳定蛋白。陆地棉是四倍体,具有A、D 2个亚基因组,染色体定位分析发现,这33个GhCNGC蛋白有15个存在于A亚基因组上,18个存在于D亚基因组上。由系统发育分析结果可知,GhCNGC家族成员分为I、Ⅱ、Ⅲ、Ⅳ等4个组,第Ⅳ组又分为Ⅳ a、Ⅳ b 2个亚组,与拟南芥CNGC家族分类相似[25]。这些数据表明,基因在进化过程中发生了分离。不同分组中包含的家族成员数量不同,可能由于在基因家族进化过程中,基因复制、基因丢失扮演着重要角色,从而创造新的基因和不同的功能[26],以优化植物的适应性。分析基因差异表达图谱发现,GhCNGC10、GhCNGC11、GhCNGC27、GhCNGC29在花托、花萼、雌蕊中表现出差异表达,表明它们可能参与到陆地棉的生殖过程中。而GhCNGC10、GhCNGC11、GhCNGC17、GhCNGC29等基因在多个组织中均有较高表达量,可能由于这些基因参与了多个代谢通路。
本研究以生物信息学和比较基因组学方法,分析了关于结构域,外显子-内含子结构以及系统进化树和表达分析的全面信息。已知植物CNGC对多种非生物刺激起着关键作用,包括冷胁迫、盐胁迫、激素应答、发育和光信号传导等[27],这些信息可以用于分析蛋白质相互作用网络,可为改善陆地棉的抗逆性、分子育种、纤维品质等提供重要参考。
4 结论
综上所述,本研究通过生物信息学手段从陆地棉基因组中全面鉴定出33个CNGC基因,分别位于A、D亚基因组中的CNGC家族中,分别有15、18个成员。本研究还分析了系统发育、保守基序和外显子等,以转录组数据为基础分析了该家族成员在不同组织中的差异性表达。总体来看,对陆地棉基因组中GhCNGC基因家族的分析对植物CNGC功能研究提供了综合概述,并为进一步阐明GhCNGC基因家族基因间相互作用的机制提供了前景。
参考文献:
[1]DeFalco T A,Marshall C B,Munro K,et al. Multiple calmodulin-binding sites positively and negatively regulate Arabidopsis CYCLIC NUCLEOTIDE-GATED CHANNEL12[J]. Plant Cell,2016,28(7):1738-1751.
[2]Chen J Q,Yin H,Gu J P,et al. Genomic characterization,phylogenetic comparison and differential expression of the cyclic nucleotide-gated channels gene family in pear (Pyrus bretchneideri Rehd.)[J]. Genomics,2015,105(1):39-52.
[3]Zelman A K,Dawe A,Berkowitz G A. Identification of cyclic nucleotide gated channels using regular expressions[J]. Methods in Molecular Biology,2013,1016:207-224.
[4]Nawaz Z,Kakar K U,Saand M A,et al. Cyclic nucleotide-gated ion channel gene family in rice,identification,characterization and experimental analysis of expression response to plant hormones,biotic and abiotic stresses[J]. BMC Genomics,2014,15(1):853.
[5]Hao L D,Qiao X L. Genome-wide identification and analysis of the CNGC gene family in maize[J]. Peer J,2018,6:e5816.
[6]Moon J Y,Belloeil C,Ianna M L,et al. Arabidopsis CNGC family members contribute to heavy metal ion uptake in plants[J]. International Journal of Molecular Sciences,2019,20(2):413.
[7]Kakar K U,Nawaz Z,Kakar K,et al. Comprehensive genomic analysis of the CNGC gene family in Brassica oleracea:novel insights into synteny,structures,and transcript profiles[J]. BMC Genomics,2017,18(1):869.
[8]Li Q Q,Yang S Q,Ren J,et al. Genome-wide identification and functional analysis of the cyclic nucleotide-gated channel gene family in Chinese cabbage[J]. 3 Biotech,2019,9(3):114.
[9]Saand M A,Xu Y P,Munyampundu J P,et al. Phylogeny and evolution of plant cyclic nucleotide-gated ion channel (CNGC) gene family and functional analyses of tomato CNGCs[J]. DNA Research,2015,22(6):471-483.
[10]Lu M,Zhang Y Y,Tang S K,et al. AtCNGC2 is involved in jasmonic acid-induced calcium mobilization[J]. Journal of Experimental Botany,2016,67(3):809-819.
[11]Ali R,Ma W,Lemtiri-Chlieh F,et al. Death don’t have no mercy and neither does calcium:Arabidopsis CYCLIC NUCLEOTIDE GATED CHANNEL2 and innate immunity[J]. Plant Cell,2007,19(3):1081-1095.
[12]Tunc-Ozdemir M,Rato C,Brown E,et al. Cyclic nucleotide gated channels 7 and 8 are essential for male reproductive fertility[J]. PLoS One,2013,8(2):e55277.
[13]Tunc-Ozdemir M,Tang C,Ishka M R,et al. A cyclic nucleotide-gated channel (CNGC16) in pollen is critical for stress tolerance in pollen reproductive development[J]. Plant Physiology,2013,161(2):1010-1020.
[14]Gao F,Han X W,Wu J H,et al. A heat-activated calcium-permeable channel-Arabidopsis cyclic nucleotide-gated ion channel 6-is involved in heat shock responses[J]. Plant Journal,2012,70(6):1056-1069.
[15]晁江濤,孔英珍,王 倩,等. MapGene2Chrom基于Perl和SVG语言绘制基因物理图谱[J]. 遗传,2015,35(1):91-97.
[16]Bailey T L,Boden M,Buske F A,et al. MEME SUITE:tools for motif discovery and searching[J]. Nucleic Acids Research,2009,37:202-208.
[17]Chen C J,Chen H,Zhang Y,et al. TBtools:an integrative Toolkit developed for interactive analyses of big biological data[J]. Molecular Plant,2020,13(8):1194-1202.
[18]Moeder W,Urquhart W,Ung H,et al. The role of cyclic nucleotide-gated ion channels in plant immunity[J]. Molecular Plant,2011,4(3):442-452.
[19]Wang L X,Li M,Liu Z G,et al. Genome-wide identification of CNGC genes in Chinese jujube (Ziziphus jujuba Mill.) and ZjCNGC2 mediated signalling cascades in response to cold stress[J]. BMC Genomics,2020,21(1):191.
[20]Cui Y M,Lu S,Li Z,et al. CYCLIC NUCLEOTIDE-GATED ION CHANNELs 14 and 16 promote tolerance to heat and chilling in rice[J]. Plant Physiology,2020,183(4):1794-1808.
[21]Nawaz Z,Kakar K U,Ullah R,et al. Genome-wide identification,evolution and expression analysis of cyclic nucleotide-gated channels in tobacco (Nicotiana tabacum L.)[J]. Genomics,2019,111(2):142-158.
[22]Guo J,Islam M A,Lin H C,et al. Genome-wide identification of cyclic nucleotide-gated ion channel gene family in wheat and functional analyses of TaCNGC14 and TaCNGC16[J]. Front Plant Sci,2018,9:18.
[23]Zhang T Z,Hu Y,Jiang W K,et al. Sequencing of allotetraploid cotton (Gossypium hirsutum L. acc. TM-1) provides a resource for fiber improvement[J]. Nature Biotechnology,2015,33(5):531-537.
[24]Khaldi N,Shields D C. Shift in the isoelectric-point of milk proteins as a consequence of adaptive divergence between the milks of mammalian species[J]. Biol Direct,2011,6:40.
[25]Mser P,Thomine S,Schroeder J I,et al. Phylogenetic relationships within cation transporter families of Arabidopsis[J]. Plant Physiology,2001,126(4):1646-1667.
[26]Chauve C,Doyon J P,El-Mabrouk N. Gene family evolution by duplication,speciation,and loss[J]. Journal of Computational Biology,2008,15(8):1043-1062.
[27]Fu Y P,Duan X Y,Tang C L,et al. TaADF7,an actin-depolymerizing factor,contributes to wheat resistance against Puccinia striiformis f. sp. tritici[J]. Plant J,2014,78(1):16-30.
