应用光谱分析方法测定牛肝菌的产地和不同部位矿物质含量
2020-12-04陈凤霞杨天伟李杰庆刘鸿高范茂攀王元忠
陈凤霞,杨天伟,李杰庆,刘鸿高,范茂攀*,王元忠
1. 云南农业大学资源与环境学院,云南 昆明 650201 2. 云南省热带作物科学研究所,云南 景洪 666100 3. 云南农业大学农学与生物技术学院,云南 昆明 650201 4. 云南省农业科学院药用植物研究所,云南 昆明 650200
引 言
云南省地形地貌复杂,独特的立体气候条件使得云南野生菌种类丰富多样[1]。 野生食用菌备受消费者喜爱,因具有较大的食用价值和经济价值而逐渐国际化[2]。 美味牛肝菌则是世界著名野生食用菌,也被称为“王者牛肝菌”,其肉质细腻、味道鲜美,富含多种功能性物质,兼具食药用价值; 其菌在云南分布广、产量高,但受地形气候的影响,不同产地的美味牛肝菌品质差异明显,营养物质和化学成分含量具有差异性[3-4]。 传统的菇类鉴别方法鉴别野生食用菌,主要以产地、子实体形状、菌体颜色、气味以及分泌物来进行区分,但牛肝菌种类多,种间形态较为相似,仅凭传统方法鉴别难以达到预期效果[5]。 我国是野生菌生产和出口大国,市面上销售的野生食用菌优掺劣、真掺假、中毒等安全问题突出,相关部门难以管控。 寻找一种准确快速的野生食用菌鉴别方法有重要性意义。
光谱技术具有低成本、易操作、稳定可靠等特点,在食品分析中应用较为广泛。 Giraudo[6]等根据光谱特征结合化学计量学成功对咖啡豆进行了产地鉴别; 有报道在牛肝菌产地鉴别中使用了电感耦合等离子体原子发射光谱法与红外光谱法; Cebi[7]等利用傅里叶变换衰减全反射红外光谱法对明胶橡皮糖进行分类的效果明显。 数据融合是将多种数据信息整合、优化从而提高信息的采集能力与可信度[8]。 大多数单一光谱信息提取可能会因为环境、仪器等原因导致结果产生偏差,而数据融合结合光谱技术则可以解决单一光谱信息的片面性。 Pizarro[9]等使用光谱指纹图谱结合PLS-DA的数据融合成功对西班牙不同产地特级初榨橄榄油进行了分类。 有研究采用FTIR与UV-Vis结合中级数据融合成功对牛肝菌进行了种类和产地鉴别。 以上研究均采用光谱技术、数据融合等方式对食品进行分类分析并取得了较好的结果。
本研究采用FTIR-MIR、FTIR-NIR、UV-Vis、元素等多元数据融合结合PLS-DA和SVM模型分类方法,对美味牛肝菌进行产地溯源研究,寻找最佳的产地鉴别方法。 为美味牛肝菌质量管控与优质选择提供借鉴方法,保护消费者权益。
1 实验部分
1.1 材料
不同产地美味牛肝菌样品详细采集地点见表1,样品经云南农业大学刘鸿高教授鉴定为成熟期美味牛肝菌。 采集的样品用陶瓷刀刮去表面泥土、杂物并用自来水清洗干净,将样品的盖和柄分开切片后置于50 ℃烘箱内烘干,粉碎后存放于聚乙烯自封袋备用。
1.2 数据信息采集
FTIR-MIR: 称取(1.50±0.20) mg样品粉末与(100.00±2.00) mg溴化钾置于玛瑙研钵中混合研磨后用模具压成透明薄片。 Frontier型傅里叶变换红外光谱仪(Perkin Elmer 公司,USA)采集光谱范围为4 000~400 cm-1,累计扫描16次,仪器分辨率为4 cm-1。 FTIR-NIR: 取(20.00±0.50) g样品均匀混合,置于玻璃器皿中压缩,Antaris Ⅱ型傅里叶变换近红外光谱仪(Thermo Fisher公司,USA)在波数为10 000~4 000 cm-1范围扫描,分辨率为4 cm-1,信号累计扫描64次。 UV-Vis: TU-1901紫外-可见分光光度计预热1 h,加入甲醇的石英比色皿进行扫描扣除背景值。 将(100.00±0.20) mg样品粉末与10 mL甲醇置于试管中进行溶解并超声40 min采集并截取特征吸收峰光谱范围200~400 cm-1之间的变量。 元素: 取美味牛肝菌盖、柄样品(200.00±0.10) mg与5 mL硝酸和2 mL过氧化氢置于消解罐中加盖密封,放入微波消解仪,其中升温时间与保温时间为5 min、功率为1 500 kW、温度依次以120,150,170和180 ℃。 消解结束后转入比色管用超纯水进行25 mL定容。 利用相同的方法测茶叶标准物GBW07605与空白样品。 所用数据每个样品一式三份,取平均值。
1.3 数据处理
使用OMNIC8.0软件对光谱数据进行基线校正和吸光度转换,将光谱数据用SIMCA13.0软件分别进行平滑(Savitzky-Golay)、二阶导数(second derivatives,SD)、标准正态变换(standard normal variables, SNV)等预处理。 采用Kennard-Stone算法筛选2/3(83)数据作为训练集1/3(41)数据作为预测集。 利用SIMCA13.0与MATLABR2018a分别建立PLS-DA与SVM模型。 通过模型正确率来筛选最佳的产地鉴别结果。
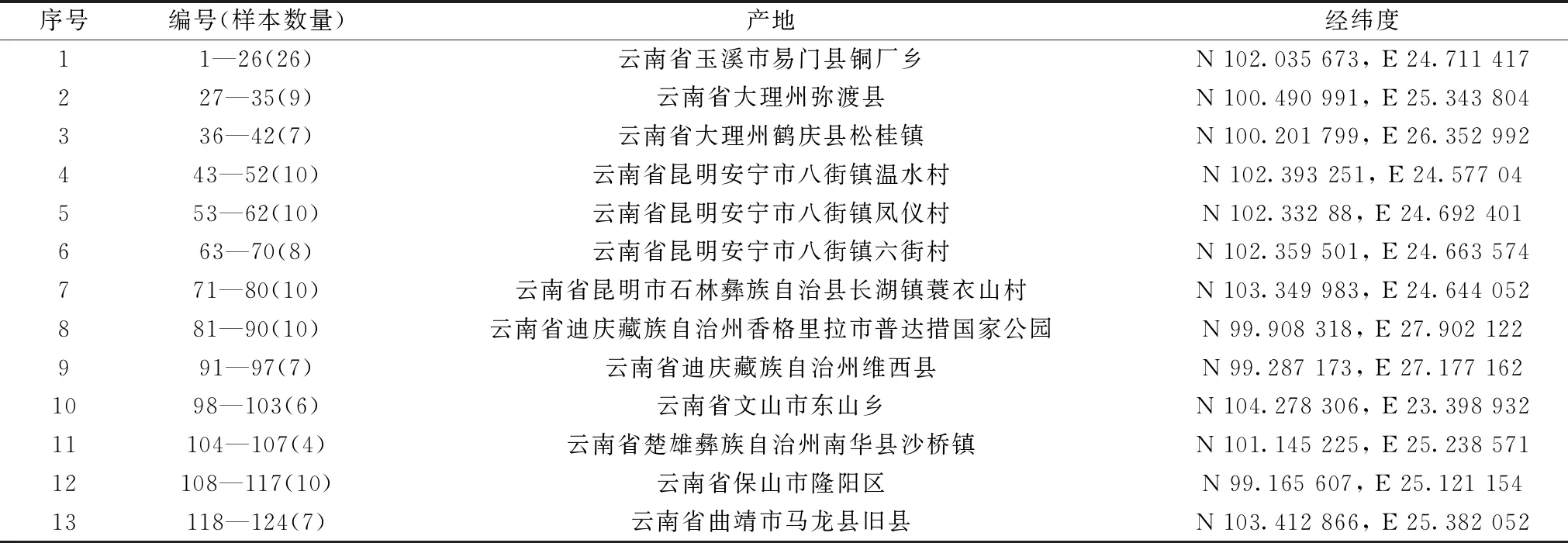
表1 美味牛肝菌产地来源信息Table 1 The geographical origin information of Boletus edulis
2 结果与讨论
2.1 元素测定方法
茶叶标准物质GBW07605元素测定结果如表2,其中回收率在91%~106%之间,测定值与真实值结果接近。 表明此方法对美味牛肝菌元素含量的测定准确可靠。
2.2 美味牛肝菌矿质元素分析
13个产地美味牛肝菌菌盖与菌柄矿质元素含量范围如表3所示分别是Ba: 17.50~159.44,15.72~181.98 mg·kg-1; Ca: 104.74~467.72,115.27~571.58 mg·kg-1; Cr: 17.70~524.24,10.09~354.27 mg·kg-1; Cu: 24.03

表2 茶叶标准物GBW07605的元素测定结果Table 2 The element determination results ofcertified reference material GBW07605
~53.15,15.68~77.93 mg·kg-1; K: 7 142.86~14 701.82,5 636.27~14 279.42 mg·kg-1; Mg: 421.44~976.09,246.24~924.63 mg·kg-1; Na: 17.89~716.17,18.84~887.34 mg·kg-1; Ni: 9.66~60.32,8.98~76.02 mg·kg-1; P: 4 414.57~7 311.13,2 198.33~2 423.69 mg·kg-1; Sr: 2.10~143.60,3.99~176.87 mg·kg-1; V: 4.31~54.09,5.28~60.31 mg·kg-1; Zn: 86.24~165.06,39.90~162.54 mg·kg-1。 美味牛肝菌中富含K,P,Mg,Na和Ca等矿质元素。 大部分产地美味牛肝菌矿质元素含量菌盖大于菌柄,除个别产地不同部位元素含量差异明显外,菌盖和菌柄之间矿质元素含量差异幅度较小。 K在美味牛肝菌中含量最高,其中云南省昆明安宁市八街镇凤仪村美味牛肝菌菌盖K元素已达到14 701.82 mg·kg-1; 其次是P,范围在2 198.33~7 311.13 mg·kg-1之间,最大值与最小值之间相差3.33倍。 不同产地Cr含量差距较大,云南省楚雄彝族自治州南华县沙桥镇美味牛肝菌菌盖是云南省文山市东山乡美味牛肝菌菌盖的29.62倍; 不同产地V含量最大值与最小值也相差13.99倍。 美味牛肝菌相同产地不同部位之间,不同产地相同元素之间存在差异性。 这有可能与美味牛肝菌不同部位富集差异及云南不同的环境、地形、土壤情况等因素有关。
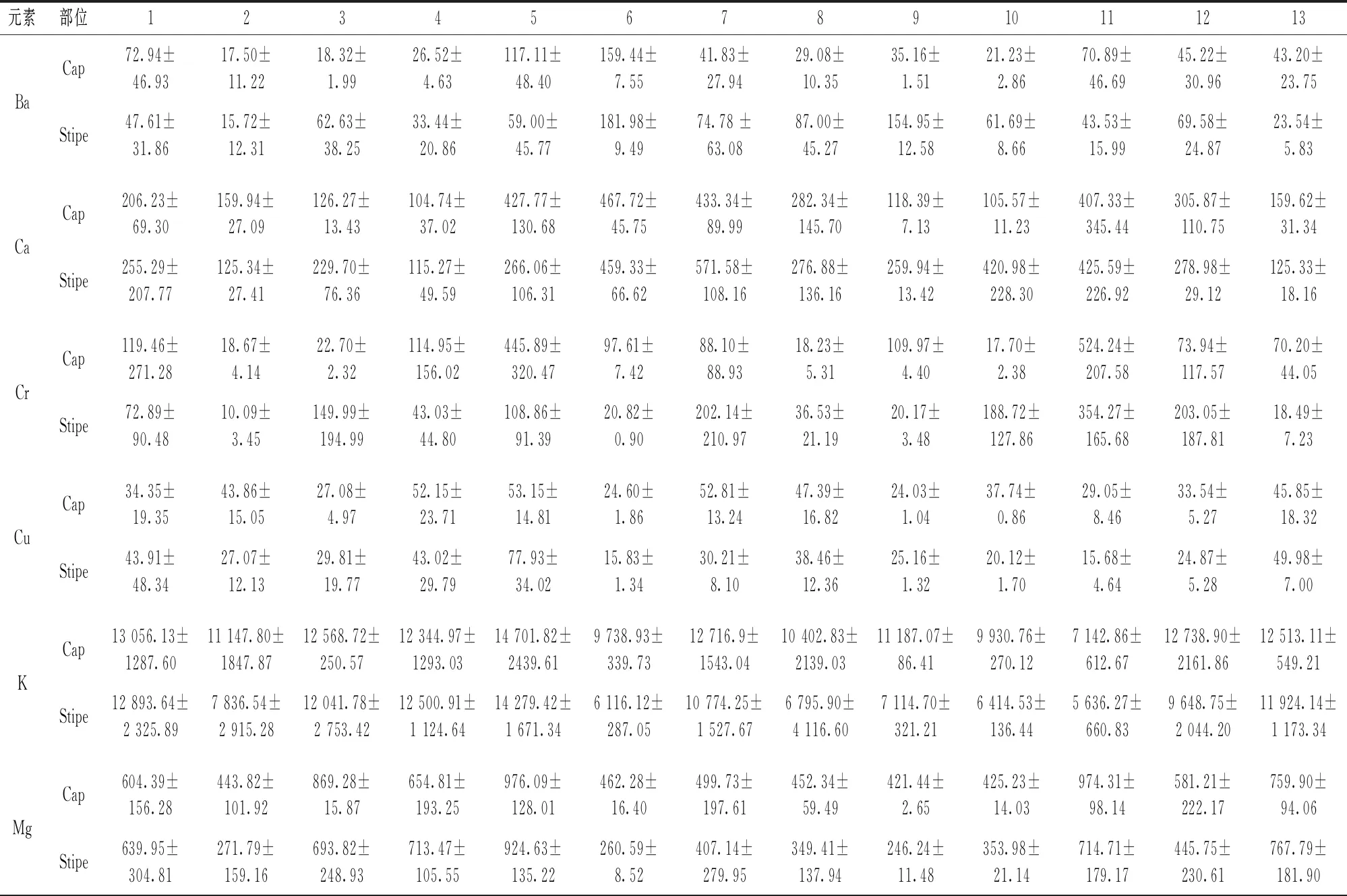
表3 美味牛肝菌不同部位矿质元素含量(Mean±SD)Table 3 Elements concentrations(Mean±SD)in Cap and Stipe of Boletus edulis
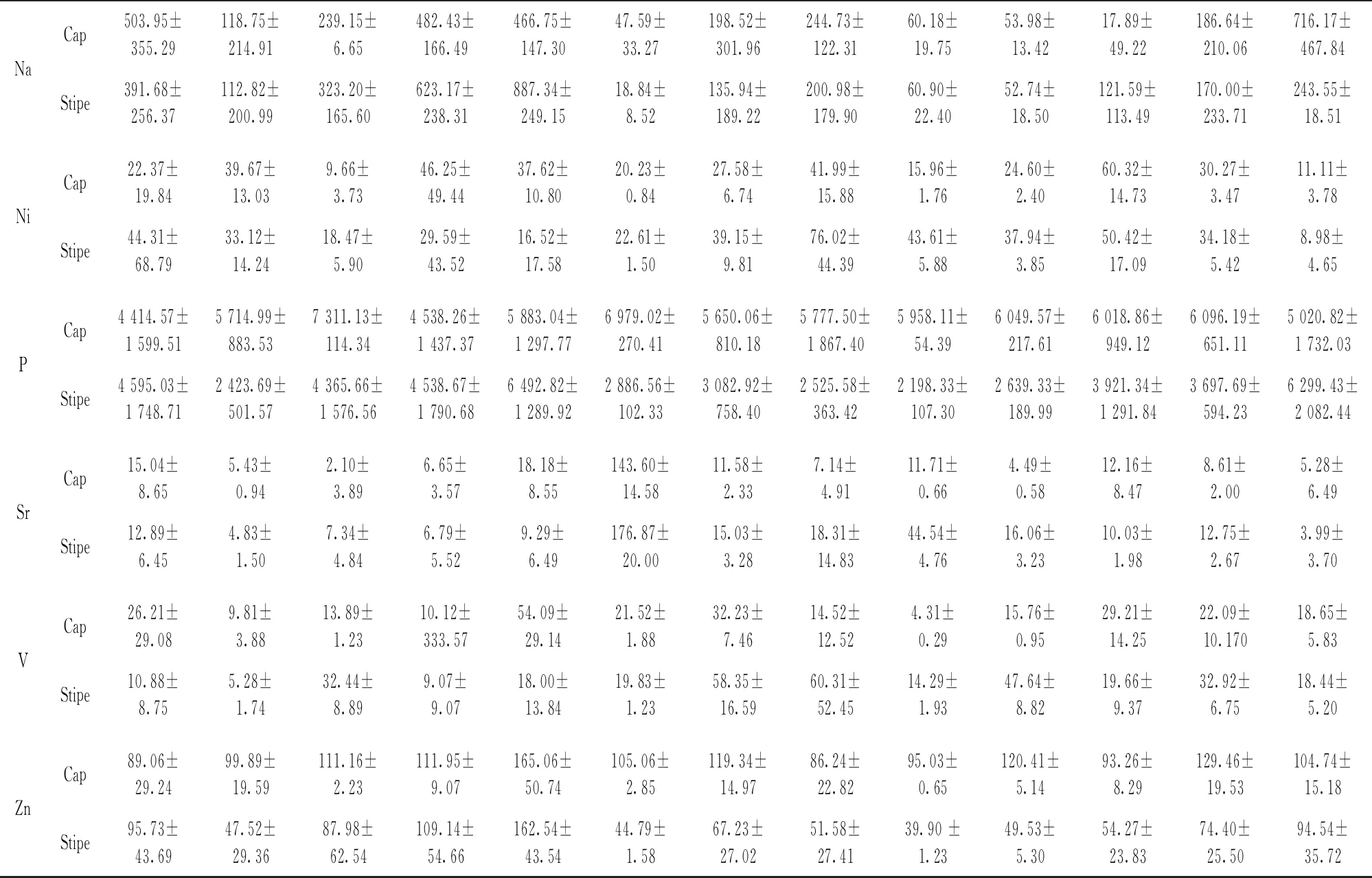
续表3
2.3 光谱分析

2.4 主成分提取
主成分分析是从数据中提取重要的信息,降低数据集维数的思想将多个相互关联的数值转化为少数几个综合指标来进行分析[10]。 用SIMCA13.0软件分别对FTIR-MIR、FTIR-NIR、UV-Vis、元素(盖)、元素(柄)提取特征值>1和Q2最大时的主成分。 由表4可知,其中FTIR-MIR前16个主成分的平均特征值为7.19,累计贡献率达到了0.835; UV-Vis前17个主成分的平均特征值为6.52,累计贡献率为0.667。 提取五种数据的主成分并组合为一个新的数据矩阵,为中级融合进行预备工作。

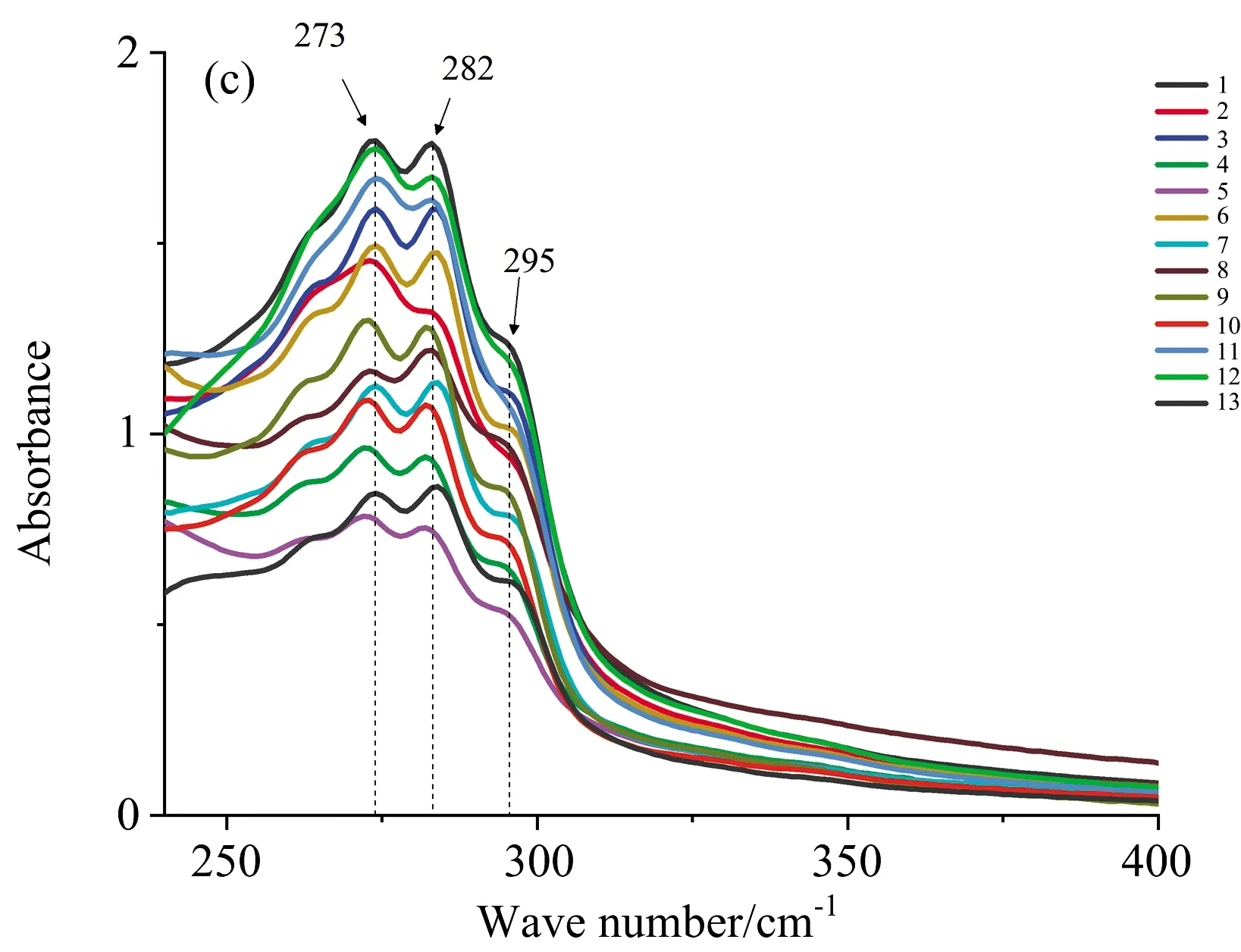
图1 13个产地美味牛肝菌的FTIR-MIR,FTIR-NIR,UV-Vis平均光谱图
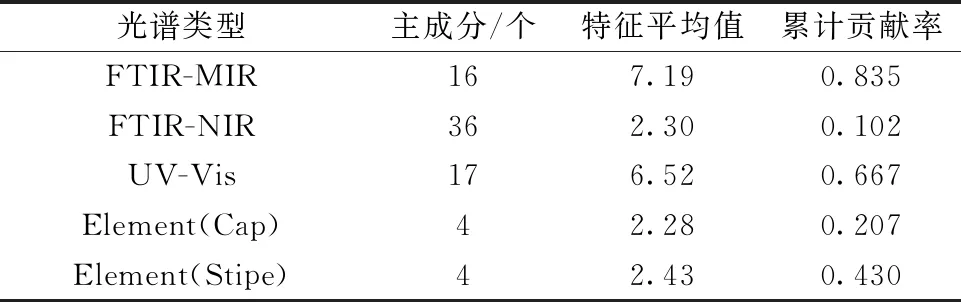
表4 5种数据主成分提取信息Table 4 The 5 data principal componentsextract information
2.5 PLS-DA
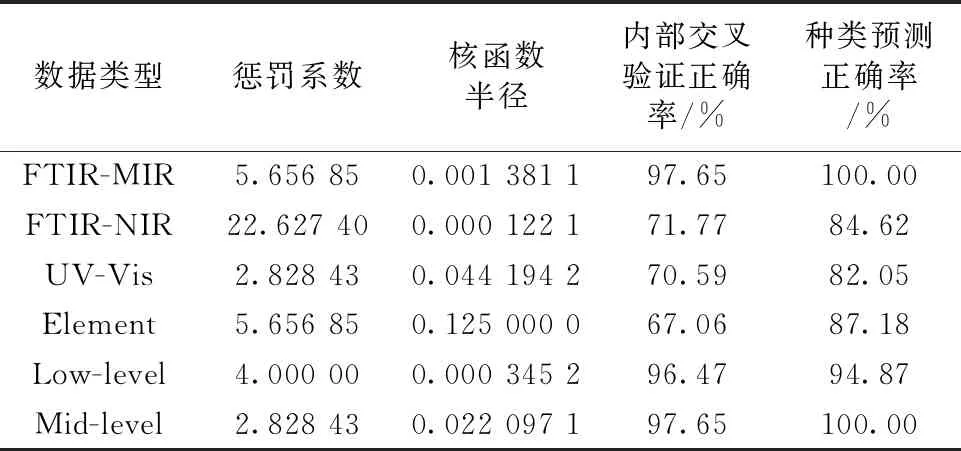
PLS-DA是一种有师监督的多变量统计分析方法[11]。 模型中参数校正均方根误差(RMSEE)、交叉验证均方根误差(RMSECV)、预测均方根误差(RMSEP)参与校正模型评价,其中RMSEE、RMSECV、RMSEP的值小于1且值越接近0代表模型效果越佳。 在参数RMSEP小于RMSECV时,可避免过拟合风险的出现。 对124个美味牛肝菌样品的五种数据进行PLS-DA分析,如表5所示,经过数据融合后的产地鉴别效果比单一数据鉴别效果更佳。 在单一数据PLS-DA分析中,FTIR-MIR光谱的训练集与预测集分别为97.44%与92.94%,在90.00%以上,高于其他单一数据。 其次为UV-Vis,主成分个数为12,训练集与预测集为94.12%和76.92%。 FTIR-NIR光谱与元素数据的预测集分别为74.36%和58.97%。 为提高模型的准确性,增加数据量,将单一的数据矩阵进行组合,形成初级融合。 在初级融合中,训练集和预测集达到了100.00%和94.87%,显著性提高了产地鉴别的准确率。 根据表4对五种数据提取具有代表性的主成分并进行中级融合,在中级融合中,训练集和预测集均可达到100.00%的正确率,RMSEE,RMSECV和RMSEP的参数值均在0.25以下且RMSEP 表5 PLS-DA模型参数结果Table 5 The PLS-DA model results of Parameters Hottelling T2检测法为美国统计学大师哈罗德·霍特林所发明,是一种常见的多变量检验法[12]。 为检验本数据融合模型的准确性,采用多元统计分析Hottelling T2检测法对初级融合与中级融合进行异常值检验。 图2(a)和(c)为初级融合和中级融合的Hottelling T2异常值检验结果,其中T2Crit(99.00%)为38.820 8和33.667,T2Crit(95.00%)为30.371 1和26.060 8。 超出T2Crit(99.00%)置信区间的样品则为异常样品,图中所有样品的初级融合与中级融合都没有超过T2Crit(99.00%)置信区间,表明在模型不存在异常值,具有准确性与可信性。 图2(b)和(d)为初级融合与中级融合得分图,初级融合得分图中13个产地美味牛肝菌相同产地可大致聚在一起,中级融合得分图的产地分类效果明显高于初级融合,训练集和预测集均达到100.00%。 图2 初级融合和中级融合的Hottelling T2置信图与得分图(a): 初级融合Hottelling T2置信图; (b): 初级融合得分图; (c): 中级融合Hottelling T2置信图; (d): 中级融合得分图Fig.2 Hotelling T2 confidence map and score map for primary and intermediate fusion(a): Hotelling T2 confidence map with primary fusion; (b): Primary fusion score map; (c): Intermediate fusion hottelling T2 confidence map; (d) Intermediate fusion score map SVM是数据中以分类或回归分析的监督式学习模型算法,在世界上被广泛应用[13]。 对数据先进行Kennard-Stone分类,分为2/3预测集和1/3训练集,再采用SVM网格搜索最佳的c和g并对四种单一数据以及初级融合与中级融合数据进行分析,结果表明: 数据错分类数从低到高排列中,中级融合=FTIR-MIR>初级融合>元素>FTIR-NIR>UV-Vis; 错分类数依次为0,0,2,5,6和7。 中级融合和FTIR-MIR全部分类正确,效果优于初级融合、元素、FTIR-NIR和UV-Vis。 说明在美味牛肝菌产地鉴别中,光谱种类的选择和数据融合方法对鉴别效果具有重要意义。 表6列出了四种单一模型与两种数据融合模型的惩罚系数、核函数半径、内部交叉验证正确率和种类预测正确率。 其中FTIR-NIR,UV-Vis和元素的内部交叉验证正确率和种类预测正确率较低,正确率均在90.00%以下。 表明大多数单一数据的SVM模型效果不佳; 但FTIR-MIR模型与中级融合模型的内部交叉验证正确率和种类预测正确率都可达到97.65%与100.00%。 在数据融合中,中级融合效果高于初级融合,表明经过筛选后的数据避免了大部分无效信息干扰,提高了分类模型正确率。 通过表5和表6可知,在PLS-DA分类模型和SVM分类模型中,数据融合建立的分类模型效果显著优于单一数据模型。 PLS-DA分类模型效果优于SVM分类模型。 PLS-DA分类模型可以作为美味牛肝菌产地鉴别的最佳方法。 表6 SVM模型预测结果Table 6 The results of SVM models 采集美味牛肝菌样品的FTIR-MIR,FTIR-NIR和UV-Vis光谱信息和不同部位矿质元素,对光谱信息进行预处理,使用Kennard-Stone算法把数据分为2/3的训练集合1/3的预测集后对单一数据和数据融合进行PLS-DA模型和SVM模型。 结果表明,美味牛肝菌不同部位和不同产地之间元素含量具有差异。 原始光谱图中不同产地美味牛肝菌的光谱特征具有差异,可进行产地区分。 对优化后的数据建立PLS-DA和SVM,经过数据融合后的建模效果比单一数据建模更好。 中级融合PLS-DA模型的训练集和预测集达到了100.00%,可区分不同产地的美味牛肝菌。 多光谱数据结合不同部位矿质元素建立PLS-DA模型可用于野生食用菌产地溯源研究。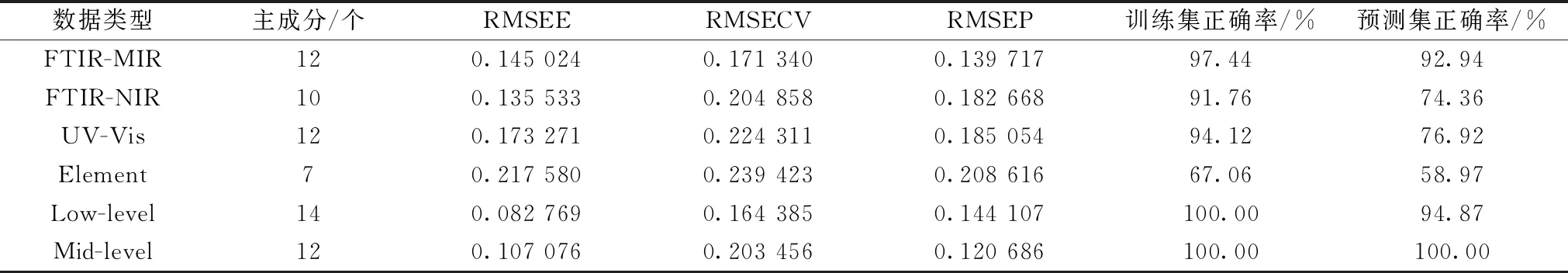

2.6 SVM
3 结 论
