统计学方法在生物信息学分析中的应用
2020-11-25刘壮张悦
刘 壮 张 悦
(中国医科大学《中国卫生统计》杂志 沈阳 110122)
1 引言
20世纪80年代末人类基因组计划启动,基因组学测序数据迅猛增加,随之兴起生物信息学这门新的交叉学科。伴随生物学和医学的迅速发展,特别是人类基因组计划的顺利推进,产生海量生物学数据,特别是生物分子数据积累速度在不断快速增加[1-2]。由此产生的数据具有丰富内涵,隐藏着很多生物学知识。如何充分利用这些数据,通过合理分析和处理揭示其内涵,获得对人类有意义的信息,为生物学科研工作者带来挑战。
2 生物信息学与医学统计学概述
2.1 生物信息学
包含基因组信息获取、处理、存储、分配、分析和解释的所有方面,是基因组学研究不可分割的一部分;是当下自然科学和技术科学领域中“基因组”、“信息结构”和“复杂性”这3个重大科学问题的有机结合[3-5]。生物信息学研究是为了揭示基因组信息结构的复杂性及遗传语言的根本规律,人类在认识自身的基础上可以丰富和发展现有的生物学和信息科学,推动学科群发展,使其成为自然科学中多学科交叉的新领域。
2.2 医学统计学
2.2.1 概述 从20世纪20年代起,统计学理论与方法日益广泛地被生物医学研究工作者所应用。随着流行病学、基因组学、蛋白质组学、代谢组学等学科迅猛发展,促使统计学与这些学科的交叉融合,对医学统计学研究人员提出很多实践中的新课题。为解决这些新课题,统计学家在对经典统计理论研究和认识的基础上不断探索和发展统计新理论和新方法。医学统计学研究内容主要包括3个方面:统计设计、统计分析和其他复杂分析方法。
2.2.2 统计设计 包括对资料收集、整理和分析全过程的设想和安排。在设计前,研究者必须明确的重要问题包括研究目的、研究总体、研究对象、研究内容、样本量、干预措施和研究结果等。在研究设计的构思过程中还应注意几个关键问题,例如抽样方法、控制偏倚和设置对照方法等。
2.2.3 统计分析 主要包括统计描述和统计推断两个部分。统计描述是指用合适的统计图表或统计方法对数据资料的分布状态、数量特征和随机变量之间关系进行估计和测定。统计推断是指在一定的可信程度下由样本信息推断总体特征,包括由样本统计指标(统计量)来推断总体相应指标(参数),即参数估计;由样本差异来推断总体之间是否可能存在差异,即假设检验。
2.3 其他复杂分析方法
在理论统计研究方面,涉及各种概率分布研究、分布偏差的有效性推定以及综合评价方法与理论的研究;在应用统计研究方面,涉及综合评价方法及其应用、统计预测理论与模型研究、各种多元统计方法及其应用的研究、生存时间与生存质量的研究、计算机辅助诊断与治疗模型的研究等。对于这些方面,医学统计学都有相应统计分析方法。
3 统计学方法在生物信息学分析中的应用
3.1 概述
生物信息学中的许多分析方法基本原理都是医学统计学方法的应用和拓展[6-7]。目前生物信息学中常见的问题有序列相似性分析、基因表达分析、基因转录调控网络分析和序列结构与模式识别分析等,本文将介绍这4类问题中统计方法的应用情况。
3.2 序列相似性
3.2.1 概述 在分子生物学研究中,对于待研究的碱基序列或由此翻译得到的氨基酸序列,往往需要在数据库搜索到具有一定相似性的同源序列,以推测该未知序列可能属于哪个基因家族,具有哪些生物学功能。序列比较结果一般要经过统计学检验才能判断是否具有显著意义[8]。
3.2.2 Monte Carlo仿真法 将序列中的符号随机改变后再在同样条件下计算新的配准得分,重复约100次后计算样本配准得分的均值和标准差,常被用来判断一对序列配准得分值的统计显著性。在随机序列配准积分符合正态分布的假设下,结果显著性由配准得分高于均值多少个标准差的数目(Z值)决定。当Z值为3.1、4.3和5.2SD单位时,配准积分的随机出现概率分别是10-3、10-5和10-7。通常认为当Z值>5SD时,两个被比较的序列在进化上相关;当Z值在3~5SD之间时,如果两者在其他方面有相类似的证据可表明两者同源;当Z值<3SD时,表示两者不同源。
3.2.3 Karlin-Altschul公式 由于各得分随机变量是在大量分值数据中的最大值(最优配准),正态性假设不尽合理,因此Karlin和Altschul提出计算BLAST得分显著性的Karlin-Altschul公式。Vingron和Watterman将此公式推广为适用于计算局部配准得分统计显著性的公式,将序列长度作为其一个参数。对两个序列a、b,BLAST发现的高分区匹配域称为HSPs(high scoring pairs)ai…ai+k与bj…bj+k。最佳HSP得分H(a,b)超过阈值t的概率为:
P(H(a,b)>t)≈1-e-rnmpt
(1)
式中r和p可以通过解一个方程或直接计算得到,m、n分别是两个序列的长度。式(1)反映HSPs得分高于阈值t的数目近似为Poisson分布。
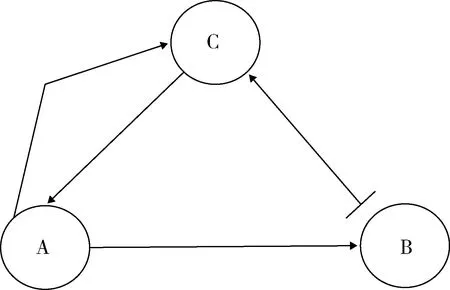
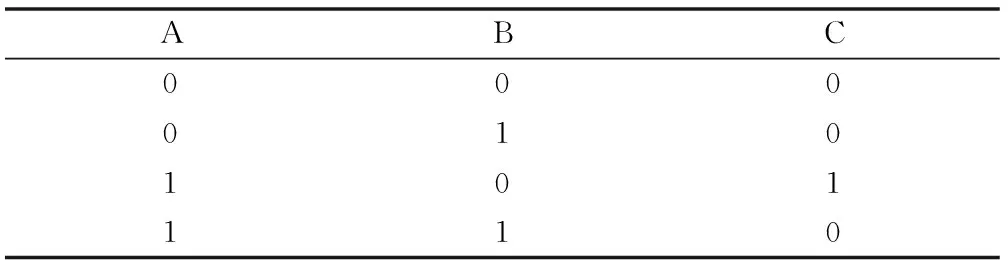
3.2.4 非重叠局部亚优化配准(Non-overlapping Local Suboptimal Alignment,NOLSA) 那些使局部Smith-Waterman配准的期望分值随着被比较序列的长度而呈对数关系增长的罚分称为强gap罚分。在强gap罚分的情况下,Karlin-Altschul公式近似适用于局部配准分析。Waterman和Eggert提出NOLSA算法,其中任何一对已经在一种配准中使用过的残疾不再在接下去的较小得分的配准中使用。此算法在每次进行新的次优配准时不必重新计算整个动态规划矩阵,只需重做上一次配准的一个领域,得到的次优配准间的依赖性较低。最优NOLSA是Smith-Waterman配准。记ω(t)表示分支不小于阈值t的NOLSAs数目,可以用Waterman-Eggert算法计算NOLSAs,直到第1个NOLSA分值 P(ω(t)>t)≈1-e-rnmpt (2) 3.3.1 概述 随着生命科学进入后基因组时代,基因芯片技术所面临的挑战早已不再是基因表达芯片本身,而是在于发展实验设计方法以对基因表达进行时空全面探索[9]。数据分析与挖掘对其来说是最大挑战。基因芯片表达实验产生海量数据,隐藏着丰富信息,通过数据统计或可视化方法可以发现新的知识。聚类分析是目前运用最多的一种表达数据分析方法。一块基因芯片上往往载有成千上万个基因,一次实验可同时检测这些基因的表达情况。应用同一种芯片在不同条件下(如不同时间、细胞等)进行基因表达实验,搜集表达数据,将原始数据放在一起,生成一个数据表格。表格每一行代表一个基因,每一列代表在不同实验条件下得到的基因表达强度。表格中每一行数据可作为一个向量,聚类分析是将这些向量按照相似程度进行归类。 3.3.2 分层聚类分析[10-11]在分层聚类情况下,数据被看作是一种二元树结构,在最高层上所有数据同属于一个类。其原理与树的分叉结构相似,类被一分为二,相似的类被保留在同一个子类中,不相似的类则被分开。在进行聚类分析时,从类的每个元素出发将类的集合分为只含有两个类的一组二元类对合集。每个时间中一个类对被合二为一,这样类的数目就减少一个,连续向后进行此过程,最终得到树图的数据分层结构。 3.3.3 K-Means聚类[12]在数据划分上不考虑类的分层结构问题。将R矩阵的P列数组聚为K个类,具体方法如下:(1)随机将R1,R2,…,RP分配到K个类中。(2)计算K个类的重心Y1,Y2,…,YK。(3)按照由1到P的顺序计算R1,R2,…,RP到重心Y1,Y2,…,YK间的距离,Ri将分配到距离最近的类中。(4)如果Ri被分配到一个新的类中,则重新计算两个受影响的类的重心。(5)重复步骤(3),直到不再有新的类划分出现。 3.4.1 概述 基因芯片表达数据不仅可用于分析基因表达的时空规律、研究基因功能,还可用于分析基因间的相互关系和基因转录调控网络。单一基因表达结果受其他基因影响,而这个基因同时能影响其他基因表达,这种相互影响、制约的关系构成复杂基因表达调控网络。基因调控网络的研究意义在于通过建立基因转录调控网络统计模型,对某个物种或组织的全部基因的表达关系进行整体分析和研究,分析基因间相互作用。 3.4.2 布尔网络模型 一种以有向图为基础的离散系统,是基因调控分析中最简单的一种模型。在此模型中每个基因只有两种状态,“开”表明基因转录表达,形成基因产物;“关”则表明基因未转录。基因间的相互关系可表示为: (3) 即如果基因A表达,而且基因B不表达,则基因C表达,其网络图,见图1。在布尔网络模型中各个基因状态的集合是整个系统的状态,当系统从一个状态转换为另一个状态时,各基因下一时刻的状态由其连接输入机器布尔规则确定。布尔规则用“真值表”的形式表示,当基因A和基因B处于不同状态时,基因C的状态随之发生变化,见表1。 图1 布尔网络模型 表1 基因C真值 3.4.3 线性组合模型 一种连续网络模型,在此模型中假设基因之间的相互作用是线性的,一个基因的表达值是若干个其他基因表达值的加权和。线性组合模型可表示为: Xi(t+△t)=∑WijXj(t) (4) 其中Xi(t+△t)是基因i在t+△t时刻的表达水平,Xj(t)是基因j在t时刻的表达水平,为Wij代表基因j的表达水平对基因i的影响。在这种基因相互关系表达形式中还可以增加其他数据项,以模拟基因调控的真实情况。 结构复杂的蛋白质实际上是由一些相同或不同的结构域缔结而成,每一结构域承担一定功能,各结构域协同作用体现了蛋白质总的生物学功能。测定大量的蛋白质结构可简化为对数量、残基数目较少的结构域结构测定,了解它们如何组装成完整的蛋白质,需要发展新的检索结构域的模式匹配方法。频率表法最先用于核酸序列特殊信号的模式识别,随后逐渐应用于蛋白质结构域的模式匹配分析中。由于蛋白质的结构域通常由几十个或几百个残基组成,属于同一类结构域的序列的类似性可能很小。结构域保守区决定了结构域的同源,因此其存在确定了结构域的存在,可以用结构域的保守顺序直接分析蛋白质与蛋白质超家族的类似性,增加检测敏感性。 作为连接生命科学和信息科学的新兴学科,生物信息学发展前景广阔。而统计学作为生物信息学分析的重要工具,可以探查和提取数据之间的因果关系,揭示数据内涵,从而获得更多有价值的信息。本文通过介绍序列相似性分析、基因表达分析、基因转录调控网络分析和序列结构与模式识别分析中统计学方法的应用,为科研人员学习系统的生物分析技术提供理论依据。3.3 基因表达
3.4 基因转录调控网络

3.5 序列结构与模式识别
4 结语
