基于深度学习的大豆豆荚类别识别研究
2020-09-24闫壮壮闫学慧虞江林张战国胡振邦蒋鸿蔚辛大伟齐照明刘春燕武小霞陈庆山朱荣胜
闫壮壮 闫学慧 石 嘉 孙 凯 虞江林 张战国 胡振邦 蒋鸿蔚 辛大伟 李 杨 齐照明 刘春燕 武小霞 陈庆山 朱荣胜,*
基于深度学习的大豆豆荚类别识别研究
闫壮壮2闫学慧2石 嘉2孙 凯2虞江林2张战国1胡振邦3蒋鸿蔚3辛大伟3李 杨1齐照明3刘春燕3武小霞3陈庆山3朱荣胜1,*
1东北农业大学文理学院, 黑龙江哈尔滨 150030;2东北农业大学工程学院, 黑龙江哈尔滨 150030;3东北农业大学大豆研究所, 黑龙江哈尔滨 150030
作物表型调查是作物品种选育过程中的一项关键工作。传统表型调查主要依靠人力, 使得表型调查的结果难以达到自动化、高精度、高可靠性的要求。在大豆的表型调查中, 对豆荚类别的正确识别是豆荚个数、长度和宽度等表型准确提取的关键和前提。本文针对成熟期大豆豆荚的图片, 通过利用深度学习迁移5种不同的网络模型[AlexNet、VggNet (Vgg16, Vgg19)、GoogleNet、ResNet-50], 对一粒荚、二粒荚、三粒荚、四粒荚进行识别。为提高训练速度和准确率, 本试验微调模型, 选择不同的优化器(SGD、Adam)对网络模型进行优化。结果表明, 在针对豆荚辨识问题中, Adam的性能优于SGD, 而Vgg16网络模型搭配Adam优化器, 豆荚类别的测试准确率达到了98.41%, 在所选的网络模型中体现了最佳的性能。在十折交叉验证试验中也体现了Vgg16网络模型具有良好的稳定性。因此本研究认为Vgg16网络模型可以应用到实际的豆荚识别中, 为进一步实现豆荚表型自动提取提供一条重要的解决途径。
大豆育种; 豆荚辨别; 深度学习; 迁移学习
作物表型组学的研究目的是获取高质量、高精度和可重复的植物表型数据, 而高通量、高准确性成为其特点[1-2]。基于高精准、高通量的表型数据与基因型结合分析能够得出比传统方法(人工表型调查)更准确、更普遍的生物学规律。随着人口数量增加和气候的异常变化, 快速培育出产量更高、适应性更强的作物新品种成为了育种家们面临的最大挑战。此外, 随着人类基因组计划的实施、展开和延续, 大量生物物种基因被获得, 其中包括大量的作物物种, 如水稻、大豆、玉米等, 相较于基因组学的蓬勃发展, 作物表型组学的研究才刚刚开始[3-4]。与基因工程相比, 表型的研究处于滞后局面, 传统表型测量依靠人工测量和统计, 不仅成本很高, 而且会对植物造成破坏并产生主观性误差。结合现有的遗传信息, 这些落后的表型分析程序和处理植物表型的技术, 既不能对表型进行彻底地功能分析, 也不能绘制出基因型到表型之间的功能图谱[5], Shakoor等[6]的试验也表明, 高通量、高精度的表型获取技术能加快作物改良和新品种的育成, 对产量提高和抗病性研究至关重要。
成熟期大豆豆荚种类的识别是豆荚长度、宽度和所含籽粒个数等表型性状获取的前提[7], 豆荚数量、类别、籽粒数直接影响大豆的产量。目前对于豆荚种类的考察主要依靠专业的人员, 工作量反复单一, 消耗大量的人力物力, 且难以实现高通量。根据这一问题, 本文以机器学习中的深度学习[8]来对大豆豆荚进行辨识, 实现豆荚表型性状的高通量获取。深度学习对作物表型特征进行提取, 从海量的原始复杂表型中自动学习高层次特征, 既不需要进行大量的病灶分割, 也不需要人工特征提取。Mohanty等[9]训练了一个用于识别14种植物和26种植物疾病的深度学习模型, 在植物种类和植物疾病诊断中达到了99.35%。Cheng等[10]利用深度学习对玉米复杂背景下虫害的识别, 能够帮助农业工作者进行病虫害的检测和分类。Amara等[11]运用深度学习对香蕉叶片疾病进行分类。Dyrmann等[12]使用卷积神经网络实现了对植物种类的识别。Ubbens[13]使用深度学习对莲座科植物叶片进行计数。Uzal等[14]构建卷积神经网络模型对豆荚的瘪实度进行识别。深度学习对复杂作物表型具有很强的分析和处理能力, 可以帮助育种家理解基因到表型的转化关系。
本文基于SGD和Adam两种优化算法, 迁移学习了5种深度网络模型, 尝试找到一种更稳定更准确的豆荚类别识别网络模型。该网络模型将应用在大豆表型过程中, 解决豆荚识别问题, 加快豆荚表型数据提取, 为育种选种提供表型数据支撑。
1 材料与方法
1.1 试验材料
分别选取5个品种东农251、东农252、东农253、黑农48、黑农51的大豆豆荚作为试验材料。每个品种选取8株, 共计40株。所有试验材料均于2018年5月种植于东北农业大学大豆试验基地。
首先对40株大豆的豆荚进行拆分拍摄, 经图像分割得到单张单荚图片。由大豆专家对分割的豆荚图片进行识别和分类, 制作成数据集, 用于训练网络模型和测试网络。其中原始数据中一粒荚158张、二粒荚489张、三粒荚1008张、四粒荚340张, 共计1995张, 经数据增强后共计8484张。再拆分为2部分, 随机选取80%作为训练集, 用于训练卷积神经网络模型(AlexNet、VggNet、GoogleNet、ResNet-50)[15-18], 其余20%作为验证集, 用于测试网络识别准确率。
1.2 试验方法
基于迁移学习的方法, 迁移5种不同的深度学习模型, 微调网络模型的全连接层和分类层用于豆荚识别。首先, 选择Adam、SGD 2种不同训练算法, 优化学习率、批量大小、L2等超参数进行初步训练, 对比训练网络模型的训练精度, 选出性能较高的训练算法。然后, 高性能的训练算法再与5种网络模型(AlexNet、Vgg16、Vgg19、GoogleNet、ResNet-50)进行组合试验, 根据识别准确率和损失值选择出对豆荚识别最佳网络模型, 用于豆荚种类的识别与分类。
1.3 图像分割
拍摄拆分后的大豆豆荚, 利用Matlab软件分割图片[19-20]。首先, 将拍摄好的RGB图像进行灰度化处理, 再对灰度化图像进行二值处理, 由于在自然环境下拍摄照片存在着噪声的影响, 还需对图片进行去噪处理, 再利用边缘检测算子对图像中的豆荚进行边缘检测, 最后经过BoundingBox函数[21]对豆荚的位置进行定位和分割, 得到单张单荚图片。卷积神经网络的输入图片需要固定尺寸大小, AlexNet模型的输入图片大小为227´227, VggNet、GoogleNet、ResNet-50等网络模型的输入图片大小为224´224, 因此还需要调整分割图片的像素。
1.4 数据增强
卷积神经网络存在过拟合问题。过拟合使模型泛化能力变差, 不能很好运用在全新数据集的检测, 本试验采用数据增强来降低过拟合。数据增强就是通过对现有数据集进行简单的随机变换(旋转、平移、调色调、加噪声、缩放等), 增加数据集, 从而让模型学习更多的特征, 提高模型的泛化能力达到降低过拟合的效果。本文通过使用Python搭建TensorFlow深度学习框架[22]对图片进行顺逆旋转角度、以及调亮度、饱和度、对比度等, 从而达到数据增强, 增强后一粒荚1836张、二粒荚1436张、三粒荚1589张、四粒荚3618张, 总计8484张。以二粒荚为例增强效果如图1所示。

图1 数据增强
a: 左右翻转时针旋转90度; b: 逆时针旋转90度; c: 调亮度; d: 左右翻转; e: 上下翻转; f: 调整饱和度; g: 逆时针旋转180度。
a: rotate 90 degrees left and right clockwise; b: rotate 90 degrees counterclockwise; c: adjust brightness; d: rotate left and right; e: rotate up and down; f: adjust saturation; g: rotate 180 degrees counterclockwise.
1.5 迁移学习
近几年, 迁移学习[23-24]逐渐成为深度学习中一个热门的领域。由于训练一个新的高性能的卷积神经网络主要包含网络结构设计、超参数优化、防止过拟合等过程, 同时还需要大量数据对网络进行训练, 这一过程往往十分漫长。而实际问题是我们拥有很小的数据集不足以训练一个全新的网络, 又想利用深度学习的高性能表型分析能力, 于是迁移学习应运而生。迁移学习简单来说就是利用模型已经学习的知识来学习相关领域的新知识。本文利用迁移预训练网络模型的方法进行试验[这些预训练模型已在大型数据集(ImageNet)大赛[25]上训练好, 具有很强的鲁棒性和泛化能力], 通过冻结网络权重、微调网络分类层的方法, 解决豆荚识别问题。
1.6 深度学习模型
1.6.1 AlexNet网络模型 AlexNet是由Hinton和他的学生Krizhevsky在2012年ILSVRC大赛上提出的深度卷积神经网络模型, 在图像分类上top-5错误率为15.3%, 获得该届大赛的冠军[15]。AlexNet网络模型包含65,000个神经元、5个卷积层、3个全连接层和1个softmax分类器。前7层都包含Relu激活函数, 第1层、第2层、第5层运用最大池化保留最大特征值来降低图片大小、减小参数。Dropout是降低过拟合很好的措施[26], 因此在前2个全连接层中也加入Dropout来降低过拟合的影响。AlexNet网络模型最后全连接层被微调成个4神经元, 代替原有的1000个神经元。微调结构如图2所示, AlexNet网络模型的输入图片是227´227像素的图片。

图2 AlexNet网络结构
1.6.2 VggNet网络模型 VggNet网络模型在2014的ILSVRC大赛上获得定位项目的冠军和图像分类项目的亚军, 在图像分类上top-5错误率为7.5%[16], 相比于Alexnet有了很大程度上的提高。VggNet网络模型就是在AlexNet的基础上加深了网络结构, 统一采用3´3的卷积核, 取代了原来的11´11和5´5的卷积核。随着网络的不断加深, 参数也不断的增加, 以Vgg16网络模型为例, 卷积层参数约为14,714,688, 而在分类器上更是有200多万个参数[16], 也增加了网络模型训练难度。为了加速运算, VggNet网络模型采用NVIDIA生产的GPU搭载CUDA和CUDNN进行加速训练。本试验通过冻结VggNet网络模型的卷积层, 修改网络的全连接层和分类层进行豆荚辨识。
1.6.3 GoogleNet网络模型 GoogleNet网络模型获得2014年ILSVRC大赛冠军模型, 图像分类的top-5错误率为 6.7%[17]。GoogleNet不仅仅将网络的深度增加到22层, 同时也增加了网络的宽度。虽然网络加深、加宽, 参数却是AlexNet网络模型参数的一半, 原因是引入了Inception结构[27]。Inception结构由1´1、3´3、5´5的卷积核构成, 通过多种卷积核提取图像不同尺度的信息, 最后进行融合, 可以更好地提取图像特征。而引入了1´1的卷积核起到降低维度、减少参数的作用。网络模型为了防止过拟合添加了Dropout结构, 原理是随机选取部分神经元参与训练, 使网络模型轻量化来达到降低过拟合的效果。全连接层参数量过大, 也很容易过拟合并降低训练速度, 因此采用全局平均池化(AvgPooling)代替全连接层, Lin等[28]也提到全局平均池化是对整个网络在结构上进行正则化, 从而达到降低过拟合的目的。而GoogleNet在后面又加入一个全连接层, 其主要目的是为了方便调节模型, 本文也是微调最后的全连接层与分类层进行试验。
1.6.4 ResNet网络模型 Kaiming等[18]提出, ResNet (Residual Neural Network)也称之为残差网络, 并在ILSVRC2015大赛中取得冠军, 图像分类的top-5错误率仅有3.57%, 相比于其他网络结构, 性能有很大程度上的提高。网络中采用了独具一格的残差模块作为ResNet的基本组成部分, 每个单元都可以用公式[18]表示;

1.7 训练算法
SGD[29]、Adam[30]优化算法, 搭配AlexNet、Vgg16、GoogleNet、ResNet-50网络模型试验, 选取高性能优化器。其中SGD算法的参数设置: 批量大小设置为64、初始学习率为0.0001、动量为0.9、迭代次数为2000次, 每50次迭代进行验证1次。Adam参数设置: 批量大小设置为64、初始学习率为0.0001、学习率衰减值为0.9, 迭代次数为2000次。
为了更好的拟合数据和训练网络, 本试验在SGD、Adam中添加L2正则化[31-32]来防止网络的过拟合, 提高模型的泛化能力。L2参数正则化又被称作权重衰减, 如公式[32](2);

正则项使得权重衰减更加接近原点[31], 网络模型变得更加简单, 从而达到优化参数的目的, 本试验取L2=0.0001进行试验。
1.8 模型评价指标
为评估网络模型的稳定性和分类性能, 本文采用十折交叉验证(10-fold cross-validation)方法评估网络模型稳定性、混淆矩阵评价网络分类性能、F1-score衡量模型总体性能。真阳性(true positive, TP): 模型正确预测真实荚类; 真阴性(true negative, TN): 模型预测其它荚类为非真实荚类; 假阳性(False Positive, FP): 模型不正确把其他荚类预测成真实荚类; 假阴性(False Negative, FN): 模型把真实荚类错误预测成其它荚类。验证指标基于准确率(Accuracy)、召回率(Recall)、精确率(Precision)和F1分数(F1-score)等, 具体公式如下:

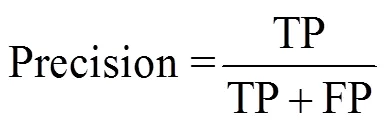


1.9 试验器材
所有试验均在Windows 10中实现, 使用Matlab2018a, 并搭载NVIDIA Titan Xp 12G的GPU显卡对试验进行加速。
2 结果与分析
2.1 优化算法结果
本文首先对训练算法进行选择, 搭配AlexNet、Vgg16、GoogleNet、ResNet-50四个网络模型, 分别用SGD和Adam进行训练, 对比2种算法的性能, 以豆荚的识别准确率(Accuracy)为标准。从图3可以看出, 针对豆荚识别, Adam的训练算法要优于SGD算法。其中AlexNet网络模型和GoogleNet网络模型在搭配Adam算法时性能明显优于SGD。在Vgg16网络模型和ResNet-50网络模型的对比中, 试验的准确率也高于SGD算法。因此, 本试验选取Adam做为优化算法, 进行网络模型的训练。
2.2 模型选择结果
选择AlexNet、Vgg16、Vgg19、GoogleNet、ResNet-50五种不同的迁移模型, 搭配Adam进行组合试验, 经过优化参数后对豆荚进行识别。对比不同模型试验结果, 以验证准确率和训练时间为标准, 选择出最优的模型。从图4和表1可以看出, Vgg16网络模型的验证准确率最高(98.41%), 其次是Vgg19验证准确率(98.35%), ResNet-50表现出最差的识别准确率(87.15%)。网络模型的训练需要花费大量时间, 模型训练时间最长的是Vgg19 (255.23 min), 时间最短是GoogleNet (18.13 min)。而GoogleNet与Vgg16相比, Vgg16训练时间更长, 但准确率提高了2.58%, 表明Vgg16网络模型是对一粒荚、二粒荚、三粒荚、四粒荚识别的最优迁移模型。

图3 深度网络模型在不同算法下的表现
a: AlexNet模型在Adam算法和SGD算法下的验证准确率; b: Vgg16模型在Adam算法和SGD算法下的验证准确率; c: GoogleNet模型在Adam算法和SGD算法下的验证精准确率; d: ResNet-50模型在Adam算法和SGD算法下的验证准确率。横轴为迭代次数, 纵轴为验证准确率。
a: the verification accuracy of the AlexNet model under the Adam algorithm and SGD algorithm; b: the verification accuracy of the Vgg16 model under the Adam algorithm and SGD algorithm; c: the verification accuracy of the GoogleNet model under the Adam algorithm and SGD algorithm; d: ResNet-50 The verification accuracy of the model under Adam algorithm and SGD algorithm. The horizontal axis is the number of iterations, and the vertical axis is the verification accuracy.

图4 模型准确率和损失图
a: 5种模型搭配Adam训练算法在相同数据集下的验证准确率; b: 5种模型搭配Adam训练算法在相同数据集下的验证损失。
a: the verification accuracy of the five models with the Adam training algorithm under the same data set; b: the verification loss of the five models with the Adam training algorithm under the same data set.
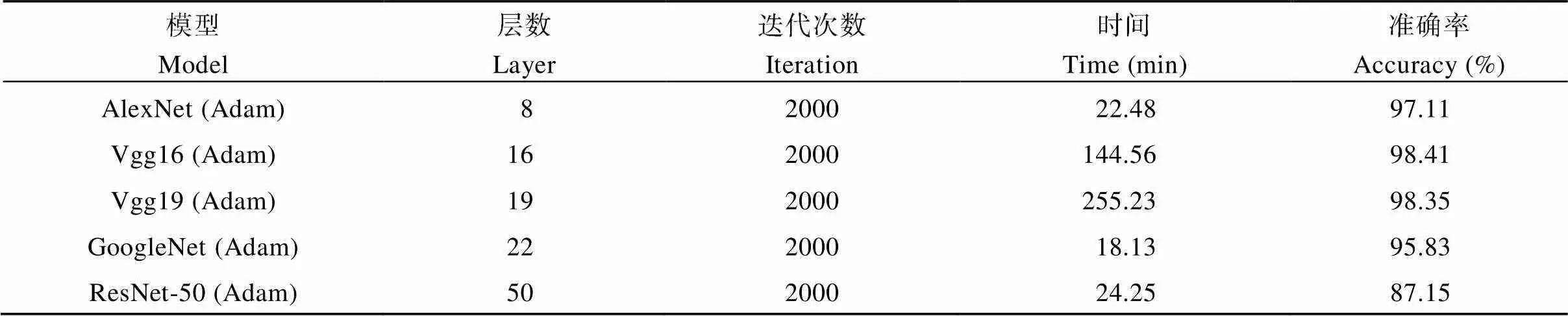
表1 不同迁移模型搭配Adam的试验结果
2.3 最优网络模型分析
为进一步评估Vgg16网络模型分类性能与稳定性, 本试验采用十折交叉验证(10-fold cross-validation)方法评估网络模型稳定性和分类性能。在十折交叉验证试验中, 把原始的豆荚数据随机分割为10个不重合的子数据集, 然后做10次Vgg16网络模型的训练和验证。每一次, 选取9份子数据集作为训练集, 1份子数据集作为验证集。在10次的训练和验证中, 每次用来验证模型的子数据集都不同。最后, 根据这10次验证集的验证精度标准差和验证误差均值来评估模型的稳定性, 结果如表2。
从表2可以看出, Vgg16网络模型的平均验证准确率96.97%, 十折交叉验证精度标准差为0.0085, 从验证标准差和平均验证损失结果来看, Vgg16网络模型搭配Adam算法具有良好的鲁棒性。
采用混淆矩阵分析精确率(Precision)、召回率(Recall)以及F1分数评估Vgg16分类性能。精确度是测试结果中正确部分的百分比, 反映模型的准确度。召回率是测试结果的正确部分与实际正确部分的百分比, 反映模型的灵敏度。F1分数为精确率与召回率的调和平均值, 其取值范围在0到1之间, 用于衡量模型总体性能。从图5和表3可以看出, 网络模型对各类豆荚识别精确率在95.6%以上, F1-score均值为97.2%模型总体性能良好。综合十折交叉验证和混淆矩阵分析, Vgg16网络模型针对豆荚分类问题具有很好的稳定性和良好的分类性能。表明Vgg16网络模型是对一粒荚、二粒荚、三粒荚、四粒荚识别的最优迁移模型。

表2 十折交叉验证结果

图5 模型混淆矩阵热力图
混淆矩阵中列为预测类别, 行为真实类别。图最右边的列为精确率, 底部的行为召回率。
The columns in the confusion matrix are predicted class, and the rows are true class. The rightmost column of the figure is the precision, and the bottom row is the recall.
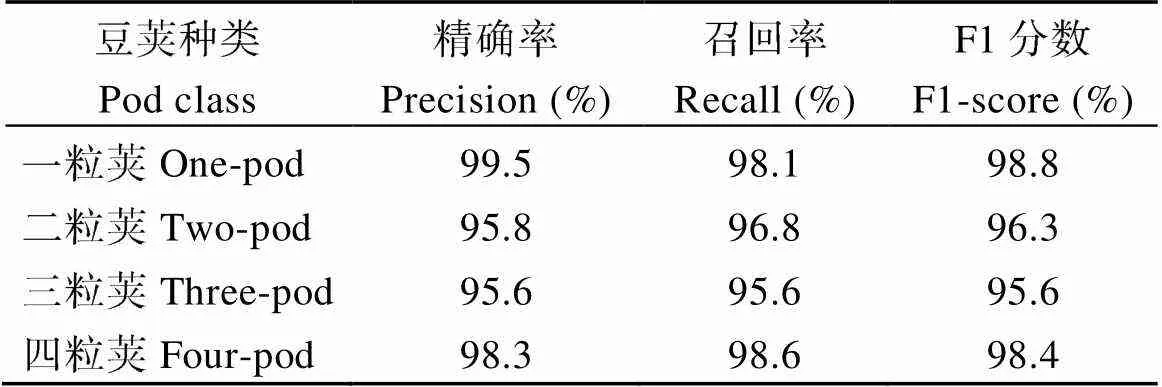
表3 模型的分类性能
3 讨论
成熟期大豆豆荚种类的识别是豆荚长度、宽度和所含籽粒个数等表型性状获取的前提。为完成对豆荚种类的自动识别, 本研究以各类豆荚的图像为对象, 基于SGD和Adam两种优化算法, 迁移学习了5种深度网络模型, 尝试找到一种更稳定更准确地豆荚类别识别网络模型。本研究表明, 结合了Adam的Vgg16网络模型验证准确率达到了98.41%, 在所有测试网络中表现最好, 十折交叉验证也表明该模型具有良好的稳定性。
本研究分别对2种优化算法(SGD, Adam)和5个网络模型(AlexNet、Vgg16、Vgg19、GoogleNet、ResNet-50)进行选择。针对优化算法的选择, Ruder[33]对比研究表明, 训练更深更复杂的神经网络和处理离散数据时, Adam等自适应算法效果更好, 速度更快, 而SGD算法具有更好的收敛性, 但训练速度慢, 特别是在大型数据集。本研究在此基础之上, 选择SGD、Adam优化算法对不同网络模型进行训练发现, Adam训练的模型识别准确率更高, 更适合豆荚识别。在网络模型选择方面, 迁移5种预训练模型进行训练, 搭配Adam进行实验。我们发现针对同一数据集, 不同迁移模型识别效果不同, Arnal[34]和Kamilaris等[35]都认为数据集大小和多样性会对迁移学习有效性产生影响, 而Mehdipour等[36]则认为, 学习率、批量大小等超参数也会对迁移模型产生影响。更丰富的训练样本数据集和改进优化超参数能够对网络进行更进一步的优化。
高精度豆荚类别识别网络模型的建立是进一步获取精准表型的关键, 如高精度豆荚分类网络模型结合目标区域识别算法可以大大降低豆荚长和豆荚宽性状获取的错误率, 而豆荚类别的正确识别可以为籽粒个数和大小的估算提供重要支持, 进而成为产量估算的基础。另一方面, 通过迁移学习的方式是可以找到高分辨率的豆荚类别辨识工具的, 为研究者尝试解决其他农业研究中存在的识别和分类问题提供了一个重要的途径。
4 结论
本文以大豆豆荚表型提取平台为基础, 获取豆荚原始的数据, 以深度学习中的迁移学习为技术手段, 解决了大豆豆荚的识别问题。为了提高网络模型的准确率并减少训练时间, 本研究分别进行了优化算法的选择和超参数优化, 并选取合适的迁移模型。在试验的过程中Vgg16网络模型搭配Adam整体表现出更佳优异的性能, 准确率高达98.41%, 十折交叉验证和混淆矩阵分析Vgg16网络模型针对豆荚分类问题具有很好的稳定性和良好的分类性能。实现了对豆荚的高通量精准识别, 这一模型有利于简化大豆考种过程, 同时为大豆育种工程提供大量的豆荚表型数据, 加快选种和育种进程, 也为深度学习运用到农业的问题研究提供参考。
[1] Tester M, Langridge P. Breeding technologies to increase crop production in a changing world., 2010, 327: 818–822.
[2] Ribaut J M, Vicente M D, Delannay X. Molecular breeding in developing countries: challenges and perspectives., 2010, 13: 213–218.
[3] Furbank R T, Tester M. Phenomics-technologies to relieve the phenotyping bottleneck., 2011, 16: 635–644.
[4] Cannon S B, Jackson M S A. Three sequenced legume genomes and many crop species: rich opportunities for translational genomics., 2009, 151: 970–977.
[5] Rahaman M M, Chen D J, Gillani Z, Klukas C, Chen M. Advanced phenotyping and phenotype data analysis for the study of plant growth and development., 2015, 6: 619.
[6] Shakoor N, Lee S, Mockler T C. High throughput phenotyping to accelerate crop breeding and monitoring of diseases in the field., 2017, 38: 184–192.
[7] 邱丽娟. 大豆种质资源描述规范和数据标准. 北京: 中国农业出版社, 2006. pp 64–74. Qiu L J. Specification and Data Standard of Soybean Germplasm Resources. Beijing: China Agriculture Press, 2006. pp 64–74 (in Chinese).
[8] Lecun Y, Bengio Y, Hinton G. Deep learning., 2015, 521: 436.
[9] Mohanty S P, Hughes D P, Salathé M. Using deep learning for image-based plant disease detection., 2016, 7: 1419.
[10] Cheng X, Zhang Y, Chen Y, Wu Y, Yue Y. Pest identification via deep residual learning in complex background., 2017, 141: 351–356.
[11] Amara J, Bouaziz B, Algergawy A. A deep-learning based approach for banana leaf diseases classification., 2017, 266: 79–88.
[12] Dyrmann M, Karstoft H, Midtiby H S. Plant species classification using deep convolutional neural network., 2016, 151: 72–80.
[13] Ubbens J, Cieslak M, Prusinkiewicz P, Stavness I. The use of plant models in deep learning: an application to leaf counting in rosette plants., 2018, 14: 6.
[14] Uzal L C, Grinblat G L, Namías R, Larese M G, Bianchi J S, Morandi E N, Granitto P M. Seed-per-pod estimation for plant breeding using deep learning., 2018, 150: 196–204.
[15] Krizhevsky A, Sutskever I, Hinton G. ImageNet classification with deep convolutional neural networks. In: Advances in Neural Information Processing Systems. New York: Curran Associates, 2012. pp 1097–1105.
[16] Karen S, Andrew Z. Very deep convolutional networks for large-scale image recognition. In: Bengio Y, Lecun Y, eds. 3rd International Conference on Learning Representations. San Diego: Conference Track Proceedings, 2015.https://arxiv.org/abs/1409. 1556.
[17] Christian S, Liu W, Jia Y Q, Pierre S, Scott R, Dragomir A, Dumitru E, Vincent V, Andrew R. Going deeper with convolutions. In: IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Boston: IEEE Computer Society, 2015. pp 1–9.
[18] He K M, Zhang X Y, Ren S Q, Sun J. Deep residual learning for image recognition. In: IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Las Vegas: IEEE Computer Society, 2016. pp 770–778.
[19] 张德丰. MATLAB数字图像处理. 北京: 机械工业出版社, 2012. pp 276–285. Zhang D F. MATLAB Digital Image Processing. Beijing: Mechanical Industry Press, 2012. pp 276–285 (in Chinese).
[20] 张铮, 倪红霞, 苑春苗, 杨立红. 精通MATLAB数字图像处理与识别. 北京: 人民邮电出版社, 2017. pp 253–289.Zhang Z, Ni H X, Yuan C M, Yang L H. Proficient in MATLAB Digital Image Processing and Recognition. Beijing: People’s Posts and Telecommunications Press, 2017. pp 253–289 (in Chinese).
[21] Lempitsky V, Kohli P, Rother C, Sharp T. Image segmentation with a bounding box prior. In: IEEE 12th International Conference on Computer Vision. Kyoto: IEEE Computer Society, 2009. pp 277–284.
[22] Abadi M, Barham P, Chen J M, Chen Z F, Davis A, Dean J, Devin M, Ghemawat S, Irving G, Isard M, Kudlur M, Levenberg J, Monga R, Moore S, Murray D, Steiner B, Tucker P, Vasudevan V, Warden P, Zhang X Q. TensorFlow: a system for large-scale machine learning. In: 12th Symposium on Operating Systems Design and Implementation. Symposium: USENIX Association, 2016. pp 265–283.
[23] Weiss K, Khoshgoftaar T M, Wang D D. A survey of transfer learning., 2016, 3: 9.
[24] Pan S J, Qiang Y. A survey on transfer learning., 2010, 22: 1345–1359.
[25] Russakovsky O, Deng J, Su H, Krause J, Satheesh S, Ma S, Huang Z H, Karpathy A, Khosla A, Bernstein M, Berg A C, Li F F. ImageNet large scale visual recognition challenge., 2015, 115: 211–252.
[26] Srivastava N, Hinton G, Krizhevsky A, Sutskever I, Salakhutdinov R. Dropout: a simple way to prevent neural networks from overfitting., 2014, 15: 1929–1958.
[27] Christian S, Vincent V, Sergey I, Jonathon S, Zbigniew W. Rethinking the inception architecture for computer vision. In: IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Las Vegas: IEEE Computer Society,2016. pp 2818–2826.
[28] Lin M, Chen Q, Yan S. Network in network. In: Bengio Y, LeCun Y, eds. International Conference on Learning Representations. Banff: Conference Track Proceedings, 2014. https://arxiv.org/abs/ 1312.4400.
[29] Duchi J, Hazan E, Singer Y. Adaptive subgradient methods for online learning and stochastic optimization., 2011, 12: 257–269.
[30] Kingma D, Ba J. Adam: a method for stochastic optimization. In:Bengio Y, LeCun Y, eds. 3rd International Conference on Learning Representations. San Diego: Conference Track Proceedings, 2015. https://arxiv.org/abs/1412.6980v8.
[31] Ng A Y. Feature selection, L1 vs. L2 regularization, and rotational invariance. In: Proceedings of the Twenty-first International Conference on Machine Learning. New York: Association for Computing Machinery, 2004. pp 78–86.
[32] Goodfellow I, Bengio Y, Courville Y. Deep Learning. Cambridge: MIT Press, 2016 [2019-11-26].http://www.deeplearningbook.org.
[33] Ruder S. An overview of gradient descent optimization algorithms., 2016, abs/1609.04747. https://arxiv.org/abs/1609. 04747.
[34] Arnal B J G. Impact of dataset size and variety on the effectiveness of deep learning and transfer learning for plant disease classification., 2018, 153: 46–53.
[35] Kamilaris A, Prenafeta-Boldú F X. Deep learning in agriculture: a survey., 2018, 147: 70–90.
[36] Mehdipour G M, Yanikoglu B, Aptoula E. Plant identification using deep neural networks via optimization of transfer learning parameters., 2017, 235: 228–235.
Classification of soybean pods using deep learning
YAN Zhuang-Zhuang2, YAN Xue-Hui2, SHI Jia2, SUN Kai2, YU Jiang-Lin2, ZHANG Zhan-Guo1, HU Zhen-Bang3, JIANG Hong-Wei3, XIN Da-Wei3, LI Yang1, QI Zhao-Ming3, LIU Chun-Yan3, WU Xiao-Xia3,CHEN Qing-Shan3, and ZHU Rong-Sheng1,*
1College of Arts and Sciences, Northeast Agricultural University, Harbin 150030, Heilongjiang, China;2Engineering College Northeast Agricultural University, Harbin 150030, Heilongjiang, China;3Soybean Research Institute, Northeast Agricultural University, Harbin 150030, Heilongjiang, China
Crop phenotype investigation is a key task in the selection and breeding of crop varieties. The traditional phenotypic survey mainly relies on human labors, which makes the results of the phenotypic survey difficult to meet the requirements of automation, high precision and high reliability. In the investigation of soybean phenotypes, the correct identification of pod types is the key and premise for the accurate extraction of phenotypes such as the number, length and width of pods. This study focused on the pictures of mature soybean pods by using deep learning to migrate five different network models [AlexNet, VggNet (Vgg16, Vgg19), GoogleNet, ResNet-50], to identify one-pod, two-pod, three-pod, and four-pod. In order to improve training speed and accuracy, this experiment fine-tuning the model and selected different optimizers (SGD, Adam) to optimize the network model. Adam’s performance was better than SGD in the problem of pod identification. With the Vgg16 network model and the Adam optimizer, the test accuracy of the pod category reached 98.41%, which reflected the best performance in the selected network model. In the 10-fold cross-validation test, the Vgg16 network model had good stability. Therefore, this study indicates that the Vgg16 network model can be applied to the actual identification of pods, and provide an important solution for further automatic extraction of pod phenotypes.
soybean breeding; pod identification; deep learning; transfer learning
10.3724/SP.J.1006.2020.94187
本研究由国家自然科学基金项目(31471516)和国家自然科学基金青年项目(31400074)资助。
This study was supported by the National Natural Science Foundation of China (31471516) and the National Natural Science Foundation of China Youth Project (31400074).
朱荣胜, E-mail: rshzhu@126.com, Tel:0451-55191945
E-mail: zhuangyanneau@163.com
2019-11-26;
2020-07-02;
2020-07-13.
URL: https://kns.cnki.net/kcms/detail/11.1809.S.20200713.1153.004.html
