基于生物信息学分析F13A1基因及蛋白质
2020-07-17王淮杨健康
王淮 杨健康


摘要:目的 利用生物信息学方法对F13A1及其编码蛋白质进行分析,为F13A1的实验研究提供理论基础和新线索。方法 从UCSC(http://genome.ucsc.edu/index.html)数据库检索“F13A1”,下载F13A1蛋白序列,截取转录起始点上游2000 bp碱基序列作为启动子区域进行分析,分析F13A1编码蛋白的理化性质、亚细胞定位、信号肽和跨膜区域、蛋白质结构、相互作用网络及GO注释。结果 F13A1包含多个启动区域,不存在CpG岛,但存在SRF、Sp1和AP-1等转录因子结合位点。F13A1编码蛋白是由732个氨基酸组成的无信号肽、无跨膜区域的亲水蛋白,其主要定位于细胞核,二级结构以随机卷曲为主。另外蛋白F13B、FGA、FGG、F2、FGB、FN1、SERPINF2、VWF、VEGFA、F5可能与F13A1存在相互作用。结论 F13A1基因存在多个启动子区域,拥有多个转录因子结合位点;F13A1蛋白与F13B、FGA、FGG、F2等多个蛋白质相互作用,其参与组成内质网腔、血小板α颗粒内腔以及细胞内细胞器腔等多种细胞成分,同时在血液凝固、纤维蛋白溶解和血小板活化等多种生理過程发挥重要作用。
关键词:F13A1;启动子;蛋白质;生物信息学
中图分类号:R394.3 文献标识码:A DOI:10.3969/j.issn.1006-1959.2020.11.017
文章编号:1006-1959(2020)11-0052-06
Abstract:Objective To analyze F13A1 and its encoded protein using bioinformatics methods, and provide theoretical basis and new clues for the experimental study of F13A1. Methods Search "F13A1" from the UCSC (http://genome.ucsc.edu/index.html) database, download the F13A1 protein sequence, and intercept the 2000 bp base sequence upstream of the transcription start point as a promoter region for analysis,the physicochemical properties, subcellular localization, signal peptide and transmembrane region, protein structure, interaction network and GO annotation of F13A1 encoded protein were analyzed.Results F13A1 contains multiple promoter regions, there is no CpG island, but there are transcription factor binding sites such as SRF, Sp1 and AP-1. The protein encoded by F13A1 is a hydrophilic protein composed of 732 amino acids with no signal peptide and no transmembrane region. It is mainly located in the nucleus, and the secondary structure is dominated by random coils. In addition, proteins F13B, FGA, FGG, F2, FGB, FN1, SERPINF2, VWF, VEGFA, F5 may interact with F13A1.Conclusion The F13A1 gene has multiple promoter regions with multiple transcription factor binding sites; the F13A1 protein interacts with multiple proteins such as F13B, FGA, FGG, and F2, and is involved in the formation of the endoplasmic reticulum cavity, platelet αparticle cavity, and cells Various cellular components such as the inner organelle cavity play an important role in various physiological processes such as blood coagulation, fibrinolysis and platelet activation.
Key words:F13A1;Promoter;Protein;Bioinformatics
人凝血因子XⅢ(CoagulationFactor XⅢ,FXⅢ)又称纤维蛋白稳定因子,主要功能是共价交联纤维蛋白纤维,稳定纤维蛋白凝块[1]。F13A1基因编码人凝血因子XⅢ的A链。遗传性凝血因子XⅢ纯合或复合杂合缺乏症是一种罕见的严重出血性疾病,2016年Thomas A等[2]首次报道1例覆盖整个外显子12的新的纯合子F13A1缺失的病例,通过实验确定是由内含子11和内含子12的6 bp微同源序列引起。研究表明[3],F13A1具有上调血浆IL-6水平的功能,在痤疮发病过程中扮演重要角色;F13A1基因变异与血浆FXⅢA水平对急性心肌梗死的长期预后具有显著的作用[4]。本文通过生物信息学的方法对F13A1基因及其编码蛋白质进行一系列预测分析,期望能为今后的研究提供线索及方向。
1材料与方法
1.1资料来源 F13A1基因及编码蛋白的序列获取登录UCSC(http://genome.ucsc.edu/index.html)数据库,在GeneSorter中检索“F13A1”,截取转录起始点上游2000 bp[5]碱基序列作為启动子区域进行分析,同时下载F13A1蛋白序列。
1.2方法 本文运用到的分析软件见表1,所有软件都应用默认参数分析F13A1编码蛋白的理化性质、亚细胞定位、信号肽和跨膜区域、蛋白质结构、相互作用网络及GO注释。
2结果
2.1 F13A1基因特征 人F13A1基因总长为176750 bp,在染色体上的位置是chr6:6144085-6320834,包含15个外显子和14个内含子,该基因编码的蛋白质包含732个氨基酸残基。
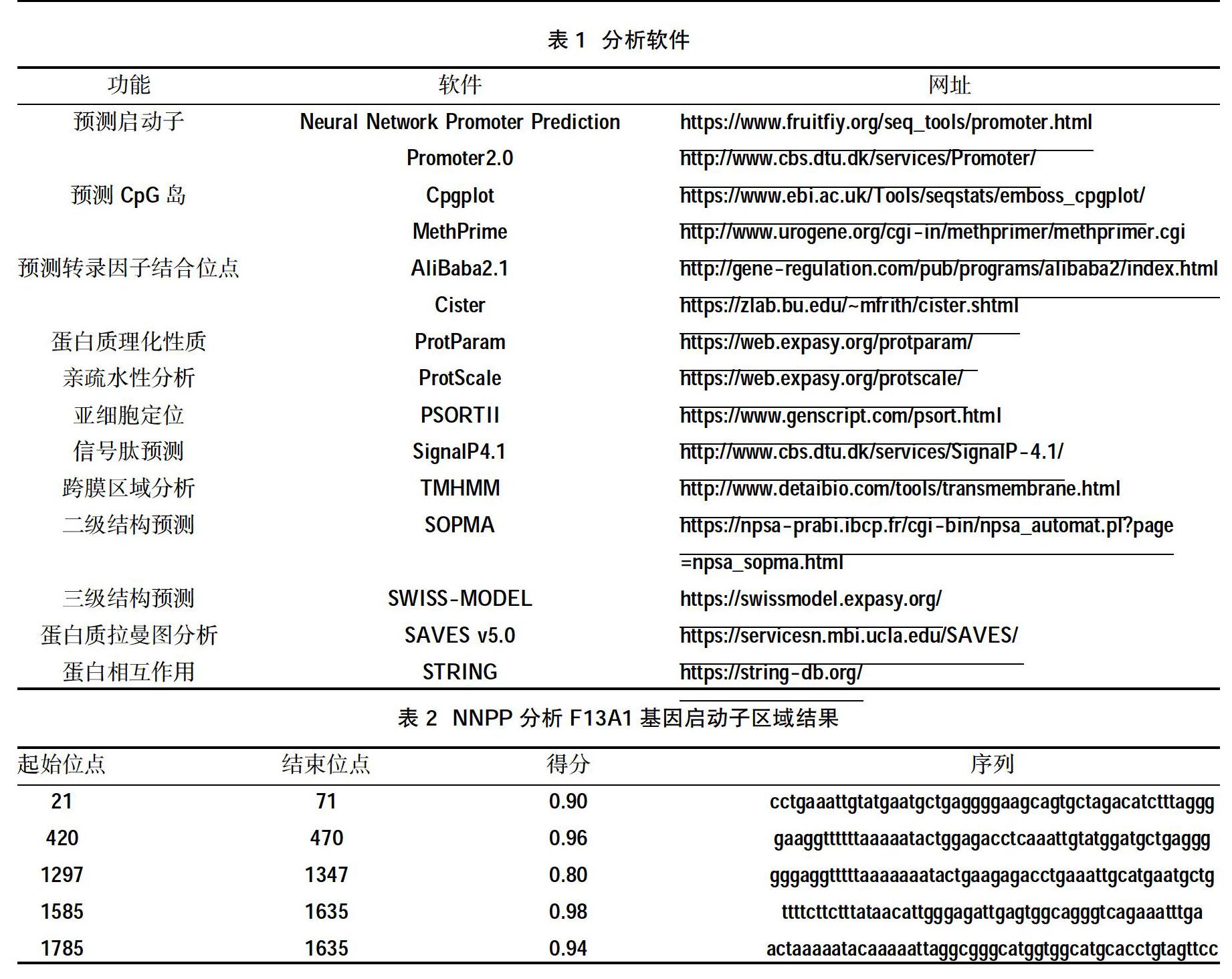
2.2 F13A1基因启动子分析 Promoter2.0分析结果显示,F13A1基因上游存在3个启动子区域,分别位于200 bp,800 bp和1900 bp处,均为临界性预测。NNPP分析结果显示,F13A1基因上游存在5个启动子区域,见表2。推测F13A1基因至少包含3个启动子区域,其中以1585~1635 bp,420~470 bp和1785~1635 bp可能性最大。
2.3 F13A1基因CpG岛预测 在观察值/预期值>0.6,G%+C%>50%,长度>100 bp条件下,运用在线软件Cpgplot和MethPrime对F13A1基因进行CpG岛预测,均未发现甲基化岛,推测F13A1基因启动子区域不包含CpG岛,见图1、图2。
2.4 F13A1基因转录因子结合位点预测 AliBaba2.1的预测结果共有255个,主要包含Sp1、Ap-1、SRF、NF-1、USF等。Cister预测F13A1基因共有27个转录因子结合位点,表3为概率最大的前5个。推测F13A1基因转录因子结合位点至少包含SRF、Sp1和AP-1,此为两个软件的共同预测结果,相对可靠。
2.5 F13A1蛋白质的理化性质分析 ProtParam分析结果显示,人F13A1蛋白质共有732个氨基酸残基,分子质量为83267.30 Da,理论等电点值为5.75,属酸性蛋白质。分子式为C3711H5743N1015O1110S29,原子总数为11608。在732个氨基酸残基中带负电荷的氨基酸残基(Asp+Glu)的数量为95,带正电荷的氨基酸残基(Arg+Lys)的数量为83。F13A1蛋白质的不稳定系数为37.30,属于稳定蛋白质,脂肪系数为79.00,总的平均亲水性为-0.391,属于亲水性蛋白。Protscale分析结果显示,位于201位的谷氨酸(E)亲水性最强,分值为-3.411;位于631位的脯氨酸(P)疏水性最强,分值为2.244。可知F13A1蛋白的疏水区域少于亲水区域,属于亲水蛋白,该结果与ProtParam结果一致,可信性高,见图3。
2.6 F13A1蛋白的亚细胞定位、信号肽和跨膜结构域预测 PSORTII对F13A1蛋白的亚细胞定位的预测结果显示,位于细胞核的可能性为34.8%,位于线粒体的可能性为26.1%,位于细胞质的可能性为26.1%,位于分泌系统囊泡的可能性为13.0%。SignalP4.1的预测结果显示,C、Y、S的最大值分别为0.109、0.108、0.125,S-mean的值为0.107,D值为0.108,可知F13A1蛋白不存在剪切位点,不是分泌蛋白,见图4。TMHMM预测F13A1蛋白的跨膜区域结果显示,该蛋白不存在跨膜区域,不属于膜蛋白,见图5。
2.7 F13A1蛋白二级结构与三级结构预测 SMOPA预测F13A1蛋白质的二级结构结果显示,随机卷曲共有358个,占48.91%;延伸链共有204个,占27.87%;α螺旋共有131个,占17.90%;β转角有39个,占5.33%。其中大部分氨基酸处于随机卷曲状态,F13A1蛋白整体处于比较松散的状态,见图6。
蛋白质的高级结构决定其生理功能,采用同源建模法预测蛋白质的三级结构,将F13A1蛋白序列提交至SWISS-MODEL,得到其高级结构见图7。其中预测模型A采用的模板为1f13.1.A,覆盖率为100%,序列相似度为63%;预模型B采用的模板为3ohx.1.B,覆盖率为15%,序列相似度为26%;预测模型C采用的模板为2ice.1.aA,覆盖率为9%,序列相似度为30%。综合比较,预测模型A的可信度最高
为验证模型A的可信度,通过蛋白质拉曼图在线分析软件SAVES v5.0对预测模型的氨基酸的二面角进行分析,判断预测模型的可靠性,得到结果见图8。预测结构中所涉及的1279个氨基酸,其中1149个处于最佳区域,即红色区域;126个处于允许区域,即黄色区域;另有4个处于允许区域范围之外,说明该预测模型中几乎所有的氨基酸均形成合理的二面角,构成的蛋白质高级结构稳定可信。
