基于时间序列的DNA 特征分析
2020-06-22王楚雯方宝琦
王楚雯 方宝琦 许 瑶
(大连民族大学理学院,辽宁 大连116600)
1 概述
生物学的相关信息量革命性的爆炸,产生了对海量生物信息进行处理的需求,而计算机技术的革命性发展,形成了处理海量生物信息的能力。生物信息学是从大量生物信息中提取生物学知识的学科,其研究了DNA、RNA 和蛋白质分子,这些大分子包含了所有物种遗传及其进化的信息。如何在DNA 中探索更多的生物信息是有难度的,碱基在基因库中的增长是迅速的,利用线粒体DNA 进行分析是最有效、最快速的方法,线粒体DNA 是在生物系统研究中应用最为广泛的遗传物质之一。线粒体DNA 较核DNA 进化速率快,并在遗传过程不发生基因重组、倒位、易变等突变,严格遵守母系遗传方式的特点。在此本文对线粒体中携带的mtDNA 的一般属性进行分析,随机选取30 个哺乳动物的线粒体DNA 序列,利用短记忆ARIMA 模型进行建模,探究不同物种间的系统关系及特征。
2 模型介绍
短记忆ARIMA 模型:
具有如下结构的模型称为求和自回归移动平均模型,简称为ARIMA(p,d,q)模型:
其中,非负整数d 为求和阶数,Φ(B)=1-φ1B-…-φpBp,为平稳可逆ARMA(p,q) 模型的自回归系数多项式;Θ(B)=1-θ1B-…-θqBq为平稳可逆ARMA 模型的移动平均系数多项式。
d 阶差分算子:
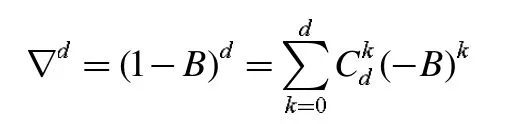
显然,ARIMA 模型实质就是差分运算与ARMA 模型的组合,说明只要任意序列只要通过适当阶数的差分实现差分后平稳,就可以对差分序列进行ARMA 模型拟合了。
ARIMA 模型建模的基本步骤为:①判断观察值序列的平稳性。②对原序列进行一阶差分运算。对序列进行平稳性检验、白噪声检验、残差序列检验、模型预测。如果序列非平稳则重新建立模型。
3 线粒体DNA 的研究分析
随机抽取30 个哺乳动物的线粒体DNA,对于线粒体DNA携带的mtDNA 序列进行研究。首先对选取的数据进行初步处理:抽取mtDNA 序列中第四个位置的碱基为研究对象,将DNA中的四种核苷酸A、T、C、G,分别用编号为1,2,3,4 进行碱基的替换,将DNA 字符串转换为数值型变量,即DNA 序列时序化。对30 个哺乳动物中的人类、马、长须鲸、大猩猩、猩猩五种线粒体DNA 携带的mtDNA,建立短记忆ARIMA 模型进行DNA 序列的拟合。下面以人为例。
3.1 mtDNA 序列的平稳性及随机性检验
对human 的mtDNA 序列时序化后的时间序列,进行绘制时序图观察序列的平稳性,如图1 所示,70 个数据的时序图上下波动较大,波动范围有界,但波动有明显趋势性而无周期性,可知为非平稳序列。再对其进行1 阶差分,可看到图二为human 的一阶差分时序图,可看出有平稳性;如图3,4 所示的是human 的mtDNA 序列的自相关图和偏自相关图,可以看出的是差分后的时序图上下波动,但自相关系数、偏自相关系数始终非零,均具有拖尾性。DNA 序列的纯随机检验p 值在延迟6 阶和12 阶后分别为7.543e-06,3.01e-08,均小于显著性水平0.05,故拒绝原假设,认为差分后的human 的DNA 序列为平稳非白噪声序列。

图1 人的时序图

图2 人的一阶差分的时序图

图3 一阶差分自相关图
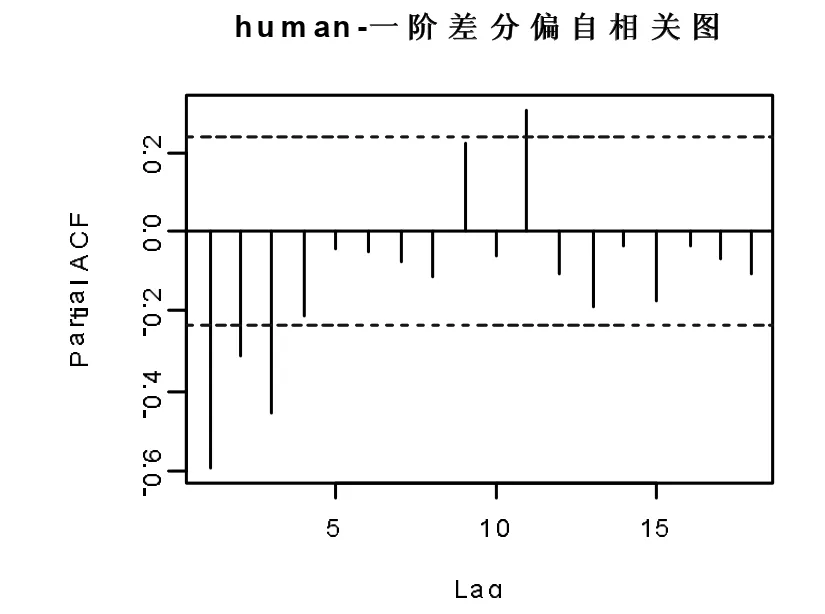
图4 一阶差分偏自相关图
3.2 模型识别及检验
对于平稳非白噪声序列的人的mtDNA 序列,进行短记忆ARIMA(p,d,q)模型的识别,其中d=1,由1 阶差分序列的时序图、自相关图和偏自相关图都表明,差分后的数据具有平稳性,且能看出的是自相关系数在延迟1 阶后都具有拖尾性,故我们首先初步确定ARTMA(1,1,1)模型来拟合mtDNA 的时间序列,拟合的模型结果为:
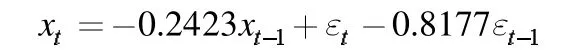
其中aic 值为210.95,再对残差序列做白噪声检验,白噪声检验结果表明, 延迟 6 阶和 12 阶的p 值分别为0.4465,0.09382,其值均大于0.05,因此模型成立,即ARIMA(1,1,1)模型拟合成功,但并不是最优模型,重新建立ARIMA(3,1,1)模型,得到拟合模型为:

其中aic 值为209.73<210.95,再做残差序列的白噪声检验,其结果表明,延迟延迟6 阶和12 阶的p 值分别为0.9999,0.1856,其值均大于0.05,因此模型成立,通过aic 值可以看出,ARIMA(3,1,1)模型为最优模型。
3.3 其他物种DNA 的数据分析
对于我们随机选取的其他4 个物种线粒体DNA 携带的mtDNA 进行同样的模型建立,看是ARIMA 模型是否同样能够适用并且高度拟合,其他的4 个物种分别是马、长须鲸、大猩猩、猩猩,对于这四个物种的mtDNA 序列时序化后的时间序列,进行模型识别、参数估计、模型检验。首先同样先用ARIMA(1,1,1)模型来拟合其他四个物种的mtDNA 的时间序列,再建立ARIMA(3,1,1)模型来逼近ARIMA(1,1,1)模型,可以得到是ARIMA(3,1,1)模型依旧是最优模型,其模型拟合结果为表1。其中模型残差检验在延迟6 阶和12 阶的p 值均大于显著水平0.05,即模型拟合成功,说明利用短记忆ARIMA 模型进行建模,可以探究不同物种间的系统关系及mtDNA 序列特征。

表1 4 条不同物种mtDNA 序列ARIMA 模型

表2 5 条不同物种mtDNA 序列预测值与真实值对比表
3.4 模型预测
对于上述5 个拟合成功的ARIMA(p,d,q)模型,我们对其mtDNA 序列的后五个碱基(71-75)进行预测,来验证短记忆ARIMA(p,d,q)模型是否对线粒体DNA 携带的mtDNA 具有有效性。对于模型的预测值我们将其与真实值进行比对,来检验ARIMA 模型是否高度拟合。下表为五个物种mtDNA 的预测值与真实值对比表。可以见得五个物种的mtDNA 序列的平均误差分别是(见表2)。
4 结果分析
本文通过运用短记忆ARIMA 模型,能清楚看到对于物种之间线粒体DNA 携带mtDNA 的特征,结构以及之间的物种联系。
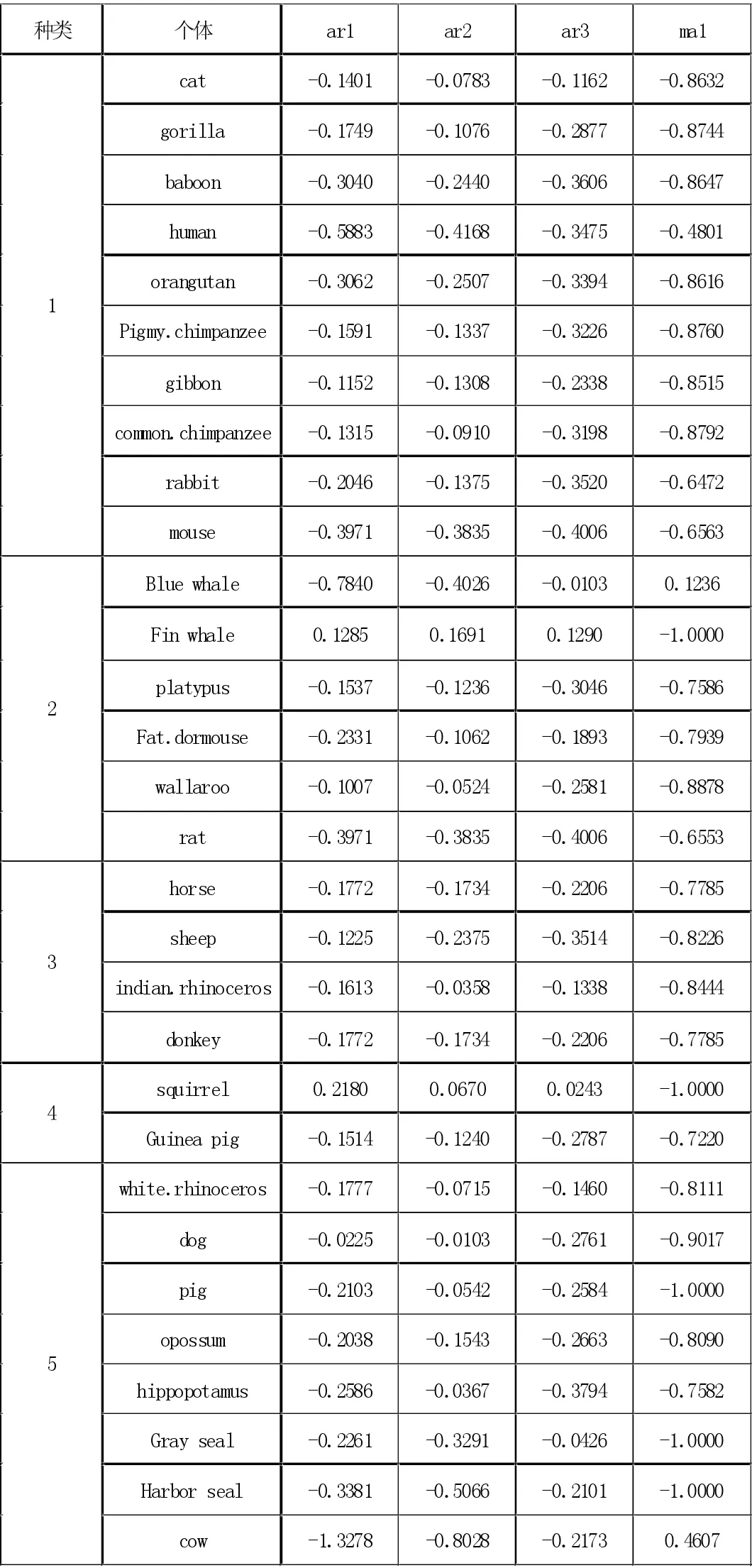
表3 30 个物种mtDNA - ARIMA 模型表
对于30 个物种的mtDNA 再次进行建模分析之间是否有亲属关系以及物种间的相似度,下表为30 条mtdna 根据模型ARIMA 拟合所得参数结果。从表中我们得出30 个线粒体DNA大致分为五大类:
①猫、大猩猩、狒狒、人、猩猩、小黑猩猩、长臂猿、普通黑猩猩、兔子、老鼠、蓝鲸、长须鲸、睡鼠、鸭嘴兽、大袋鼠、鼠;其中大猩猩、狒狒、人类、猩猩、小黑猩猩、长臂猿、普通黑猩猩是有共同特性的。根据生物学知识,人和猩猩的基因差异只有0.75%,按照生物的形态结构、功能以及亲缘关系,它们都被分属于动物界脊椎动物门哺乳纲灵长目类。对于其他物种因为我们所选取的是DNA 片段并不全面,会有片面的判断,因而会造成错误分类,出现误差。
②马、羊、印度犀牛、驴;其中除了羊、马、印度犀牛以及驴都属于动物界脊椎动物门哺乳纲奇蹄目类。
③豚鼠、松鼠;其中豚鼠、松鼠都被分属于动物界脊椎动物门哺乳纲啮齿目类。
④白犀牛、狗、猪、负鼠、河马、灰海豹、斑海豚、牛。第四类没有显著特性,这8 个物种也不具备相似的亲缘关系。
从上述结果分析来看,30 个物种都可以用ARIMA(p,d,q)模型进行有效合理的拟合,不排除个别个体的差异性,拟合结果都较好,则表明模型建立的合理,如此一来,我们可以利用此模型更准确地估计随机时序发展变化的规律并且对其进行研究,利于我们生物学进行根深一步的发展和探索。
对于物种之间DNA 的检验不仅仅只局限于线粒体DNA 的研究,也不仅仅局限于这30 个物种,生物信息学所含括的还有很多,都可以运用时间序列建立模型进行探索。物种间的遗传和进化还存在于RNA,蛋白质等大分子中,同样可以利用短记忆模型。本文基于时间序列对DNA 特性的分析,其结果是DNA分子具有短记忆性,在物种间的联系可将生物圈分为不同类别。
