基于群优化拟合及临床数据的癌症lncRNA预测技术研究
2020-01-03王波李玲玲刘佰泉陶佰睿李敬有
王波 李玲玲 刘佰泉 陶佰睿 李敬有

摘 要:提出一种群优化拟合方法,通过测试函数和优化模型,分析验证了其具有较好的优化能力。采用群优化拟合方法计算了关键特征集合,并结合临床数据提出了一种基于群优化拟合及临床数据的癌症lncRNA预测方法。该方法在关键特征集合的基础上采用判别分析完成预测,预测过程中采用马氏统计距离的最小原则。实验结果表明,该方法获得了较好的收敛性能,在精确度、召回率和F1-Score三个指标上都达到了较好的预测结果。
关键词:群优化拟合;临床数据;lncRNA预测技术;癌症
中图分类号:TP391 文献标识码:A 文章编号:2096-4706(2020)16-0008-05
Prediction Technology Study of Cancer lncRNA Based on Swarm Optimization Fitting and Clinical Data
WANG Bo1,2,LI Lingling3,LIU Baiquan3,TAO Bairui4,LI Jingyou4
(1.College of Computer and Control,Qiqihar University,Qiqihar 161006,China;2.College of Computer Science and Technology,Harbin Engineering University,Harbin 150001,China;3.Network Information Center,Qiqihar University,Qiqihar 161006,China;4.School of Communications and Electrical Engineering,Qiqihar University,Qiqihar 161006,China)
Abstract:In this paper,a method of swarm optimization fitting was proposed,which was proved to have good optimization ability by test function and optimization model. The pivotal feature set was calculated by the method of swarm optimization fitting,and a prediction method of cancer lncRNA based on swarm optimization fitting and clinical data was proposed in combination with clinical data. The method used discriminant analysis to complete the prediction based on pivotal feature set,and the Mahalanobis statistical distance principle was adopted in the prediction process. Experimental results show that this method achieved good convergence performance and good prediction results in accuracy,recall rate and F1-Score.
Keywords:swarm optimization fitting;clinical data;lncRNA prediction technology;cancer
0 引 言
lncRNA是一種不具有编码功能且长度大于200个核苷酸的RNA。研究表明很多复杂的疾病都与lncRNA的变异或异常表达相关,在分子层面对致病lncRNA的研究可以找到致病的生物靶标和药物靶标[1,2]。目前研究lncRNA与肺癌、乳腺癌、前列腺癌、结肠直肠癌、胃癌、膀胱癌和宫颈癌等有密切关系[3-5],各种相关数据库也在逐步完善[6-9]。本文提出采用群优化拟合方法完成关键特征集合的计算,并结合临床数据实现了lncRNA与疾病关联的预测,实验表明该方法有较好的预测性能。本文受黑龙江省教育厅基本科研业务专项,齐齐哈尔大学科学研究类项目的支持,目前已经完成与疾病关联的lncRNA预测技术的相关研究,完成数据的整理和预测模型测试与调试,实验结果良好。
1 lncRNA关键特征选择
将研究对象抽象为lncRNA向量lncRNA={lncRNAi,i∈
[1,N]},N个lncRNAi中的关键特征选择是进行与疾病关联预测lncRNA的预处理过程,设每个lncRNAi的影响度为influence-degreei,influence-degreei的动态调整会得的lncRNA的不同的总体评价值ΛlncRNA,lncRNA的ΛlncRNA的计算公式如下:
当ΛlncRNA达到最大值的时候,取前Γ个lncRNAi为关键特征集合lncRNAiΦ(Φ表示为关键特性),lncRNAiΦ的总体评价值ΛlncRNAΦ的计算公式为:
这样求解lncRNA关键特征选择,抽象为一个最优化问题,求得ΛlncRNA达到最大值采用群优化拟合方法实现。
2 LncRNA预测
在进行lncRNA预测之前,要将lncRNAiΦ与临床数据进行关联,本文研究用到的临床数据来自于TCGA数据。与lncRNAiΦ关联的临床数据为clinical-,最终预测数据集合为ΦlncRNA-clinical=lncRNAiΦ∪clinical-。
2.1 预测数据集的平滑处理
预测数据集合ΦlncRNA-clinical的数据集中会有一些缺失或者噪声数据,因此需要对ΦlncRNA-clinical进行数据的平滑处理,如果不对ΦlncRNA-clinical进行平滑处理,会使算法执行异常或者出现执行的结果偏差较大等情况。对数据的平滑处理可以选用均值平滑和边界平滑。对于缺失数据和噪声数据通过不同的方法完成平滑处理,平滑处理有两个准则:
准则1(缺失-边界):对于缺失数据,ΦlncRNA-clinical具有整体性和局部性,往往缺失数据的局部性可能对其真实值的影响更大,所以对于缺失数据的平滑处理选用边界平滑。
准则2(噪声-均值):对于噪声数据,分析ΦlncRNA-clinical的数据分布特征发现,它的方差较大,说明数据的波动较大。此时如果选用边界平滑,会出现如果待处理数据的边界恰好是波动最大值,往往这样的数值有存在异常的可能性,所以此时用边界平滑的的方法会使这种异常的出现概率提升。所以针对噪声数据选择均值平滑,用噪声异常值总体的均值对噪声数据进行平滑处理。
上述平滑技术可以使ΦlncRNA-clinical更加完整,可提高算法的执行精度。
2.2 群优化拟合
群优化拟合方法的目标是使拟合函数?最大或者最小,本研究拟合函数?为ΛlncRNA。通过群体的仿生运动,从而实现?的优化。群拟合优化方法有下文所述三类运动方式。
2.2.1 方式1:散漫随机运动
该运动方式为在进化初期为了得到全局最优值,群体执行散漫随机运动,个体可以根据自身的方向倾向性,而自行运动,这样也使整个群体的运动区域具有全局性,可以保证在全局范围内寻优。
2.2.2 方式2:原地避让运动
由于个体在散漫随机运动的过程中,可能会有不同的个体在某一时刻恰好运动到系统同一地点,由于某个地点只能允许一个个体占有,此时就发生了碰撞,那么需要其中一个个体执行原地避让运动,个体中能量最高的占有这个位置,而能量较低的其他个体要原地避让,等待下一时刻搜寻运动地点。
2.2.3 方式3:域内群聚运动
在进化的后期,由于此时若再执行散漫随机运动,可能会使优化趋势被破坏,所以这个时候在选择下一时刻运动的位置时,应该考虑此时群体聚集的趋势中心点的位置,应该向这个中心点运动。这样群聚的方向即为最优解的方向。
2.2.4 强制机制
在群体的进化运动过程中会遇到个体盲选的情况,就是該个体不知道未来时刻的运动位置方向,此时我们需要执行强制机制。强制机制的原理是:个体沿着逆时针方向旋转(1≤integer(θ)≤Ω),在所有试探的方向中选择一个最佳的位置;设个体为ΦlncRNA-clinicali,当前位置为locationi,按角度 旋转的位置为locationi|。设第ω个位置为最佳位置的表示公式为:
群优化拟合算法描述如下,群优化拟合流程图如图1所示。
步骤1:种群的初始化,初始化迭代次数及参数,设置拟合函数?的公告板。
步骤2:判断当前迭代次数d是否大于最大迭代次数D的1/2,如果是转到步骤3,否则转到步骤4。
步骤3:执行域内群聚运动,更新群体的全部个体的信息。
步骤4:执行散漫随机运动,更新群体的全部个体的信息。
步骤5:判断当前状态中是否碰撞,如果是转到步骤6,否则转到步骤7。
步骤6:执行原地避让运动,对碰撞个体不做更新操作,其余个体执行更新操作。
步骤7:判断当前状态中是否有盲选,如果是转到步骤8,否则转到步骤9。
步骤8:执行强制机制,个体沿着逆时针方向旋转 (1≤integer(θ)≤Ω),在所有试探的方向中选择一个最佳的位置。
步骤9:更新公告板,获得当前最优值。
步骤10:判断是否达到了最大迭代次数,如果是转到步骤11,否则转到步骤2。
步骤11:算法终止,输出最优值。
2.3 三种运动的四个机制
群优化拟合的散漫随机运动、原地避让运动和域内群聚运动具有不同的运动机制。
机制1:散漫随机运动,由于其运动速度与运动方向都具有很大的随机性,这样可以增加解空间的基数,基数越大寻优的可能空间就越大。
机制2:散漫随机运动还具有动态性,可在不同时刻动态变化个体的运动速度和运动方向。
机制3:原地避让运动,在整个进化过程中加入了压抑机制,因为种群都处于活跃状态会使碰撞概率更大,可能算法会频繁地解决处理碰撞,导致算法的负载过重,性能严重下降。引入原地避让运动就是为了减低碰撞概率,减轻算法的负载。这里选择原地避让而没有选择变化位置的避让,原因是在进化过程中为了保证局域寻优结果,所以选择原地避让是最好方案,若选择其他位置进行避让则无法保证当前寻优结果的准确性。
机制4:域内群聚运动,这个机制在进化的后期执行,这里采用的是聚类的思想,即往往个体运动的方向是群体选择最多的运动方向,而这个方向可能是最优的结果方向。
2.4 群优化拟合的实例化
群优化拟合方法用于计算关键特征选择,需要将群优化拟合方法进行实例化,种群个体为lncRNA集合,在这集合中我们需要计算出关键特征。每一个个体就是某一个lncRNA,任意一个lncRNA执行群优化拟合方法中的三种运动。在群优化拟合方法中的lncRNA,除了本身表达值之外,还有两个附加信息,就是lncRNA的位置和方向,此时lncRNA可以理解为是一个三维向量。其中,三维向量的位置信息用于马氏统计距离的判定使用,在移动的过程中,下一时刻的位置发生变化,该向量的位置信息会更新变化。由于个体的周围会存在着若干个移动中心点,那么个体需要根据方向再结合马氏统计距离,综合判定下一时刻要移动的位置。此外,群优化拟合的目标实例化为拟合函数?,这里要求解的是拟合函数?的最大值,在?达到最大值时,为最终的最优解。整个群优化拟合过程中都是以?最大值为目标,所以每次迭代都要更新?,每一个寻优的动作都是以?最大为准则,当算法达到了最大迭代次数后,算法结束。
2.5 lncRNA预测
本文提出了基于群优化拟合及临床数据的癌症lncRNA预测方法(Prediction method of cancer lncRNA based on swarm optimization fitting and clinical data,PCL-SOF-CD),PCL-SOF-CD采用群优化拟合方法计算了关键特征集合,在关键特征集合的基础上采用判别分析完成lncRNA预测。首先根据已知预测标签的数据,分别计算各个预测标签的中心点;其次,对于任意一个学习数据判别它与中心点的马氏距离;最后,根据距离最小原则完成预测。
3 实验性能分析与讨论
3.1 群优化拟合的进化性能
本文出的群优化拟合方法在计算关键特征集合lncRNAiΦ的前Γ个lncRNAi时,寻优曲线如图2所示,该方法在150代的时候,就趋于平稳,获得较好的收敛性能。
3.2 群优化拟合的优化能力
为了进一步验证群优化拟合方法的优化能力,选用了如表1所示的三个测试函数来分析优化能力,其中Griewank和Rastrigin为高维度测试函数,Rosenbrock为不确定维度测试函数,三个测试函数的最优值均为0。其中,xi为第i个变量,i为xi的个数;D为维度。
图3显示了三个测试函数的迭代曲线,其中Rosenbrock在迭代220次时达到收敛,Griewank在迭代580次时达到收敛,Rastrigin在迭代700次时达到收敛,这说明群优化拟合方法达到了较好的收敛性能。
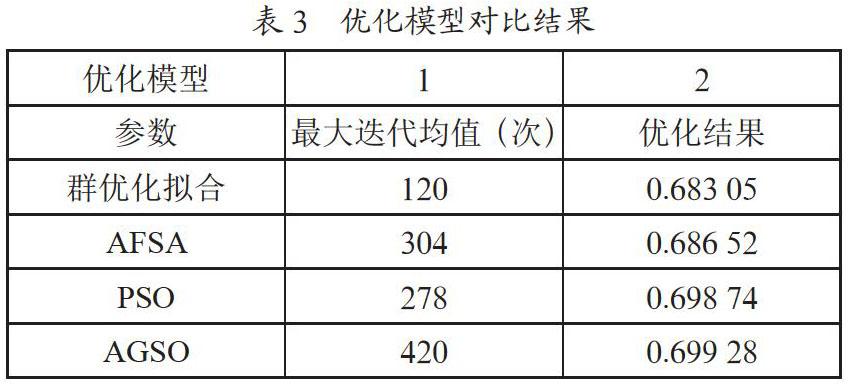
同时如表2所示,选用2个优化模型综合分析群优化方法的优化能力(对比方法为AFSA、PSO和AGSO)。
优化结果如表3所示,对于优化模型1,群优化拟合在迭代120次时达到了最优值,AFSA在迭代304次時达到了最优值,PSO在迭代278次时达到了最优值,AGSO在迭代420次时达到了最优值,显然群优化拟合求解速度最快。对于优化模型2,群优化拟合的优化结果为0.683 05,AFSA的优化结果为0.686 52,PSO的优化结果为0.698 74,AGSO的优化结果为0.699 28,显然群优化拟合方法的求解精度最高。
3.3 PCL-SOF-CD算法性能
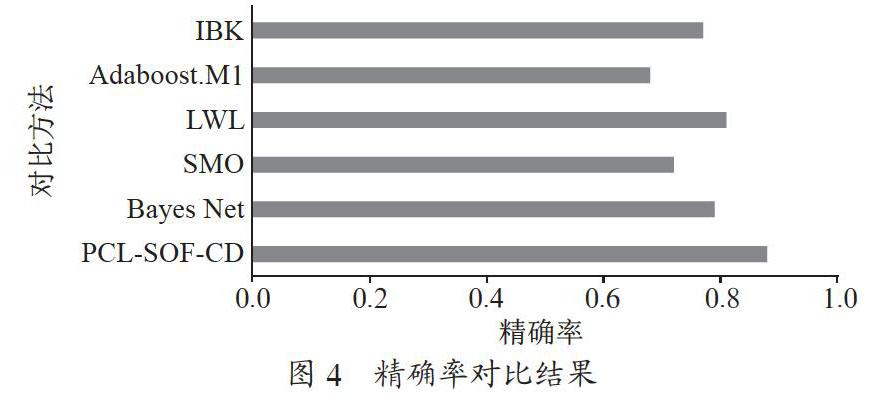
本文提出的PCL-SOF-CD与5个对比方法进行了对比分析,这5个对比方法为:Bayes Net、SMO、LWL、Adaboost.M1和IBK。实验执行了10折交叉验证,对比指标为精确率、召回率和F1-Score。如图4所示,PCL-SOF-CD的精确率为0.88,Bayes Net为0.79,SMO为0.72,LWL为0.81,Adaboost.M1为0.68,IBK为0.77,综上可见PCL-SOF-CD精确率最高。
如图5所示,PCL-SOF-CD的召回率为0.81,Bayes Net为0.71,SMO为0.68,LWL为0.66,Adaboost.M1为0.63,IBK为0.74,综上可见PCL-SOF-CD的召回率最高。
如图6所示,PCL-SOF-CD的F1-Score为0.84,Bayes Net为0.75,SMO为0.70,LWL为0.73,Adaboost.M1为0.65,IBK为0.75,综上可见PCL-SOF-CD的F1-Score最高。根据对比结果可以得知,PCL-SOF-CD在精确率、召回率和F1-Score三个指标上都达到了较好的预测性能。
4 结 论
本文提出一种群优化拟合方法,定义了该方法的3种运动方式:散漫随机运动、原地避让运动和域内群聚运动。基于群优化拟合方法计算了关键特征集合,并结合临床数据采用判别分析实现了lncRNA与疾病的关联预测,提出了一种基于群优化拟合及临床数据的癌症lncRNA预测方法,实验表明该方法具有很高的推广价值。
参考文献:
[1] WASHIETL S,KELLIS M,GARBER M. Evolutionary dynamics and tissue specificity of human long noncoding RNAs in six mammals [J].Genome Research,2014,24(4):616-628.
[2] GUTTMAN M,RINN J L. Modular regulatory principles of large non-coding RNAs [J].Nature,2012,482(7385):339-346.
[3] HUARTE M. The emerging role of lncRNAs in cancer [J].Nature Medicine,2015,21(11):1253-1261.
[4] LI J,XUAN Z Y,LIU C N. Long Non-Coding RNAs and Complex Human Diseases [J].IJMS,2013,14(9):18790-18808.
[5] CHEN X,SUN Y Z,GUAN N N,et al. Computational models for lncRNA function prediction and functional similarity calculation [J].Briefings in functional genomics,2019,18(1):58-82.
[6] JANG S Y,KIM G,PARK S Y,et al. Clinical significance of lncRNA-ATB expression in human hepatocellular carcinoma [J].Oncotarget,2017,8(45):78588-78597.
[7] MIAO Y,SUI J,XU S Y,et al. Comprehensive analysis of a novel four-lncRNA signature as a prognostic biomarker for human gastric cancer [J].Oncotarget,2017,8(43):75007-75024.
[8] MO X B,WU L F,ZHU X W,et al. Identification and evaluation of lncRNA and mRNA integrative modules in human peripheral blood mononuclear cells [J].Epigenomics,2017,9(7):943-954.
[9] ZHANG Y L,LI X B,HOU Y X,et al. The lncRNA XIST exhibits oncogenic properties via regulation of miR-449a and Bcl-2 in human non-small cell lung cancer [J].Acta Pharmacologica Sinica,2017,38(3):371-381.
作者简介:王波(1980—),男,汉族,黑龙江齐齐哈尔人,副教授,博士生,研究方向:与复杂疾病关联的lncRNA预测技术。
