不同耐盐性高粱在盐逆境下的比较转录组分析
2019-12-19张飞王艳秋朱凯张志鹏朱振兴卢峰邹剑秋
张飞,王艳秋,朱凯,张志鹏,朱振兴,卢峰,邹剑秋
不同耐盐性高粱在盐逆境下的比较转录组分析
张飞,王艳秋,朱凯,张志鹏,朱振兴,卢峰,邹剑秋
(辽宁省农业科学院高粱研究所,沈阳 110161)
【】土壤盐渍化是制约作物生产的重要非生物胁迫因子之一,高粱耐盐性强,进行高粱耐盐基因挖掘及分子机制研究是开发和利用盐渍土壤的有效途径,通过转录组测序分析与高粱耐盐相关的基因调控机制和代谢通路,挖掘高粱耐盐潜力。通过以筛选出的极耐盐品种八叶齐和盐极敏感品种PL212为试验材料,采用盆栽沙培,在播种后20 d(5叶期)采用180 mmol·L-1的 NaCl 溶液漫灌模拟盐逆境,盐胁迫48 h后取幼叶,并连同对照(未经过盐处理)的同期幼苗共4个样品提取RNA,进行转录组测序,采用qRT-PCR方法对测序结果进行验证。耐盐和盐敏感材料分别在盐渍和非盐渍处理下的4个样品间共检测到1 338个差异表达基因,包括819个上调基因和519个下调基因。聚类分析发现在应答盐渍胁迫逆境时,5个依赖性氧合酶超家族蛋白、4个富含半胱氨酸的激酶、3个谷胱甘肽S-转移酶和3个重金属运输/解毒超家族蛋白相关基因表现出明显的上调表达和下调表达,还发现1个K+转运蛋白基因在耐盐调节中起着重要作用。GO分析发现在15 418个基因中获得4 528个有效GO注释条目,同时耐盐和盐敏感材料在遭受盐逆境时的生物过程、细胞组分和分子功能3个方面均存在较大差异。生物过程中代谢过程、细胞过程耐盐材料明显高于盐敏感材料,耐盐材料的生理过程中较盐敏感材料增加了多生物过程和定位这两个过程,很可能与耐盐材料盐抗性较强密切相关。差异基因KEGG分析结果显示耐盐和盐敏感材料在对照和盐渍胁迫条件下的苯丙烷类生物合成、苯丙氨酸代谢、类黄酮生物合成3个途径中差异基因表达较多,可能是造成耐盐和盐敏感材料耐盐性差异较大的重要原因。高粱耐盐调控基因表达涉及生物过程、细胞组分和分子功能多个方面,生物过程和定位这两个过程是提高高粱耐盐性的关键;苯丙烷类生物合成、苯丙氨酸代谢、类黄酮生物合成3个途径的基因表达很可能是造成盐害的重要原因。
高粱;耐盐;转录组;差异基因表达;生理调控
0 引言
【研究意义】土壤盐渍化是一个全球性的资源和环境问题,严重制约着作物的生产。中国盐渍化土壤面积约为3 600万hm2,占全国可利用土地的4.88%[1],且盐渍化土壤面积呈逐年上升趋势,越来越多的土地由于盐渍化程度过高而无法利用,造成土地资源的荒废[2]。因此,如何利用盐渍化土地进行作物栽培及生产已成为农业发展的重要需求[3-4]。高粱是公认的耐盐性较强的作物,发掘高粱耐盐基因,研究高粱耐盐的代谢途径,挖掘高粱耐盐潜力对促进盐渍土地高粱生产及盐渍土地资源的开发利用都尤为重要。【前人研究进展】前人研究表明,土壤盐渍化对作物具有多重危害,盐胁迫会引起作物生理代谢紊乱,如光合效率下降、呼吸作用加剧、蛋白质合成受阻、有毒物质积累及加速衰老和死亡等[4]。在作物耐盐基因及分子机制研究方面,学者们开展了大量研究。曾有研究表明植物的耐盐性涉及多个基因,由多种机制协调作用[5]。也有报道指出盐胁迫应答相关的基因涉及离子转运、细胞防御、生理代谢及细胞生长等诸多方面,这些基因以不同方式协同作用抵御盐渍逆境,如编码与光合作用相关的基因、渗透调节基因、自由基清除酶基因和液泡区域化酶基因等[6]。随着生物技术的发展和耐盐机理研究的不断深入,Liu等[7]发现盐胁迫下从水稻中分离的2个水通道蛋白基和在根部的表达增强;此外,一些与渗透调节相关的基因也被发现与耐盐相关,如与果聚糖、脯氨酸、糖醇、甘氨酸等生物合成相关的基因、SOS[8];也有研究表明Zn/Cu SOD是定位于叶绿体中的超氧化物歧化酶,转Zn/Cu SOD基因烟草在氧化胁迫下较对照植株耐盐性有所增强[9]。另外,近年来,众多学者针对植物耐盐代谢机制开展了大量研究,其中,以生理机制研究为主,主要包括植物对盐胁迫的感应及信号传导、Na+运输、盐胁迫下的解毒途径等几个方面[10]。【本研究切入点】尽管在其他作物耐盐基因挖掘及耐受性分子机制方面取得了一定的研究进展,但对高粱耐盐性的研究仍停留在形态和生理研究水平上,在高粱耐盐基因挖掘和代谢途径等方面研究较少。【拟解决的关键问题】本研究以筛选出的对盐胁迫反应极端的2个典型高粱品种为材料,通过转录组研究分析高粱耐盐基因表达及基因调控路径和网络,以挖掘高粱耐盐潜力,为耐盐品种培育和盐碱地高效利用提供技术参考。
1 材料与方法
1.1 试验设计
试验以筛选出的对盐胁迫反应极为典型的2个材料八叶齐(极耐盐型)和PL212(极盐敏感型)为试验材料。于2017年在辽宁省农业科学院(沈阳)人工气候室进行,昼/夜温度为25℃/18℃,湿度为48%,黑暗12 h/光照12 h,光照强度为1 085 μmol·(m2·s1)-1。试验采取盆栽方式,塑料盆大小为:直径20 cm,盆深18 cm,每盆装0.8 kg过筛细沙,其中细沙先用清水洗干净,再用双蒸水清洗干净并晾干。采用称重法对土壤水分进行检测,待细沙含水量为20%时开始播种,每盆6穴,每穴2株,待出苗后每盆定苗2株。
1.2 用于转录组测序样品的处理
播种后20 d(5叶期),分别用蒸馏水漫灌(对照,CK)和180 mmol·L-1的NaCl盐溶液漫灌模拟盐胁迫对八叶齐(极耐盐型)和PL212(极盐敏感型)进行处理,处理时间48 h。以下分别简称为八叶齐对照(CK- tolerant)、八叶齐盐处理(CK-sensitive)、八叶齐盐处理(salt-tolerant)和PL212盐敏感(salt-sensitive)。之后提取处理和对照幼叶样品的RNA。
RNA提取及质量检测:采用Trizol(Invitroge)试剂提取叶片总RNA[11],并用DNaseⅠ(TaKaRa)去除DNA。利用1.2%琼脂糖凝胶电泳进行RNA条带初步分析;利用NanoDrop(ThermoFisher)检测浓度等参数;利用Agilent 2100 Bioanalyzer(Agilent)检测RNA样品的完整度。完整度RIN≥7.0,且挑选浓度等指标合格者进行高通量测序。
1.3 Illumina测序
转录组测序委托生物科技有限公司完成。简要流程如下:样品提取总RNA后,利用带有Oligo (dT)的磁珠富集mRNA,其后将mRNA打断成短片段,再以片段后的mRNA为模板,合成cDNA第一链,继而合成cDNA第二链,纯化并经末端修复、加碱基A,加测序接头,再经琼脂糖凝胶电泳回收目的大小片段,并进行PCR扩增,从而完成整个文库制备工作,构建好的文库上机进行测序。
1.4 测序数据生物信息学分析
测序数据进行生物信息学处理,首先进行测序原始数据的处理得到去除接头等序列的clean data。而后将这些数据进行参考基因组比对,将序列定位到高粱基因组上,过滤掉不能定位到基因组上序列,然后基于这些序列进行测序质量的评估和基因表达量等分析。
1.5 差异基因筛选及验证
1.5.1 基因表达定量 首先使用Htseq软件提取基因的read数目,利用Reads Per RPKMKilo bases per Million reads方法计算基因表达量。其公式为:

其中,total exon reads/mapps (millions)为所有read数中有百分之多少map到这个基因,除以基因长度,就可以获得基因单位长度有百分之多少的total mapped read有表达。转录组测序中的基因表达水平用值表示。
1.5.2 差异表达基因筛选 通过比较不同样本间的数据从而筛选出差异表达基因,后续分析中的差异基因表达模式聚类分析,使用DESeq进行差异基因分析。在差异表达基因检测过程中,将Fold Change≥2且FDR<0.01作为筛选标准。差异倍数(fold change)表示两样品(组)间表达量的比值。错误发现率(false discovery rate,FDR)是通过对差异显著性值(-value)进行校正得到的。采用了公认的Benjamini- Hochberg校正方法对原有假设检验得到的显著性值(-value)进行校正,并最终采用FDR作为差异表达基因筛选的关键指标。
1.5.3 差异表达基因功能注释和富集分析 基因注释的数据库包括:non-redundant(Nr)、nucleotide(Nt)、Swiss-Prot、the Kyoto Encyclopedia of Genes and Genomes(KEGG)、the Cluster of Orthologous Groups(COG)和GO数据库[12]。
1.5.4 基因验证 选取试验中检测到的8个差异表达基因进行qRT-PCR分析,20 d 苗龄对盐敏感及耐盐品种植株进行盐胁迫处理48 h,每个生物学重复挑选3株长势一致植株混样进行地上部分总RNA提取。qRT-PCR采用3次生物学重复,3次技术重复。选取EXCEL 2007软件进行统计分析,采用Student T测验进行两两检测差异性,对测序结果进行验证。总RNA提取如1.2所示,利用试剂盒PrimeScript RT reagent Kit(大连宝生物)合成cDNA。采用primer 3.0软件设计定量引物,产物长度片段在100—300 bp。定量PCR试验采用罗氏定量PCR仪器LightCycler® 480II(Roche),使用SYBR Premix EX Taq试剂(大连宝生物)。
2 结果
2.1 测序数据的质量分析
通过对耐盐材料对照(CK-tolerant)、盐敏感材料对照(CK-sensitive)、耐盐材料盐处理(Salt-tolerant)和盐敏感材料盐处理(Salt-sensitive)4个样本的录数据信息分析(表1)。过滤后4个样品的read平均长度变化幅度为147.41—147.64 bp,基因长度分布规律一致,过滤后总碱基数量258.2亿,占原始总碱基数量96.8%,且在4个样品间的变幅为61.35—64.89亿,较为均匀。另外,4个样品测得数据过滤后GC含量在52.18%—53.28%,CycleQ20值均超过95%,CycleQ30值也在90%左右。4个样品中,过滤后碱基中N的数量平均为4.37。测得的数据准确度较高,数据质量很好,利于后期数据的分析。
2.2 基因表达数量及比例分析
分别统计了耐盐和盐敏感材料在盐胁迫和对照条件下不同值基因的数量以及基因的表达比例(表2)。共获取83 265个基因,采用RPKM方法标准化对测序深度作了归一化,并对基因长度也作了归一化,对不同长度的基因在不同测序深度下得到的基因表达水平进行了估计。其中值大于0.1阈值基因数量达到了89.7%。在单个基因的表达方面,值大于0.1阈值的基因表达比例为92.4%,表达水平较高。另外,4个样品测得基因的长度分布规律一致,基因数目和单个所占比例均符合测序标准,有利于对基因表达进行深度分析。
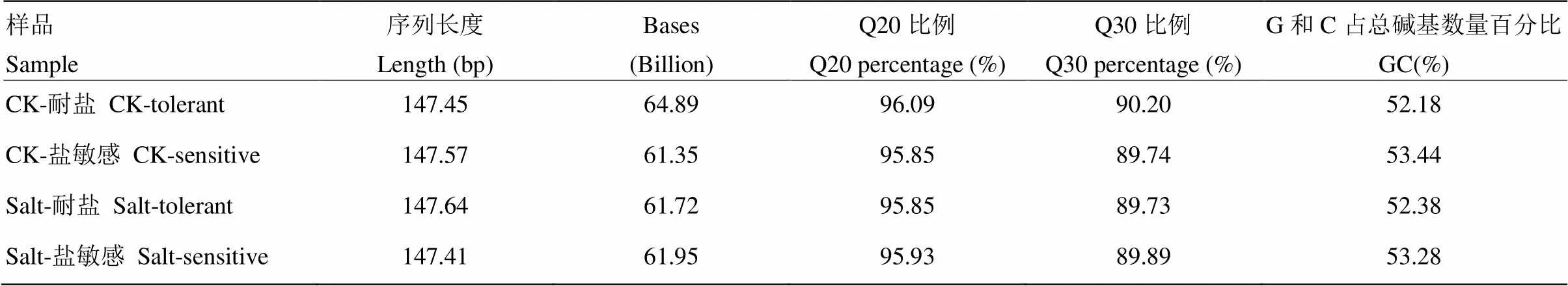
表1 测序数据质量统计
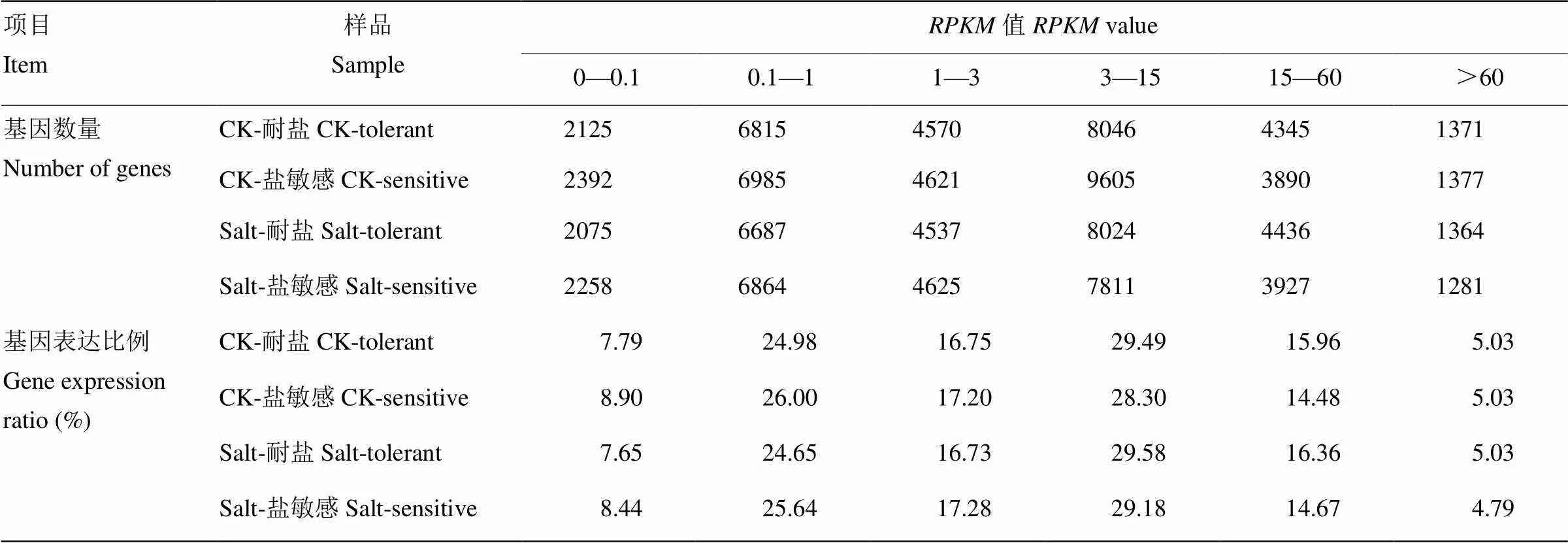
表2 不同表达水平区间的基因数量及比例统计
2.3 差异表达基因的筛选
将参试的4个样品定位到的差异表达基因进行筛选,对DESeq检测的结果按照差异显著性标准(差异基因表达变化2倍以上且FDR<0.01)进行筛选,统计基因显著性差异表达情况(图1)。在4个样品间共检测到1 338个差异表达基因,包括819个上调基因和519个下调基因(表3)。耐盐品种在盐胁迫下共有184个差异表达基因,包括122个上调基因和62 个下调基因;而盐敏感品种盐处理下仅检测到121个差异表达基因,盐胁迫下差异基因数量耐盐品种远多于盐敏感品种。通过维恩图(图2)分析,发现只在耐盐品种中检测到的差异基因为159个,而仅在盐敏感品种中检测到的差异基因68个,说明盐胁迫造成了2个材料的基因表达变化。

表3 盐胁迫下4个高粱样品间的差异表达基因

图1 差异基因火山图

图2 盐处理和对照(CK)下高粱品种差异基因表达维恩图
2.4 耐盐相关差异表达基因的功能分析
盐胁迫条件下,高粱的耐盐性强弱很大程度上取决于耐盐材料的差异基因表达,故以4个样品间共检测到的1 338个差异表达基因的聚类分析结果为基础,对应答盐渍胁迫逆境的耐盐材料特有的159个差异表达基因进行功能注释(图3),通过差异性检验和与高粱苗期耐盐代谢基因功能的综合分析,筛选出4类基因在耐盐应答中起着关键作用,分别为5个Fe离子氧合酶超家族蛋白、4个富含半胱氨酸的类受体激酶(RLK)、3个谷胱甘肽转移酶和3个重金属运输/解毒超家族蛋白。另外,基因功能注释发现钾转运蛋白、过氧化物酶超家族蛋白也发生了相应变化。涉及具体基因信息详见表4。

图3 与盐胁迫相关的高粱差异表达基因注释

表4 高粱耐盐相关基因及功能描述
2.5 盐胁迫相关基因的GO分析
对4个样品的差异表达基因进行GO分析,发现耐盐和盐敏感材料在遭受盐逆境和对照条件下的生物过程、细胞组分和分子功能3个方面存在较大差异(图4)。GO分析结果显示,在15 418个基因中获得4 528个GO注释条目(表5)。在分子功能方面,结合和催化活性基因数量耐盐材料明显高于盐敏感材料。在生物过程方面代谢过程、细胞过程等方面耐盐材料明显高于盐敏感材料。另外,值得注意的是耐盐材料的较盐敏感材料增加了多生物过程和定位这两个参数,这也进一步说明了耐盐材料其耐盐性较强可能与这两个过程相关。

图4 高粱耐盐基因GO分析柱状图
2.6 差异基因KEGG富集及代谢途径分析
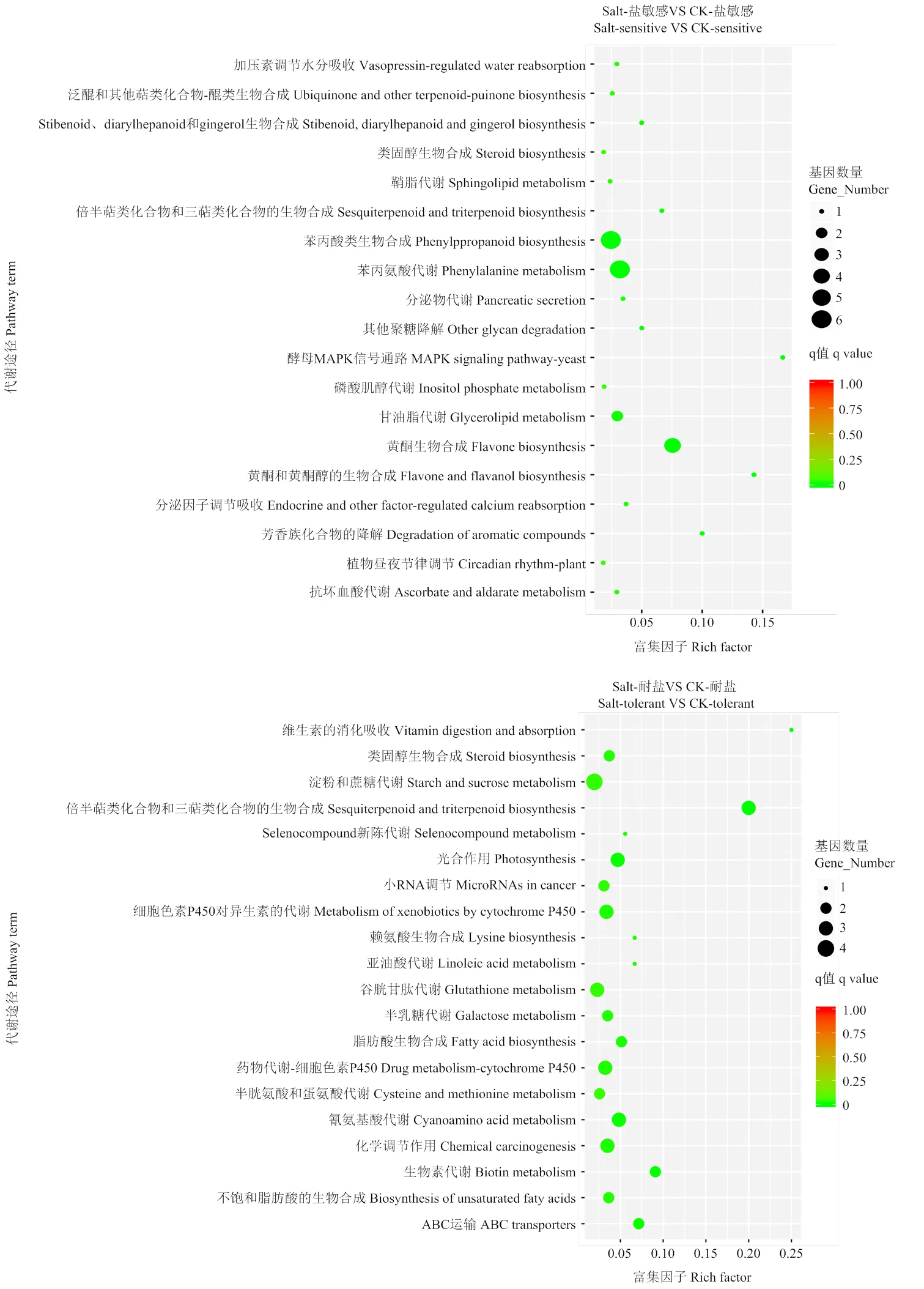
通过差异基因KEGG分析,发现耐盐和盐敏感材料在对照和盐渍胁迫条件下各生理代谢途径差异基因数量差别很大(图5)。值得注意的是,在耐盐材料中,盐胁迫下显著富集了细胞色素P450、谷胱甘肽代谢等有关抗氧化逆境的代谢途径。另外耐盐材料在蔗糖代谢、光合作用、倍半萜类化合物和三萜类化合物的生物合成等16个代谢途径都有显著富集。而盐敏感材料主要集中在一些代谢途径如苯丙烷类生物合成、苯丙氨酸代谢、类黄酮生物合成3个途径中差异基因较多,其他路径基因数量很少。表明盐胁迫下抗氧化代谢途径、光合调控途径和糖代谢途径等可能增加植物抗氧化和调节渗透能力,也可能是耐盐品种具有较强耐盐调节适应的重要原因。
纵坐标为富集的GO term,横坐标为该差异基因个数。不同颜色用来区分生物过程、细胞组分和分子功能
The ordinate is the enriched GO term, and the abscissa is the number of the differential genes. Different colors are used to distinguish biological processes, cellular components, and molecular functions
图5 差异基因KEGG富集散点图
Fig. 5 KEGG enrichment scatter plot of differential gene
2.7 应用qRT-PCR对测序结果的验证
为确认测序结果的准确性,随机选取试验中检测到的8个差异表达基因进行qRT-PCR分析,以盐敏感对材料对照(CK-sensitive)值为1作基准进行比较。通过转录组测序结果和qRT-PCR分析结果比较表明赖氨酸组氨酸转运蛋白(Sobic.007G025900)、而氧化应激蛋白(Sobic.004G279700)、钾转运蛋白(Sobic. 002G220600)、糖转运蛋白(Sobic.002G201900)、液泡铁转运蛋白(Sobic.002G194600)、果胶甲酯酶抑制剂超家族蛋白(Sobic.001G323801)、磷酸盐应答家族蛋白(Sobic.004G229200)、果胶乙酰酯酶家族蛋白(Sobic.003G384700)8个基因的RPKM值与转录组分析数据差异均不显著,且变化趋势基本一致,表明qRT- PCR分析结果与测序结果吻合,转录组测序结果准确。

**,P<0.01;*,P<0.05。因差异均不显著,故图中未作标注
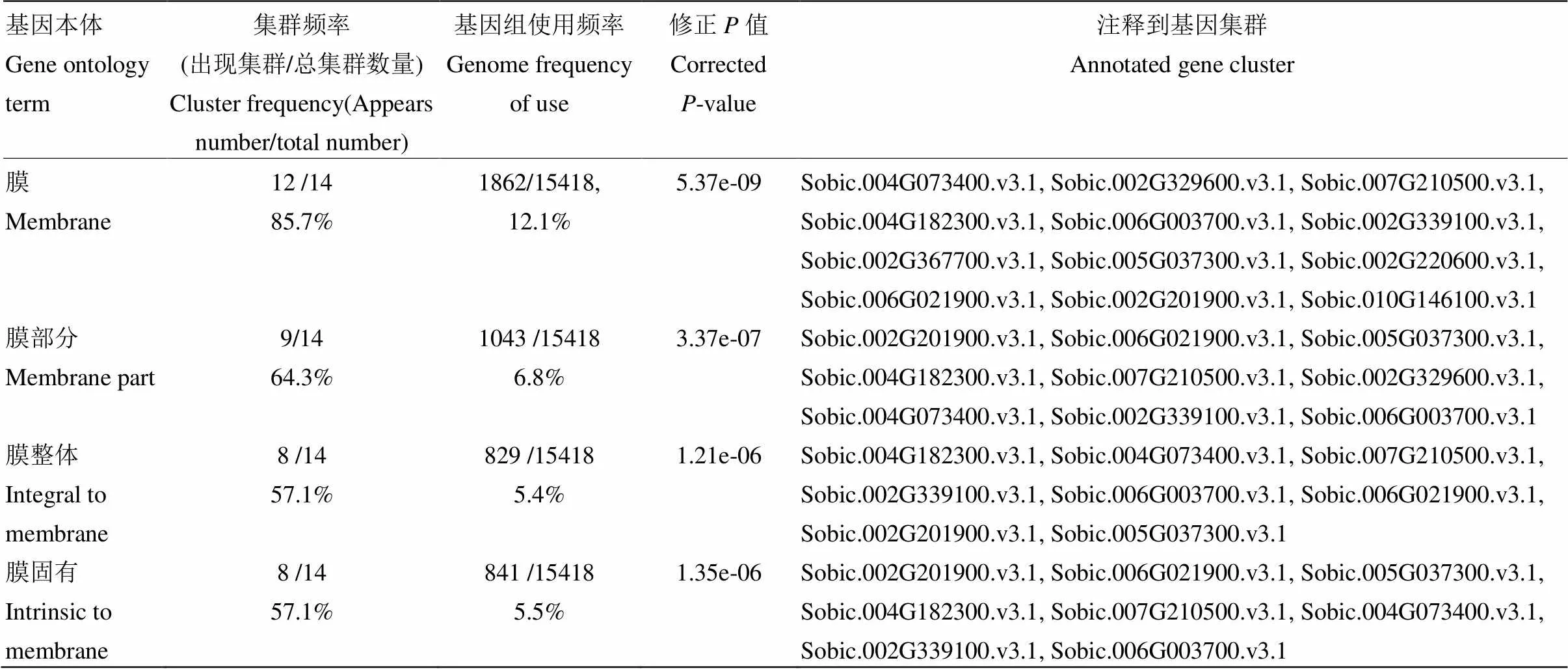
表5 高粱耐盐基因分布及注释到基因
基因集群为值小于0.05注释 Gene clusters havevalues less than 0.05 annotations
3 讨论
3.1 耐盐涉及基因
高粱耐盐是一个极其复杂的过程,耐盐调节涉及许多相关基因的调控。本研究以极耐盐和盐极敏感的2个极端高粱品种为材料,通过转录组测到与耐盐相关的差异基因涉及植物细胞氧化应激、离子转运蛋白运输、渗透调节物质转运等多个逆境生理调节关键环节。本研究与盐逆境下甜瓜[13]、紫花苜蓿[14]、棉花[15]和番茄[16]等的调控基因表达与渗透调节、离子转运途径相关的结果相类似,也与岳小红等[17]对野大麦应答盐渍胁迫研究发现的候选基因的表达模式与脯氨酸等渗透调节物质密切相关的结果相吻合。
3.2 耐盐基因表达
本研究发现在盐应答时5个Fe离子氧合酶超家族蛋白、4个富含半胱氨酸的类受体激酶、3个谷胱甘肽转移酶和3个重金属运输/解毒超家族蛋白相关基因表现出上调和下调,另外,钾转运蛋白、过氧化物酶超家族蛋白也发生了相应变化。分析可能是由于在盐渍逆境下,高粱幼苗代谢受阻,Fe离子氧合酶超家族蛋白变化活跃,盐敏感材料超氧自由基活动加剧,盐逆境下抗氧化系统失调,导致铁离子转运紊乱;富含半胱氨酸的类受体激酶的变化表明在盐胁迫下蛋白质在接受和感知信号中受影响,并影响信号传递。此研究结果与Hernandez等[18]研究并提出的盐渍条件下甘蓝抗氧化系统变化活跃的结论基本一致,同时Xu等[19]对盐渍胁迫下水稻渗透调节系统的研究也得出类似的结果。另外,本研究还发现在遭遇盐渍胁迫时,重金属等可与细胞生物大分子重要成分发生共价结合,对机体造成损害。谷胱甘肽S-转移酶可与其结合后,可防止发生此种共价结合,起到解毒作用。重金属运输/解毒超家族蛋白相关基因的变化也进一步解释了这个论断。综合分析这几类基因主要集中在抗氧化系统、渗透调节和离子化转运生理过程,同时盐渍环境下重金属毒害相关基因变化活跃,有可能是在成盐害的重要原因,但耐盐调控与重金属运输和解毒机制紧密相关方面前人尚未见报道。
3.3 耐盐表达基因功能
盐胁迫下耐盐和盐敏感材料在遭受盐逆境时的生物过程、细胞组分和分子功能3个方面均存在较大差异。GO分析发现生物过程中代谢过程、细胞过程耐盐材料明显高于盐敏感材料,耐盐材料的生理过程中较盐敏感材料增加了多生物过程和定位这两个过程。此类研究在盐逆境下陆地棉[20]、紫花苜蓿[21]、棉花[22]和番茄[23]上也曾对进行过报道,另外Wambua等[24]和Netondo等[25]对盐渍条件下高粱的生理表现分析结果与本研究结论也基本一致。本研究中还发现在分子功能方面,结合和催化活性差异表达基因数量较多,同时多生物过程和定位这两个过程与耐盐调控这关系密切。本研究结果与前人指出的植物分子功能及基因表达是造成耐盐性差异的主要原因的结论相吻合[26-27]。
3.4 耐盐基因代谢途径
盐胁迫下,耐盐和盐敏感高粱代谢通路有所不同。本研究KEGG分析结果显示耐盐和盐敏感材料在对照和盐渍胁迫条件下在苯丙烷类生物合成、苯丙氨酸代谢、类黄酮生物合成3个途径中差异基因较多,可能是造成两个材料盐耐盐性差异的重要原因。目前,苯丙氨酸和类黄酮生物合成途径与耐盐性相关已有研究得出类似结论[17],但关于类黄酮生物合成途径在耐盐调节中尚未见报道,是否是高粱耐盐特有代谢途径还有待于进一步研究。同时,本研究结果与白子彧等[28]研究的非生物胁迫下Na+的运输通道和路径与苯丙氨酸代谢密切相关的结论相吻合,同时,也有报道指出Na+/H+转运蛋白对Na+的运输途径存在影响,且其在不同作物和生育时期存在差异[29-31]。此外,有学者指出Na+运输途径不同将造成光合系统发生变化,影响光合物质生产[13]。
4 结论
高粱耐盐调控过程较为复杂,耐盐调控基因涉及生物过程、细胞组分和分子功能3个方面,其中多生物过程和定位这两个过程对高粱的耐盐调节起着关键作用;重金属运输和解毒相关基因在盐胁迫时变化活跃,可能与耐盐调节相关;在生理代谢途径方面,盐敏感材料苯丙烷类生物合成、苯丙氨酸代谢、类黄酮生物合成3个途径过量表达很可能是造成盐害的重要原因。
[1] 王佳丽, 黄贤金, 钟太洋. 盐碱地可持续利用研究综述. 地理学报, 2011(66): 673-684.
WANG J L, HUANG X J, ZHONG T Y. A review of the sustainable use of saline-alkali soils., 2011(66): 673-684. (in Chinese)
[2] MUNNS R, TESTER M. Mechanisms of salinity tolerance.,2008(59): 651-681.
[3] REDDY P S, REDDY D S, SIVASAKTHI K. Evaluation of sorghum [(L.)] reference genes in various tissues and under abiotic stress conditions for quantitative real-time PCR data normalization., 2016(7): 529-536.
[4] KAFI M, ASADI H, GANJEALI A. Possible utilization of high-salinity waters and application of low amounts of water for production of the halophyteas alternative fodder in saline agroecosystems., 2010(97): 139-147.
[5] LI M, YUYAMA N, LUO L. In silico mapping of 1758 new SSR markers developed from public genomic sequences for sorghum., 2009(24): 41-47.
[6] GUAN Y A, WANG H L, QIN L. QTL mapping of bio-energy related traits in sorghum., 2011(182): 431-440.
[7] LIU J, ZHU J K.A calcium sensor homolog required for plantsalt tolerance., 1998(280): 1943-1945.
[8] GUO Y, HALFTER U, ISHITANI M. Molecular characterization of functional domains in the protein kinase SOS2 that isrequired for plant salt tolerance., 2002(13): 1383-1400.
[9] ROXAS V P, LODHI S A, GARRETT D K. Stress tolerance in transgenic tobacco seedlings that overexpress glutathione S-transferase/glutathione peroxidase,, 2000, 41(11): 1229-1234.
[10] 安静, 张荃. 拟南芥液泡膜Na+/H+逆向转运蛋白的研究进展. 生命科学, 2006, 18(3): 273-278.
AN J, ZHANG Q. Advances in the study of Na+/H+antiporter in the tonoplast of., 2006, 18(3): 273-278. (in Chinese)
[11] ZHANG C Z, YANG H, H. LI H, DAI H. Detection of strawberry RNA and DNA viruses by RT-PCR using total nucleic acid as a template., 2007(155): 431-436.
[12] Romualdi C, Bortoluzzi S, D'Alessi F, Danieli G A. IDEG6: a web tool for detection of differentially expressed genes in multiple tag sampling experiments., 2003(12): 159-162.
[13] 赵丽娜, 张芙蓉, 莫霏, 黄隆堂, 张屹东. 甜瓜盐碱逆境生理响应及相关基因研究进展. 上海农业学报, 2016, 32(6): 176-180.
ZHAO L N, ZHANG F R, MO F, HUANG L T, ZHANG Y D. Research progress on the physiological response and related genes of melon-alkali stress in melon., 2016, 32(6): 176-180. (in Chinese)
[14] 董蔚, 邬培祥, 杨宁, 刘锡江, 宋玉光. 紫花苜蓿盐胁迫响应WRKY转录因子的克隆及表达特征分析. 植物生理学报, 2018, 54(9): 1481-1489.
DONG W, WU P X, YANG N, LIU X J, SONG Y G. Cloning and expression analysis of WRKY transcription factors in response to salt stress of alfalfa., 2018, 54(9): 1481-1489. (in Chinese)
[15] 黄芳, 徐珍珍, 孟珊, 刘静, 汪保华, 沈新莲. 盐胁迫下棉花LTR-反转座子的转录激活及在耐盐相关基因发掘中的应用. 江苏农业学报, 2017, 33(6): 1220-1226.
HUANG F, XU Z Z, MENG S, LIU J, WANG B H, SHEN X L. Transcriptional activation of cotton LTR-reflexion under salt stress and its application in salt tolerance related genes discovery., 2017, 33(6): 1220-1226. (in Chinese)
[16] 张国儒, 庞胜群, 郭晓珊, 单淑玲. 加工番茄耐盐突变体耐盐相关基因的转录组分析. 分子植物育种, 2018, 16(18): 5884-5896.
ZHANG G R, PANG S Q, GUO X S, SHAN S L. Transcriptome analysis of salt-tolerant genes in tomato salt-tolerant mutants., 2018, 16(18): 5884-5896. (in Chinese)
[17] 岳小红, 曹靖, 耿杰, 李瑾, 张宗菊, 张琳捷. 盐分胁迫对啤酒大麦幼苗生长、离子平衡和根际pH变化的影响. 生态学报, 2018, 38(20): 7373-7380.
YUE X H, CAO J , GENG J, LI J, ZHANG Z J, ZHANG L J. Effects of salt stress on growth, ion balance and rhizosphere pH of malting barley seedlings., 2018, 38(20): 7373-7380. (in Chinese)
[18] HERNANDEZ M, FERNANDEZ-GARCIA N, DIAZ-VIVANCOS P. A different role for hydrogen peroxide and the antioxidative system under short and long salt stress inroots., 2010(61): 521-535.
[19] XU D, DUAN X, WANG B. Expression of a late embryogenesis abundant protein gene, HVA1, from barley conferred tolerance to water deficit and salt stress in transgenic rice., 1996, 110(1): 249-257.
[20] 端木慧子, 陶鑫, 王建慧, 韦恒, 李海英, 马春泉. 甜菜M14品系盐胁迫转录组数据库的转录因子分析. 黑龙江大学工程学报, 2017, 8(4): 48-54.
DUANMU H Z, TAO X, WANG J H, WEI H, LI H Y, MA C Q. Transcription factor analysis of salt stress transcriptome database of sugar beet M14 strain., 2017, 8(4): 48-54. (in Chinese)
[21] 张晓钗, 李亮, 何宁芳, 龚雪晴, 主朋月, 王晓阳. 不同盐度胁迫下杜氏盐藻全转录组测序及注释. 微生物学报, 2019, 3(1): 1-20.
ZHANG X C, LI L, HE N F, GONG X Q, ZHU P Y, WANG X Y. Sequencing and annotation of the whole transcriptome ofunder different salinity stresses., 2019, 3(1): 1-20. (in Chinese)
[22] 王春霞, 王全九, 刘建军, 苏李君, 单鱼洋, 庄亮. 灌水矿化度及土壤含盐量对南疆棉花出苗率的影响. 农业工程学报, 2010, 26(9): 28-33. WANG C X WANG Q J, LIU J J, SU L J, SHAN Y Y, ZHUANG L. Effects of irrigation salinity and soil salinity on cotton emergence rate in southern Xinjiang., 2010, 26(9): 28-33. (in Chinese)
[23] 陈冠旭, 秦贵龙, 李恩广, 赵春梅, 乔利仙, 王晶珊, 隋炯明. 花生蛋白磷酸2C家族基因的鉴定和盐胁迫响应分析. 华北农学报, 2018, 33(3): 71-77.
CHEN G X, QIN G L, LI E G, ZHAO C M, QIAO L X, WANG J S, SUI J M. Identification and salinity stress-responsive analysis of PP2C genes in peanut.,2018, 33(3): 71-77. (in Chinese)
[24] WAMBUA J M, MAKOBE M N, NJUE E M. Hydroponic screening of sorghum (L. Moench) cultivars for salinity tolerance., 2017, 12(2):269-277.
[25] NETONDO G W, ONYANGO J C, BECK E. Sorghum and salinity II gas exchange and chlorophyll fluorescence of sorghum under salt stress., 2004, 44: 806-811.
[26] QUINTERO F J, OHTA M, SHI H. Reconstitution in yeast of theSOS signaling pathway for Na+homeostasis., 2002(99): 9061-9066.
[27] 戴漪晨, 黄铎, 王福玲, 林汉明. 组学在大豆耐盐研究中的应用. 土壤与作物, 2015, 4(1): 1-11.
DAI Y C, HUANG D, WANG F L, LIN H M. Application of omics in salt tolerance research of soybean., 2015, 4(1): 1-11. (in Chinese)
[28] 白子彧, 丁博, 李杨, 陈小强, 李迎霞, 杜亚军, 郭雨, 谢晓东. 小麦盐应答基因TaSR1的生物信息学鉴定及表达验证. 麦类作物学报, 2017, 37(3): 307-311.
BAI Z Y, DING B, LI Y, CHEN X Q, LI Y X, DU Y J, GUO Y, XIE X D. Bioinformatics identification and expression verification of wheat salt response gene TaSR1., 2017, 37(3): 307-311. (in Chinese)
[29] BLUMWALD E, POOLE R J. Na+/H+antiport in isolated tonoplast vesicles from storage tissue of., 1985(78): 163-167.
[30] LIANG Y, SUN W, ZHU Y G. Mechanisms of silicon-mediated alleviation of abiotic stresses in higher plants: a review., 2007(147): 422-428.
[31] 彭振, 何守朴, 龚文芳, 潘兆娥, 贾银华, 卢艳丽, 杜雄明. 陆地棉幼苗NaCl胁迫下转录因子的转录组学分析. 作物学报, 2017, 43(3): 354-370.
PENG Z, HE S P, GONG W F, PAN Z E, JIA Y H, LU Y L, DU X M. A Transcriptomic analysis of transcription factors in upland cotton seedlings under NaCl stress., 2017, 43(3): 354-370. (in Chinese)
Comparative transcriptome analysis of different salt tolerance sorghum (L. Moench) under salt stress
ZHANG Fei, WANG YanQiu, ZHU Kai, ZHANG ZhiPeng, ZHU ZhenXing, LU Feng, ZOU JianQiu
(Sorghum Institute, Liaoning Academy of Agricultural Sciences, Shenyang 110161)
Soil salinization is one of the important abiotic stress factors that restricts crop production. Understanding the salt-tolerant mechanism of sorghum may provide a novel avenue to utilize saline soil for sorghum production. The objective of this study was to explore gene regulation mechanisms and metabolic pathways that related to salt tolerance of sorghum by transcriptome sequencing.The salt-tolerant genotype Bayeqi and salt-sensitive genotype PL212 were planted in plastic pots. At five-leaf stage (20 days after sowing), plants were treated with 180 mmol L-1NaCl. Forty-eight hours after treatment, leaves treated by NaCl and unstressed control were sampled and were used for RNA extraction and transcriptome sequencing. Sequencing results were verified by qRT-PCR.Results showed that a total of 1 338 deferentially expressed genes, including 819 up-regulated and 519 down-regulated genes were detected. Cluster analysis revealed that in response to salt stress, five dependent oxygenase superfamily proteins, four cysteine-rich RLKs, three Glutathione S-transferase and three heavy metal transport/detoxification superfamily protein-related genes were significant up–regulated and/or down-regulated, and one K+ion transporter gene was also found to play an important role in salt-tolerance regulation. GO analysis found that 4 528 valid GO annotation entries were obtained from 15 418 genes, and salt-tolerant and salt-sensitive materials showed significant difference in biological processes, cellular components and molecular functions under salt stress treatment. The salt-tolerant materials exhibited obviously higher metabolic processes and cellular processes than salt-sensitive materials. Compared with salt-sensitive materials, multiple biological processes and localization processes were increased in salt-tolerant genotype, which might be the reasons of salt-tolerance. KEGG analysis showed that the salt-tolerant and salt-sensitive materials had more differential gene expression in phenylpropanoid biosynthesis, phenylalanine metabolism and flavonoid biosynthesis under control and salt stress conditions, which may be an important reason for the weak salt tolerance of sensitive materials.The expression of salt-tolerant genes in sorghum is involved in many aspects of biological processes, cellular components and molecular functions. The gene expression in multiple processes and localization processes contributes to the salt tolerance, while excessive gene expression in phenylpropanoid biosynthesis, phenylalanine metabolism, and flavonoid biosynthesis likely contributes to the damage under salt stress.
sorghum; salt tolerance; transcriptome; differential gene expression; physiological regulation
10.3864/j.issn.0578-1752.2019.22.006

2019-06-14;
2019-08-12
国家现代农业产业技术体系(CARS-06-13.5-A11,CARS-06-13.5-A22)、辽宁省自然科学基金(2019-MS-197)
张飞,Tel:024-31029903;E-mail:zhangfei19821121@163.com。通信作者邹剑秋,E-mail:jianqiuzou@126.com。卢峰,E-mail:lufeng740202023@163.com
(责任编辑 李莉)
