医学影像人工智能辅助诊断的样本增广方法
2019-10-31魏小娜李英豪王振宇李皓尊汪红志
魏小娜 李英豪 王振宇 李皓尊 汪红志


摘 要:針对不同领域人工智能(AI)应用研究所面临的采用常规手段获取大量样本时耗时耗力耗财的问题,许多AI研究领域提出了各种各样的样本增广方法。首先,对样本增广的研究背景与意义进行介绍;其次,归纳了几种公知领域(包括自然图像识别、字符识别、语义分析)的样本增广方法,并在此基础上详细论述了医学影像辅助诊断方面的样本获取或增广方法,包括X光片、计算机断层成像(CT)图像、磁共振成像(MRI)图像的样本增广方法;最后,对AI应用领域数据增广方法存在的关键问题进行总结,并对未来的发展趋势进行展望。经归纳总结可知,获取足够数量且具有广泛代表性的训练样本是所有领域AI研发的关键环节。无论是公知领域还是专业领域都进行样本增广,且不同领域甚至同一领域的不同研究方向,其样本获取或增广方法均不相同。此外,样本增广并不是简单地增加样本数量,而是尽可能再现小样本量无法完全覆盖的真实样本存在,进而提高样本多样性,增强AI系统性能。
关键词:人工智能;医学影像;辅助诊断;样本增广
中图分类号:TP391.41
文献标志码:A
Methods of training data augmentation for medical image artificial intelligence aided diagnosis
WEI Xiaona1, LI Yinghao2, WANG Zhenyu1, LI Haozun2, WANG Hongzhi1,2*
1.Shanghai Key Laboratory of Magnetic Resonance (East China Normal University), Shanghai 200062, China;
2.School of Physics and Material Science, East China Normal University, Shanghai 200062, China
Abstract:
For the problem of time, effort and money consuming to obtain a large number of samples by conventional means faced by Artificial Intelligence (AI) application research in different fields, a variety of sample augmentation methods have been proposed in many AI research fields. Firstly, the research background and significance of data augmentation were introduced. Then, the methods of data augmentation in several common fields (including natural image recognition, character recognition and discourse parsing) were summarized, and on this basis, a detailed overview of sample acquisition or augmentation methods in the field of medical image assisted diagnosis was provided, including X-ray, Computed Tomography (CT), Magnetic Resonance Imaging (MRI) images. Finally, the key issues of data augmentation methods in AI application fields were summarized and the future development trends were prospected. It can be concluded that obtaining a sufficient number of broadly representative training samples is the key to the research and development of all AI fields. Both the common fields and the professional fields have conducted sample augmentation, and different fields or even different research directions in the same field have different sample acquisition or augmentation methods. In addition, sample augmentation is not simply to increase the number of samples, but to reproduce the existence of real samples that cannot be completely covered by small sample size as far as possible, so as to improve sample diversity and enhance AI system performance.
Key words:
Artificial Intelligence (AI); medical image; aided diagnosis; sample augmentation
0 引言
人工智能(Artificial Intelligence, AI)作为当今最为热门的话题之一,是研究、开发用于模拟、延伸和扩展人的智能的理论、方法、技术及应用系统的一门新技术科学。AI是计算机科学的一個分支,它企图了解智能的实质,并生产出一种新的能以人类智能相似的方式作出反应的智能机器,该领域的研究包括机器人、语言识别、图像识别、自然语言处理等。
对所有领域AI产品的研发而言,训练样本的获取都是关键问题之一。训练样本好比AI的粮食,没有粮食,再好的AI算法也无法实现其价值[1];AI产品的质量是由其所拥有的训练样本数量决定的[2],训练样本越多,AI就越智能。那么质量达到多少才算足够呢?99.99%的准确率表示万无一失,但如果是自动驾驶,万无一失意味着无人驾驶技术的失败,准确率要训练到至少百万无一失可能才算合格。所以Google的自动驾驶[3]项目每天都有数百辆数据采集车行驶在路上采集数据。每辆车每天采集数据量达24TB,一年可采集一万英里的路况数据。尽管如此,300万英里的实际路况数据,外加10亿英里的模拟软件驾驶数据,才能达到每1000英里的脱离率为0.2(平均而言人类需要每5000英里干预一次行使)。AlphaGo熟读16万局网络棋谱后,战胜了围棋冠军柯洁。AlphaZero[4-5]通过基本围棋规则自弈300万盘,无需搜集网络棋谱,性能超越AlphaGo,本质上是自我进行的样本增广。
目前AI的技术开发有个误区,即认为AI主要做神经网络构建和编程工作。事实上,AI从业者2/3以上的工作量都在获取或标注训练样本。Google等大公司不断向世界分享他们在算法和神经网络结构上的最新成果,但对其训练数据集却很少公开,即使公开也只是其中很少一部分[6]。由此可见,神经网络构建和算法对于AI固然重要,但如何获取更多的训练样本可能更重要。目前AI领域对于算法和网络构建关注极高,而关注训练样本的人却很少。对于公知领域样本的勾画和标注,普通劳务人员即可完成,因而出现了一批专门为AI服务的数据公司,甚至是产业园。对于需要专业知识的样本获取和标注,依靠通用数据公司是无法完成的。AI应用如果按训练样本获取或标注是否需要专业性可分为公知领域和专业领域。按获取样本量的大小可分为大样本和小样本领域。目前较为成熟的AI研究和应用主要集中在公知大样本领域,其原因与样本获取和加工处理标注相对较为容易不无关系。但即使是公知大样本领域(如基于人脸识别的安防、基于语音识别的同译、基于场景识别的自动驾驶等),仍然采用了各种样本增广方法来提高训练准确率。
当前AI在医疗领域的发展可谓是风生水起,而影像已然成为AI在医疗领域落地的主要突破口[7]。鉴于影像种类的多样性和复杂性,收集医疗数据是一个耗时耗力且会耗费大量资金的过程,而且需要研究人员与专业医生合作才能完成[8]。AI影像的一个重要特征是需要大量标准图像和异常标注样本图像的输入[9],输入学习的样本种类越多,AI的适应性和准确性就越高,对具体图像的分析判断能力就越强,所得结果越准确。但在AI医学影像研究领域,样本获取存在着诸多限制[10]:1)医疗数据合法使用的法规政策尚不明确。现有医疗数据的安全规定,过于笼统缺少细则,缺乏针对性和可操作性。2)医院和医生提供医疗数据的动力不足。有些医院和医生对于提供少量标注样本与公司进行合作科研是欢迎的,但对于需要花费大量时间和精力提供大量标注样本进行产品开发存在严重的动力不足现象。3)现有医疗数据标准化没有形成,质量参差不齐。就磁共振成像(Magnetic Resonance Imaging, MRI)图像而言,尽管每家医院都有海量的MRI影像,但这些影像主要是针对某个型号设备的,其种类受制于设备提供的序列和默认序列参数。且不同厂家设备的检查序列也有差异,没有标准化,技师也有序列或图像表现偏好。因此采用某家公司的某种设备产出的MRI样本进行训练,会出现样本种类不足、对其他厂商设备或其他种类MRI图像诊断效果不佳的现象。另外,人体疾病有2000多种,目前AI还只能对单一疾病进行训练,因此总会存在一些疾病种类样本不足的问题。若AI学习的图像样本不足,其适应性和准确性就难以提高,这也是AI+MRI较少出产品的原因之一。
本文首先介绍了几种公知领域(包括自然图像识别、字符识别、语义分析)的样本增广方法;然后介绍了医学影像辅助诊断方面的样本获取或增广方法,包括X光片、X线计算机断层成像(Computed Tomography, CT)图像、MRI图像的样本增广方法;最后对不同领域样本增广方法存在的问题进行了总结,并对未来发展趋势进行了展望。
1 公知领域的样本增广方法
1.1 自然图像识别
目前,图像识别大多是借助计算机技术进行的,图像识别是AI的一个重要领域,而深度学习又是近十年来人工智能领域取得的最重要突破之一[11]。与传统的机器学习方法相比,深度学习极其依赖大规模训练数据,它需要大量数据去理解潜在的数据模式。
深度学习在计算机视觉领域最具影响力的突破发生在2012年, Krizhevsky小组采用深度学习赢得了ImageNet[12-13]图像分类比赛[14]。他们对原始图像采用裁剪、水平翻转及颜色调整的方法来增广训练样本,有效减轻了过拟合现象,使top-5误差率由26.172%降到15.315%,分类准确率超出第二名10%以上。2014年Chatfield等[15]在基于卷积神经网络的图像分类任务中,也使用了水平翻转及裁剪与水平翻转相结合的增广方法;但他们的裁剪操作不同于Krizhevsky小组,他们从整幅图像中进行裁剪然后水平翻转,此操作比从一幅256×256图像中心裁剪所获得的性能更好。受Krizhevsky小组启发,Raitoharju等[16]在AI识别无脊椎动物的训练样本时,采用旋转和镜像的增广方法,该方法类似多角度拍摄样本照片来实现样本增广;虽然训练精确度提升了3%~6%,但增广后的样本量依然很少,可否采用其他方法,如添加噪声[17]、抖动或裁剪某些身体部位(如头部或尾部)等来进行数据增广,仍需继续探究。由此可见,Krizhevsky小组的数据增广方法已然成为了数据增广领域最基本、最普遍的方法,而后的一些方法大多是在此基础上,取长补短,进一步优化改进,当然也出现了一些新的方法。
如文献[18-20]中将自然图像与众多著名艺术品相结合,产生了新风格高质量的新图像,该方法类似于选择照相机不同色调模式采集图像实现数据增广;文献[21]通过随机化原图像颜色、纹理和对比度,同时保留其几何形状和语义内容,将原图像风格进行转移来增加样本量。与传统的旋转、随机裁剪等增广技术相比,该方法可生成更多语义一致且多样化的训练数据。但该方法受传统神经类型风格转移能力的限制,无法实现复杂的变换,如昼夜或季节转换等。
为了解决上述问题,文献[22-23]中使用生成式对抗网络(Generative Adversarial Network, GAN)[24-26],实现了夏日风光的场景与冬季风景的相互转换,呈现了同一景象在不同季节下的情形。其中文献[23]采用的对抗网络,还能实现视觉上相似的两个目标之间的转移,图像风格的转移以及由绘画图像得到照片图的转换。但此处的转移不同于文献[18],这里是学习模仿一整套艺术作品的风格,而非一件选定的艺术作品的风格。该方法在涉及颜色和纹理变换的任务中所得效果较好,但对于需要几何变换的任务却收效甚微,仍需进一步完善。
事实上,在获取自然状态下的样本时,有时样本会被遮挡,为了使模型更好地应对这一影响因素,Zhong等[27]提出了一种简单且实用的无参数数据增强方法——随机擦除,即随机擦除随机选择的图像矩形区域,并以随机值擦除其像素值,进而产生具有不同遮挡程度的图像。该方法使CIFAR10的top-1误差率从3.72%降到了3.08%,CIFAR100的誤差率从18.68%降到了17.65%。随机擦除与随机裁剪、随机水平翻转具有一定的互补性,综合应用这几种方法,可取得更好的模型表现。在将来的研究中,可以考虑将该方法用于目标检索和人脸识别任务。2018年Google科学家Cubuk等[28]提出了一种新的数据增广方法:AutoAugment,这是一种自动化的增广工具,其独特之处在于它一改以往手动设计增强策略,只用强化学习就能从数据本身找出最佳图像增强策略。既可提高训练效果,又可消除研究人员寻找、制作数据集的烦恼。
除以上方法外,迁移学习[29-30]可极大缓解深度学习中数据不足引起的问题。其基本思想[31]是:先从其他数据源训练得到模型,然后利用少量的目标数据对模型进行微调。对于自然图像,可以利用文献[32]中基于网络的深度迁移方法,先对训练好的模型(如VGGNet[33]、ResNet[34]、Inception V3[35]等)进行预训练,然后用目标图像进行微调。文献[36]在基于卷积神经网络对无脊椎动物分类的任务中,就是先用ImageNet图像进行预训练,然后用无脊椎动物的较小数据集进行微调,并获得了很好的分类结果。该方法可大大减少训练样本量,并缩短训练时间。但也存在一些困难,如需要一个相对大规模的预训练数据;如何选择合适的预训练模型;如何判断需要多少额外数据来训练模型,等等。虽然存在许多困难,但随着深度神经网络的发展,迁移学习将被广泛用于解决许多具有挑战性的问题。
1.2 字符识别
字符识别是模式识别的一个重要应用领域,为了解决训练样本匮乏问题,采用数据增广技术生产数据是目前增加样本数量及多样性的有效途径[37]。
对于字符识别,也可采用平移、旋转、尺寸缩放、水平及垂直拉伸变形的方法[38],及Simard等[39]采用的仿射变换(如平移、旋转和倾斜)和弹性变形。事实上,1996年Yaeger等[40]就已提出与Simard等相似的笔画扭曲增广技术,通过倾斜、旋转和缩放等对字符进行微小改变。后来,Bastien等[41]提出了包括局部弹性变形、对比度变化、灰度变换、添加各种噪声及改变字符厚度[42]等19种手写伪样本生成方法,针对NIST-19手写数据集,生成了超过8.19亿的巨大样本,有效解决了训练样本不足的问题。但该方法最大的缺点是,操作较复杂、工作量较大。此外,文献[43]对原有Google字体库中的样本采用字符间距调整、添加下划线和投影畸变的数据生产方法,并使用扩展后的数据集研究自然场景下的文本识别,达到了90.8%的识别率。鉴于上述几何变形方法,文献[44]在研究满文字符识别时,除采用与文献[41]相似的弹性变形、仿射变换、模糊变换及添加噪声等方法外,还采用光照不均、褪色变换、背景融合及形态学处理的方法。这几种方法不仅能增加训练样本量,也能很好反映真实存在的情形,如褪色变换,类似于文档长期放置产生的褪色现象;背景融合,通过为单一字符图像添加不同类型的背景,以此模拟字符实际使用的环境;利用形态学处理中的膨胀和腐蚀操作,则可以再现出不同粗细笔画书写的字符图像。显然,这些增广方法对于其他字符的样本增广也同样适用,同时也说明,样本增广并不是只关注样本数量,所增广的样本还应符合真实存在的情形。
由于手写体汉字存在结构复杂、词汇量大、相互相似度高、不同书写风格差异大等问题,文献[45]利用三角函数构成非线性函数,通过选择合适的变形参数,将给定的手写汉字变形为24种不同的书写风格,并通过实验证明了该方法的有效性。该方法是否适用于其他领域,如形状匹配、目标识别等,值得进一步研究。文献[46]使用余弦函数对汉字图像进行变换,使用不同的余弦函数对原始图像处理,所得汉字的书写风格也就不同。该方法不仅增加了样本数量,一定程度上与提高了样本质量。但该方法的缺陷在于,无法保证变换后的图像都足够好,在使用新生成的样本前,需选出并丢弃不好的样本。后续工作中,需继续探究如何同时提升生成样本的规模和质量。
此外,文献[47]在研究合成样本对训练字符分类器进行数据增广带来的益处时,对比了在数据空间采用数据扭曲[48](对字符图像应用仿射变换和弹性变形创建扭曲数据)和在特征空间合成过采样技术[49-50]两种创造额外训练数据的方法。对于手写体字符的识别,在数据空间采用弹性变形进行数据增广效果较好。对于某些机器学习问题,有时无法确定原始数据样本的转换确实保留了标签信息,此时可在特征空间进行数据增广,但模型性能在训练集与测试集上存在一定的差距,而且通过合成数据对分类器进行扩增训练,得到的性能很可能受实际数据等效量训练的约束。相比而言,GAN所受约束较少,并能生成大量的训练样本。文献[51]提出了一种基于GAN网络的DeLiGAN模型,将该模型在MNIST数据集中进行实验,结果证明该模型能够在数据量有限的条件下,生产一系列多样化的图片。与普通GAN模型[24]相比,该模型具有很好的稳定性,可避免变形伪迹的发生,产生的样本具有更好的多样性。但该模型的建立包含一些简化的假设,限制了模型对复杂分布的估测能力,为提高模型的泛化能力,仍需对所涉及的参数继续优化调整。以上方法可有效增广手写字符的样本量,但对于离线字符[52],由于缺少书写时的动态信息,通常很难生成一组包含足够变化笔迹的字符。为了提高离线字符分类器的识别性能,文献[53]提出了一种基于人工增广实例的支持向量机离线字符识别训练方法,文献[54]结合扭曲模型使用映射函数从现有训练样本中生成大量伪样本。文献[53]的基本思想是:1)对真实字符的每一个笔画作仿射变换;2)对人造字符的每一个笔画作仿射变换,且这些变换都是在主成分分析(Principal Component Analysis, PCA)[55]的基础上合成的。该方法可以生成一组包含足够变化笔迹的人造字符,有效解决收集大量数据时耗时费资的问题。但该方法的数据增广操作要在PCA基础上才能取得很好的效果,若不使用PCA,识别率将会下降,分类时间也将变长,造成此现象的具体原因仍需进一步探讨。
1.3 语义分析
语义分析的目标是通过建立有效的模型和系统,实现在各个语言单位的自动语义分析,从而实现理解整个文本表达的真实语义[56]。显而易见,建立有效的模型是至关重要的,而数据增广是提高模型性能的有效方法,在计算机视觉领域已得到了广泛探讨。诸如翻转、旋转和改变RGB强度等是视觉系统常见的做法[14],除此之外,添加噪声、随机插值一对图像等方法[17]在前面的工作中也提到过。然而,这些方法并不能直接用于语义增广,因为语言中的单词顺序可能会形成严格的句法语法意义,因此,其相应的增广方法也就有所不同。
2016年斯坦福大学计算机科学系Robin和PercyJia等[57]在神经语义分析的研究中,提出了数据重组的增广思想,即从给定的原始训练集中,归纳出一种高精度的同步上下文无关文法,用以捕获语义分析中常见的重要条件独立属性。与经典的数据增广方法(如图像转换和添加噪声等只改变输入不改变输出)不同,该方法在对输入语言进行变换操作的同时也改变输出,使新的输入与新的输出相匹配,进而生成更多的训练样例,有效提升模型精确度。此增广思想是否具有更广泛的适用性值得进一步探讨。同年,Xu等[58]在利用深度递归神经网络对两个实体间关系进行分类的工作中,提出了一种利用句子关系的方向性进行数据增广的技术,该技术可以在不使用外部数据资源的情况下提供额外的数据样本,有效缓解数据稀疏问题并保持较深的网络,提高了模型性能,对分类任务作出了一定贡献。Jiang等[59]也于同年提出了一种为罕见语义关系训练增广数据的方法。其主要思想是:利用Co-training,在每次循環中,用有标注的数据对两个语义分析器进行初步训练,然后用这两个分析器对未标注文档进行分类,并产生对应的语义树,最后经过筛选,把置信度最高的数据加入到最初有标注的数据中,进行继续循环。该方法对罕见语义关系可达到很高的识别性能,但对常见的语义关系则是无效的。因此,对该方法的研究仍需进一步完善,以提高其对常见语义关系的识别性能。
2017年Fadaee等[60]为提高低资源语言对的翻译质量,提出了翻译数据增强方法,即通过改变平行语料库中已有的句子来增广训练数据。他们利用在大量单语数据上训练的语言模型,生成包含罕见单词的新句子对,并在翻译过程中生成更多生词,从而提高翻译质量。该方法对增广低频单词是有效的,但是否适用于一般的翻译任务仍无法确定。与Fadaee等的工作类似,2018年Kobayashi[61]提出了一种被称为contextual augmentation的数据增广方法。该方法通过使用双向语言模型,在给定要扩展的原始单词上下文的情况下,用对某个单词进行预测的单词来替换该单词。与基于同义词的增广方法相比,该方法生成了与原始文本标签兼容的各种单词,并改进了神经分类器,且该方法不受特定任务知识的限制,可用于不同领域的分类任务。同年,哈尔滨工业大学社会计算与信息检索研究中心,提出了面向任务的对话系统中语言理解模块的数据增广问题[62]。他们利用训练数据中与一个语句具有相同语义的其他句子,提出了基于序列到序列生成的数据增广框架,并创新性地将多样性等级结合到话语表示中以使模型产生多样化的语句数据,而且这些多样化的新语句有助于改善语言理解模块;但该方法是否存在一定的限制性,是否具有广泛的适用性,仍需进一步探讨。
以上方法单独作用在各自的研究任务中都取得了很好的效果,若将其与别的方法相结合,是否可以取得更好的效果仍需进一步探究。近年来,GAN[24]引起了大量的研究关注。它产生对抗性例子的能力对数据增广很具吸引力,但由于语义分析具有一定的特殊性,如何将GAN用于语义分析仍是一个有待解决的问题。
以上介绍了公知大样本领域的数据增广方法,可以看出,该领域的样本增广方法相对较为广泛,涉及面也较广。虽然该领域AI的应用和研究相对已经较为成熟,但依然没有一套通用的增广方法,对于不同的任务,需根据实际情况选择合适的方法。因此,在今后的研究中仍需研究者提出更多有创新性、实用性的增广方法。
2 AI+医学影像诊断研究中的样本增广
2.1 AI+X光片病理判定的样本获取方法
为了给研究界提供足够的训练数据,美国国家医学图书馆提供了两组公开的Postero-anterior(PA)胸片数据集[63]:MC(Montgomery County chest X-ray)集和深圳集,以促进计算机辅助诊断肺部疾病的研究。两组数据集中的影像资料分别来自美国马里兰州蒙哥马利县卫生署及中国深圳第三人民医院,这两组数据集都包含有结核表现的正常和异常胸部X光片。并且已有出版物[64-65]将这两组数据集用于结核自动筛选和肺分割。其中在结核自动筛选实验中,这两组数据集的准确率分别达到了曲线下面积87%和90%,虽然检测效能仍低于人类水平,但与放射科医生的表现已相当接近[64]。虽然这两组数据集对于结核自动筛选可取得很好的效果,但对于检测异常胸部X光片,仅依靠这两组数据集是不够的,深度学习领域依然存在数据稀缺,及对标记数据的依赖性。
为了解决这一问题,文献[66]介绍了一个更大的胸部X光片公开数据集Open I[67],它包含来自印第安纳患者护理网络的3955份放射学报告和来自医院图片存档和通信系统的7470份相关的胸部X光片。该数据集免费开放,研究人员可将该数据集用于训练计算机学习如何检测和诊断疾病,辅助医生作出更好的诊断决策。但该数据集所包含的胸透图像报告没有定量的疾病检测结果,若将该数据集用于相关模型的训练,是否会有影响还需进一步探讨。
考虑到Open I数据集存在的问题,Wang等[68]在关于胸部X光片的诊断和病理位置定位一文中,通过自然语言处理方法从医院图像存档和通信系统中,提取报告内容获取标签,构建了一套医院规模的弱监督医学图像数据集ChestX-ray14,该数据集包含112120个单独标注的14种不同胸部疾病的正面胸部X光片。后来,Wang等对这个数据集中的8种疾病图像进行研究,构建了ChestX-ray8数据集。同时,斯坦福大学吴恩达教授团队,使用ChestX-ray14数据集训练Chex Net模型[69]进行肺炎诊断,并用随机水平翻转来增加训练数据量。经充分训练的模型,能通过胸部X光片判断病人是否患有肺炎,且在敏感性和特异性肺炎的检测任务上,其表现能力已超过了专业放射科医师。但该数据集的缺点在于,胸部X线放射学报告可能不被公开分享;数据集中的图片都是正面胸片,而背部扫描的胸片有时对诊断来说也是至关重要的;此外,该数据集中的标签不是由放射科医生直接提供,而是由放射科医生的文本报告自动生成的,因此难免会出现一些错误的标签。
除此之外,吴恩达教授团队在检测异常肌肉骨骼时,开源了MURA数据集[70],并用该数据集训练卷积神经网络,用以寻找并定位X光片的异常部位。MURA是目前最大的X光片数据集之一,它包含源自14982项病例的40895张肌肉骨骼X光片。基于该数据集,该团队开发了一个有效预测异常肌肉骨骼的模型,流程如图1所示。
经充分训练后,将模型的表现能力与专业放射科医生进行对比。结果发现,该模型在诊断手指和手腕X光片异常情况时,其表现比放射科医生好,但对其他部位(如肩膀、肱部、肘部、前臂、手掌)的诊断则比放射科医生差。值得注意的是,MURA数据集中的四万张图像来自近15000篇论文,其中9067篇为正常上肢肌肉骨骼X光片的研究,5915篇是异常研究。即该团队不是直接从医院获取数据,而是从公开渠道获取样本。该方法的最大优点是所受限制较少,不足之处是需要搜集和阅读大量的资料,且获得的样本质量参差不齐。表1列出了一些公开可用的医疗放射图像数据集。
综上可知,在深度学习领域,X光片样本的获取主要是从一些公开的医疗图像数据集中得到,然后结合一些简单的变换操作来增加训练样本量。在今后的研究中,仍需研究者开源出更大的数据集,以满足AI对于临床疾病的相关研究。
2.2 AI+CT图像样本增广方法
由于CT图像属于单参数成像,主要反映组织密度差异,所有图像均属于一类图像,即标准图像只有一种,因此,对于CT图像可以采用经典的数据增广方法[14]。文献[71]公开了一种基于3D全连接卷积神经网络的CT图像肺结节检测系统,该项发明在构建训练集时,对区域训练集的图像以标签的方式分为负样本(无结节)和正样本(有结节),同时对有限数量的正样本采用平移、旋转、缩放、镜像等几何变换作数据多样性增广,类似于医生通过不同的视角、不同的上下文去分析结节区域。采用该发明技术方案可实现结节自动检测,无需任何人工干预,并能有效提高结节检测的召回率,大幅降低假阳性病灶,获得肺结节病灶区域的像素级定位、定量、定性结果。这项发明对于临床CT图像肺结节的检测具有极大的帮助,但文中所用的数据增广方法只有一些基本的几何变换,仅使用这些简单的变换方法,还不足以提升模型的泛化能力。
为解决上述问题,上海交通大学人工智能实验室[72]利用深度学习搭建的肺结节自动定位筛查系统,能有效检测CT影像中包含的微小结节、磨玻璃等各类结节,并降低假阳性误诊的发生。他们在数据处理上,除了对图像采用旋转、平移等几何变换外,还利用GAN[24]对数据预处理,从随机噪声中产生新的结节正样本,学习生成新形态的结节样本,深度增广数据多样性,有效提升了模型泛化能力,使模型更好地处理不同形态的结节特征,达到很好的检测效果。此为该团队在数据处理方面与文献[71]的最大区别之处,同时也是其成功之处所在。
此外,Frid-Adar等[73]利用GAN自动生成合成医学图像,并用于肝脏病变的分类任务。图2[73]显示了三种肿瘤病变图像,(a)真实病变图像,(b)合成病变图像,其中顶行为囊肿病变图像,中间行为转移肿瘤病变图像,底行为血管瘤病变图像。
具体训练过程为:1)先用传统增广方法(如平移、旋转、翻转和缩放)创建更大的数据集,然后将其用于训练GAN;2)用GAN生成合成图像作为数据增广的额外资源;3)将传统增广方法得到的图像与GAN生成的合成图像相结合,用于训练病灶分类器。
图3[73]显示了肝脏病变分类精确度随训练集的变化情况。由图可知,使用合成图像增加训练集,比仅用传统方法增加训练集在性能上提高了7%,这也进一步证明了用GAN生成合成图像来增加训练集的有效性和可行性。
GAN最初是由Goodfellow等[24]提出的一種生成式模型,首次将其应用于合成医学图像生成领域的是Nie等[74],为了从给定MRI图像中生成更真实的CT图像,他们使用了GAN进行训练。GAN可以生成视觉上真实的图像,从图2可以看
出合成的病变图像与真实病变图像在视觉上没有很大差别,且该方法对于数据的增广可以达到很好的效果。
2.3 MRI图像的样本增广方法
MRI图像的特点决定了其具有不同于其他医学图像的特殊性,MRI图像的多样性,决定了AI+医学影像在核磁影像方面的结合较为罕见。相对而言,研究MRI图像样本增广方法的人较少,但依然有研究者提出了一些可行的数据增广方法,如文献[75]在利用卷积神经网络对阿尔兹海默症进行识别的研究中,采用了Krizhevsky小组[14]所用的图像变换的增广方法。其具体操作是:在预处理后的一个MRI图像中选择多个中心点提取多幅2.5D图像,这样一张图就可以增益为多幅图片,然后使用卷积神经网络对增益后的图像进行训练和识别,进而共同判决该MRI的分类。该方法不仅能从MRI中提取足够的信息,并能有效抑制卷积神经网络的过拟合问题。但操作过程中多个中心点的位置选择是一个难点,而且该方法是否适用于其他病症MRI图像的数据增广依然未知。此外,Thyreau等[76]在对海马体进行分割时,开发了一种数据增强系统,该系统通过改变输入影像的几何形状、边界对比度和一般强度来增加训练样本量,生成每个图像和相应目标的多种变化。他们使用这种数据增强方案,能够从一个样本图生成4个合成样本图,有效增加训练样本量。但数据增广过程中对目标掩码图只进行了几何变换操作,且该系统是作用于高精度输入图像上的,对于低精度输入图像是否可以取得同样的效果仍需进一步探索。
近年来,在医学图像分割中U-net网络备受关注,为了提高模型的准确度和精度,Dong等[77]在利用该网络进行脑肿瘤检测和分割时,考虑到肿瘤没有明确的形状,简单的变换方法,如翻转、旋转、平移和缩放,仅能改变图像的位移场,并不能创建形状不同的训练样本,而剪切操作虽然可以在水平方向上轻微扭曲肿瘤的整体形状,但仍不足以获取足够的可变训练数据。因此他们采用了与Ronneberger等[78]分割细胞时使用的相似的数据增广方法——弹性变形[17],来生成更多任意形状且与实际情形相吻合的训练数据,有效提高了网络的性能,并取得了很好的分割效果。值得注意的是,他们用于训练模型的数据来自公开数据集(BRATS 2015[79]),将文中的增广方法用于具有各向异性分辨率的临床数据,是未来的研究方向之一。
鉴于医学影像数据集的不平衡性,文献[80]利用GAN来合成异常MRI图像的方法进行数据增广。其基本思想是:利用两个公开的MRI数据集(ADNI和BRATS),训练生成GAN网络,进而合成具有脑肿瘤的异常MRI图像。他们用合成的图像作为数据增广的一种形式,演示了其对改进肿瘤分割性能的有效性,并证明了当对合成数据和真实受试者数据进行训练时,可以获得类似的肿瘤分割效果。该方法为深度学习在医学影像领域面临的两个最大挑战(病理学发现的发生率较低及共享患者数据受到限制),提供了一个潜在的解决方案。
以上方法虽然能有效解决样本数量问题,但在解决样本多样性方面,所取得的效果并不是很理想。文献[10]介绍了一种MRI图像样本的自动增广和批量标注软件平台(DMRIAtlas),既可以解决样本数量问题,也能增加样本多样性,其工作原理是:通过定量MRI成像技术获取正常志愿者和少量阳性病例重点病灶区的物理信息,然后利用虚拟MRI成像技术对正常或病灶区信息进行虚拟数据采集和成像,基于不同的成像序列和参数,输出不同种类、不同表现的大量MRI图像。借助该软件生成的图像可以是不同分辨率、不同信噪比、不同权重、不同b值的MRI图像,可极大增广训练样本的种类和数量。
图4为使用该软件对正常脑部组织同一层面采用不同序列经虚拟扫描得到的T1WI、T2WI、T1-FLAIR、T2-FLAIR和STIR的图像[10]。
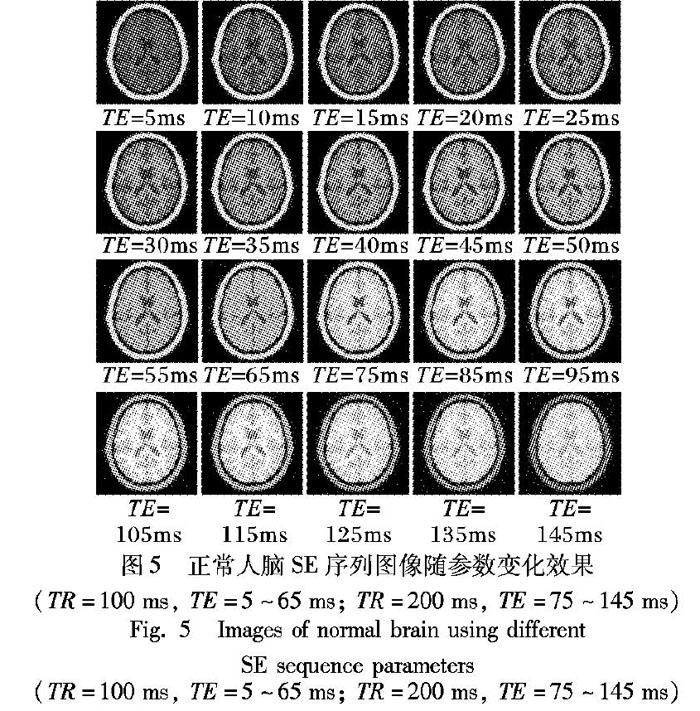
为了弥补设备差异或技师水平差异等带来的实际图像差异,对同种序列选取不同的序列参数,可以得到对比度和信噪比逐渐变化的图像效果。图5为正常人脑SE序列图像随参数变化(TR=100ms和200ms, TE=5~145ms)的效果图[10]。
显而易见,通过类似上述的简单操作,即可得到庞大的样本数据。在医学影像诊断这一专业小样本领域,该软件作为一种通用型MRI图像样本增广工具,只需采集一定数量的某种疾病不同程度阳性病例的物理信息数据,然后借助专业影像医生的勾画标注,或通过一些简单的操作(如调节不同的参数、选取不同的断面等),即可得到大量的样本,极大降低了成本,解决了样本数量与种类不足的问题,具有很好的应用前景。
上述内容总结梳理了医学影像中X光片、CT图像和MRI图像的样本获取或增广方法。当然,AI在医学影像诊断中的应用并非只针对这三类图像,其他医学图像,如医学光学图像,同样也涉及到AI的应用,并且已有学者提出了AI应用于该类图像时相应的数据增广方法。比如,Vasconcelos等[81]在皮肤病变分析黑色素瘤的检测任务中,采用了几何变换增广、颜色变换增广以及基于专家知识的数据增广方法,进行人工创建样本;Ciresan等[82]在2012年的乳腺癌组织影像有丝分裂检测挑战赛中,应用了任意旋转和镜像方法来创建额外的训练实例;等等。然而,仅从本文所述医学图像的数据增广方法中就可看出,不同医学图像的样本获取或增广方法是各不
相同的。由于医学影像本身的复杂性,目前的方法多是针对某一具体任务而言的。因此,对于不同图像、不同研究方向,需根据具体知识采用针对性的增广方法。
3 结语
本文对AI在三类公知大样本领域及专业小样本领域(医学影像识别和辅助诊断)的样本增广方法进行了全面分析。通过分析总结可知,绝大多数AI应用领域都要进行样本增广,且不同领域甚至同一领域的不同研究对象,其样本获取或增广方法也是截然不同的。
此外,样本增广不是为了简单地增加样本数量,而是尽可能再现小样本量无法完全覆盖的真实样本存在,进而提高样本多样性,增强AI系统性能,因此,需根据具体领域知识采用针对性的增广方法,使增广的数据尽可能呈现出真实情况下所出现的情形,不能一味为了增广数据而增广,必须从实际出发,与实际情形相吻合。
目前AI领域对于算法和网络构建的提升及改进关注度特别高,相对而言,关注训练样本的人却很少。实际上,充足的训练数据对AI研发起着至关重要的作用,运用合适的方法进行样本增广就起到了举足轻重的作用。但由于人们对样本增广的关注度还不够高,现在仍处于发展阶段,依然有一些问题值得进一步深入研究:
1)对于公知大样本领域,虽然该领域获取样本的方法相对较多、较为成熟,但对于不同的任务,依然没有一套统一的增广方法。对于不同的任务,需根据具体情况选取合适的增广方法,不可盲目增广,要从实际出发,使增广的数据尽可能再现出真实样本的存在。在未来的研究中,是否可以探索出一些有效且通用的样本增广方法或开发出一种通用的样本增广工具值得进一步探究。
2)对于医疗影像领域而言,由于數据规模比较小,获取样本的途径也较少且存在各种困难,因此,对样本增广方法多样性和有效性的研究,将成为该领域研究的热点,同时也是急需进一步完善的难点所在;在今后的研究中,需倡导研究者开发出更多针对某一类医学图像通用的样本增广工具(诸如DMRIAtlas软件)。此外,医学领域往往不仅仅依靠图像来诊断,结合临床信息、检验报告等非图像数据的多模态学习也是值得关注的方向。
3)现有数据增广方法中,同时适用于公知大样本领域和医疗影像领域的方法少之又少。对于医疗影像这一小样本领域,由于其有效的数据增广技术相对较少、较不成熟,因此,可对公知大样本领域中较为成熟的增广方法进一步探究,以验证其能否用于某些医学图像的增广。
尽管对样本增广方法的研究还存在许多问题,但它对AI产品的研发产生的影响不容小觑。更加多样、更有效且适用范围更广的增广方法,能够带来更多有效的样本,并能对AI系统的性能起到很大的提升作用。尤其对于医疗影像这一小样本领域,如何利用有效的增广技术获取足够丰富且高质量的影像数据,对提升诊断准确度起到了至关重要的作用。总之,对样本增广方法的研究是一个值得进一步探索的领域,在未来的研究中一定会更加成熟。
参考文献
[1]HACKER NOON. Big challenge in deep learning: training data. Artificial intelligence for real problems: deep systems.ai.[EB/OL].[2019-05-04]. https://www.jianshu.com/p/2a3388d8c9c3
HACKER NOON. Big challenge in deep learning [EB/OL]. [2018-11-04]. https://hackernoon.com/%EF%B8%8F-big-challenge-in-deep-learning-training-data-31a88b97b282.
[2]ELIZEBETH G. AI firms lure academics [J]. Nature, 2016, 532(4): 422-423.
[3]ALEXIS C. MADRIGAL. Inside Waymos secret world for training self-driving cars [EB/OL]. [2019-01-04]. The Atlantic Daily. AUG 23,2017. https://www.yahoo.com/news/inside-waymo-apos-secret-world-152456397.html.
[4]SILVER D, SCHRITTWIESER J, SIMONYAN K, et al. Mastering the game of Go without human knowledge [J]. Nature, 2017, 550(7676): 354-359.
[5]SILVER D, HUBERT T, SCHRITTWIESER J, et al. A general reinforcement learning algorithm that masters chess, shogi and Go through self-play [J]. Science, 2018, 362(6419): 1140-1144.
[6]TING D S W, LIU Y, BURLINA P, et al. AI for medical imaging goes deep [J]. Nature Medicine, 2018,24(5): 539-540.
[7]李纲,徐鼎梁.AI+医疗:如何做好一只被风吹上天的猪[EB/OL].[2019-05-04] 财经,2018(3)(LI G, XU D L.AI+ medicine: how to make a pig that is blown to the sky [EB/OL].[2019-05-04] finance and economics,2018(3))www.sohu.com/a/222070262_487521
李綱,徐鼎梁.AI+医疗:如何做好一只被风吹上天的猪[EB/OL]. [2019-01-04]. www.sohu.com/a/222070262_487521.(LI G, XU D L. AI+ medicine: how to be a pig that is blown to the sky [EB/OL]. [2019-01-04]. www.sohu.com/a/222070262_487521.)
[8]GREENSPAN H, van GINNEKEN B, SUMMERS R M. Guest editorial deep learning in medical imaging: overview and future promise of an exciting new technique [J]. IEEE Transactions on Medical Imaging, 2016, 35(5): 1153-1159.
[9]MAXMEN A. AI researchers embrace bitcoin technology to share medical data [J]. Nature, 2018,555(7696): 293-294.
[10]汪红志,赵地,杨丽琴,等.基于AI+MRI的影像诊断的样本增广与批量标注方法[J].波谱学杂志,2018,35(4):447-456.(WANG H Z, ZHAO D, YANG L Q, et al. An approach for training data enrichment and batch labeling in AI+MRI aided diagnosis [J]. Chinese Journal of Magnetic Resonance, 2018, 35(4): 447-456.)
[11]王晓刚.深度学习在图像识别中的研究进展与展望[J].中国计算机学会通讯,2015,10(WANG X G,Research progress and prospect of deep learning in image recognition[J],Communication of the Chinese computer Federation Oct,2015)
王晓刚.深度学习在图像识别中的研究进展与展望 [EB/OL]. [2019-01-10]. http://www.360doc.com/content/15/0604/11/20625606_475573792.shtml.(WANG X G. Research progress and prospect of deep learning in image recognition [EB/OL]. [2019-01-10]. http://www.360doc.com/content/15/0604/11/20625606_475573792.shtml.)
[12]DENG J, DONG W, SOCHER R, et al. ImageNet: a large-scale hierarchical image database [C]// Proceedings of the 2009 Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2009: 248-255.
[13]RUSSAKOVSKY O, DENG J, SU H, et al. ImageNet largescale visual recognition challenge [J]. International Journal of Computer Vision, 2015, 115(3): 211-252.
[14]KRIZHEVSKY A, SUTSKEVER I, HINTON G E. ImageNet classification with deep convolutional neural networks [C]// Proceedings of the 2012 International Conference on Neural Information Processing Systems. North Miami Beach, FL, USA: Curran Associates, 2012: 1097-1105.
[15]CHATFIELD K, SIMONYAN K, VEDALDI A, et al. Return of the devil in the details: delving deep into convolutional nets [J]. Computer Science, 2014:1-12
CHATFIELD K, SIMONYAN K, VEDALDI A, et al. Return of the devil in the details: delving deep into convolutional nets [EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1405.3531.pdf.
[16]RAITOHARJU J, RIABCHENKO E, MEISSNER K, et al. Data enrichment in fine-grained classification of aquatic macroinvertebrates [C]// Proceedings of the ICPR 2nd Workshop on Computer Vision for Analysis of Underwater Imagery. Washington, DC: IEEE Computer Society, 2016: 43-48.
[17]ZHANG H Y, CISSSE M, DAUPHIN Y N, et al. Mixup: beyond empirical risk minimization [EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1710.09412.pdf.
[18]GATYS L A, ECKER A S, BETHGE M. Image style transfer using convolutional neural networks [C]// Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2016: 2414-2423.
[19]JOHNSON J, ALAHI A, LI F. Perceptual losses for real-time style transfer and super-resolution [C]// Proceedings of the 2016 European Conference on Computer Vision, LNCS 9906. Berlin: Springer, 2016: 694-711.
[20]GATYS L A, ECKER A S, BETHGE M. A neural algorithm of artistic style [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1508.06576.pdf.
[21]JACKSON P T, TAPOUR-ABARGHOUEI A, BONNER S, et al. Style augmentation: data augmentation via style randomization [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1809.05375v1.pdf.
[22]RAJ B. Data augmentation: how to use deep learning when you have limited data [EB/OL]. [2019-01-04]. https://www.kdnuggets.com/2018/05/data-augmentation-deep-learning-limited-data.html.
[23]ZHU J, PARK T, ISOLA P, et al. Unpaired image-to-image translation using cycle-consistent adversarial networks [C]// Proceedings of the 2017 IEEE International Conference on Computer Vision. Washington, DC: IEEE Computer Society, 2017: 2242-2251.
[24]GOODFELLOW I J, POUGET-ABADIE J, MIRZA M, et al. Generative adversarial nets [C]// Proceedings of the 27th International Conference on Neural Information Processing Systems. Cambridge, MA: MIT Press, 2014: 2672-2680.
[25]林懿倫,戴星原,李力,等.人工智能研究的新前线:生成式对抗网络[J].自动化学报,2018,44(5):775-792.(LIN Y L, DAI X Y, LI L, et al. The new frontier of AI research: generative adversarial networks [J]. Acta Automatica Sinica, 2018, 44(5): 775-792.)
[26]ANTONIOU A, STORKEY A, EDWARDS H. Data augmentation generative adversarial networks [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1711.04340.pdf.
[27]ZHONG Z, ZHENG L, KANG G, et al. Random erasing data augmentation [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1708.04896.pdf.
[28]CUBUK E D, ZOPH B, MANE D, et al. AutoAugment: learning augmentation policies from data [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1805.09501.pdf.
[29]Sebastian Ruder. Transfer learning—machine learnings next frontier [EB/OL]. [2019-01-04]. http://ruder.io/transfer-learning/.
[30]PAN S J, YANG Q. A survey on transfer learning [J]. IEEE Transaction on Knowledge and Data Engineering, 2010, 22(10): 1345-1359.
[31]YOSINSKI J, CLUNE J, BENGIO Y, et al. How transferable are features in deep neural networks? [J] In Advances in Neural Information Processing Systems 27 (NIPS 14),NIPS Foundation,2014.
YOSINSKI J, CLUNE J, BENGIO Y, et al. How transferable are features in deep neural networks? [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1411.1792.pdf.
[32]TAN C, SUN F, KONG T, et al. A survey on deep transfer learning [C]// Proceedings of the 2018 International Conference on Artificial Neural Networks, LNCS 11141. Berlin: Springer: 270-279.
[33]SIMONYAN K, ZISSERMAN A. Very deep convolutional networks for large-scale image recognition [EB/OL].[2019-01-04]. https://arxiv.org/pdf/1409.1556.pdf.
[34]HE K M, ZHANG X Y, REN S Q, et al. Deep residual learning for image recognition [C]// Proceedings of the 2016 IEEE Conference on Computer Vision and Pattern Recognition. Washington, DC: IEEE Computer Society, 2016:770-778.
[35]SZEGEDY C, VANHOUCKE V, IOFFE S, et al. Rethinking the inception architecture for computer vision [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1512.00567.pdf.
[36]RIABCHENKO E, MEISSNER K, AHMAD I, et al. Learned vs. engineered features for fine-grained classification of aquatic macroinvertebrates [C]// Proceedings of the 23rd International Conference on Pattern Recognition. Piscataway, NJ: IEEE, 2016: 2276-2281.
[37]金連文,钟卓耀,杨钊,等.深度学习在手写汉字识别中的应用综述[J].自动化学报,2016,42(8):1125-1141.(JIN L W, ZHONG Z Y, YANG Z, et al. Applications of deep learning for handwritten Chinese character recognition: a review [J]. Acta Automatica Sinica, 2016, 42(8): 1125-1141.)
[38]LeCUN Y, BOTTOU L, BENGIO Y, et al. Gradient-based learning applied to document recognition [J]. Proceedings of the IEEE, 1998, 86(11): 2278-2324.
[39]SIMARD P Y, STEINKRAUS D, PLATT J C. Best practices for convolutional neural networks applied to visual document analysis [C]// Proceedings of the 7th International Conference on Document Analysis and Recognition. Washington, DC: IEEE Computer Society, 2003: 958-962.
[40]YAEGER L, LYON R, WEBB B. Effective training of a neural network character classifier for word recognition [C]// Advances in Neural Information Processing Systems, 1997: 807-816.
YAEGER L, LYON R, WEBB B. Effective training of a neural network character classifier for word recognition [C]// Proceedings of the 9th International Conference on Neural Information Processing Systems. Denver, Colorado: [s.n.], 1997:807-816.
[41]BASTIEN F, BENGIO Y, BERGERON A, et al. Deep self-taught learning for handwritten character recognition [EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1009.3589v1.pdf.
[42]VARGA T, BUNKE H. Generation of synthetic training data for an HMM-based handwriting recognition system [C]// Proceedings of the 7th International Conference on Document Analysis and Recognition. Piscataway, NJ: IEEE, 2003: 618-622.
[43]JADERBERG M, SIMONYAN K, VEDALDI A, et al. Synthetic data and artificial neural networks for natural scene text recognition [EB/OL]. [2019-01-10]. https://arxiv.org/pdf/1406.2227.pdf.
[44]毕佳晶,李敏,郑蕊蕊,等. 面向满文字符识别的训练数据增广方法研究[J].大连民族大学学报,2018,20(1):73-78.(BI J J,LI M, ZHENG R R, et al. Research on training data augmentation methods for Manchu character recognition [J] . Journal of Dalian Minzu University, 2018, 20(1): 73-78.)
[45]JIN L, HUANG J, YIN J, et al. Deformation transformation for handwritten Chinese character shape correction[C]// Proceedings of the 3rd International Conference on Multimodal Interfaces, LNCS 1948. Berlin: Springer, 2000: 450-457.
[46]CHEN G, ZHANG H, GUO J. Learning pattern generation for handwritten Chinese character using pattern transform method with cosine function [C]// Proceedings of the 2006 International Conference on Machine Learning and Cybernetics. Piscataway, NJ: IEEE, 2006: 3329-3333.
[47]WONG S C, GATT A, STAMATESCU V, et al. Understanding data augmentation for classification: when to warp? [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1609.08764.pdf.
[48]BAIRD H S. Document image defect models [M]// BAIRD H S, BUNKE H, YAMAMOTO K. Structured Document Image Analysis. Berlin: Springer, 1992: 546-556.
[49]CHAWLA N V, BOWYER K W, HALL L O, et al. SMOTE: synthetic minority over-sampling technique [J]. Journal of Artificial Intelligence Research, 2002, 16(1): 321-356.
[50]DEVRIES T, TAYLOR G W. Dateset augmentation in feature space [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1702.05538.pdf.
[51]GURUMURTHY S, SARVADEVABHATLA R K, BABU R V. DeLiGAN: generative adversarial networks for diverse and limited data [C]// Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Piscataway, NJ: IEEE, 2017: 4941-4949.
[52]楊明,刘强,尹忠科,等.基于轮廓追踪的字符识别特征提取[J].计算机工程与应用 2007,43(20):207-209.(YANG M, LIU Q, YIN Z K, et al. Feature extraction in character recognition based on contour pursuit [J]. Computer Engineering and Applications, 2007, 43(20): 207-209.)
[53]MIYAO H, MARUYAMA M. Virtual example synthesis based on PCA for off-line handwritten character recognition [C]// Proceedings of the 7th International Workshop on Document Analysis Systems, LNCS 3872. Berlin: Springer, 2006: 96-105.
[54]LEUNG K C, LEUNG C H. Recognition of handwritten Chinese characters by combining regularization, Fishers discriminant and distorted sample generation [C]// Proceedings of the 10th International Conference on Document Analysis and Recognition. Piscataway, NJ: IEEE, 2009: 1026-1030.
[55]赵元庆,吴华.多尺度特征和神经网络相融合的手写体数字识别[J].计算机科学,2013,40(8):316-318.(ZHAO Y Q, WU H. Hand written numeral recognition based on multi-scale features and neural network [J]. Computer Science, 2013, 40(8): 316-318.)
[56]张敏,韩先培,张家俊,等.中文信息处理发展报告(2016)[R].北京:中国中文信息学会,2016.(ZHANG M, HAN X P, ZHANG J J, et al. Chinese information processing development report (2016)[R]. Beijing: Chinese Information Society, 2016.)
[57]JIA R, LIANG P. Data recombination for neural semantic parsing [C]// Proceedings of the 54th Annual Meeting of the Association for Computational Linguistics. Stroudsburg, PA: Association for Computational Linguistics, 2016: 12-22.
[58]XU Y, JIA R, MOU L, et al. Improved relation classification by deep recurrent neural networks with data augmentation [EB/OL].[2019-01-04]. https:arxiv.org/pdf/1601.03651.pdf.
[59]JIANG K, CARENINI G, NG R T. Training data enrichment for infrequent discourse relations [EB/OL]. [2019-01-04]. https://www.aclweb.org/anthology/C16-1245.
[60]FADAEE M, BISAZZA A, MONZ C. Data augmentation for low-resource neural machine translation [C]// Proceedings of the 55th Annual Meeting of the Association for Computational Linguistics. Stroudsburg, PA: Association for Computational Linguistics, 2017: 567-573.
[61]KOBAYASHI S. Contextual augmentation: data augmentation by words with paradigmatic relations [C]. //Proceedings of the North American Chapter of the Association for Computational Linguistics: Human Language Technologies (NAACL-HLT 2018), New Orleans, Louisiana, June 1-6, 2018:452-457.
KOBAYASHI S. Contextual augmentation: data augmentation by words with paradigmatic relations [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1805.06201.pdf.
[62]HOU Y, LIU Y, CHE W, et al. Sequence-to-sequence data augmentation for dialogue language understanding [C] // The 27th International Conference on Computational Linguistics. Santa Fe, New Mexico August 20-26, 2018: 1234-1245
HOU Y, LIU Y, CHE W, et al. Sequence-to-sequence data augmentation for dialogue language understanding [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1807.01554.pdf.
[63]JAEGER S, CANDEMIR S, ANTANI S, et al. Two public chest X-ray datasets for computer-aided screening of pulmonary diseases [J]. Quantitative Imaging in Medicine and Surgery, 2014, 4(6): 475-477.
[64]JAEGER S, KARARGYRIS A, CANDEMIR S, et al. Automatic tuberculosis screening using chest radiographs [J]. IEEE Transactions on Medical Imaging, 2014, 33(2): 233-245.
[65]CANDEMIR S, JAEGER S, PALANIAPPAN K, et al. Lung segmentation in chest radiographs using anatomical atlases with nonrigid registration [J]. IEEE Transactions on Medical Imaging, 2014, 33(2): 577-590.
[66]DEMNER-FUSHMAN D, KOHLI M D, ROSENMAN M B, et al. Preparing a collection of radiology examinations for distribution and retrieval [J]. Journal of the American Medical Informatics Association, 2016, 23(2): 304-310.
[67]Open-i. Open access biomedical image search engine [DB/OL]. [2019-01-04]. https://openi.nlm.nih.gov.
[68]WANG X, PENG Y, LU L, et al. Chest X-ray8: hospital-scale chest X-ray database and benchmarks on weakly-supervised classification and localization of common thorax diseases [C]// Proceedings of the 2017 IEEE Conference on Computer Vision and Pattern Recognition. Piscataway, NJ: IEEE, 2017: 3462-3471.
[69]RAJPURKAR P, IRVIN J, ZHU K, et al. CheXNet: radiologist-level pneumonia detection on chest X-rays with deep learning [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1711.05225.pdf.
[70]RAJPURKAR P, IRVIN J, BAGUL A, et al. MURA dataset: towards radiologist-level abnormality detection in musculoskeletal radiographs [EB/OL]. [2019-01-04].https://stanfordmlgroup.github.io/competitions/mura/.
[71]程国华,陈波,季红丽.基于3D全连接卷积神经网络的CT图像肺结节检测系统[P].中国,CN 201710173432.6[P]. 2017-07-11(CHENG G H, CHEN B, JI H L. CT image pulmonary nodule detection system based on 3D fully connected convolutional neural network [P].China, CN 201710173432.6[P].2017-07-11)
程国华,陈波,季红丽. 基于3D全连接卷积神经网络的CT图像肺结节检测系统: CN201710173432.6[P/OL].2017-07-11[2019-01-04]. http://www2.drugfuture.com/cnpat/search.aspx.(CHENG G H, CHEN B, JI H L. CT image pulmonary nodule detection system based on 3D fully connected convolutional neural network: CN201710173432.6[P/OL]. 2017-07-11[2019-01-04]. http://www2.drugfuture.com/cnpat/search.aspx.)
[72]上海交通大学人工智能实验室如何用AI定位肺结节[EB/OL]。[2019-05-04].https://www.jiqizhixin.com/articles/2017-10-24
机器之心.天池大数据竞赛第一名,上海交通大学人工智能实验室如何用AI定位肺结节[EB/OL]. [2019-01-04].https://www.jiqizhixin.com/articles/2017-10-24.(Heart of Machine. How to locate pulmonary nodules with AI in artificial intelligence laboratory of Shanghai Jiaotong University which is Tianchi Big Data Competition No. 1[EB/OL]. [2019-01-04]. https://www.jiqizhixin.com/articles/2017-10-24.)
[73]FRID-ADAR M, KLANG E, AMITAI M, et al. Synthetic data augmentation using GAN for improved liver lesion classification [C]// Proceedings of the IEEE 15th International Symposium on Biomedical Imaging. Piscataway, NJ: IEEE, 2018: 289-293.
[74]NIE D, TRULLO R, LIANG J, et al. Medical image synthesis with context-aware generative adversarial networks [C]// Proceedings of the 2017 International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 10435. Berlin: Springer, 2017: 417-425.
[75]林偉铭,高钦泉,杜民.卷积神经网络诊断阿尔兹海默症的方法[J].计算机应用,2017, 32(12):3504-3508.(LIN W M, GAO Q Q, DU M. Convolutional neural network based method for diagnosis of Alzheimers disease [J]. Journal of Computer Applications, 2017, 32(12): 3504-3508.)
[76]THYREAU B, SATO K, FUKUDA H, et al. Segmentation of the hippocampus by transferring algorithmic knowledge for large cohort processing [J]. Medical Image Analysis, 2018,43: 214-228.
[77]DONG H, YANG G, LIU F, et al. Automatic brain tumor detection and segmentation using U-net based fully convolutional networks [C]// Proceedings of the 2017 Annual Conference on Medical Image Understanding and Analysis, CCIS 723. Berlin: Springer, 2017: 506-517.
[78]RONNEBERGER O, FISCHER P, BROX T. U-net: convolutional networks for biomedical image segmentation [C]// Proceedings of the 2015 International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 9351. Berlin: Springer, 2015:234-241.
[79]MENZE B H, JAKAB A, BAUER S, et al. The multimodal brain tumor image segmentation benchmark (BRATS) [J]. IEEE Transactions on Medical Imaging, 2015, 34(10): 1993-2024.
[80]SHIN H, TENEHOLTZ N A, ROGERS J K, et al. Medical image synthesis for data augmentation and anonymization using generative adversarial networks [EB/OL]. [2019-01-04]. https://arxiv.org/pdf/1807.10225.pdf.
[81]VASCONCELOS C N, VASCONCELOS B N. Increasing deep learning melanoma classification by classical and expert knowledge based image transforms [EB/OL].[2019-01-04]. https:// arxiv.org/pdf/1702.07025v1.pdf.
[82]CIRESAN D C, GIUSTI A, GAMBARDELLA L M, et al. Mitosis detection in breast cancer histology images with deep neural networks [C]// Proceedings of the 2013 International Conference on Medical Image Computing and Computer-Assisted Intervention, LNCS 8150. Berlin: Springer, 2013: 411-418.
This work is partially supported by the Shanghai Pujiang Talent Plan (17PJ1432500).
WEI Xiaona, born in 1986, M.S. candidate. Her research interests include deep learning,medical image processing.
LI Yinghao, born in 1999. His research interests include machine learning, image reconstruction.
WANG Zhenyu, born in 1996, M. S. candidate. His research interests include deep learning, medical image processing.
LI Haozun, born in 1996. His research interests include deep learning, image reconstruction.
WANG Hongzhi, born in 1975, Ph. D., associate professor. His research interests include magnetic resonance imaging technology, medical image analysis.
