代谢组学在农产品营养品质检测分析中的应用
2019-10-10许彦阳姚桂晓刘平香赵洁王昕璐孙君茂钱永忠
许彦阳,姚桂晓,3,刘平香,赵洁,王昕璐,孙君茂,钱永忠
代谢组学在农产品营养品质检测分析中的应用
许彦阳1,姚桂晓1,3,刘平香1,赵洁1,王昕璐1,孙君茂2,钱永忠1
(1中国农业科学院农业质量标准与检测技术研究所/农业农村部农产品质量安全重点实验室,北京 100081;2农业农村部食物与营养发展研究所, 北京 100081;3西安理工大学印刷包装与数字媒体学院,西安 710048)
科学检测和分析农产品营养品质对提升优质农产品的营养水平具有重要作用。由于农产品中营养成分组成复杂,已有的分析方法只能针对已知营养素的浓度、功能等进行分析,无法对农产品中存在的大量其他功能性活性物质进行分析鉴定。代谢组学技术通过高通量化学分析技术对生物样品中的小分子代谢产物进行定性和定量分析,在具有特殊营养功能的小分子物质分析中具有突出优势。代谢组学技术的引入,为农产品营养成分表征及差异性分析,产地溯源及真伪鉴别,生长储藏过程中营养成分变化规律揭示,营养功能成分作用机制研究等提供了新方法,也为膳食结构的优化调整提供了新策略。本文对代谢组学研究方法的新进展,包括样品制备、代谢物分析鉴定以及数据分析等进行了综述,总结了代谢组学技术在农产品营养成分表征及差异性分析、产地溯源及真伪鉴别、生长储藏过程中营养成分变化规律以及营养功能成分作用机制等方面的应用,旨在为我国农业高质量发展提供思路。在样品制备方法方面,首先通过快速改变样品所处的环境条件,如添加强酸、强碱或液氮冷冻等技术终止新陈代谢相关酶的活性。然后针对代谢物的极性,选择不同的提取溶剂,从而获得较高的提取率。在样品分析方法方面,核磁共振、色谱质谱和毛细管电泳质谱等技术得到了广泛地应用。其中,色谱质谱联用技术将色谱的有效分离和质谱的准确定量相结合,已经成为代谢组学中使用最广泛的分析技术。在数据处理及分析方面,无监督分析中的主成分分析和有监督分析中的正交偏最小二乘法-判别分析是目前应用最多的数据分析方法。通过通路分析软件的富集分析和拓扑分析,可以明确与代谢物差异相关性最高的代谢通路,对差异代谢产物的机制进行分析和解释。在农产品营养成分分析应用方面,通过对农产品中初生代谢产物及次生代谢产物进行全面表征,形成农产品独特的代谢指纹图谱,从而实现营养成分的差异性分析;通过非特定目标物的检测和无监督分析方法,实现不同产地农产品的组间差异鉴别和差异化合物筛选;通过农产品生长过程中关键成分的消长规律及合成机理分析,指导最佳收获期;通过体液代谢谱和生物标志物的检测,系统研究营养功能成分与生物体代谢之间的交互作用,为营养功能成分作用机制研究及膳食指导提供有价值的信息。
代谢组学;农产品;营养;品质
0 引言
当前,我国进入消费结构转型升级和供给侧结构性改革的关键时期,面对“吃饱”到“吃好”的需求转变,需要发展高品质、高营养的农产品[1-2]。农产品的品质和营养一般根据产品中营养素的种类、含量以及营养素功能进行判定。农产品中的营养素[3]主要包括蛋白质、脂肪、碳水化合物、维生素、矿物质元素、膳食纤维、水及其他各类微量功能活性成分等8大类,通过对8类物质成分含量和组分的检测,建立食用农产品的营养成分数据库,从而评价农产品的质量。同时,针对不同农产品中营养素的特定功能性成分,如不饱和脂肪酸类、多酚类、黄酮类、多糖类、多肽类等物质进行降血糖、降血压、抗衰老、抗动脉硬化等功能研究,评价农产品的营养功能。然而,农产品中营养成分组成复杂,已有的分析方法只能针对已知营养素的浓度、功能等进行分析,无法对农产品中存在的大量其他功能性活性物质进行分析鉴定。
代谢组学以分子量在1 000以下的小分子为研究对象,通过对动态多参数代谢应答反应进行定量测量,研究生物体内源性和外源性代谢物整体构成及代谢途径变化的新技术[4-6],在疾病研究[7]、药物开发[8]、毒性评价[9]、环境科学[10]等领域已经得到较为广泛的应用。由于代谢组学可以在全景角度揭示生物系统、生理状态等优势,近年来被逐渐应用于农产品的营养品质研究。通过代谢组学分析,为农产品营养成分表征及差异性分析,产地溯源及真伪鉴别,生长储藏过程中营养成分变化规律揭示,营养功能成分作用机制研究等提供新方法[11-12],也为膳食结构的优化调整提供新策略。
1 代谢组学研究进展
代谢组学技术根据研究目的和研究层次不同分为非靶向代谢组学和靶向代谢组学[13]。非靶向代谢组学是对生物系统中代谢物进行全面系统地分析,尽可能多地定性和相对定量生物体系中的代谢物,获取大量数据并对其进行处理,进而分析获得差异性代谢物;靶向代谢组学通常针对某个代谢通路或已知的某些特定代谢物,进行高通量检测和定量分析,主要用于验证差异性代谢物。应用代谢组学技术开展农产品营养品质检测分析研究主要包括样品的收集和制备,代谢物分离、检测和鉴定,数据统计分析等过程。
1.1 代谢组学样品制备方法
样品的收集和制备是代谢组学分析的基础,主要包括样品采集、淬灭、储存、提取等[14-15],旨在迅速、有效终止样品新陈代谢反应,最大限度地去除样本中杂质(如蛋白质、糖类、脂肪等),同时能较为完整地保留样品中的整体代谢产物或特异性目标代谢产物[16]。
代谢组学技术的研究对象包括组织、器官、提取液等,分析对象种类多,结构、性质差异大,要获得稳定可重复的试验结果,需考虑试验目的、仪器平台、分析方法等要求,选择合适的样品处理方法。目前,主要通过快速改变样品所处的环境条件,如添加强酸、强碱或液氮冷冻等手段终止新陈代谢相关酶的活性。采用的前处理技术主要包括超声波提取[17]、固相萃取[18]、固相微萃取[19]、分散液液微萃取[20]、超临界流体萃取[21]等,使用的提取剂主要包括甲醇、乙醇、乙腈、异丙醇、氯仿、水以及混合溶剂等。Vargas等[22]比较了不同萃取剂、不同温度和运输条件对代谢物提取效果的影响,结果显示,提取溶剂对代谢产物具有显著影响。
代谢物的极性不同,提取溶剂也应不同。如极性代谢物的提取溶剂主要为含水的有机溶剂,如甲醇/水溶液、乙腈/水溶液、氯仿/甲醇/水溶液等[23]。脂溶性代谢物常用的提取溶剂主要有氯仿、甲基叔丁醚和二氯甲烷等[23-24],获得较高提取率的关键是氯仿/甲醇/水的比例。BLIGH等[25]在FOLCH等[26]提取方法的基础上进行了进一步的优化,指出提取鳕鱼组织中脂质的氯仿/甲醇/水的最佳比例为1﹕2﹕0.8。鉴于氯仿具有高毒性,作为其替代物的二氯甲烷逐渐被用于脂溶性代谢产物的提取[27]。此外,使用氯仿/甲醇法提取脂质代谢产物时,目标物位于下方的氯仿层,操作时需穿过上方水层,操作不方便。MATYASH等[28]基于甲基叔丁醚密度小于水的原理,提出了以甲基叔丁醚代替氯仿或二氯甲烷提取脂质的方法,使得脂质所在的甲基叔丁醚层位于水层上方,简化了脂质提取的操作过程。
生物体内的部分代谢物极性强、挥发性低,如氨基酸、脂肪酸、胺类、糖类和核苷酸类物质,前处理过程中需要进行化学衍生化,常用的衍生化方法包括硅烷化、甲酯化、烷基化、酰基化等[29]。另外,代谢物的基质不同,所选择的提取方法和提取试剂也各不相同,在实际操作过程中应结合基质特点,妥善选择前处理方法,以达到最佳提取效果。在非靶向代谢组学研究中,通常需要制备质控(QC)样品,保证分析数据的稳定性和可靠性。
1.2 代谢组学分析方法
代谢组学基于高通量分析技术对生物样品中小分子代谢产物的组成、含量及其变化进行定性和定量分析,通过代谢物信息与生物体生理变化的关联分析,寻找生物标志物。样品中的成分复杂多样,有效分离并准确鉴定各种物质是代谢组学研究的前提。目前,代谢组学主要的分析技术包括核磁共振、色谱质谱和毛细管电泳质谱等。
1.2.1 核磁共振技术 核磁共振(nuclear magnetic resonance,NMR)技术是指核磁矩不为零的原子核,在外磁场的作用下,共振吸收某一特定频率的电磁波。通过能量吸收曲线分析,判断该原子在分子中所处的位置及相对数目,从而实现定量分析和结构分析。NMR技术可以对样品实现无偏性分析,具有前处理较少、不破坏样品结构和性质等优点,能够对完好组织、生物液等进行代谢成分分析,并确定代谢物结构式[30]。适用于农产品中脂肪、多元醇类、有机酸、糖类等营养成分的检测分析。应用最为广泛的一维氢谱核磁共振(1H-NMR)对含氢代谢产物具有普适性,具有无需标准品、无损等特点,可得到丰富的样品信息,在农产品组分分析、产品质量鉴别、质量控制等方面应用广泛[31-32]。许茜等[33]采用NOESY脉冲序列对14个不同来源的固体动物胶样品进行原料来源鉴定,结果显示,1H-NMR技术能够区分不同样品的来源,判别正确率达91.67%。对复杂样品的代谢物鉴定需结合二维核磁共振波谱,如总相干转移光谱(total correlation spectroscopy,TOCSY)[34],TOCSY可以解决重叠峰的问题,能进一步提高分辨率[35]。另外,核磁共振技术与色谱结合使用可有效提高分析的灵敏度,如液相核磁共振联用(LC-NMR)解决了NMR中干扰过多等问题,有效提高了分析的检测限[36]。BRAUNBERGER等[37]综合液相质谱技术、液相核磁共振联用和离线NMR技术,分析了茅膏菜中黄酮类和鞣花酸衍生物,并解析了13种化合物的结构。
1.2.2 质谱分析技术 色谱质谱联用技术将色谱的有效分离和质谱的准确定量相结合,已经成为代谢组学中使用最广泛的分析技术。经过近年来的发展,目前,质谱分析技术主要包括四级杆质谱(quadrupole mass spectrometer, QMS)、三重四级杆质谱(triple quadrupole mass spectrometer,QQQ)、飞行时间质谱(time-of-flight mass spectrometry,TOF-MS)、四级杆飞行时间串联质谱(quadrupole time-of-flight mass spectrometry,Q-TOF-MS)、离子阱质谱(ion trap mass spectrometer,ITMS)等,所使用的电离源主要包括电喷雾电离源(electron spray ionization,ESI)[38]、大气压化学电离源(atmospheric-pressure chemical ionization,APCI)、基质辅助激光解析电离源(matrix- assisted laser desorption ionization,MALDI)[39]、大气压光电离源(atmospheric-pressure photoionization,APPI)、质子转移反应器(proton transfer reaction,PTR)[40]、实时直接分析(direct analysis in real time,DART)[41]等。相较于NMR技术,质谱的灵敏度更高,但稳定性不足,在不同仪器甚至不同日期获得的数据之间存在差异,为保证结果的稳定,需采用标准化的操作步骤[42]。
GC-MS是指气化后的样品根据各组分的熔沸点、吸附性及极性不同在色谱柱中实现分离,在质谱系统中经电离产生具有不同质荷比的离子后进行分析的技术。GC-MS技术具有分离能力强,灵敏度高,分析速度快,操作方便,标准谱库成熟等优点,通常用于挥发性、半挥发性、低分子量和热稳定化合物的鉴定和定量检测。适用于农产品中精油、酯类、类十二烷酸、类胡萝卜素、类黄酮、脂质等极性较小物质的分析。相对于NMR技术,GC-MS分析技术样品前处理较为复杂,部分代谢物在分析前需要衍生化处理,以增强挥发性[43]。MARI等[44]使用甲硅烷基化混合物,经两步衍生化后,对蕨麻植物样品中的53种代谢物进行了分析。也有学者[45]采用甲氧基胺化/三甲基硅基化衍生化后,分析了乙烯利处理后甜樱桃代谢谱变化规律。为同时分析尽可能多的代谢物,具有较高分离能力和灵敏度的质谱分析技术被逐渐应用于组学分析,如WONG等[46]采用二维气相色谱飞行时间质谱(GC- GC-TOF/MS)对桉树叶油代谢谱进行了全面分析。
LC-MS是根据样品中各组分在液相色谱柱中保留时间不同实现分离,在质谱系统中经离子源电离后产生具有不同质荷比的离子后进行分析的技术。LC-MS技术可以在常温下实现分离,相较于NMR灵敏度低、GC-MS样品处理较复杂等问题,LC-MS更灵敏,不受试样挥发性和热稳定性的限制,样品前处理简单,可对农产品中糖类、氨基酸、多酚、萜类化合物等极性较强的代谢产物进行分析[47]。GARCIA等[38]基于UPLC-ESI-QToF-MS建立了非靶向的分析方法,对生菜代谢谱进行了分析。此外,基于HPLC-QToF-MS的非靶向技术在鸡蛋蛋黄[48]及大蒜[49]小分子物质分析方面也得到了很好的应用。但LC-MS没有综合性数据库,给后期代谢物的分析造成了一定的困难与挑战。
毛细管电泳质谱(capillary electrophoresis–mass spectrometry,CE-MS)[50]是基于样品中各组分间淌度和分配行为差异实现分离,经质谱系统中电离源电离产生不同质荷比的离子,从而实现离子型化合物的分析。CE-MS技术能够检测离子型化合物,在含氨基化合物等复杂基质食品分析中有很大优势,常用于氨基酸类物质的分析。CE-MS能够同时测定丁醇衍生后的6种氨基酸(鸟氨酸、丙氨酸、γ-氨基丁酸、异亮氨酸、瓜氨酸和焦谷氨酸),该方法已成功应用于大豆油、橄榄油掺假分析[51]。
目前,代谢组学的研究大部分依赖成本相对较高的高分辨质谱或核磁技术,但LAN等[52]成功开发了HPLC-UV技术,分析了不同批次草药提取物含量,该方法简单、廉价,可以作为农产品营养成分分析的初步方法。由于农产品代谢物种类多,结构、性质差异大,仅采用单一分析手段很难实现代谢物的全景定性及定量分析,可采用多种分析手段,取长补短,尽可能全面地覆盖农产品代谢物。
1.3 代谢组学的数据处理及分析
在代谢组学分析中,代谢物数据量大、样本分析产生的数据复杂,需要能自动、无歧视分析原始数据文件[53]。为使数据更简洁、分析简便,仪器分析获得的数据通常需要预处理,主要包括信号滤噪、代谢特征提取、色谱与质谱匹配、缺失值过滤与补值、信号归一化、化合物定性定量等步骤[54-55]。经过预处理后的代谢组学数据需要借助数据分析软件实现单变量分析和多元变量等统计分析,数据分析方法包括无监督分析和有监督分析两种,其中无监督分析包括主成分分析(principal component analysis,PCA)[56]、聚类分析(hierarchical cluster analysis,HCA)[57]等。由于无监督分析法不能忽略组内误差,不利于组间差异的鉴别,需要进一步结合有监督分析突出组间差异;有监督分析主要包括偏最小二乘分析(partial least squares discrimination analysis,PLS-DA)[58]、正交偏最小二乘法-判别分析(orthogonal partial least squares discriminant analysis,OPLS-DA)等。数据分析时也可以通过数据分析软件特有的分析功能揭示代谢物上/下调情况、变量权重(variable importance,VIP值)和显著性变化,经多种组合数据分析后,可以揭示不同组别样本中小分子代谢物的差异,筛选获得差异代谢物。随着组学技术的发展,多家仪器公司结合数据采集软件开发了多种数据分析软件,如Agilent公司的MPP软件、Waters公司的QI软件、SCIEX公司的Lipid View软件等[59-61]。上述数据分析软件的成功研发给从事代谢组学研究的学者们提供了便利条件,简化了数据分析工作。
通过组学软件筛选出的差异代谢物需要进一步借助通路分析软件、网站或大量相关文献,实现生物标志物的重要性和功能性解读,了解其在生命活动过程中参与的代谢通路,继而进一步为物质靶点分析、营养功能评价等提供参考依据[62]。HMDB[63]、KEGG、METLIN[64]、NIST[65]等数据库是常用的代谢物结构鉴定和代谢通路分析的数据库。我国学者WANG等[66]也开发了代谢途径延伸(MPE)的方法快速表征代谢组学生物标志物,即通过已知的代谢途径连接未知的代谢物到特定的核心化合物,然后通过定量测定匹配代谢物的MS/MS谱进行验证,获得93种生物标志物(95%覆盖)的结构后,通过MS/MS匹配确认获得66种代谢物(70%覆盖),快速阐明了肉碱的靶向代谢网络。
2 代谢组学在农产品营养品质检测分析中的应用
代谢组学能在生物学水平上检测农产品中小分子代谢产物的差别,定性与定量分析不同品种、不同产地和不同生长储藏条件下农产品营养成分的差异。同时,经过非靶向代谢组学全面系统分析和靶向代谢组学的确证后,新的生物标志物的发现进一步推动了代谢组学在产地溯源、作用机制、育种策略以及种养方式等方面的应用,见表1。
2.1 农产品营养成分表征及差异性分析
不同农产品具有不同的特征营养成分,代谢组学技术可对农产品中初生代谢产物及次生代谢产物进行全面表征,形成农产品独特的代谢指纹图谱。ZHU等[67]采用广泛靶向的LC-MS/MS分析方法,研究了442份成熟番茄果实果皮组织中代谢物,共发现了980个不同的代谢物。BÖTTCHER等[68]采用LC/ESI-QTOF-MS技术对洋葱中的低聚果糖、氨基酸、多肽、S-半胱氨酸、黄酮及皂苷类等极性及半极性成分进行了全面的代谢轮廓分析。对葡萄代谢组的研究发现,野生品种的葡萄花色苷及芪类化合物组成比杂交品种及欧亚种葡萄复杂,欧亚种葡萄果皮和种籽中原花青素含量更高[69]。意大利‘蓝蟹’等3个品种代谢组学分析结果显示,谷氨酸、丙氨酸、甘氨酸、龙虾碱、乳酸、甜菜碱和牛磺酸等7种代谢物含量具有显著差异[70]。
农产品中代谢物种类及水平通常与其感官特征具有重要相关性,如番茄中邻甲基苯乙酮、苯甲酮等挥发性成分对番茄气味形成起关键作用[71],芜菁中L-谷氨酰胺、L-天冬酰胺与苦味呈负相关,而金缕梅糖、麦芽糖和苹果酸与苦味呈正相关,异硫氰酸烯丙酯等芥子甙降解产物与“锐”度呈正相关[72]。此外,不同农产品品种、不同组织及器官营养成分不同,如不同品种蔓越莓[73]、树莓[74]花青素的含量及水平,马铃薯皮和块茎组织[75]、番茄果肉与种子[76]代谢物种类及水平均存在较大差异,借助代谢组学分析,可以获得不同品种、组织及器官的营养成分组成及含量,从而应用于不同的农产品加工和消费。
转基因技术提高了粮食产量,但由此而来的安全问题引起广泛争论,代谢组学技术能在生物学水平上鉴别和筛选转基因与非转基因农产品代谢差异物,揭示转基因带来的本质性变化以及各种非预期变异效应[77]。RAO等[78]对转基因玉米与常规玉米中210种代谢产物进行分析,在考虑自然变异后发现4种差异代谢物。番茄经基因修饰后氨基酸和多酚等物质含量水平发生显著性改变,其中山奈酚、桂皮素苷含量水平较对照组提高10倍以上[79]。非靶向代谢组学在考虑自然变异的情况下能为转基因作物代谢产物分析提供技术手段。
2.2 农产品产地溯源及真伪鉴别
农产品的产地来源是消费者选择食品的重要依据,食品掺假等问题引起了消费者的广泛关注。代谢组学技术在非特定目标物的检测方面有着其他方法无法比拟的优势,有监督分析方法可以有效减少同一产地农产品的组内误差,实现不同产地农产品的组间差异鉴别和差异化合物筛选,为农产品产地溯源和真伪鉴别提供了重要技术手段[80-81]。ISABAL等[82]基于非靶向代谢组学方法分析了4种不同地理来源枸杞的化学成分区别,发现了蒙古枸杞中槲皮素、山奈酚糖苷、二咖啡酰奎宁酸和酚酸含量显著高于其他地区的枸杞。对捷克、中国和西班牙3个国家的大蒜进行代谢轮廓分析[83],获得了蒜氨酸、PC(16﹕0/18﹕2)和精氨酸3种标志性差异化合物。RITOTA等[84]采用高分辨率魔角旋转核磁共振技术(HRMAS-NMR)及偏最小二乘法判别分析(PLS-DA)等多元数据统计分析方法,成功实现意大利4个产地、2个品种大蒜(白皮蒜、紫皮蒜)的分类及溯源。CAMARGO[85]、VADALA[86]和SMITH等[87]也分别通过矿物质元素及痕量金属元素实现了大蒜不同产地的溯源。
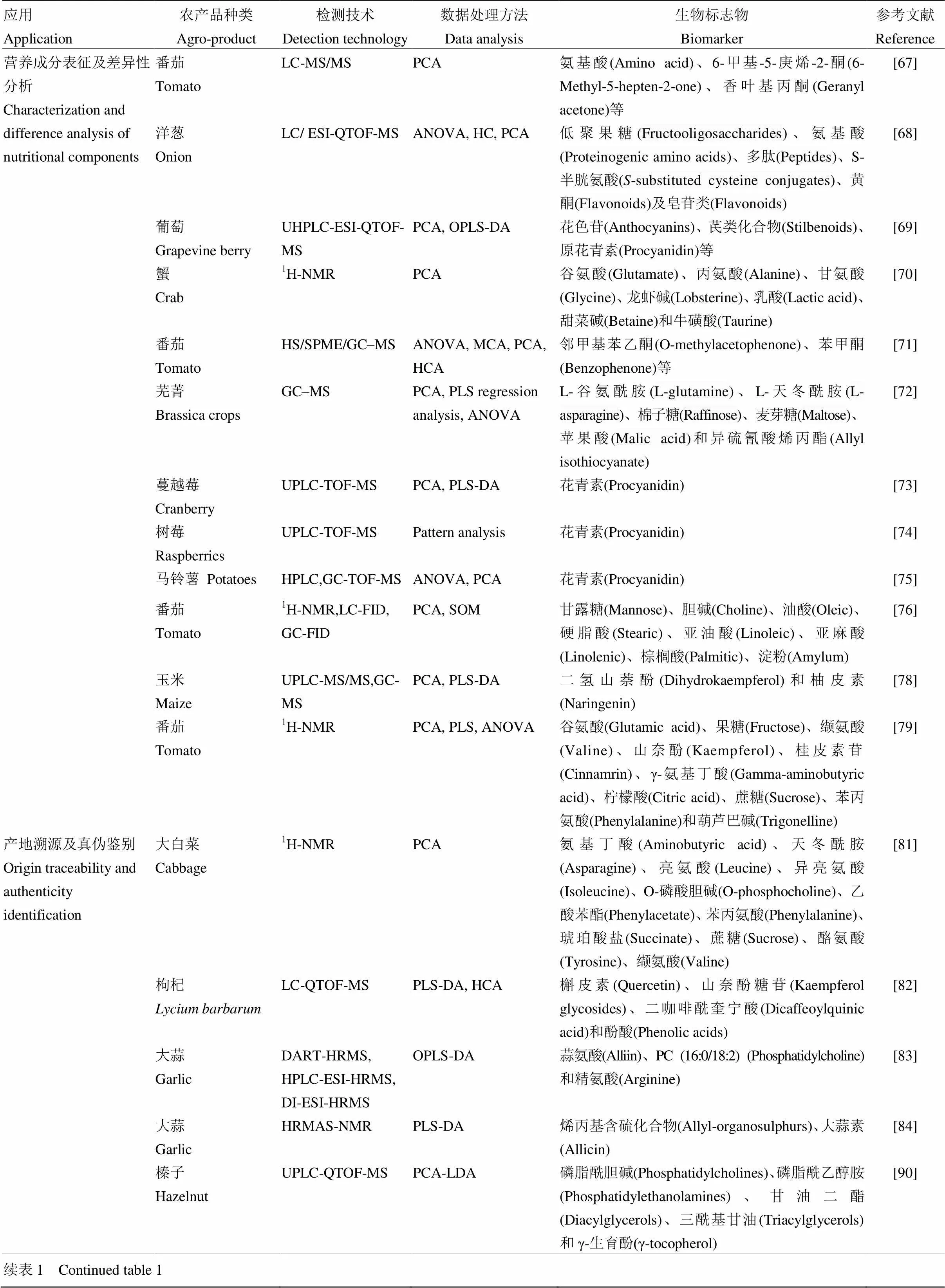
表1 代谢组学在农产品营养品质检测分析中的应用
经非靶向代谢组学筛查获得差异代谢物后,靶向代谢组学可以对产品中的特异性指标如氨基酸、挥发性成分等化合物成分进行定量分析[88-89],进一步提高模型鉴别能力。KLOCKMANN等[90]采用UPLC- QTOF-MS对来自德国、法国、意大利、土耳其和格鲁吉亚共196个榛子进行了产地溯源,筛选确定了20种关键差异代谢物(5种磷脂酰胆碱,3种磷脂酰乙醇胺,4种甘油二酯,7种三酰基甘油和γ-生育酚),PCA-LDA训练精度可达99.5%。随后,对鉴定出的差异代谢物采用LC-ESI-QqQ-MS进行了靶向分析[91],预测模型训练精度达到100%,交叉验证精度达到97%。
2.3 农产品生长储藏过程中营养成分变化规律
农产品不同生长阶段营养成分存在较大差异,研究农产品生长过程中关键成分的消长规律及合成机理,对于确定最佳收获期和改善果蔬的保质期具有重要意义。ZHANG等[92]对草莓生长过程中的挥发性成分进行了定性和定量分析,发现草莓成熟之前游离氨基酸含量逐渐下降,过熟后快速上升,涉及的代谢通路包括酯生物合成、三羧酸循环、莽草酸途径和氨基酸代谢。对‘赤霞珠’和‘梅洛’2个不同葡萄品种在生长、发育及成熟过程中营养成分变化规律及代谢途径研究发现,葡萄在坐果和颜色转化期代谢物变化较大,且在果实成熟过程中,糖类和氨基酸呈现相反的变化趋势[93]。农产品在采后、储藏过程中,营养成分会发生转化或降解,对其营养品质产生影响。OMS-OLIU等[94]鉴定发现,甘露糖、柠檬酸、葡萄糖酸和酮-1-古洛糖酸等4种代谢产物与番茄采后阶段密切相关。JOHNSON等[48]研究了不同储藏时间蛋黄内小分子代谢物的变化规律,确定胆碱为不同储藏时间的差异代谢物,随后靶向验证结果显示,12周储藏时间内胆碱水平由6.8 μg∙g-1上升至28.7 μg∙g-1。
此外,外界环境以及灌溉、施肥、管理措施等因素均会对农产品生长发育过程中营养品质的变化规律产生影响[95],借助代谢组学高通量检测技术研究不同环境条件下农产品的生物标志物,可为农产品的品质优化提供科学的指导意见。我国西南地区在大豆收获期温度低,湿度大,持续降雨天气易导致霉菌爆发,DENG等[96]研究了豆荚、种皮和子叶对霉菌的抵抗机制,发现不同组织代谢轮廓存在差异,脯氨酸、赖氨酸和硫在子叶、种皮和豆荚代谢中发挥重要作用。
2.4 农产品中营养功能成分作用机制及对代谢的影响
代谢组学通过体液代谢谱和生物标志物的检测,可以系统性地研究营养功能成分与生物体代谢之间的交互作用[97],为营养功能成分作用机制研究及膳食指导提供有价值的信息[98-99]。代谢组学为食药两用农产品中功能成分作用机制研究提供了可靠便利的方法。LIU等[100]基于雄性C57BL/6小鼠模型,研究了生姜中姜精油(GEO)和柠檬醛的抗氧化能力和肝脏保护作用,采用HPLC-QTOF-MS分析技术对小鼠血清中的代谢产物进行了分析,结果显示,服用姜精油(GEO)和柠檬醛后,因食用含酒精液体饮食而导致的D-葡糖醛酸-6,3-内酯、甘油-3-磷酸、丙酮酸、石胆酸、2-吡啶酸和前列腺素E1等异常代谢产物可恢复至正常水平,上述代谢产物的改变主要涉及碳代谢、氨基酸代谢等代谢通路。枸杞多糖(lycium barbarum polysaccharides,LBP)的降糖作用研究结果显示[101],枸杞多糖干预一个月后,2型糖尿病模型大鼠血清中丙氨酸、胸腺嘧啶脱氧核苷酸含量有所上升,氨基酸代谢和核苷酸代谢通路的确认为进一步研究LBP的降糖作用机制提供了参考。除小鼠和大鼠模型外,也有学者采用细胞模型分析农产品中营养素的作用机制,如FUJIMURA等[102]基于人脐静脉内皮细胞,研究了不同品种绿茶抑制肌球蛋白调节轻链(MRLC)磷酸化的活性,分析获得了能够显著抑制MRLC磷酸化的品种,并发现多酚含量与其独特的代谢特征和生物活性相关。
此外,通过对膳食摄入后人体尿液、血液、粪便等生物样本的研究,代谢组学可以了解机体对单一物质或复杂物质的代谢应答[103-104],从而为全面考察农产品摄入对机体代谢的影响提供参考[105-106]。边会喜[107]基于小鼠模型研究了苦瓜及苦瓜的不同提取组分对降低血脂的作用,通过对尿液样本的NMR代谢组分析识别了37种差异代谢物,确定了泛酸和辅酶a合成、淀粉和蔗糖代谢、三羧酸循环等3条主要作用途径,发现苦瓜的正丁醇提取物和胰岛素对小鼠体重及多种脂肪组织的抵抗效应高于其他提取物。人体尿液样品的代谢谱分析发现[108],适量饮用白茶使机体内马尿酸和柠檬酸上调、肌酐下降。1H-NMR光谱技术结合多元统计分析研究素食、少肉、多肉3种饮食模式对人体代谢的影响显示,肌酸、肉碱、乙酰肉碱和三甲胺-N-氧化物等代谢物在多肉饮食模式中含量较高,而对-羟基苯基在素食饮食模式下含量较高[109]。
3 展望
随着组学技术的不断发展和完善,代谢组学技术将在农产品营养品质检测分析中发挥越来越重要的作用,该类技术在提升农产品营养的同时还有助于提升农产品品质,对农产品育种策略、种养模式调整和膳食指导等方面具有重要指导意义。然而,当前阶段组学分析中存在样品分析结果不稳定、仪器分析范围局限和数据库不完善等诸多挑战,因此,如何优化代谢组学的样品前处理技术,建立高通量的检测技术,如何使数据处理过程更加标准和规范,以便获得更多更准确的代谢物信息,在取样方法、分析技术研发和数据库构建等方面,都需要进一步的研究。另外,基于代谢组学技术发现了许多农产品中的生物标志物,但对农产品中生物活性成分的作用机制研究还不够系统和深入,需结合基因组学、转录组学和蛋白组学等组学技术,形成系统生物学数据链,通过多组学联合分析技术,从表型-通路等多角度解释功能活性成分的作用机制,为膳食结构的优化调整提供技术手段。
[1] 吴永宁. 我国食品安全科学研究现状及"十三五"发展方向. 农产品质量与安全, 2015(6): 3-6.
WU Y N. The research status of food safety science in China and the development direction in “13th Five-year Plan”., 2015(6): 3-6. (in Chinese)
[2] 唐华俊. 中国营养型农业发展正当其时. 高科技与产业化, 2018, 266(7): 3.
TANG H J. China's nutritious agriculture is developing at the right time., 2018, 266(7): 3. (in Chinese)
[3] 韩娟, 孙君茂, 秦玉昌. 农产品质量与营养功能风险评估研究方向探讨. 农产品质量与安全, 2016(2): 45-48.
HAN J, SUN J M, QIN Y C. Discussion on the research direction of risk assessment in agricultural product quality and nutrition function., 2016(2): 45-48. (in Chinese)
[4] 许国旺, 路鑫, 杨胜利. 代谢组学研究进展. 中国医学科学院学报, 2007, 29(6): 701-711.
XU G W, LU X, YANG S L. Recent advances in metabonomics., 2007, 29(6): 701-711. (in Chinese)
[5] NICHOLSON J K, LINDON J C, HOLMES E. ''Metabonomics'': understanding the metabolic responses of living systems to pathophysiological stimuli via multivariate statistical analysis of biological NMR spectroscopic data., 1999, 29(11): 1181-1189.
[6] FIEHN O. Metabolomics–the link between genotypes and phenotypes., 2002, 48(1/2): 155-171.
[7] COCCI P, MOSCONI G, PALERMO F A. Changes in expression of microRNA potentially targeting key regulators of lipid metabolism in primary gilthead sea bream hepatocytes exposed to phthalates or flame retardants., 2019, 209: 81-90.
[8] EVERETT J R. Pharmacometabonomics in humans: A new tool for personalized medicine., 2015, 16(7): 737-754.
[9] POTRATZ S, TARNOW P, JUNGNICKEL H, BAUMANN S, VON BERGEN M, TRALAU T, LUCH A. Combination of metabolomics with cellular assays reveals new biomarkers and mechanistic insights on xenoestrogenic exposures in MCF-7 Cells., 2017, 30(4): 883-892.
[10] JIANG G T, KANG H Y, YU Y Q. Cross-platform metabolomics investigating the intracellular metabolic alterations of HaCaT cells exposed to phenanthrene., 2017, 1060: 15-21.
[11] ZOTTI M, DE PASCALI S A, DEL COCO L, MIGONI D, CARROZZO L, MANCINELLI G, FANIZZI F P.1H NMR metabolomic profiling of the blue crab () from the Adriatic Sea (SE Italy): a comparison with warty crab (), and edible crab ()., 2016, 196: 601-609.
[12] CHEN J P, CHAN P H, LAM C T W, LI Z G, LAM K Y C, YAO P, DONG T T X, LIN H Q, LAM H, TSIM K W K. Fruit of ziziphus jujuba (Jujube) at two stages of maturity: distinction by metabolic profiling and biological assessment., 2015, 63(2): 739-744.
[13] CEVALLOS-CEVALLOS J M, REYES-DE-CORCUERA J I, ETXEBERRIA E, DANYLUK M D, RODRICK G E. Metabolomic analysis in food science: A review., 2009, 20(11): 557-566.
[14] 赵春霞, 许国旺. 基于液相色谱-质谱技术的代谢组学分析方法新进展. 分析科学学报, 2014, 30(5): 761-766.
ZHAO C X, XU G W. Progress of metabonomics technique based on liquid chromatography-mass spectrometry.,2014, 30(5): 761-766. (in Chinese)
[15] 马宁, 杨亚军, 刘希望, 李剑勇. 基于液质平台代谢组学生物样本的采集和制备. 中国兽医学报, 2017, 37(6): 1193-1200.
MA N, YANG Y J, LIU X W, LI J Y. Biological sample collection and preparation for metabonomic study with LC-MS platform.,2017, 37(6): 1193-1200. (in Chinese)
[16] KNOLHOFF A M, CROLEY T R. Non-targeted screening approaches for contaminants and adulterants in food using liquid chromatography hyphenated to high resolution mass spectrometry., 2016, 1428: 86-96.
[17] LIU Y Y, HU X L, BAO Y F, YIN D Q. Simultaneous determination of 29 pharmaceuticals in fish muscle and plasma by ultrasonic extraction followed by SPE-UHPLC-MS/MS., 2018, 41(10): 2139-2150.
[18] ANDRADE-EIROA A, CANLE M, LEROY-CANCELLIERI V, CERDà V. Solid-phase extraction of organic compounds: A critical review (Part I)., 2016, 80: 641-654.
[19] WANG C H, SU H, CHOU J H, HUANG M Z, LIN H J, SHIEA J. Solid phase microextraction combined with thermal-desorption electrospray ionization mass spectrometry for high-throughput pharmacokinetics assays., 2018, 1021: 60-68.
[20] SHAMSIPUR M, YAZDANFAR N, GHAMBARIAN M. Combination of solid-phase extraction with dispersive liquid–liquid microextraction followed by GC–MS for determination of pesticide residues from water, milk, honey and fruit juice., 2016, 204: 289-297.
[21] GIL-RAMIREZ A, AL-HAMIMI S, ROSMARK O, HALLGREN O, LARSSON-CALLERFELT A K, RODRíGUEZ-MEIZOSO I. Efficient methodology for the extraction and analysis of lipids from porcine pulmonary artery by supercritical fluid chromatography coupled to mass spectrometry., 2019, 1592: 173-182.
[22] VARGAS L H G, NETO J C R, RIBEIRO J A D, RICCI-SILVA M E, SOUZA M T, RODRIGUES C M, OLIVEIRA A E, ABDELNUR P V. Metabolomics analysis of oil palm () leaf: evaluation of sample preparation steps using UHPLC-MS/MS., 2016, 12(10): 153.
[23] 徐佳, 刘其南, 翟园园, 单进军, 张丽. 细胞代谢组学样品前处理研究进展. 中国细胞生物学学报, 2018, 40(3): 418-425.
XU J, LIU Q N, ZHAI Y Y, SHAN J J, ZHANG L. The Research development of sample pretreatment in cell metabolomics., 2018, 40(3): 418-425. (in Chinese)
[24] WANG X, XU Y, SONG X, JIA Q, ZHANG X, QIAN Y, QIU J. Analysis of glycerophospholipid metabolism after exposure to PCB153 in PC12 cells through targeted lipidomics by UHPLC- MS/MS., 2019, 169: 120-127.
[25] BLIGH E G, DYER W J. A rapid mmethord of total lipid extraction and purification., 1959, 37(8): 911.
[26] FOLCH J, LEES M, SLOANE STANLEY G H. A simple method for the isolation and purification of total lipides from animal tissues., 1957, 226(1): 497-509.
[27] SARAFIAN M H, GAUDIN M, LEWIS M R, MARTIN F P, HOLMES E, NICHOLSON J K, DUMAS M E. Objective set of criteria for optimization of sample preparation procedures for ultra-high throughput untargeted blood plasma lipid profiling by ultra performance liquid chromatography-mass spectrometry., 2014, 86(12): 5766-5774.
[28] MATYASH V, LIEBISCH G, KURZCHALIA T V, SHEVCHENKO A, SCHWUDKE D. Lipid extraction by methyl-tert-butyl ether for high-throughput lipidomics., 2008, 49(5): 1137-1146.
[29] MOROS G, CHATZIIOANNOU A C, GIKA H G, RAIKOS N, THEODORIDIS G. Investigation of the derivatization conditions for GC-MS metabolomics of biological samples., 2017, 9(1): 53-65.
[30] 焦宏. 核磁共振技术在代谢组学中的应用. 山西医药杂志, 2011(4): 335-336.
JIAO H. Application of nuclear magnetic resonance technology in metabolomics., 2011(4): 335-336. (in Chinese)
[31] 王超, 涂文志, 王穆, 让蔚清. 代谢组学分析技术及代谢物鉴定. 国际药学研究杂志, 2010, 37(5): 355-360.
WANG C, TU W Z, WANG M, RANG W Q. Metabonomics analytical technologies and metabolite identification., 2010, 37(5): 355-360. (in Chinese)
[32] 夏建飞, 梁琼麟, 胡坪, 王义明, 罗国安. 代谢组学研究策略与方法的新进展. 分析化学, 2009, 37(1): 136-143.
XIA J F, LIAGN Q L, HU P, WANG Y M, LUO G A. recent trends in strategies and methodologies for metabonomics., 2009, 37(1): 136-143. (in Chinese)
[33] 许茜, 王磊, 张杰, 许卉. 基于核磁代谢组学的阿胶原料来源鉴别. 食品科技, 2018, 43(1): 316-319.
XU Q, WANG L, ZHANG J, XU H. Origin identification of Colla Corii Asini based on1H-NMR metabolomics., 2018, 43(1): 316-319. (in Chinese)
[34] BRENNAN L. NMR-based metabolomics: From sample preparation to applications in nutrition research., 2014, 83: 42-49.
[35] SANDUSKY P, APPIAH-AMPONSAH E, RAFTERY D. Use of optimized 1D TOCSY NMR for improved quantitation and metabolomic analysis of biofluids., 2011, 49(3/4): 281-290.
[36] STURM S, SEGER C. Liquid chromatography-nuclear magnetic resonance coupling as alternative to liquid chromatography-mass spectrometry hyphenations: curious option or powerful and complementary routine tool?, 2012, 1259(19): 50-61.
[37] BRAUNBERGER C, ZEHL M, CONRAD J, FISCHER S, ADHAMI H R, BEIFUSS U, KRENN L. LC-NMR, NMR, and LC-MS identification and LC-DAD quantification of flavonoids and ellagic acid derivatives in drosera peltata., 2013, 932(15): 111-116.
[38] GARCIA C J, GARCíA-VILLALBA R, GARRIDO Y, GIL M I, TOMáS-BARBERáN F A. Untargeted metabolomics approach using UPLC-ESI-QTOF-MS to explore the metabolome of fresh-cut iceberg lettuce., 2016, 12(8): 138.
[39] SARABIA L D, BOUGHTON B A, RUPASINGHE T, VAN DE MEENE A M L, CALLAHAN D L, HILL C B, ROESSNER U. High-mass-resolution MALDI mass spectrometry imaging reveals detailed spatial distribution of metabolites and lipids in roots of barley seedlings in response to salinity stress., 2018, 14(5): 1-16.
[40] FARNETI B, KHOMENKO I, CAPPELLIN L, TING V, ROMANO A, BIASIOLI F, COSTA G, COSTA F. Comprehensive VOC profiling of an apple germplasm collection by PTR-ToF-MS., 2015, 11(4): 838-850.
[41] KIM H J, SEO Y T, PARK S-I, JEONG S H, KIM M K, JANG Y P. DART–TOF–MS based metabolomics study for the discrimination analysis of geographical origin ofroots collected from Korea and China., 2015, 11(1): 64-70.
[42] GONG Z G, HU J, WU X, XU Y J. The Recent developments in sample preparation for mass spectrometry-based metabolomics., 2017, 47(4): 1-7.
[43] BEALE D J, PINU F R, KOUREMENOS K A, POOJARY M M, NARAYANA V K, BOUGHTON B A, KANOJIA K, DAYALAN S, JONES O A H, DIAS D A. Review of recent developments in GC-MS approaches to metabolomics-based research., 2018, 14(11): 152.
[44] MARI A, LYON D, FRAGNER L, MONTORO P, PIACENTE S, WIENKOOP S, EGELHOFER V, WECKWERTH W. Phytochemical composition ofL. analyzed by an integrative GC-MS and LC-MS metabolomics platform., 2013, 9(3): 599-607.
[45] SMITH E D, WHITING M D, RUDELL D R. Metabolic profiling of ethephon-treated sweet cherry (L.)., 2011, 7(1): 126-133.
[46] WONG Y F, PERLMUTTER P, MARRIOTT P J. Untargeted metabolic profiling ofspp. leaf oils using comprehensive two-dimensional gas chromatography with high resolution mass spectrometry: Expanding the metabolic coverage., 2017, 13(5): 46.
[47] TOFFALI K, ZAMBONI A, ANESI A, STOCCHERO M, PEZZOTTI M, LEVI M, GUZZO F. Novel aspects of grape berry ripening and post-harvest withering revealed by untargeted LC-ESI-MS metabolomics analysis., 2011, 7(3): 424-436.
[48] JOHNSON A E, SIDWICK K L, PIRGOZLIEV V R, EDGE A, THOMPSON D F. Metabonomic profiling of chicken eggs during storage using high-performance liquid chromatography–quadrupole time-of-flight mass spectrometry., 2018, 90(12): 7489-7494.
[49] MOLINA-CALLE M, SáNCHEZ DE MEDINA V, CALDERóN- SANTIAGO M, PRIEGO-CAPOTE F, LUQUE DE CASTRO M D. Untargeted analysis to monitor metabolic changes of garlic along heat treatment by LC-QTOF MS/MS., 2017, 38(18): 2349-2360.
[50] IBáñEZ C, SIMó C, GARCíA-CAñAS V, CIFUENTES A, CASTRO- PUYANA M. Metabolomics, peptidomics and proteomics applications of capillary electrophoresis-mass spectrometry in foodomics: A review., 2013, 802: 1-13.
[51] BAN E, PARK S H, KANG M J, LEE H J, SONG E J, YOO Y S. Growing trend of CE at the omics level: The frontier of systems biology-an update., 2012, 33(1): 2-13.
[52] LAN K, ZHANG Y, YANG J Y, XU L. Simple quality assessment approach for herbal extracts using high performance liquid chromatography-UV based metabolomics platform., 2010, 1217(8): 1414-1418.
[53] FIEHN O. Metabolomics--the link between genotypes and phenotypes., 2002, 48(1/2): 155-171.
[54] 徐淑玲, 魏芳, 董绪燕, 陈洪. 脂质组学在脂质膳食营养与健康研究中的应用. 中国食物与营养, 2017, 23(11): 5-10.
XU S L, WEI F, DONG X Y, CHEN H. The Application of lipidomics in lipids dietary nutrition and health research., 2017, 23(11): 5-10. (in Chinese)
[55] GIKA H G, THEODORIDIS G A, PLUMB R S, WILSON I D. Current practice of liquid chromatography-mass spectrometry in metabolomics and metabonomics., 2014, 87: 12-25.
[56] GUO Q, WU W, MASSART D L, BOUCON C, JONG S D. Feature selection in principal component analysis of analytical data., 2002, 61(1): 123-132.
[57] ARROIO A, HONóRIO K M, SILVA A B F D. A theoretical study on the analgesic activity of cannabinoid compounds., 2004, 709(1): 223-229.
[58] BOULESTEIX A L, STRIMMER K. Partial least squares: a versatile tool for the analysis of high-dimensional genomic data., 2007, 8(1): 32-44.
[59] DAYGON V D, PRAKASH S, CALINGACION M, RIEDEL A, OVENDEN B, SNELL P, MITCHELL J, FITZGERALD M. Understanding the Jasmine phenotype of rice through metabolite profiling and sensory evaluation., 2016, 12(4): 63.
[60] CAI Y, WENG K, GUO Y, PENG J, ZHU Z-J. An integrated targeted metabolomic platform for high-throughput metabolite profiling and automated data processing., 2015, 11(6): 1575-1586.
[61] RAFIEI A, SLENO L. Comparison of peak-picking workflows for untargeted liquid chromatography/high-resolution mass spectrometry metabolomics data analysis., 2015, 29(1): 119-127.
[62] 王昕璐. 基于脂质组学的PCB153和PCB95对PC12细胞联合毒性效应研究[D]. 中国农业科学院, 2018.
WANG X L. PCB153 and PCB95 in combined exposure modulate PC12 cells as defined by targeted lipidomics analysis [D]. Chinese Academy of Agricultural Sciences, 2018. (in Chinese)
[63] WISHART D S, JEWISON T, GUO A C, WILSON M, KNOX C, LIU Y, DJOUMBOU Y, MANDAL R, AZIAT F, DONG E. HMDB 3.0-The human metabolome database in 2013., 2013, 41: 801-807.
[64] SMITH C A. METLIN: A metabolite mass spectral database., 2005, 27(6): 747-751.
[65] PAUPIèRE M J, MüLLER F, LI H, RIEU I, TIKUNOV Y M, VISSER R G F, BOVY A G. Untargeted metabolomic analysis of tomato pollen development and heat stress response., 2017, 30(2): 81-94.
[66] WANG L, YE H, SUN D, MENG T, CAO L J, WU M Q, ZHAO M, WANG Y, CHEN B Q, XU X W, WANG G J, HAO H P. Metabolic pathway extension approach for metabolomic biomarker identification., 2017, 89(2): 1229-1237.
[67] ZHU G T, WANG S C, HUANG Z J, ZHANG S B, LIAO QBG, ZHANG C Z, LIN T, QIN M, PENG M, YANG C K, CAO X, HAN X, WANG X X, VAN DER KNAAP E, ZHANG Z H, CUI X, KLEE H, FERNIE A R, LUO J, HUANG S W. Rewiring of the fruit metabolome in tomato breeding., 2018, 172(1/2): 6-8.
[68] BöTTCHER C, KRäHMER A, STüRTZ M, WIDDER S, SCHULZ H. Comprehensive metabolite profiling of onion bulbs () using liquid chromatography coupled with electrospray ionization quadrupole time-of-flight mass spectrometry., 2017, 13(4): 35.
[69] NARDUZZI L, STANSTRUP J, MATTIVI F. Comparing wild American grapes with: A metabolomics study of grape composition., 2015, 63(30): 6823-6834.
[70] ZOTTI M, COCO L D, PASCALI S A D, MIGONI D, VIZZINI S, MANCINELLI G, FANIZZI F P. Comparative analysis of the proximate and elemental composition of the blue crab callinectes sapidus, the warty crab eriphia verrucosa, and the edible crab cancer pagurus., 2016, 2(2): e00075.
[71] CORTINA P R, SANTIAGO A N, SANCE M M, PERALTA I E, CARRARI F, ASIS R. Neuronal network analyses reveal novel associations between volatile organic compounds and sensory properties of tomato fruits., 2018, 14(5): 57.
[72] FUKUDA T, OKAZAKI K, WATANABE A, SHINANO T, OKA N. GC–MS based metabolite profiling for flavor characterization of brassica crops grown with different fertilizer application., 2015, 12(2): 20.
[73] BROWN P N, MURCH S J, SHIPLEY P. Phytochemical diversity of cranberry (Aiton) cultivars by anthocyanin determination and metabolomic profiling with chemometric analysis., 2012, 60(1): 261-271.
[74] CARVALHO E, FRANCESCHI P, FELLER A, HERRERA L, PALMIERI L, ARAPITSAS P, RICCADONNA S, MARTENS S. Discovery of A-type procyanidin dimers in yellow raspberries by untargeted metabolomics and correlation based data analysis., 2016, 12(9): 144.
[75] INOSTROZA-BLANCHETEAU C, DE OLIVEIRA SILVA F M, DURáN F, SOLANO J, OBATA T, MACHADO M, FERNIE A R, REYES-DíAZ M, NUNES-NESI A. Metabolic diversity in tuber tissues of native Chiloé potatoes and commercial cultivars ofssp.L., 2018, 14(10): 138.
[76] MOUNET F, LEMAIRE-CHAMLEY M, MAUCOURT M, CABASSON C, GIRAUDEL J-L, DEBORDE C, LESSIRE R, GALLUSCI P, BERTRAND A, GAUDILLèRE M, ROTHAN C, ROLIN D, MOING A. Quantitative metabolic profiles of tomato flesh and seeds during fruit development: complementary analysis with ANN and PCA., 2007, 3(3): 273-288.
[77] 张晓磊, 张瑞英. 代谢组学及其在农作物研究中的应用. 生物技术通讯, 2018, 29(3): 446-450.
ZHAGN X L, ZHANG R Y. Metabolomics and its application in the crop research., 2018, 29(3): 446-450. (in Chinese)
[78] RAO J, YANG L T, GUO J C, QUAN S, CHEN G H, ZHAO X X, ZHANG D B, SHI J X. Metabolic changes in transgenic maize mature seeds over-expressing the., 2016, 35(2): 429-437.
[79] LE GALL G, COLQUHOUN I J, DAVIS A L, COLLINS G J, VERHOEYEN M E. Metabolite profiling of tomato () using1H NMR spectroscopy as a tool to detect potential unintended effects following a genetic modification., 2003, 51(9): 2447-2456.
[80] LI Y, PANG T, LI Y, WANG X L, LI Q H, LU X, XU G W. Gas chromatography-mass spectrometric method for metabolic profiling of tobacco leaves., 2011, 34(12): 1447-1454.
[81] KIM J, JUNG Y, SONG B, BONG Y S, RYU D H, LEE K S, HWANG G S. Discrimination of cabbage (ssp.) cultivars grown in different geographical areas using1H NMR-based metabolomics., 2013, 137(1): 68-75.
[82] BONDIA-PONS I, SAVOLAINEN O, TöRRöNEN R, MARTINEZ J A, POUTANEN K, HANHINEVA K. Metabolic profiling of Goji berry extracts for discrimination of geographical origin by non- targeted liquid chromatography coupled to quadrupole time-of-flight mass spectrometry., 2014, 63(Part B): 132-138.
[83] HRBEK V, REKTORISOVA M, CHMELAROVA H, OVESNA J, HAJSLOVA J. Authenticity assessment of garlic using a metabolomic approach based on high resolution mass spectrometry., 2018, 67: 19-28.
[84] RITOTA M, CASCIANI L, HAN B Z, COZZOLINO S, LEITA L, SEQUI P, VALENTINI M. Traceability of Italian garlic (L.) by means of HRMAS-NMR spectroscopy and multivariate data analysis., 2012, 135(2): 684-693.
[85] CAMARGO A B, RESNIZKY S, MARCHEVSKY E J, LUCO J M. Use of the Argentinean garlic (L.) germplasm mineral profile for determining geographic origin., 2010, 23(6): 586-591.
[86] VADALà R, MOTTESE A F, BUA G D, SALVO A, MALLAMACE D, CORSARO C, VASI S, GIOFRè S V, ALFA M, CICERO N, DUGO G. Statistical analysis of mineral concentration for the geographic identification of garlic samples from sicily (Italy), Tunisia and Spain., 2016, 5(1): 20.
[87] SMITH R G. Determination of the country of origin of garlic () using trace metal profiling., 2005, 53(10): 4041-4045.
[88] DU H Y, FU J L, WANG S Q, LIU H L, ZENG Y C, YANG J R, XIONG S B.1H-NMR metabolomics analysis of nutritional components from two kinds of freshwater fish brain extracts., 2018, 8(35): 19470-19478.
[89] 陈羽红, 张东杰, 张桂芳, 王颖, 王长远. 代谢组学技术在食品产地溯源中的研究进展. 粮食与饲料工业, 2016, 12(7): 16-19.
CHEN Y H, ZHANG D J, ZHANG G F, WANG Y, WAGN C Y. Review on metabonomics techniques in food origin traceability., 2016, 12(7): 16-19. (in Chinese)
[90] KLOCKMANN S, REINER E, BACHMANN R, HACKL T, FISCHER M. Food fingerprinting: metabolomic approaches for geographical origin discrimination of hazelnuts () by UPLC-QTOF-MS., 2016, 64(48): 9253-9262.
[91] KLOCKMANN S, REINER E, CAIN N, FISCHER M. Food targeting: Geographical origin determination of hazelnuts () by LC-QqQ-MS/MS-based targeted metabolomics application., 2017, 65(7): 1456-1465.
[92] ZHANG J J, WANG X, YU O, TANG J J, GU X G, WAN X C, FANG C B. Metabolic profiling of strawberry (×Duch.) during fruit development and maturation., 2011, 62(3): 1103-1118.
[93] CUADROS-INOSTROZA A, RUíZ-LARA S, GONZáLEZ E, ECKARDT A, WILLMITZER L, PEñA-CORTéS H. GC-MS metabolic profiling of Cabernet Sauvignon and Merlot cultivars during grapevine berry development and network analysis reveals a stage- and cultivar-dependent connectivity of primary metabolites., 2016, 12(2): 39.
[94] OMS-OLIU G, HERTOG M L A T M, VAN DE POEL B, AMPOFO- ASIAMA J, GEERAERD A H, NICOLAï B M. Metabolic characterizationof tomato fruit during preharvest development, ripening, and postharvest shelf-life., 2011, 62(1): 7-16.
[95] MARTINS N, PETROPOULOS S, FERREIRA I C F R. Chemical composition and bioactive compounds of garlic (L.) as affected by pre- and post-harvest conditions: A review., 2016, 211: 41-50.
[96] DENG J C, YANG C Q, ZHANG J, ZHANG Q, YANG F, YANG W Y, LIU J. Organ-specific differential NMR-based metabonomic analysis of soybean [(L.) Merr.] fruit reveals the metabolic shifts and potential protection mechanisms involved in field mold infection., 2017, 8(508).
[97] 许腾, 张玥, 张海丽, 辛凤姣, 王艳, 王凤忠. 代谢组学技术在营养学研究中的应用. 中国食物与营养, 2017, 23(11): 11-16.
XU T, ZHANG Y, ZHAGN H L, XIN F J, WANG Y, WANG F Z. Applications of metabonomics in nutriology research., 2017, 23(11): 11-16. (in Chinese)
[98] 张双庆, 黄振武. 营养代谢组学技术在营养学研究中的应用. 卫生研究, 2013, 42(6): 1041-1046.
ZHAGN S Q, HUANG Z W. Application of nutritional metabolomics technology in nutrition research., 2013, 42(6): 1041-1046. (in Chinese)
[99] 何庆华, 任萍萍, 王玉兰. 代谢组学在营养学研究中的应用. 食品科学, 2011, 32(5): 317-320.
HE Q H, REN P P, WANG Y L. Applications of metabolomics in nutrition research: A review., 2011, 32(5): 317-320. (in Chinese)
[100] LIU C T, RAGHU R, LIN S H, WANG S Y, KUO C H, TSENG Y F J, SHEEN L Y. Metabolomics of ginger essential oil against alcoholic fatty liver in mice., 2013, 61(46): 11231-11240.
[101] 唐华丽, 夏惠, 王锋, 孙桂菊. 枸杞多糖作用于2型糖尿病大鼠的血清代谢组学研究. 食品科学, 2017, 38(13): 160-166.
TANG H L, XIA H, WANG F, SUN G J. Serum metabonomics study of type 2 diabetic rats administrated with lycium barbarum polysaccharides., 2017, 38(13): 160-166. (in Chinese)
[102] FUJIMURA Y, KURIHARA K, IDA M, KOSAKA R, MIURA D, WARIISHI H, MAEDA-YAMAMOTO M, NESUMI A, SAITO T, KANDA T, YAMADA K, TACHIBANA H. Metabolomics-driven nutraceutical evaluation of diverse green tea cultivars,. 2011, 6(8): e23426.
[103] ACCARDI C J, WALKER D I, UPPAL K, QUYYUMI A A, ROHRBECK P, PENNELL K D, MALLON C T, JONES D P. High-resolution metabolomics for nutrition and health assessment of armed forces personnel., 2016, 58(8S Suppl 1): S80-S88.
[104] RAI A, SAITO K, YAMAZAKI M. Integrated omics analysis of specialized metabolism in medicinal plants., 2017, 90(4): 764-787.
[105] 侯绍英, 黄放放, 刘鑫妍, 彭雪. 基于UPLC-MS技术的人体内芒果苷代谢组学研究. 哈尔滨医科大学学报, 2016, 50(4): 315-318.
HOU S Y, HUANG F F, LIU X Y, PENG X. Metabolomics study of mangiferin in human based on UPLC-MS., 2016, 50(4): 315-318. (in Chinese)
[106] LLORACH R, URPI-SARDA M, TULIPANI S, GARCIA-ALOY M, MONAGAS M, ANDRES-LACUEVA C. Metabolomic fingerprint in patients at high risk of cardiovascular disease by cocoa intervention., 2013, 57(6): 962-973.
[107] 边会喜. 基于代谢组学与多光谱成像技术对苦瓜影响高脂饮食肥胖小鼠能量代谢的有效组分的研究[D]. 合肥: 合肥工业大学, 2016.
BIAN H X. The energy metabolism study of effective componets from bitter melon (Momordica charantia) on DIO mice based on metabolomics and multispectral imaging techonogy [D]. Hefei: Hefei University of Technology, 2016. (in Chinese)
[108] TAKIS P G, ORAIOPOULOU M E, KONIDARIS C, TROGANIS A N. (1)H-NMR based metabolomics study for the detection of the human urine metabolic profile effects oftea ingestion., 2016, 7(9): 4104-4115.
[109] STELLA C, BECKWITH-HALL B, CLOAREC O, HOLMES E, LINDON J C, POWELL J, VAN DER OUDERAA F, BINGHAM S, CROSS A J, NICHOLSON J K. Susceptibility of human metabolic phenotypes to dietary modulation., 2006, 5(10): 2780-2788.
Review on the Application of Metabolomic Approaches to Investigate and Analysis the Nutrition and Quality of Agro-products
XU YanYang1, YAO GuiXiao1, 3, LIU PingXiang1, ZHAO Jie1, WANG XinLu1, SUN JunMao2, QIAN YongZhong1
(1Institute of Quality Standards and Testing Technology for Agro-Products, Chinese Academy of Agricultural Sciences/Key Laboratory of Agro-Product Quality and Safety, Ministry of Agriculture and Rural Affairs, Beijing 100081;2Institute of Food and Nutrition Development, Ministry of Agriculture and Rural Affairs, Beijing 100081;3Xi’an University of Technology, Xi’an 710048)
Scientific evaluation of the nutrition and quality of agricultural products is essential for improving the nutrition level of agro-products. Because of the complex composition of nutrients in agro-products, the existing analytical methods can only analyze the concentration and function of known nutrients but cannot analyze and identify a large number of unknown functional substances. On the basis of high-throughput chemical analyses, metabolomics can qualitatively and quantitatively analyze endogenous and exogenous metabolites of biological samples. Therefore, metabolomics has outstanding advantages in the analysis of small molecular substances with special nutritional functions in agricultural products; it has advantages like providing new methods for the characterization and differential analysis of nutrient components, traceability and authenticity of identification, variation analysis of functional substances during growth and storage, and the effect mechanisms of functional components. It also provides new strategies for structural optimization of dietary requirements. In this paper, the recent advances in metabolomics research, including sample preparation, metabolite analysis, data processing, differential metabolite identification, and metabolic pathway analysis were reviewed. This work summed up the application of metabolomics in the characterization and difference analysis of metabolites, traceability and authenticity identification of origin, metabolite variation in the process of storage, and the evaluation of nutritional functions to provide theoretical bases and practical references for high-quality agricultural development in China. In the field of sample preparation, the activity of metabolism-related enzymes is first terminated by rapidly changing the environmental conditions, such as adding strong acid (alkali) or freezing in liquid nitrogen. Different extraction solvents are selected based on the polarities of the metabolites to obtain a higher extraction rate. In the field of sample analysis methods, technologies, such as nuclear magnetic resonance spectroscopy, chromatography mass spectrometry and capillary electrophoresis-mass spectrometry, have been widely used. Among them, the combination of chromatography and mass spectrometry has become the most commonly used analytical technique in metabolomics. In the field of data processing and analysis, principal component analysis and orthogonal partial least squares-discriminant analysis are the most common data analysis techniques. Through enrichment and topological analysis, the metabolic pathway with the highest correlation to differential metabolites can be identified, and the reason of differential metabolites can be explained and analyzed. In the field of evaluation of nutrition and quality of agricultural products, through the comprehensive characterization of primary metabolites and secondary metabolites in agricultural products, unique fingerprints of agricultural products are established and used for differential analysis, whereas through non-specific target analysis and unsupervised analysis methods, differences between groups and relating metabolites can be identified. Via concentration analysis of key components in the growth process of agricultural products, the best harvest periods can be provided. Interaction studies between functional components and metabolism of organisms based on the detection of humoral metabolism and biomarkers can provide valuable information for dietary guidance.
metabolomics; agro-product; nutrition; quality
10.3864/j.issn.0578-1752.2019.18.009

2019-02-22;
2019-04-23
国家自然科学基金(31701519)、科技部食品安全关键技术研发专项(2017YFC1600705)
许彦阳,Tel:010-82106539;E-mail:xuyanyang@caas.cn。
钱永忠,Tel:010-82106298;E-mail:qianyongzhong@caas.cn。通信作者孙君茂,Tel:010-82109887;E-mail:sunjunmao@caas.cn
(责任编辑 赵伶俐)
