植物染色质重塑复合体研究进展
2019-08-27郭明欣何亚莉许可可赵旭升
郭明欣 何亚莉 许可可 赵旭升
摘要 染色质重塑复合体在基因的转录调控方面发挥重要作用。综述了近年来国内外学者在染色质重塑复合体的组成、结构、分类及功能分析方面的最新研究概况。重点介绍了植物领域染色质重塑复合体核心亚基的研究进展,并分析了目前影响该领域发展的原因及解决对策,同时还对应用前景进行了展望。
关键词 染色质重塑复合体;转录调控;核心亚基
中图分类号 Q943文献标识码 A
文章编号 0517-6611(2019)13-0016-04
doi:10.3969/j.issn.0517-6611.2019.13.005
开放科学(资源服务)标识码(OSID):
Research Advance on Chromatin Remodeling Complex in Plants
Abstract Chromatin remodeling complex play a vital role in transcriptional regulation. In this paper, we reviewed the components, structure, classification, functional analysis of chromatin remodeling complex. Furthermore, we focus on the core subunits of chromatin remodeling in plants. Moreover, the new trends and prospects of chromatin remodeling complex were also discussed.
Key words Chromatin remodeling complex;Transcriptional regulation;Core subunits
染色质的基本结构单元是核小体,核小体包括4种组蛋白即H2A、H2B、H3、H4,每一种组蛋白各有2个分子,形成为一个组蛋白八聚体。146 bp的DNA围绕组蛋白八聚体形成核小体的核心结构。连接核小体之间的DNA链上由组蛋白H1 结合,由此形成了11 nm的核小体串珠状结构。核小体经过进一步的折叠及组装形成染色质的高级结构[1]。染色质的折叠和包装使染色质可以合适的大小存在于真核细胞核里。然而,染色质的高度紧密使得一些基因的正常转录受到抑制。现在已知有2类染色质的修饰复合物:一类是ATP依赖的染色质重塑复合体(ATP-dependent chromatin remodeling complex),其依靠核心亚基ATPase水解ATP为整个复合物提供能量。该复合体可以移动核小体的位置,改变DNA和组蛋白之间的互作,使转录起始因子可以结合在DNA上[2-4]。另一类是染色质的共价化学修饰,一般是对组蛋白末端“尾巴”进行乙酰化、磷酸化、甲基化和泛素化等修饰[5]。已有的研究表明,染色质重塑复合体通过调控相关基因的表达参与了植物的生长发育以及对外界逆境响应等众多过程。目前对于染色质重塑复合体的研究已逐渐成为研究基因表达调控的热点。基于此,笔者综述了染色质重塑复合体的组成、结构、分类以及功能,为后续的研究提供参考,并在核心亚基的功能解析方面进行了展望。
1 染色质重塑复合体的组成和结构
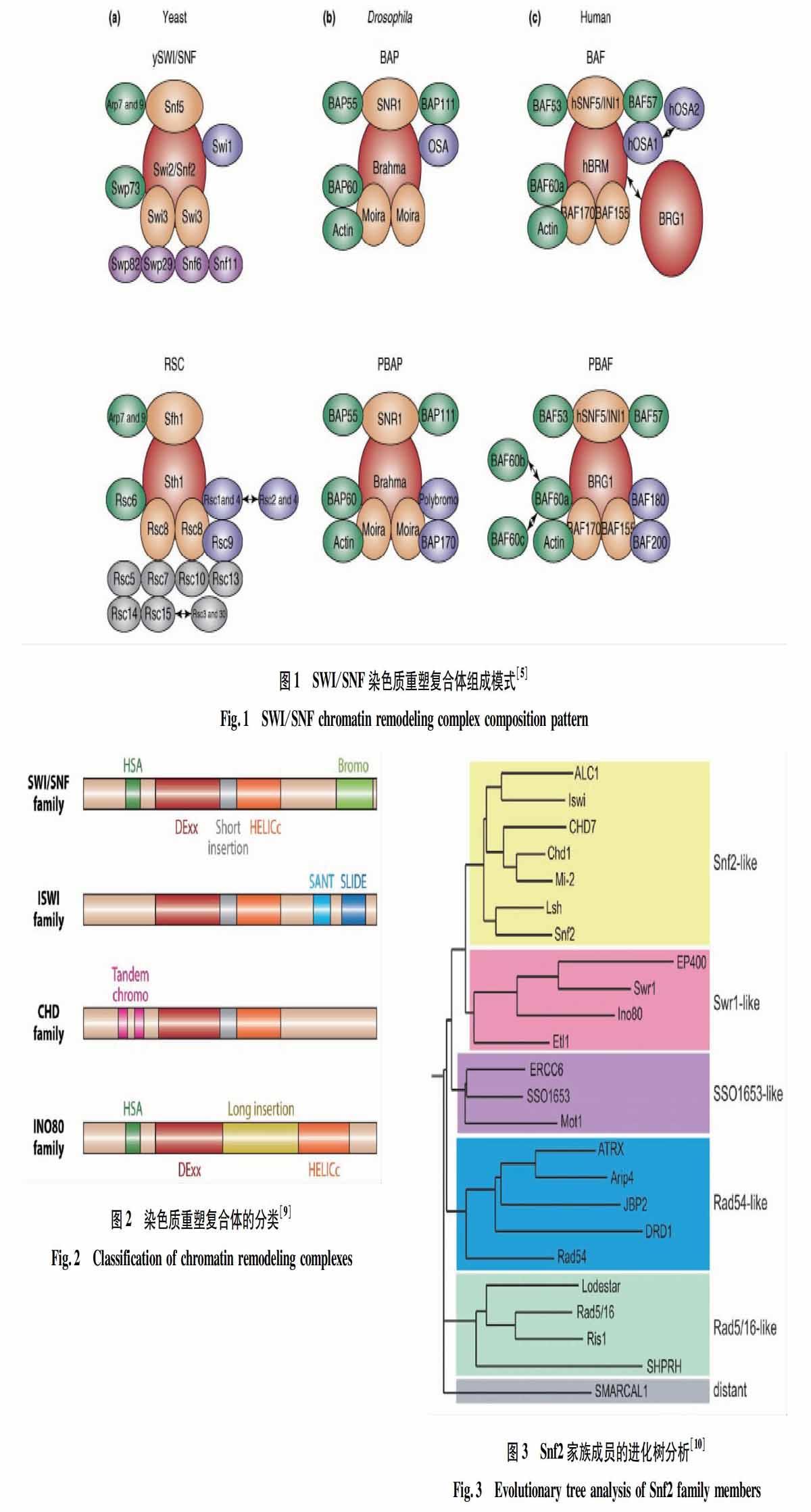
ATP 依赖的染色质重塑复合体是由多个亚基组成的复合体。核心亚基是属于 Snf2家族的ATPase。由于复合体有很多的亚基组成,而且有些亚基分子量很小。另外,有些核心亚基,比如酵母的 Sth1、Isw1 等参与了几个结构相似、功能不同的重塑复合体[6-7],因此直接纯化染色质重塑复合体变得非常困难,也阻碍了对该类复合体功能的深入研究。目前,利用低分辨率的电子显微镜(low resolution electron microscopy)已经确定了少数染色质重塑复合体的组成。如图1所示[5],复合体的结构像一个大的碗,碗的中央部位收缩以使复合体能够结合在核小体上[8]。
2 染色质重塑复合体的分类
由于很多染色质重塑复合体的组成和生化功能是未知的,因此很难根据生化功能对其进行分类。传统的分类方法,是根据Snf2蛋白所包含的2个主要功能域DEXDc和HELICc之间插入氨基酸的长度,把染色质重塑复合体分为4类,包括SWI/SNF、 Mi-2/CHD、ISWI、INO80/SWR1(图2)[9]。这是一种过于简化的、比较模糊的分类方法。
Snf2家族的蛋白都含有DEXDc和HELICc这2个结构域(Domain),这2个结构域组合在一起发挥ATPase的作用。Flaus等[10]利用来自不同物种的1 300个Snf2家族的蛋白,通过对蛋白的DEXDc和HELICc所包含的及2个功能域之间的氨基酸序列进行多重序列比对建立了进化树,把Snf2家族分为5个类群(group)、23 个亚家族(subfamily)(图3)。
3 染色质重塑复合体的功能
目前对染色质重塑复合体的功能研究主要集中在動物和微生物领域,比如人类、斑马鱼、果蝇、小鼠、或线虫。在模式植物拟南芥、水稻等有少量的相关研究报道,但总体研究进展较其他领域远为落后。
染色质重塑复合体在生物体中的功能主要体现在3个方面。第一,调节基因的转录,起始或抑制基因的转录。大部分ATP依赖的染色质重塑复合体能够改变染色质结构激活基因的转录[11-14],但是Mi-2/NuRD亚家族的染色质重塑复合体却抑制基因的转录。Mi-2/NuRD不仅具有改变核小体位置的功能,还具有组蛋白去乙酰化的功能,使处于转录激活状态的基因中的组蛋白发生去乙酰化,从而导致转录受到抑制[15]。第二,染色质重塑复合体参与DNA修复和减数分裂时姐妹染色体的联会和分离。酵母的RSC、SWI/SNF 和拟南芥的BRM参与了DNA的损伤修复[16-17];酵母的RSC参与了减数分裂时姐妹染色体的联会和分离[18]。第三,当生物面对内在或外在条件改变时,需要染色质重塑复合体来完成转录重组(transcriptional reprogramming)。
酵母中的SWI/SNF是第一个被发现的染色质重塑复合体,其核心亚基Swi2/Snf2的相关突变体是通过2个独立的遗传途径筛选到的。swi2突变体表现交配缺陷的表型(mating type switching defective phenotypes);而snf2突变体表现蔗糖不发酵的表型(sucrose nonfermenting phenotypes),2个突变体是酵母中同一个基因 YOR290C突变所致。后续研究表明,Swi2/Snf2与其他9个亚基组成一个大的复合体发挥作用(图1)[5]。之后,又陆续在酵母、人类、果蝇等生物体中发现了一些染色质重塑复合体,如酵母的RSC、ISW1a、ISW1b、ISW2、CHD1、INO80、SWR1[10,19-20];人类的BAF、PBAF、NURF、CHRAC、ACF、CHD1、NuRD、INO80、SRCAP、TRRAP[10];果蝇的BAP、PBAP、NURF、CHRAC、ACF、CHD1、Mi-2、Tip60、Pho-dINO80[10]。这些染色质重塑复合体参与了生物体的很多过程,包括交配、合子分裂及胚胎的早期发育和器官的发生(神经系统、心脏系统、表皮系统、肌肉系统等[5,9]。
在植物中,到目前为止还没有一个完整的染色质重塑复合体的组成被发现。但是有一些核心亚基的功能已经被研究,比如拟南芥的AtBRM[21-24]、AtSYD[25-28]、AtCHR12[29-30]、AtCHR23[29],以及水稻的OsCHR4[31]、OsALT1[32]。这些核心亚基参与了植物的很多发育过程,包括营养生长、生殖生长以及植物对生物和非生物胁迫的响应。
3.1 AtBRM
AtBRM是果蝇DmBRM的同源基因。DmBRM是Snf2类型的染色质重塑复合体BAP、PBAP的核心亚基。AtBRM编码的蛋白由 2 193个氨基酸组成,含有 DEXDc、HELICc、BROMO 3个功能域。对AtBRM的功能研究表明,AtBRM调控着拟南芥的营养生长、生殖生长及对非生物胁迫的响应。AtBRM的RNAi转基因植株表现为株高变矮、叶片卷曲、叶片与花序分生组织变小、开花期提前,并导致了花器官形态的改变如花瓣和雄蕊变小、花药不能成熟、结实率降低[21]。brm突变体则表现了更严重的发育缺陷,纯合突变体完全不育[22]。
合成和积累种子储藏蛋白(seed storage proteins)是种子成熟过程中的一个重要方面。编码种子储藏蛋白相关的基因主要在成熟的种子中表达,而在营养器官如叶片中没有转录产物。但是,在brm突变体的叶片中,能够检测到与种子储藏蛋白相关基因如At2S2、At2S3、At2S5、At7S1等的表达[23]。进一步研究表明,AtBRM与AtSWI3C呈现较强互作,与AtSWI3B微弱互作,而与AtSWI3A和AtSWI3D没有互作[22]。而且,swi3c突变体与brm有相似的表型,而且在swi3c突变体叶片中At2S2、At2S3、At7S1等基因的表达明显上调。SWI/SNF复合体的组成亚基Snf5在拟南芥中的只有一个同源基因AtBSH,bsh突变体与brm有相似的表型,At7S1的表达在bsh突变体叶片中也明显上调[23]。另外,brm、swi3c、bsh突变体在苗期对植物激素ABA都表现高度敏感,其中AtBRM的功能丧失导致AtABI5的表达上调,使brm突变体的耐旱能力明显增强[24]。
3.2 AtSYD
AtSYD 是Snf2类型的染色质重塑复合体的核心亚基,编码的蛋白由3 583个氨基酸组成,含有DEXDc和HELICc 2个结构域。对AtSYD的功能研究表明,AtSYD 调控着拟南芥全生育期的生长发育以及对生物胁迫的响应,该基因的功能丧失造成syd突变体株高变矮、生长缓慢、叶片极性缺失、次级花序减少。同时,AtSYD与AtLFY共同调控茎端分生组织(shoot apical meristem,SAM)的识别[25],并通过直接调控WUS的表达而影响顶端分生组织的大小[26]。
植物花器官的发育由A、B、C、D、E这5类基因控制,其中B、C类基因控制着雄性和雌性生殖器官的发育,因而显得尤为重要。在拟南芥中存在一类多梳蛋白,它们会抑制B、C类基因的表达。最近的研究表明,AtSYD和AtBRM能够克服多梳蛋白的抑制而激活B类基因AP3和C类基因AG的表达,而且AtBRM和AtSYD可以直接分别结合在AP3和AG的启动子区域[27]。
另外,AtSYD 还参与了生物胁迫的信号转导。AtSYD调控乙烯(ethylene)和茉莉酸(jasmonic acid,JA)信号通路中一些基因如PDF1.2a、MYC2、VSP2的表达。在syd突变体中,PDF1.2a、MYC2、VSP2的表达明显下调。与野生型相比,syd 对真菌病原物番茄灰霉病菌(Botrytis cinerea)敏感,但是对细菌病原物假单胞杆菌(Pseudomonas syringae)表现不敏感,表明AtSYD对病原菌具有选择抗性[28]。
3.3 AtCHR12、AtCHR23
AtCHR12、AtCHR23又分别叫做MINU1、MINU2。AtCHR12编码的蛋白由1 102 个氨基酸组成,而AtCHR23编码的蛋白由1 064个氨基酸组成。AtCHR12与AtCHR23都含有DEXDc和HELICc这2个结构域,具有81.7% 的同源性。由此,AtCHR12和AtCHR23表现功能冗余,单个基因功能丧失的chr12或chr23突变体在表型上没有明显变化。与此相比,chr12、chr23双突变体则表现为株高变矮、根系变短、生长缓慢、生育期延长、花药数目减少、结实率降低[29]。
AtCHR12负向调控拟南芥对非生物胁迫的耐性。在正常生长条件下,chr12突变体的表型与野生型没有差异,但在干旱、高温、盐胁迫的情况下,突变体则表现了明显的耐性。在干旱和高温胁迫的情况下,chr12的株高高于野生型,而过量表达该基因的转基因植株的株高则较野生型降低。另外,在25、50 mM NaCl胁迫时,chr12突變体的根长较野生型明显增加[30]。
3.4 OsCHR4
OsCHR4屬于Mi-2亚家族的核心亚基,编码的蛋白由2 259个氨基酸组成,含有1个PHD功能域、2个CHROMO功能域、1个DEXDc功能域和1个HELICc功能域。OsCHR4在水稻近轴段叶肉细胞叶绿体的发育中发挥重要作用。chr4突变体叶片的远轴端生长正常,而近轴端叶色变黄。进一步的研究表明,chr4突变体叶片近轴端叶肉细胞的叶绿体存在缺陷,其质体的生长和类囊体膜的形成被阻碍。OsCHR4定位在细胞核内,其转录产物主要分布在根端分生组织、花、维管束以及叶肉细胞中[31]。
3.5 OsALT1
OsALT1属于推测的Snf2家族的核心亚基,编码的蛋白由1 228个氨基酸组成,含有1个DEXDc功能域、1个HELICc功能域和1个RING finger结构域。OsALT1在水稻苗期的耐碱性及生长发育方面发挥重要的调节作用。alt1突变体在苗期表现非常明显的耐碱性,同时,在正常栽培条件下,alt1突变体的最典型特征是根系较短,同时地上部有一定程度的矮化。
系统进化分析结果表明,ALT1属于Snf2家族中Ris1亚家族。亚细胞定位显示ALT1是一个核定位蛋白。GUS染色和定量RT-PCR结果显示,ALT1在根、茎、叶片、叶鞘、幼穗中均有表达。与ALT1的耐碱负向调节功能相一致,ALT1的表达受到碱胁迫显著抑制。对ALT1耐碱机理的研究表明,ALT1主要通过调控氧化胁迫的损伤防御在水稻耐碱中发挥作用,揭示了植物耐碱的一个新的分子机制[32]。
4 展望
经过10多年的研究,生物中染色质重塑复合体的研究取得了较大的进展,部分染色质重塑复合体的结构、组成及核心亚基和附属亚基的功能被解析,其作用机制也越来越清楚。然而在植物领域,染色质重塑复合体的研究却相对缓慢,以模式植物拟南芥和水稻为例,拟南芥基因组中有41个Snf2家族的成员,目前仅有4个成员的功能被解析;水稻基因组中有42个Snf2家族的成员,目前仅有2个成员的功能被解析。导致这种现象的原因,一方面可能是Snf2家族基因内部存在功能冗余的现象,比如拟南芥AtCHR12和AtCHR23,因此不太容易筛选到表型发生明显改变的突变体;另一方面,Snf2家族成员编码的蛋白分子量均较大(大部分基因编码的蛋白长度在1 000 aa以上),这在一定程度上限制了通过反向遗传学的手段研究基因的功能。
近年来,基因组编辑(Genome editing)技术的出现,特别是CRISPR/Cas9技术在基因敲除中的广泛应用,为研究Snf2家族的基因功能提供了一个非常好的途径。CRISPR/Cas9实现了同时对多个基因的敲除[33],对于研究存在功能冗余的基因家族非常有用。此外,随着部分Snf2家族耐逆功能的解析[32,34],应用这些基因进行分子改良与新品种选育,将会是一个很好的研究方向。
参考文献
[1] TREMETHICK D J.Higherorder structures of chromatin:The elusive 30 nm fiber[J].Cell,2007,128(4):651-654.
[2] HAVAS K,WHITEHOUSE I,OWENHUGHES T.ATPdependent chromatin remodeling activities[J].Cellular and molecular life sciences,2001,58(5/6):673-682.
[3] HARGREAVES D C,CRABTREE G R.ATPdependent chromatin remodeling:Genetics,genomics and mechanisms[J].Cell research,2011,21(3):396-420.
[4] CLAPIER C R,CAIRNS B R.The biology of chromatin remodeling complexes[J].Annual review of biochemistry,2009,78:273-304.
[5] KWON C S,WAGNER D.Unwinding chromatin for development and growth:A few genes at a time[J].Trends genetics,2007,23(8):403-412.
[6] CAIRNS B R,SCHLICHTER A,ERDJUMENTBROMAGE H,et al.Two functionally distinct forms of the RSC nucleosomeremodeling complex,containing essential AT hook,BAH and bromodomains[J].Molecular cell,1999,4(5):715-723.
[7] VARY J C,GANGARAJU V K,QIN J,et al.Yeast Isw1p forms two separable complexes in vivo[J].Molecular cellular biology,2003,23(1):80-91.
[8] HOTA S K,BARTHOLOMEW B.Diversity of operation in ATPdependent chromatin remodelers[J].Biochimica biophysica acta,2011,1809(9):476-487.
[9] CLAPIER C R,CAIRNS B R.The biology of chromatin remodeling complexes[J].Annual review of biochemistry,2009,78:273-304.
[10] FLAUS A,MARTIN D M A,BARTON G J,et al.Identification of multiple distinct Snf2 subfamilies with conserved structural motifs[J].Nucleic acids research,2006,34(10):2887-2905.
[11] NARLIKAR G J,FAN H Y,KINGSTON R E.Cooperation between complexes that regulate chromatin structure and transcription[J].Cell,2002,108:475-487.
[12] MARTENS J A,WINSTON F.Recent advances in understanding chromatin remodeling by Swi/Snf complexes[J].Current opinion in genetics & development,2003,13:136-142.
[13] HSIAO P W,FRYER C J,TROTTER K W,et al.BAF60a mediates critical interactions between nuclear receptors and the BRG1 chromatinremodeling complex for transactivation[J].Molecular and cellular biology,2003,23(17):6210-6220.
[14] PRECHT P,WURSTER A L,PAZIN M J.The SNF2H chromatin remodeling enzyme has opposing effects on cytokine gene expression[J].Molecular immunology,2010,47(11/12):2038-2046.
[15] DENSLOW S A,WADE P A.The human Mi2/NuRD complex and gene regulation[J].Oncogene,2007,26:5433-5438.
[16] CHAI B,HUANG J,CAIRNS B R,et al.Distinct roles for the RSC and Swi/Snf ATPdependent chromatin remodelers in DNA doublestrand break repair[J].Genes development,2005,19(14):1656-1661.
[17] SHAKED H,AVIVIRAGOLSKY N,LEVY A A.Involvement of the Arabidopsis SWI2/SNF2 chromatin remodeling gene family in DNA damage response and recombination[J].Genetics,2006,173(2):985-994.
[18] HUANG J,HSU J M,LAURENT B C.The RSC nucleosomeremodeling complex is required for Cohesin's association with chromosome arms[J].Molcular cell,2004,13(5):739-750.
[19] VARY J C,JR,GANGARAJU V K,QIN J,et al.Yeast Isw1p forms two separable complexes in vivo[J].Molecular cellular biology,2003,23(1):80-91.
[20] JIANG Y J,WANG X,BAO S L,et al.INO80 chromatin remodeling complex promotes the removal of UV lesions by the nucleotide excision repair pathway[J].Proceedings of the national academy of sciences of the USA,2010,107(40):17274-17279.
[21] FARRONA S,HURTADO L,BOWMAN J L,et al.The Arabidopsis thaliana SNF2 homolog AtBRM controls shoot development and flowering[J].Development,2004,131(20):4965-4975.
[22] HURTADO L,FARRONA S,REYES J C.The putative SWI/SNF complex subunit BRAHMA activates flower homeotic genes in Arabidopsis thaliana[J].Plant molcular biology,2006,62(1/2):291-304.
[23] TANG X R,HOU A F,BABU M,et al.The Arabidopsis BRAHMA chromatinremodeling ATPase is involved in repression of seed maturation genes in leaves[J].Plant physiology,2008,147:1143-1157.
[24] HAN S K,SANG Y,RODRIGUES A,et al.The SWI2/SNF2 chromatin remodeling ATPase BRAHMA represses abscisic acid responses in the absence of the stress stimulus in Arabidopsis[J].Plant cell,2012,24(12):4892-4906.
[25] WAGNER D,MEYEROWITZ E M.SPLAYED,a novel SWI/SNF ATPase homolog,controls reproductive development in Arabidopsis[J].Current biology,2002,12(2):85-94.
[26] KWON C S,CHEN C,WAGNER D.WUSCHEL is a primary target for transcriptional regulation by SPLAYED in dynamic control of stem cell fate in Arabidopsis[J].Genes development,2005,19(8):992-1003.
[27] WU M F,SANG Y,BEZHANI S,et al.SWI2/SNF2 chromatin remodeling ATPases overcome polycomb repression and control floral organ identity with the LEAFY and SEPALLATA3 transcription factors[J].Proceedings of the national academy of sciences of the USA,2012,109(9):3576-3581.
[28] WALLEY J W,ROWE H C,XIAO Y M,et al.The chromatin remodeler SPLAYED regulates specific stress signaling pathways[J].PLoS Pathogens,2008,4(2):1-8.
[29] SANG Y,SILVAORTEGA C O,WU S,et al.Mutations in two noncanonical Arabidopsis SWI2/SNF2 chromatin remodeling ATPases cause embryogenesis and stem cell maintenance defects[J].The plant journal,2012,72(6):1000-1004.
[30] MLYNROV L,NAP J P,BISSELING T.The SWI/SNF chromatinremodeling gene AtCHR12 mediates temporary growth arrest in Arabidopsis thaliana upon perceiving environmental stress[J].The plant journal,2007,51(5):874-885.
[31] ZHAO C F,XU J M,CHEN Y,et al.Molecular cloning and characterization of OsCHR4,a rice chromatinremodeling factor required for early chloroplast development in adaxial mesophyll[J].Planta,2012,236(4):1165-1176.
[33] GUO M X,WANG R C,WANG J,et al.ALT1,a Snf2 family chromatin remodeling ATPase,negatively regulates alkaline tolerance through enhanced defense against oxidative stress in rice[J].PLoS One,2014,9(12):1-25.
[33] ZHANG Z J,MAO Y F,HA S,et al.A multiplex CRISPR/Cas9 platform for fast and efficient editing of multiple genes in Arabidopsis[J].Plant cell reports,2015,35:1519-1533.
[34] KIM J M,TO T K,NISHIOKA T,et al.Chromatin regulation functions in plant abiotic stress responses[J].Plant,cell and environment,2010,33:604-611.
