食管鳞癌和腺癌转录组差异基因综合分析
2019-07-26李雨濛马佳康任凯凯孙金旗张亚丽
李雨濛,马佳康,任凯凯,李 南,王 健,孙金旗,张亚丽,马 军
0 引 言
食管癌是世界上最常见的恶性肿瘤之一,发病率位于第8位,占所有癌症死亡率的第6位。中国食管癌病例占全球一半以上,每年约有15万人死于这一疾病,是我国癌症相关死亡的第4大病因。食管癌在组织病理学和流行病学上存在较大程度的差异。食管癌主要有食管鳞癌和食管腺癌2个亚型,我国食管鳞癌的发病率达90%以上,而西方国家以食管腺癌为主[1]。食管鳞癌主要发生在食管上段和中段,与吸烟和酗酒关系密切;食管腺癌主要发生在和胃相连的食管下部,与肥胖、胃反流和Barrett食管有关[2]。食管腺癌的男女发病率亚洲地区为4∶1,北美地区达8∶1;食管鳞癌全球发病率男女比为2.7∶1,从西亚地区的1.2∶1到东欧的7.8∶1有较大的变化[3]。但食管鳞癌和食管腺癌在分组特征方面有多大的差异目前并不明确。本研究分析了TCGA数据库中食管鳞癌、食管腺癌的差异基因,构建了相关的ceRNA网络,筛选出了一批生存相关的lncRNA、miRNA及mRNA。
1 材料与方法
1.1 TCGA数据下载及差异基因筛选从TCGA(https://cancergenome.nih.gov/)数据库下载食管鳞癌、腺癌的mRNA、lncRNA、miRNA测序数据及临床信息。截止2018年10月10日,数据库中食管鳞癌mRNA及lncRNA测序数据92例,其中81例食管鳞癌组织样本及11例癌旁正常组织样本,miRNA食管鳞癌测序数据108例,包括食管鳞癌组织样本95例,癌旁正常组织样本13例;食管腺癌mRNA及lncRNA测序数据91例,其中80例食管腺癌组织样本及11例癌旁正常组织样本,miRNA食管腺癌测序数据102例,包括食管腺癌组织样本89例、癌旁正常组织样本13例。使用R语言“edgeR”包筛选差异基因,筛选条件为:log 2-fold change(logFC)>2且P<0.05。
1.2 食管鳞癌和食管腺癌的ceRNA网络构建筛选出的差异mRNA、lncRNA、差异miRNA在miRcode数据库中进行配对。利用starBase在线软件(http://starbase.sysu.edu.cn/)对筛选出的差异miRNA进行靶基因预测,miRDB、miRTarBase、TargetScan3个数据库预测靶基因,从而得到lncRNA-miRNA-mRNA的ceRNA调控网络,利用Cytoscape v3.5.1软件进行作图。
1.3 miRNA靶基因预测及GO、KEGG功能富集分析利用在线数据库miRWalk(http://mirwalk.umm.uni-heidelberg.de/)预测miRNA调控的靶基因。人类miRNA识别靶基因的seed区域比较短,预测到的靶基因较多,预测结果在targetscan,miRDB等数据库进行进一步的筛选。GO、KEGG使用Cytoscape 中的“ClueGO”、“CluePedia”软件包完成,筛选标准为P<0.05。
1.4 利用生存分析寻找和生存相关的差异基因利用TCGA数据库中所有食管癌的生存时间,采用的是R语言中的“Survival”包,对差异的lncRNA、miRNA、mRNA进行生存分析。以P≤0.05为差异有统计学意义。
2 结 果
2.1 食管鳞癌和腺癌差异基因筛选及ceRNA网络构建TCGA数据库中食管鳞癌的转录组测序数据分析结果显示,共筛选出差异lncRNA1160个、mRNA 2064个、miRNA 69个;腺癌共筛选出差异lncRNA 709个、mRNA 1490个、miRNA 81个。食管鳞癌中,73个lncRNA、9个miRNA及13个mRNA参与构建了ceRNA网络;食管腺癌中,51个lncRNA、10个miRNA及14个mRNA参与构建了ceRNA网络。食管鳞癌和食管腺癌的ceRNA网络分析结果显示:PVT1、LINC00524、miR-204、miR-383、HOXC8、NTRK2等在食管鳞癌和腺癌中均起重要调控作用。见图1。
2.2 miR-204靶基因预测及GO、KEGG功能分析miR-204-5p经miRWalk共预测到13227个调控靶基因,在targetscan,miRDB数据库共筛选出232个靶基因。GO功能分析显示38个富集,主要参与:细胞-基质黏附的调节、细胞膜投射形态发生、β-连环蛋白结合等过程。KEGG分析显示4个相关通路:Hedgehog信号通路、寿命调节通路、雌激素信号通路、松弛素信号通路。见图2。
2.3 食管鳞癌和腺癌ceRNA中差异基因生存分析食管鳞癌和腺癌相关的ceRNA网络中的差异基因生存分析结果显示,食管鳞癌中与患者生存相关的 差 异 lncRNA 包 括 LINC00261、MLIP-IT1、LINC00504,差异miRNA包括hsa-mir-338,未发现与生存相关的mRNA。食管腺癌中与生存相关的差异lncRNA包括CYP1B1-AS1、HOTAIR,差异mRNA包括IL11、NTRK2、ANGPT2、PBK,未发现与生存相关的miRNA。
2.4 食管鳞癌和腺癌共有差异基因分析食管鳞癌和腺癌共有的差异lncRNA上调占15.4%(150个),下调占26.8%(158个);miRNA上调占24.2%(22个),下调占27.6%(8个);mRNA上调占20.5%(234个),下调占23.7%(418个)。食管鳞癌和腺癌中对上述共有差异基因进行生存分析,结果显示与食管鳞癌和腺癌生存均相关的lncRNA为AL138789.1;miRNA为hsamir-105-2、hsa-mir-105-1、hsa-mir-767;mRNA 为KIF18A、PBK、GPER1,见图3。
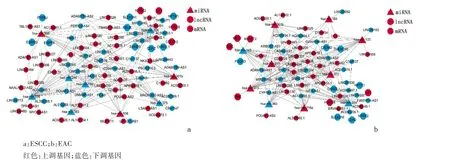
图1 食管鳞癌及腺癌相关ceRNA网络Figure 1 Competitive endogenous RNA(ceRNA)networks for ESCC(a)and EAC(b)
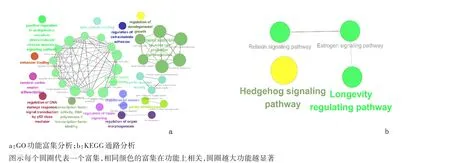
图2miR-204靶基因GO及KEGG功能分析Figure 2 GO analysis of the functional enrichments(a)and KEGG analysis of the signaling pathways(b)of the miR-204 target gene
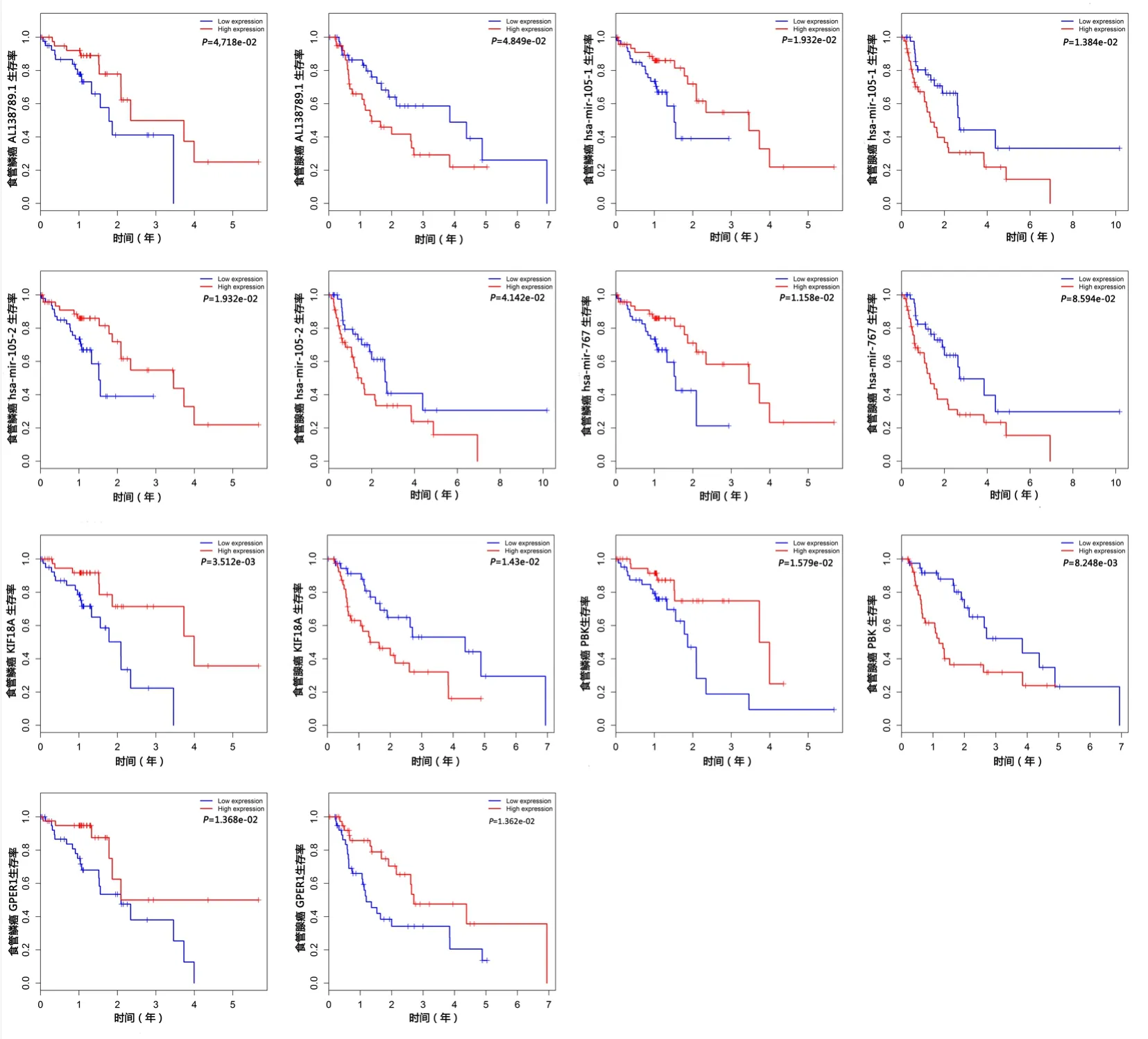
图3 食管鳞癌和腺癌共有差异表达基因生存分析图Figure 3 Venn diagram and Kaplan-Meier survival curves for differentially expressed genes in both ESCC and EAC
3 讨 论
EAC和ESCC存在着明显的流行病学和组织病理学差异,预示着EAC和ESCC可能存在明显的差异,但ESCC与EAC在分子特征方面的差异目前并不明确。我们从TCGA数据库ESCC及EAC测序数据中筛选出差异RNA,并成功构建了lncRNA-miRNA-mRNA调控网络,这可能对研究食管癌的发生、发展相关机制提供新的方法。
ceRNA网络分析发现了一些在ESCC和EAC均起重要调控作用的基因。Li PD等研究表明PVT1作为miR-203和LASP1的分子海绵可以促进ESCC的进展[6]。一项关于食管癌的研究表明:miR-204可以促进Sox4的降解,但这一作用可以被lncRNA UCA1所抑制进而促进食管癌细胞的增殖[7]。miR-204靶基因的GO、KEGG分析也表明其可能参与ESCC及EAC的形成与进展。对ceRNA网络中的差异基因进行Kaplan-Meier生存分析筛选出了一批与ESCC及EAC生存相关的差异基因。其中,LINC00261和miR-338已被证实参与ESCC的形成与进展[8-9],本研究分析结果与其相同且发现它们与患者的预后相关。既往研究表明HOTAIR、PBK、NTRK2与ESCC相关,本研究中发现它们也可能参与EAC的发生、发展并且与患者预后相关[10-12]。总之,本研究发现了一些与ESCC、EAC患者生存相关的差异基因,可能作为食管癌患者预后的生物标志物。
本研究发现ESCC和EAC在分子特征方面存在差异。为了寻找与ESCC和EAC生存均相关的差异基因,本研究将分别将ESCC和EAC中的这些差异基因进行生存分析,结果显示:AL138789.1、hsa-mir-105-2、hsa-mir-105-1、hsa-mir-767、KIF18A、PBK、GPER1与ESCC和EAC患者预后均相关,除GPER1在ESCC和EAC中的生存趋势一致,其余差异基因在ESCC和EAC中的生存趋势则相反。GPER1作为膜性雌激素受体的一种,介导了雌激素靶器官癌症的发生发展,尤其是在乳腺癌中可以与雌激素和表皮生长因子相互作用使雌激素具有表皮生长因子的作用[13],同时使乳腺癌细胞产生耐药[14]。本研究分析结果显示该基因在ESCC和EAC肿瘤组织中表达均降低,并且与患者预后不良相关,可以作为食管癌预后的生物标志物。在非小细胞肺癌和肝细胞肝癌中,miR-105-1表达量降低且与高表达患者相比生存期明显缩短[15-16]。ESCC的生存分析与其一致,高表达患者生存期较低表达者更长;但在EAC患者中的生存趋势相反。KIFl8A作为驱动蛋白家族的成员之一,其在维持微管动态和参与细胞有丝分裂中扮演着重要的角色。研究表明,KIF18A在肝细胞肝癌、结直肠癌、乳腺癌中表达增高,在调控肿瘤细胞增殖、迁移、侵袭方面发挥着重要作用[17-19],但在胃癌中表达下调且与患者不良预后相关[20]。本研究分析结果显示:KIF18A在ESCC和EAC中均增高,但在高表达的ESCC患者中预示着更长的生存时间,但在高表达的EAC中则相反。
总之,本研究分析表明ESCC和EAC可能存在着明显的分子差异,筛选出的生存相关差异基因对于ESCC和EAC患者预后的预测作用相反,我们将在今后的实验中对具体的作用机制进行探讨。
